2. 新疆生产建设兵团第6师农业科学研究所,新疆 五家渠 831300;
3. 山西大学生物工程学院,太原 030006
2. Institute of Agricultural Science, Sixth Division, Xinjiang Production and Construction Corps, Wujiaqu 831300, China;
3. College of Bioengineering, Shanxi University, Taiyuan 030006, China
乙烯 (Ethylene) 是植物内源激素,对植物生长发育具有重要调控作用。具有呼吸跃变的果实在呼吸跃变前产生基础水平的少量乙烯,被称为系统Ⅰ乙烯,调控植物正常生长、发育和应对胁迫反应等。系统Ⅰ乙烯具有自抑制机制。随着果实成熟,伴随呼吸跃变产生大量乙烯,被称为系统Ⅱ乙烯。系统Ⅱ乙烯具有自催化机制,促进花的凋亡和果实成熟[1]。
乙烯的生物合成遵循甲硫氨酸 (Methionine,Met)→S-腺苷甲硫氨酸 (S-Adenosyl methionine,SAM)→1-氨基环丙烷1-羧酸ACC (1-aminocyclopropane-1-carboxylic acid,ACC)→乙烯的途径。ACC是合成乙烯的直接前体,由ACC合成酶ACS (ACC synthase) 催化SAM形成,这个过程被认为是乙烯合成的限速步骤,ACS是乙烯合成的限速酶。番茄 (Solanum lycopersicum) 是呼吸跃变型果实,其ACS (SlACS) 由多基因家族编码,SlACS1A和SlACS6主导系统Ⅰ乙烯的合成,负责番茄果实在呼吸跃变前产生基础水平的少量乙烯,SlACS2和SlACS4主导系统Ⅱ乙烯的合成[2],在果实成熟过程中伴随呼吸跃变产生大量乙烯,使得番茄成熟过程难以控制,果实易过熟导致腐烂变质。
通过基因工程手段抑制番茄SlACS2的表达,主要是把SlACS2的反义RNA基因[3-4]或RNAi基因[5-6]导入番茄,可以减缓番茄果实中的乙烯合成,迟滞果实过熟,延长货架期,提高耐贮运性能。上述利用常规转基因技术,导入的外源基因在植物基因组上的整合位置具有随机性,容易出现一些非预期效应,如转基因整合到宿主基因的“阅读框”,使其不能有效表达,影响基因工程育种效率。民众对于转基因的安全性疑虑,也增加了转基因作物评价和应用成本。
CRISPR-Cas9是一种新型的基因组定点编辑系统[7-8],其通过一段20 nt的sgRNA (Single guide RNA) 与基因组同源序列互补配对引导Cas9蛋白结合于基因组的特异性位点,在sgRNA 3’端PAM (Protospacer adjacent motif,前间区序列邻近基序) 元件上游的第3~4 bp处剪切,造成DNA双链断裂 (Doubled-strand breaks, DSB)。不同CRISPR-Cas9的PAM序列有所不同,经典酿脓链球菌 (Streptpcpccus pyogenes) 中的PAM元件序列为5’-NGG-3’。因此,CRISPR-Cas9系统中靶位点的选择是由20 nt的sgRNA及3’端的PAM序列共同决定的。
Cas9蛋白切割造成的DNA双链断裂有两条修复途径[9],即非同源末端连接 (Non-homologous end joining, NHEJ) 和同源重组 (Homologous recombination, HR)。非同源末端连接是主要修复途径,同源重组只发生在特定的细胞周期和类型中。非同源末端连接容易出错,修复过程中会引入短的缺失或插入,抑制或修饰特定基因功能。与诱发突变相比,CRISPR-Cas9系统为定点突变,效率高、可控性好。CRISPR-Cas9系统通过植物基因组原位编辑来抑制或修饰基因功能,无需整合新基因,比转基因技术更为安全可控,易于消除民众安全性疑虑。
CRISPR-Cas9系统中,20 nt的sgRNA及其3’端的PAM元件总长度仅23 nt,易在基因组的非目标区段出现同源序列。另外,Cas9核酸酶可能切割与sgRNA不完全匹配的序列,导致脱靶效应[10-11]。规避脱靶效应和提高编辑效率是应用CRISPR-Cas9系统的关键。本文设计、筛选出了番茄SlACS2基因的sgRNA,以期为利用CRISPR-Cas9技术抑制SlACS2基因表达、提高番茄的耐贮运性能提供依据。
1 材料与方法 1.1 SIACS2数字表达谱的建立以番茄功能基因组数据库网站 (http://ted.bti.cornell.edu/cgi-bin/) 中RNA-seq data的数据作基础,建立SlACS2基因的基于RNA-seq的数字表达谱。
1.2 SlACS2染色体定位和基因组结构的确定利用NCBI (http://www.ncbi.nlm.nih.gov/) 核酸数据库查询获得SlACS2的cDNA,以此作query,搜索番茄基因组数据库得到相应的gDNA序列和所在染色体信息。通过序列比对确定SlACS2的基因组结构以及外显子所在染色体位置。
1.3 sgRNA的设计和选择根据CRISPR-Cas9靶点设计原则,利用CRISPRdirect ((https://crispr.dbcls.jp/)[12], 2015) 筛选SlACS2外显子区域的sgRNA序列,即3’端有PAM元件的20个连续的碱基序列,PAM元件设定为NGG [13],sgRNA的结构为5’-(N)20NGG-3’,N为任意核苷酸。利用CRISPR-P (http://cbi.hzau.edu.cn/crispr/) [14]对sgRNA的靶向特异性进行综合评分。
2 结果与分析 2.1 基于RNA-seq的SlACS2数字表达谱分析以番茄功能基因组RNA-seq数据库为基础,分析了SlACS2基因 (TC125329) 在不同组织以及不同生长时期的的表达模式 (见图 1)。结果显示,SlACS2在番茄的种子、胚根、顶端分生组织、不同时期的幼苗、根系、叶片、花蕾、绿果中均无表达,在子房和愈伤组织中有低水平表达,在破色期的果实和成熟的红果实中有较高水平的表达。上述基于RNA-seq的表达谱分析证实了SlACS2是与果实成熟相关的基因,利用CRISPR-Cas9技术对该基因进行原位编辑,有可能减缓番茄内源乙烯的产生,迟滞番茄过熟,提高贮运品质。

|
图 1 基于RNA-seq的SlACS2表达谱 Figure 1 The digital expression profile of SlACS2 based on RNA-sequence 注:1-子房; 2-半绿果实; 3-成熟绿果实; 4-破色期果实; 5-成熟红果实; 6-花蕾 (0-3 mm); 7-花蕾 (3-8 mm); 8-开花前花蕾 (8 mm); 9-完全展开花; 10-发育阶段花的混合物; 11-野生番茄花粉; 12-感染假单胞菌叶片; 13-假单胞菌叶片; 14-混合诱导叶片; 15-植株的顶端分生组织 (4-6周龄); 16-植株顶端分生组织 (8周龄); 17-花前根; 18-挂果期根; 19-营养匮乏的根; 20-完全吸涨5 d后的胚根; 21-完全吸涨7 d后的幼苗; 22-休眠种子; 23-愈伤组织。 PRKM:每百万reads中来自于特定基因每千碱基长度的reads数。 |
从GenBank搜索到SlACS2序列 (GenBank Accession:NM_001247249.2),mRNA长度1 855 nt,cds位于181~1 629 nt之间,长度1 458 nt。
以SlACS2的cds作Querry,在番茄的基因组数据库进行Blast。比对结果表明,SlACS的gDNA位于番茄的1号染色体 (GenBank Accession:NC_015438.2),位置在88 456 011~88 453 490 nt之间,长度2 522 nt,由4个外显子、3个内含子组成 (见图 2),前3个外显子均较短,位于SlACS2的5’端,可在每个外显子区域设计sgRNA,规避sgRNA分布在外显子和内含子连接处。

|
图 2 SlACS2的基因组结构和染色体位置 Figure 2 SlACS2 enomic costructure and its location on the chromosome |
SlACS2第1外显子长度171 nt,位于染色体88 456 011~88 455 841 nt之间。根据CRISPR-Cas9靶点设计原则,设定sgRNA长度为20 nt,下游的PAM元件为NGG。CRISPRdirect在线工具分析表明,第1外显子含有18条sgRNA (见表 1)。CRISPR-P分析表明,这些sgRNA的靶向特异性综合得分在29~49。
| 表 1 SlACS2基因第1外显子含有的sgRNAs Table 1 sgRNAs of exon 1 in SlACS2 gene |
在CRISPR-Cas9系统中,sgRNA的转录通常由U6或者U3启动子驱动,二者驱动转录的活性相对较高。U6或者U3启动子属于Pol Ⅲ型启动子[15],连续的TTTT序列会使转录终止。在第1外显子的18条sgRNA中,有9条 (sgRNA1-1、-4、-5、-6、-8、-9、-11、-12、-18) 含有连续的TTTT序列,可排除选择。另外,sgRNA1-2的GC含量较低,sgRNA1-7除了在靶位点外,在番茄5号染色体上也有与之完全匹配的序列,易产生脱靶效应。在其余7条sgRNA (见图 3) 中,位于外显子132~154 nt的sgRNA1-14特异性最好,该sgRNA和它包含的对特异性起重要作用的、邻近PAM的12 nt种子序列[16]在番茄基因组上均是唯一序列,其他部位没有与之完全匹配的序列。sgRNA1-14的GC含量45%,靶向特异性综合得分也最高,可作为优选sgRNA。

|
图 3 SlACS2第1外显子主要sgRNAs的分布。 Figure 3 The position of some important sgRNAS of exon 1 in SlACS2 注:括号内数值为该sgRNA所在外显子位置。 |
Cas9蛋白包含氨基端的RuvC-like结构域及位于蛋白中间位置的HNH核酸酶结构域。HNH核酸酶结构域切割与sgRNA互补配对的模板链,RuvC-like结构域切割另1条链。突变Cas9核酸酶的一个功能结构域时,则只能切割DNA双链中的一条链,形成Cas9单切口酶 (Nickase)。体内DNA单链断裂修复采用碱基切除修复途径,不能产生突变。当1对分别位于染色体正负链上、方向相反且距离较近的两个sgRNA (即“Paired-sgRNA”) 同时引导Cas9单切口酶对基因组编辑时,两个近距离的单链断裂会组成双链断裂,通过非同源末端连接修复,造成短的缺失或插入,导致移码突变;而潜在的脱靶位点处只有单链断裂,修复过程不易发生突变。因此,采用“paired-sgRNA”和Cas9单切口酶编辑策略时,酶切识别所涉及的核苷酸数就由20 nt增加到40 nt,从而提高靶位点的专一性,降低脱靶风险[17-18]。据此,采用“paired-sgRNA”和Cas9单切口酶编辑策略,对第1外显子sgRNA1-3及其互补链的sgRNA1-14进行切割,间隔66 nt,有较大概率产生专一性移码突变。
2.4 SlACS2第2、3外显子sgRNA的设计和分析SlACS2第2外显子位于染色体86 455 742~86 455 614 nt之间,长129 nt,与第1外显子相隔98 nt的内含子。CRISPRdirect在线分析表明,第2外显子有9条sgRNA序列,其GC含量均低于35%,邻近PAM 12 nt的种子序列的脱靶位点在2个以上 (见表 2),没有合适的sgRNA可供选择。
| 表 2 SlACS2基因第2外显子含有sgRNAs Table 2 sgRNAs of exon 2 in SlACS2 gene |
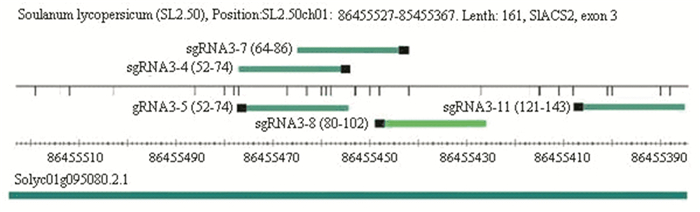
SlACS2第3外显子位于染色体86 455 528~86 455 368 nt之间,长161 nt,与第2外显子相隔85 nt的内含子,与第4外显子相隔881 nt的内含子。CRISPRdirect在线分析表明,第3外显子有11条sgRNAs。CRISPR-P分析表明,sgRNAs的靶向特异性综合得分在22~48,差异较大 (见表 3)。在这些sgRNAs中,sgRNA3-9和sgRNA3-10含有连续的TTTT序列,易使转录提前终止;sgRNA3-1、-2、-3的GC含量较低;sgRNA3-6的靶向特异性综合得分最低。在其余5条sgRNAs (见图 4) 中,sgRNA3-8特异性最好,其邻近PAM 12 nt的种子序列在番茄全基因组上是唯一序列,其他部位没有与之完全匹配的序列,靶向特异性综合得分也较高。该sgRNA 3’端第20位的碱基为鸟嘌呤G,意味着基因组编辑效率较高[19],可作为优选序列。sgRNA3-11特异性也较好,靶向特异性综合得分也较高,其12 nt的种子序列除了靶位点外,在5号染色体的1个位点上有与其完全匹配的序列,其脱靶效应可根据脱靶位点两侧的序列 (见图 5) 设计引物,对PCR产物测序予以验证。另外该sgRNA的第一碱基为G,也适合U6启动子转录,是另一优选序列。sgRNA3-8与位于互补链的sgRNA3-4间隔5 nt,sgRNA3-11与位于互补链的sgRNA3-7之间相隔34 nt,这两对sgRNA均可设计为“paired-sgRNA”,用Cas9单切口酶进行编辑。
| 表 3 SlACS2基因第3外显子含有sgRNAs Table 3 sgRNAs of exon 3 in SlACS2 gene |
|
图 4 SlACS2第3外显子主要sgRNAs的分布 Figure 4 The position of some important sgRNAS of exon 3 in SlACS2 注:括号内数值为该sgRNA所在外显子位置。 |

|
图 5 第3外显子sgRNA3-11的脱靶位点和染色体位置 Figure 5 The position of off-target site of sgRNA3-11 in exon 3 |
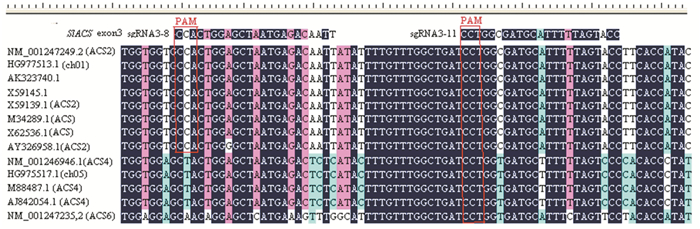
以SlACS2的第1和第3外显子作Query在GenBank中进行BLAST,搜索同源序列,进行多序列比对 (见图 6, 图 7)。比对结果表明,sgRNA1-14、sgRNA3-11在搜索到的8条序列上均完全一致,sgRNA3-8也只有1个SNP,表明本研究优选的3条sgRNA均具有较好的保守性,可用于不同品种SlACS2基因的靶向编辑。而与GenBank公布的SlACS4和SlACS6比对,sgRNA1-14均有5个SNP,其中3个位于种子序列区;sgRNA3-8分别有3个和6个SNP,且有1个SNP位于PAM的第2个核苷酸,TGG突变为TAG;sgRNA3-11有3个SNP,其中2个位于种子序列区;预示本研究优选的3条sgRNA均可规避对SlACS家族其他成员的脱靶效应。

|
图 6 SlACS2第1外显子sgRNA1-14的SNP位点 Figure 6 SNP loci on sgRNA1-14 of exon 1 in SlACS2 |
|
图 7 SlACS2第3外显子sgRNA3-8和sgRNA3-11的SNP Figure 7 SNP loci on sgRNA3-8 and sgRNA3-11 of exon 3 in SlACS2 |
科学设计sgRNA,提高基因组编辑效率和最大限度降低脱靶风险是CRISPR/Cas9系统面临的最大挑战。番茄是模式植物之一,已完成了全基因组测序,sgRNA的特异性及其脱靶位点可通过全基因组层面扫描获得相关信息。sgRNA的设计已有多个工具软件和在线分析平台,如CRISPRdirect综合考虑了sgRNA全长和12 nt及8 nt种子序列的特异性,结果输出中会标出每条sgRNA在外显子上的位置和GC含量,还将特异性高、脱靶位点少的sgRNA以及含有转录终止序列“TTTT”的sgRNA分别用亮绿色和灰色作出标示;华中农业大学作物遗传改良国家重点实验室开发的CRISPR-P则根据特定公式对每个sgRNA序列的靶向特异性进行综合评分和排序,并标示出sgRNA靶标位点中是否含限制性内切酶识别序列,以方便采用酶切法检测基因组切割效率。本研究综合运用CRISPRdirect和CRISPR-P两个在线分析平台,对SlACS2基因5’端的第1、2、3个外显子上所分布的sgRNAs进行了综合评价,筛选出3条sgRNAs优选序列,即第1外显子sgRNA1-14、第3外显子sgRNA3-8和sgRNA3-11。其中sgRNA1-14和sgRNA3-8的全序列或12 nt的种子序列在番茄基因组上均为唯一序列,但它们的5’端第1个碱基为A或C。双子叶植物sgRNA的转录一般选用U6启动子驱动,其转录活性相对较高,有明确的转录起始位点,即以“G”碱基开头。精确起始转录能消除无关DNA序列的转录,从而减少脱靶效应的产生[20-21]。因此,如果选用U6启动子,需要在这2条sgRNA的5’端添加鸟嘌呤核苷酸。有研究表明,在sgRNA的5’端额外增加两个鸟嘌呤核苷酸后能够显著提高CRISPR/Cas9系统的特异性[17],为非G起始的sgRNA应用提供了依据。
乙烯除了促进果实成熟,在植物种子萌发、叶片扩展、根毛伸长、侧根生长、开花、植株衰老等阶段均起重要作用。抑制番茄系统Ⅰ或过度抑制系统Ⅱ乙烯的生成,将影响番茄的正常生长或致使果实不能成熟。本研究从SlACS2中筛选出的3条sgRNAs与GenBank中公布的SlACS4和SlACS6的同源序列存在3~8个SNP,其中2~3个SNP分布在12 nt的种子序列区。这些SNP的存在规避了sgRNA对SlACS4和SlACS6的脱靶效应,可望达到预期目的:(1) 既抑制系统Ⅱ乙烯合成限速酶基因SlACS2的表达,又不影响SlACS4的正常表达,使系统Ⅱ乙烯保持在一定水平而不致过量,以迟滞番茄过熟;(2) 不干扰系统Ⅰ乙烯形成的SlACS6基因的正常表达,保证番茄的正常生长。生物信息学可以提供许多有用信息,但不能代替实验验证。在应用这3条优选的sgRNA改良番茄的耐贮运性状前,可先用原生质体系统验证其靶向能力和编辑效率。
CRISPR-Cas9系统通过sgRNA引导Cas9蛋白在PAM元件上游第3~4 bp处剪切,造成DNA双链断裂,在非同源末端连接修复过程中引入短的缺失或插入,使基因失活,但失活基因仍能复制和转录,消耗植物营养和能量,如能删除失活基因的完整序列或大片断,将降低植物营养和能量的消耗。近年来,已通过双sgRNA引导Cas9系统在植物[22-23]和动物[24]上删除了一段DNA序列,最大缺失片段达105 kb。本研究筛选的sgRNA1-14和sgRNA3-8特异性较好,在染色体上相隔409 nt,利用双sgRNA引导Cas9将其删除,造成DNA大片段缺失,将更彻底的抑制SlACS2基因功能。
| [1] |
MCMURCHIE E, MCGLASSON W, EAKS I. Treatment of fruit with propylene gives information about the biogenesis of ethylene[J]. Nature, 1972, 237(5352): 235–236.
DOI:10.1038/237235a0 ( 0) 0)
|
| [2] |
BARRY C, LOP-TOUS M, GRIERSON D. The regulation of 1-aminocyclopropane-1-carboxylic acid synthase gene expression during the transition from system-1 to system-2 ethylene synthesis in tomato[J]. Plant Physiology, 2000, 123(3): 979–986.
DOI:10.1104/pp.123.3.979 ( 0) 0)
|
| [3] |
OELLER P, LU M, TAYLOR L, et al. Reversible inhibition of tomato fruit senescence by antisense RNA[J]. Science, 1991, 254(5030): 437–439.
DOI:10.1126/science.1925603 ( 0) 0)
|
| [4] |
熊爱生, 姚泉洪, 李贤, 等. ACC氧化酶和ACC合成酶反义RNA融合基因导入番茄和乙烯合成的抑制[J]. 实验生物学报, 2003, 36(6): 35–41.
XIONG Aisheng, YAO Quanhong, LI Xian, et al. Double antisense ACC oxidase and ACC synthase fusion gene introduced into tomato by agrobacterium-mediated transformation and analysis the ethylene production of transgenic plants[J]. Acta Biologiae Experimentalis Sinica, 2003, 36(6): 35–41.
( 0) 0)
|
| [5] |
陈银华, 李汉霞, 叶志彪. 不同结构的外源ACO基因导入番茄对乙烯生成速率的影响[J]. 园艺学报, 2007, 34(3): 644–648.
CHEN Yinhuan, LI Hanxia, YE Zhibiao. Effects of introduction of different T-DNA structures of ACO gene to tomato genome on ethylene production rate[J]. Acta Horticulturae Sinica, 2007, 34(3): 644–648.
( 0) 0)
|
| [6] |
GUPTA A, PAL R, RAJAM M. Delayed ripening and improved fruit processing quality in tomato by RNAi-mediated silencing of three homologs of 1-aminopropane-1-carboxylate synthase gene[J]. Journal of Plant Physiology, 2013, 170(11): 987–995.
DOI:10.1016/j.jplph.2013.02.003 ( 0) 0)
|
| [7] |
CONG L, RAN F, COX D, et al. Multiplex genome engineering using CRISPR/Cas systems[J]. Science, 2013, 339(6121): 819–823.
DOI:10.1126/science.1231143 ( 0) 0)
|
| [8] |
Doudna J A, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9[J]. Science, 2014, 346(6213): 1258096–1258096.
DOI:10.1126/science.1258096 ( 0) 0)
|
| [9] |
ILIAKIS G, WANG H, PERRAULT A, et al. Mechanisms of DNA double strand break repair and chromosome aberration formation[J]. Cytogenetic & Genome Research, 2004, 104(1-4): 14–20.
DOI:10.1159/000077461 ( 0) 0)
|
| [10] |
FU Y, FODEN J, KHAYTER C, et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells[J]. Nature Biotechnology, 2013, 31(9): 822–826.
DOI:10.1038/nbt.2623 ( 0) 0)
|
| [11] |
HSU P, SCOTT D, WEINSTEIN J, et al. DNA targeting specificity of RNA-guided Cas9 nucleases[J]. Nature Biotechnology, 2013, 31(9): 827–832.
DOI:10.1038/nbt.2647 ( 0) 0)
|
| [12] |
NATIO Y, HINO K, BONO H, et al. CRISPRdirect: software for designing CRISPR/Cas guide RNA with reduced off-target sites[J]. Bioinformatics, 2015, 31(7): 1 120–1 123.
DOI:10.1093/bioinformatics/btu743 ( 0) 0)
|
| [13] |
ZHANG Y, GE X, YANG F, et al. Comparison of non-canonical PAMs for CRISPR/Cas9-mediated DNA cleavage in human cells[J]. Scientific Reports, 2014(4): 5405.
DOI:10.1038/srep05405 ( 0) 0)
|
| [14] |
LEI Y, LU L, LIU H, et al. CRISPR-P: a web tool for synthetic single-guide RNA design of CRISPR-system in plants[J]. Molecular Plant, 2014, 7(9): 1 494–1 496.
DOI:10.1093/mp/ssu044 ( 0) 0)
|
| [15] |
SHAN Q, WANG Y, LI J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system[J]. Nature Biotechnology, 2013, 31(8): 686–688.
DOI:10.1038/nbt.2650 ( 0) 0)
|
| [16] |
PATTANAYAK V, LIN S, GUILINGER J, et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity[J]. Nature Biotechnology, 2013, 31(9): 839–843.
DOI:10.1038/nbt.2673 ( 0) 0)
|
| [17] |
CHO S W, KIM S, KIM Y, et al. Analysis of off-target effects of CRISPR/Cas-derived RNA-guided endonucleases and nickases[J]. Genome Research, 2014, 24(1): 132–141.
DOI:10.1101/gr.162339.113 ( 0) 0)
|
| [18] |
RAN F, HSU P, LIU C, et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity[J]. Cell, 2013, 154(6): 1 380–1 389.
DOI:10.1016/j.cell.2013.08.021 ( 0) 0)
|
| [19] |
DOENCH J, HARTENIAN E, GRAHAM D, et al. Rational design of highly active sgRNAs for CRISPRCas9-mediated gene inactivation[J]. Nature Biotechnology, 2014, 32(12): 1262–1267.
DOI:10.1038/nbt.3026 ( 0) 0)
|
| [20] |
BELHAJ K, CHAPARRO-GARCIA A, KAMOUN S, et al. Editing plant genomes with CRISPR/Cas9[J]. Current Opinion in Biotechnology, 2015(33): 76–84.
DOI:10.1016/j.copbio.2014.11.007 ( 0) 0)
|
| [21] |
LI X, JIANG D H, YONG K L, et al. Varied transcriptional efficiencies of multiple Arabidopsis U6 small nuclear RNA genes[J]. Journal of Integrative Plant Biology, 2007, 49(2): 222–229.
DOI:10.1111/j.1744-7909.2007.00393.x ( 0) 0)
|
| [22] |
LI J, NORVILLE J, ACH J, et al. Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9[J]. Nature Biotechnology, 2013, 31(8): 688–691.
DOI:10.1038/nbt.2654 ( 0) 0)
|
| [23] |
BROOKS C, NEKRASOV V, LIPPMAN Z, et al. Efficient gene editing in tomato in the first generation using the CRISPR/Cas9 system[J]. Plant Physiology, 2014(166): 1 292–1 297.
DOI:10.1104/pp.114 ( 0) 0)
|
| [24] |
SONG Y, YUAN L, WANG Y, et al. Efficient dual sgRNA-directed large gene deletion in rabbit with CRISPR/Cas9 system[J]. Cellular & Molecular Life Sciences Cmls, 2016(73): 2959.
DOI:10.1007/s00018-016-2143-z ( 0) 0)
|
 2017, Vol. 15
2017, Vol. 15


