生物信息学的发展和测序技术的进步,使得人类在基因、蛋白质结构与功能等方面积累了海量数据。如国际千人基因组计划仅包含2 500个人的基因组信息,数据量已达到50 TB。人们希望利用数据,探讨基因变异与疾病的关系,识别用于蛋白质编码的基因,预测蛋白质的结构与功能。如何从海量数据中提取潜在信息,创造知识是生物信息学面临的一大挑战[1]。已有研究运用数据挖掘方法开展生物信息学研究。如Hua等采用支持向量机预测蛋白质亚细胞定位[2];Ernst等采用C4.5算法生成蛋白质注释等[3]。以上研究给我们的启示在于,数据挖掘方法可能是解决从海量数据中提取潜在信息问题的有力工具。
数据挖掘包括分类、聚类、回归、关联分析等诸多方法,哪些方法适用于生物信息学研究目前尚不完全清楚。一方面是由于数据挖掘是从数据中发现知识,本身带有不确定性;另一方面,数据挖掘与生物信息学的综合研究方兴未艾,尚未总结出具有普适性的研究流程。
本研究目标在于:1)找出生物信息学与数据挖掘交叉领域的领先机构和研究者;2)生物信息学研究中常用的数据挖掘方法有哪些; 3)数据挖掘方法多用来解决生物信息学中什么问题。回答以上问题,需要借助文献计量方法对目前已发表文献进行分析。
文献计量学是根据文献的各种特征数量,采用数学与统计学方法来描述评价和预测科学技术的现状与发展趋势的图书情报学分支学科[4]。该方法是了解生物信息学领域发展状况的重要工具,如宋茂海和李东方基于共词分析方法研究国内生物信息学热点领域[5]。
本研究从文献计量学视角对生物信息学与数据挖掘领域的文献进行分析,试图给出该交叉领域的研究概况,为后续生物信息学如何与数据挖掘相结合提供研究思路。
1 数据来源及处理 1.1 数据收集在web of science上以data mining、machine learning和bioinformatics、genomics等主题词检索文献,构建的检索式为:TS=("data mining" AND bioinformatics) OR TS=("machine learning" AND bioinformatics) OR TS=("data mining" AND genomics) OR TS=("machine learning" AND genomics)。
检索文献类型为Research article和Review,检索截止日期为2015年12月31日,共检索到1681篇文献。以1 681篇文献作为数据源开展后续分析,通过web of science输出全记录,包括作者、题目、关键词、摘要、年份、参考文献等。
1.2 数据分析采用Bibexcel软件,进行论文发表时间、机构、作者、关键词等信息的提取,并进行词频分析。对关键词、被引用文献作者进行共现分析。被引用文献作者这里只考虑第一作者有两个原因。第一,web of science输出参考文献记录时,默认第一作者;第二,用被引文献第一作者分析可避免多名作者合著时的重复计算[6]。共现分析作为一种信息计量方法,通过主题分析可较直观地揭示学科微观结构,其原理是当两个学科领域内的关键词在一篇文献中同时出现时,表明这两个词之间具有一定的内在关系,出现的次数越多,表明它们的关系越密切[7-8]。共现分析借助Gephi软件实现可视化[9]。可视化可通过展示事件的关联,实现隐性知识的显性化。
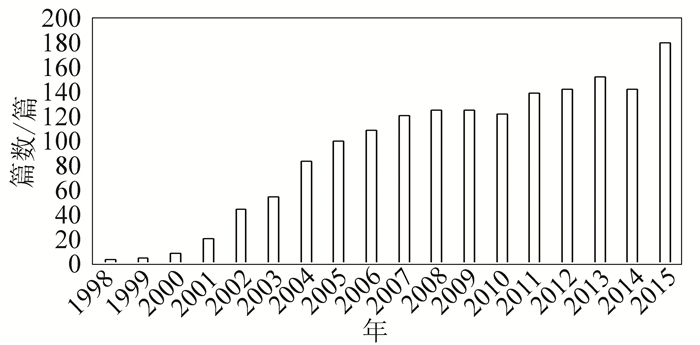
2 研究结果 2.1 频次统计 2.1.1 历年文章数量运用数据挖掘方法的生物信息学研究可上溯至1998年。该年出现四篇论文均涉及生物信息学和数据挖掘。这四篇文章分别为Eckman BA的The Merck Gene Index browser: an extensible data integration system for gene finding, gene characterization and EST data mining。Brazma A的Approaches to the automatic discovery of patterns in biosequences.Rebhan M的GeneCards:a novel functional genomics compendium with automated data mining and query reformulation support和van Ommen GJB的The Human Genome Project and the role of genetics in health care.
自1998年到2015年,运用数据挖掘方法的生物信息学研究呈现逐年增多的趋势。其中,发文量最大的是2015年,该年共有180篇文章涉及生物信息学与数据挖掘,见图 1。
|
图 1 1998~2015年发表文章数量 Figure 1 The number of literatures in years from 1998 to 2015 |
自1998~2015年,该领域发表论文的机构共有1 675个,表 1列出发文量最多的10个机构。发文量前10名的研究机构共发文279篇,占1 681篇的16.6%。
| 表 1 发文量最大研究机构前10位 Table 1 Top 10 research organization published most papers |
自1998~2015年,该领域发表论文的作者共有6 307位。表 2列出发文量前10位的作者。发文量前10名的作者共发文95篇,占1 681篇的5%。
| 表 2 发文数量最多作者前10名 Table 2 Top 10 researchers published most papers |
由表 3可知,被引用频次最高的文献是Altschul SF在1997年发表在NUCLEIC ACIDS RES上的文章,这篇论文被引用频次为126次。被引用频次排名前十的这些文献集中在1997~2001年,有2篇发表在P NATL ACAD SCI USA上,2篇发表在NUCLEIC ACIDS RES上。
| 表 3 被引频次前10名论文 Table 3 Top 10 highly cited frequency literatures |
1998~2015年,运用数据挖掘方法的生物信息学研究发表在627种期刊上。发文量最多的期刊是INT J DATA MIN BIOIN,发表文章数量占全部文章数量的4.5%。前10个发文量最大的期刊共发表 506篇,占全部文章数量的30.1%, 见表 4。
| 表 4 期刊载文量前10名 Table 4 Top 10 journals published most papers |
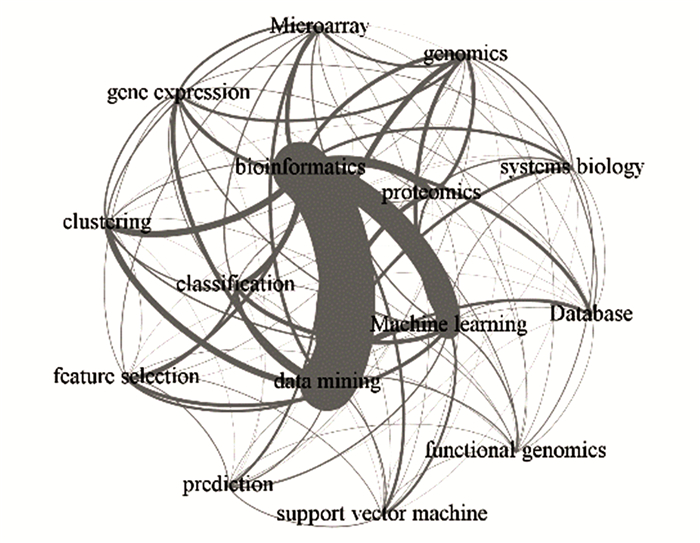
出现频次超过24次的关键词共有15个(见表 5),data mining, machine learning, classification, clustering, feature selection, support vector machine, prediction属于数据挖掘领域,bioinformatics, genomics, proteomics, gene expression, microarray, systems biology, functional genomics属于生物信息学领域,database在数据挖掘领域和生物信息学领域均有体现。
| 表 5 关键词出现频次最多前15位 Table 5 Top 15 frequency distribution of keywordss |
与data minning共同出现较多的关键词是bioinformatics (216次),machine learning (25次),clustering (25次)。与bioinformatics共同出现较多的关键词是data mining (216次),machine learning (96次),proteomics (30次),共现分析见图 2。
|
图 2 关键词共现分析 Figure 2 The analysis in co-occurrence of keywords |
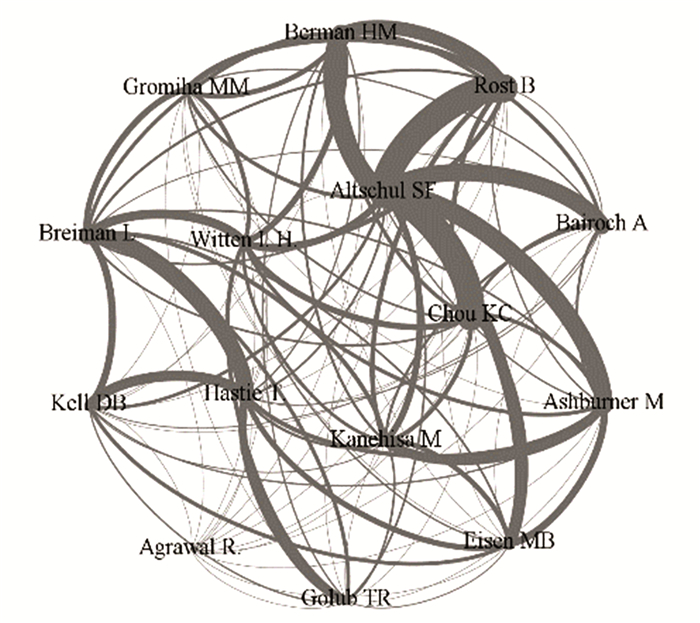
被引频次居前论文的作者网络中,存在两个明显的联系紧密的子网络,见图 3。第一个子网络中以Altschul SF为中心向外辐射,连接Berman HM,Rost B,Bairoch A,Ashburner M和Chou KC,同时Ashburner M,Chou KC又分别向外辐射连接Kanehisa M和Eisen MB。第二个子网络以Hastie T为中心向外连接Breiman L,Kell DB,Golub TR。
|
图 3 被引频次居前论文作者的共现分析 Figure 3 The analysis in co-occurrence of authors of highlycited frequency literatures |
生物信息学与数据挖掘的结合始于1998年,正值人类基因组计划完成前夕。通过该计划,积累了大量基因组测序数据。这表明生物信息学与数据挖掘的结合是受生物信息学学科发展内在要素驱动。生物学数据积累促使人们采用数据挖掘方法处理、分析数据自1998年至今,相关论文数量增加与生物学数据积累过程呈现一致趋势。随着生物学数据的继续积累,数据挖掘方法在生物信息学领域具有广阔的应用前景。
在生物信息学与数据挖掘结合的领域,美国、中国、欧盟具有优势,其中美国优势明显,发文量前10位的研究机构中美国占据4位。中国科学院是亚洲唯一进入发文量前10名的研究机构。
数据挖掘方法中机器学习、分类和聚类出现的频次较高,表明生物信息学中主要采用的数据挖掘方法是对所搜集的数据集进行类型划分和归类,这意味着数据挖掘和机器学习在生物信息学中可发挥重要作用。基因组学、基因表达、蛋白组学、微阵列、基因芯片、系统生物、功能基因组学是当前生物信息学中利用数据挖掘方法的主要领域,这与该时期基因组测序技术发展密不可分。但必须指出,随着蛋白质相关数据积累,如何从蛋白质数据中发现潜在信息可能是未来数据挖掘方法与生物信息学结合的一大热点领域。数据库是生物信息学和数据挖掘的切入点。生物信息学中的数据主要贮藏在数据库中,而数据挖掘则从数据库中调用、提取数据。这表明数据库对于生物信息学与数据挖掘的结合非常重要。未来应考虑在数据迅速积累的情况下保持数据库的及时更新与升级;同时,由于各组学数据格式并不统一,整合多种数据类型,将独立的、分散的数据库中的信息整合到一起并开发新的数据整合算法,形成标准化、全方面的信息数据库是目前该研究领域的新挑战[10]。
被引频次居前论文的作者网络中的子网络与应用和研究方向密切相关。第一个子网络擅于在研究中利用各数据库进行研究。网络中心作者Altschul SF主要研究利用blast进行DNA和蛋白质序列比对或者在DNA和蛋白质中应用一些算法[11]。由此向外连接的几位作者分别在DNA或者蛋白质领域的不同方向深入研究。Berman HM主要利用蛋白质数据库银行研究[12],Rost B主要研究蛋白质二级结构预测。Bairoch A运用SWISS-PROT数据库进行蛋白与核酸信息和结构的研究[13]。Ashburner M主要讲基因本体论,Chou KC则利用蛋白数据库,运用分类研究蛋白的亚细胞定位[14]。
第一个子网络中次级网络Kanehisa M研究基因表达本体和序列标签,Eisen MB主要研究运用分类分析全基因组表达的数据,分别与Ashburner M和Chou KC的研究相同或类似,因此Ashburner M和Kanehisa M,Chou KC和Eisen MB成为高共被引作者,组成次级子网络。
第二个子网络与基因表达或者癌症相关。Hastie T根据基因表达模式区分癌症,Breiman L运用各种算法对基因进行功能和分类预测[15],Kell DB主要研究基因组表达的功能,Golub TR主要研究癌症分子的分类。这表明研究者的研究方向相近或者相关时,他们的文章常常会一起被引用。
通过文献计量学方法,我们分析了生物信息学与数据挖掘这一交叉领域的基本情况,通过共现分析和可视化展示,生物信息学中采用的主要数据挖掘方法、相关研究内在联系得以揭示,共现分析和可视化展示的结合是理解研究领域相关进展的有力工具。
致谢 感谢Bibexcel的开发者Olle Persson教授。该软件使得数据提取过程非常高效。
| [1] |
朱杰. 生物信息学的研究现状及其发展问题的探讨[J]. 生物信息学, 2005, 3(4): 185–188.
ZHU Jie. Bioinformatics' status in quo and itsdevelopment in the future[J]. China Journal of Bioinformatics, 2005, 3(4): 185–188.
( 0) 0)
|
| [2] |
HUA S, SUN Z. Support vector machine approach for protein subcellular localization prediction[J]. Bioinformatics, 2001, 17(8): 721–728.
DOI:10.1093/bioinformatics/17.8.721 ( 0) 0)
|
| [3] |
KRETSCHMANN E, FLEISCHMANN W, APWEILER R. Automatic rule generation for protein annotation with the C4.5 data mining algorithm applied on SWISS-PROT[J]. Bioinformatics, 2001, 17(10): 920–926.
DOI:10.1093/bioinformatics/17.10.920 ( 0) 0)
|
| [4] |
赵蓉英, 许丽敏. 从文献计量学到网络计量学嬗变的可视化分析[J]. 情报科学, 2011, 29(7): 975–983.
ZHAO Rongying, XU Limin. Visualization analysis of the evolution from the bibliometrics to webometrics[J]. Infromation Science, 2011, 29(7): 975–983.
( 0) 0)
|
| [5] |
宋茂海, 李东方. 基于共词分析的国内生物信息学热点领域研究[J]. 生物信息学, 2014, 12(1): 46–52.
SONG Maohai, LI Dongfang. Hot spots analysis of China's bioinformatics based on co-word analysis method[J]. Chinese Journal of Bioinformatics, 2014, 12(1): 46–52.
DOI:10.3969/j.issn.1672-5565.2014.01.08 ( 0) 0)
|
| [6] |
周志超. 基于国内知识图谱领域高被引作者的社会网络分析[J]. 现代情报, 2012, 32(8): 97–100.
ZHOU Zhichao. Social network analysis of high cited authors based on domestic mapping knowledge domains[J]. Journal of Modern Information, 2012, 32(8): 97–100.
( 0) 0)
|
| [7] |
郭文姣, 欧阳昭连, 李阳, 等. 应用共词分析法揭示生物医学工程领域的研究主题[J]. 中国生物医学工程学报, 2012, 31(4): 545–551.
GUO Wenjiao, OUYANG Zhaolian, LI Yang, et al. Revealing theme structure of biomedical engineering using Co-Word analysis[J]. Chinese Journal of Biomedical Engineering, 2012, 31(4): 545–551.
( 0) 0)
|
| [8] |
朱安青, 周金元. 我国科技查新研究热点及趋势分析-共词分析视角[J]. 图书情报研究, 2009, 2(4): 45–49.
ZHU Anqing, ZHOU Jinyuan. Co-Word analysis of Sci-tech novelty retrieval research in China[J]. Library & Information Studies, 2009, 2(4): 45–49.
( 0) 0)
|
| [9] |
关迎晖, 向勇, 陈康. 基于gephi的可视分析方法研究与应用[J]. 电信科学, 2013(Z1): 112–119.
GUAN Yinghui, XIANG Yong, CHEN Kang. Research and application of visual analysis method based on gephi[J]. Telecommunications Science, 2013(Z1): 112–119.
DOI:10.3969/j.issn.1000-0801.2013.Z1.023 ( 0) 0)
|
| [10] |
杨健, 蔡浩洋. 肿瘤生物信息学数据库[J]. 生物技术通报, 2015, 31(11): 89–101.
YANG Jian, CAI Haoyang. The cancer-related bioinformatics databases[J]. Biotechnology Bulletin, 2015, 31(11): 89–101.
DOI:10.13560/j.cnki.biotech.bull.1985.2015.11.010 ( 0) 0)
|
| [11] |
ALTSCHUL S F, MADDEN T L, SCHÄFFER A A. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs[J]. Nucleic Acids Research, 1997, 25(17): 3389–3402.
DOI:10.1093/nar/25.17.3389 ( 0) 0)
|
| [12] |
BERMAN H M, WESTBROOK J, FENG Zukang. The protein data bank[J]. Nucleic Acids Research, 2000, 28(1): 235–242.
DOI:10.1107/so907444902003451 ( 0) 0)
|
| [13] |
BAIROCH A, APWEILER R. The SWISS-PROT protein sequence database and its supplement TrEMBL in 2000[J]. Nucleic Acids Research, 2000, 28(1): 45–48.
DOI:10.1093/nar/28.1.45 ( 0) 0)
|
| [14] |
CHOU Kuochen, ELROD D W. Protein subcellular location prediction[J]. Protein Engineering, 1999, 12(2): 107–118.
DOI:10.1093/protein/12.2.107 ( 0) 0)
|
| [15] |
BREIMAN L. Bagging predictors[J]. Machine Learning, 1996(24): 123–140.
DOI:10.1023/A:1018054314350 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


