从美国国立生物技术信息中心(National Center for Biotechnology Information, NCBI)的GenBank数据库,获取人MYBPC3基因转录本序列与cMyBP-C蛋白的氨基酸序列信息,MYBPC3基因突变位点信息来自Uniprot网站。
1.2 方法采用Clustal 2.1软件比对氨基酸序列,Mega6绘制序列系统进化树。采用SPORTⅡ软件和SobLoc网站预测cMyBP-C的亚细胞定位,NCBI的UniGene数据库分析其组织表达特异性。采用专业蛋白质分析系统Expasy (Expert Protein Analysis System)的ProtParam工具,分析cMyBP-C蛋白的理化性质,采用SignalP 4.0预测信号肽,TMHMM 2.0软件预测跨膜区域。SOPMA工具预测二级结构,并通过NCBI的Conserved Domain数据库与Smart在线预测cMyBP-C的结构域。采用STRING数据库,构建cMyBP-C与其他蛋白之间的相互作用网络。
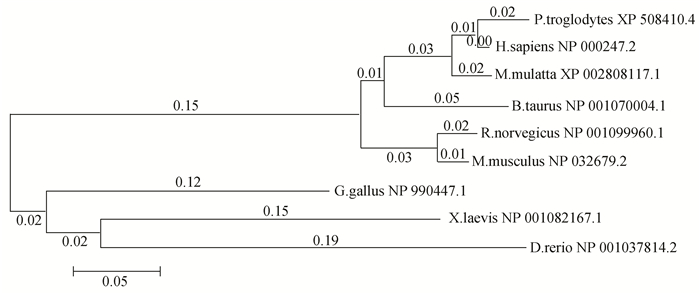
2 结果与分析 2.1 cMyBP-C蛋白多序列比对分析通过NCBI的BLAST数据库比对得到其它几种哺乳动物、鸟类、两栖类和鱼类中存在人cMyBP-C的同源蛋白,人cMyBP-C蛋白的氨基酸序列与黑猩猩、猕猴、牛、小鼠、大鼠、鸡、非洲爪蟾、斑马鱼的同源蛋白相似性分别为98.5 %、97.7 %、92 %、92 %、91.9 %、72.6 %、69.1 %、66.8 %。采用Clustal 2.1软件进行cMyBP-C同源蛋白间的多重序列比对后,通过Mega6的邻接法(Neighbor-joining, NJ)绘制序列系统进化树(见图 1),NJ法是基于序列同源性的一种算法,分支中数字表示进化距离。结果表明,人cMyBP-C蛋白和其它几种哺乳动物组成一支,与黑猩猩的同源性最高进化距离为0.02,与鸡的cMyBP-C同源蛋白进化距离为0.14,进化距离最远的是非洲爪蟾和斑马鱼分别为0.19和0.23。cMyBP-C蛋白的同源性与物种间的亲缘关系成正相关关系,该分子在物种的进化与演变中发挥一定的作用。
|
图 1 人cMyBP-C蛋白与其同源序列比对的分子进化树 Figure 1 The sequence alignment of human cMyBP-C protein and its homologous sequences |
NCBI的UniGene数据库EST结果显示,MYBPC3在以下组织中均有表达,拷贝数分别是:心脏592,肝24,肌肉18,肺14,淋巴结11,胎盘7,结蹄组织6,睾丸6,胚胎组织4,心脏中的表达量最高。通过SPORTⅡ亚细胞定位分析,cMyBP-C蛋白定位于细胞质的可能性较大(52.2%),其次分别为细胞核(30.4%),线粒体(13.0%)和分泌系统囊泡上(4.0%)。SobLoc网站分析cMyBP-C定位于细胞质,cMyBP-C是心肌细胞肌小节的组成成分,确实属于胞浆成分,而软件预测其细胞核的定位可能性也比较大,结合其在肝等其他组织中也有表达,预示cMyBP-C的未知功能值得进一步探究。
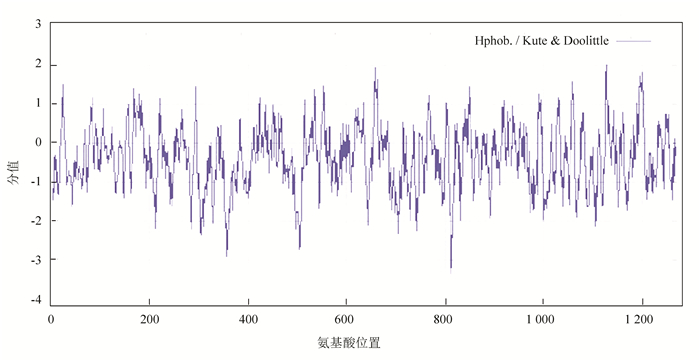
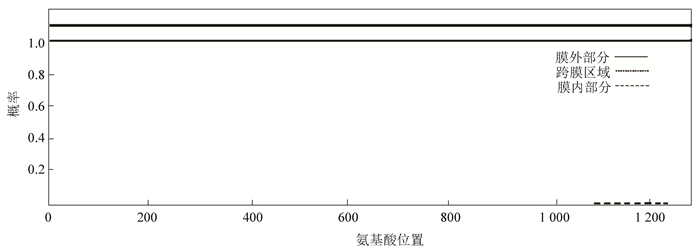
2.3 cMyBP-C蛋白质的理化性质人MYBPC3基因位于11号染色体(11p11.2)上,有3种mRNA可变剪切产物,分别是NM_000256.3、XM_011520118.1和XM_011520117.1,蛋白质产物分别是NP_000247.2,XP_011518420.1和XP_011518419.1。其中,NM_000256.3共有35个外显子,mRNA全长为3 825 nt, 编码全长为1 274个氨基酸组成的NP_000247.2产物是MYBPC3的蛋白共识编码序列,其ID为CCDS53621.1。cMyBP-C蛋白质分子式为C6254H9898N1732O1877S43,分子量为14 0762.4 Da,理论等电点预测为6.24,属酸性蛋白,cMyBP-C在哺乳动物网织红细胞内半衰期为30 h,不稳定系数为42.08,属不稳定蛋白质。PrortScale预测cMyBP-C亲水性最强的位点是第831位的赖氨酸,分值为-3.356,疏水性最强的位点是1 128位的谷氨酸,分值是2.022,cMyBP-C的整个氨基酸序列中亲水区域多于疏水区域(见图 2),Protparam预测cMyBP-C脂肪系数是79.73,总的平均亲水性为-0.392,所以其属于亲水蛋白。SignalP4.0预测cMyBP-C蛋白序列不含切割位点,无信号肽序列(见图 3)。TMHMM2.0在线分析cMyBP-C无跨膜结构域(见图 4)。采用PhosphoSitePlus分析cMyBP-C蛋白质翻译后修饰,发现K418会被乙酰化修饰,被磷酸化修饰位点有:S18、T59、S78、Y79、S86、S133、S212、Y213、T274、S275、S284、S286、T290、S304、T307、S311、Y340、Y373、S424、S427、Y548、S550、S588、T602、T607、S708、S830、Y1043、Y1119、Y1135、Y1136、Y1167、Y1172。cMyBP-C的磷酸化程度是决定心脏收缩的速度和力量的关键因素之一,探索其磷酸化动态变化会加深对心脏病理状态改变的理解。分析结果显示小鼠的cMyBP-C同源蛋白泛素化位点有K87、K102、K183、K308、K376、K391、K102、K394、K414、K446、K484、K539,针对人cMyBP-C蛋白是否会泛素化及其如何降解的问题,值得进一步探究。
|
图 2 程序分析cMyBP-C蛋白的疏水性 Figure 2 Hydrophobicity profile of the cMyBP-C protein |
|
图 3 cMyBP-C蛋白的信号肽分析结果 Figure 3 The result of cMyBP-C signal peptide anlysis |
|
图 4 cMyBP-C蛋白的跨膜结构分析 Figure 4 Transmembrane domain of cMyBP-C predicted by TMHMM |
图中的细实线、点状虚线和线性虚线分别表示膜外部分、跨膜区域和膜内部分,图中cMyBP-C蛋白位于膜外的概率几乎为100%,跨膜区域和位于膜内的区域的概率极低几乎为0,虚线与纵坐标为0的基线重叠。粗线是用来标识多肽链中跨膜区域所在位置的,因本文的cMyBP-C蛋白没有跨膜区域,所以在粗线上没有任何标记。
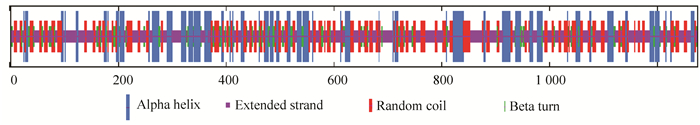
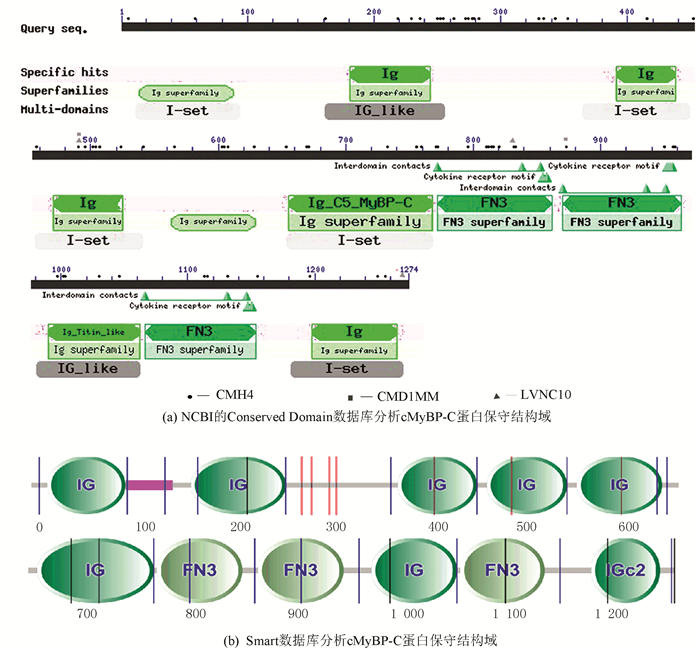
2.4 cMyBP-C蛋白质结构分析cMyBP-C蛋白的二级结构中,随机卷曲占51.73%,α-螺旋占28.18%,延伸链占20.09%(见图 5)。结构域分析表明,含有3个细胞因子受体超二级结构和3个结构域间相互作用模体,cMyBP-C属于Ⅲ型纤连蛋白超家族和免疫球蛋白超家族,含有3个Ⅲ型纤连蛋白结构域(Fibronectin type 3,FN3)和8个免疫球蛋白样结构域(Immunoglobulin,Ig)。FN3是两片反向平行的β-链形成的层状结构,一层含有3个β折叠链,另一层含4个,含FN3的多为受体蛋白。Ig由约70-110氨基酸组成,由两层反向平行的β-折叠链形成的层状结构,含9个β折叠链的称为IgV (Immunoglobulin variable),含7个β折叠链称IgC1(Immunoglobulin constant),如IgC尺寸和IgV一样则称为IgC2,其他类似结构域归为IgI (Immunoglobulin intermediate)。NCBI保守结构数据库将cMyBP-C的8个Ig结构域分为6个IgI型结构域和2个类Ig结构域,与Smart数据库预测结果基本一致,后者将C端的Ig结构域归类为Ig C2型(见图 6)。总之,cMyBP-C属于FN3蛋白超家族和Ig蛋白超家族,两者都是广泛存在于动物蛋白中的进化保守的蛋白结构域,多介导蛋白质-配体间的相互作用。
|
图 5 SOPMA预测cMyBP-C蛋白二级结构* Figure 5 Predicted secondary structure of cMyBP-C protein by SOPMA 注:彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2016年第4期doi:10.3969/j.issn.1672-5565.2016.04.04) |
|
图 6 cMyBP-C蛋白保守结构域 Figure 6 Conserved domain of cMyBP-C protein |
图 6(a)表示NCBI的Conserved Domain数据库分析结果,黑色圆点指示突变后表现为CMH4(Cardiomyopathy, familial hypertrophic 4)症状的位点,灰色方框表示突变后表现为CMD1MM (Cardiomyopathy, dilated 1MM)症状的位点,灰色三角表示突变后表现为LVNC10(Left ventricular noncompaction 10)症状的位点;图 6(b)表示Smart数据库分析结果。
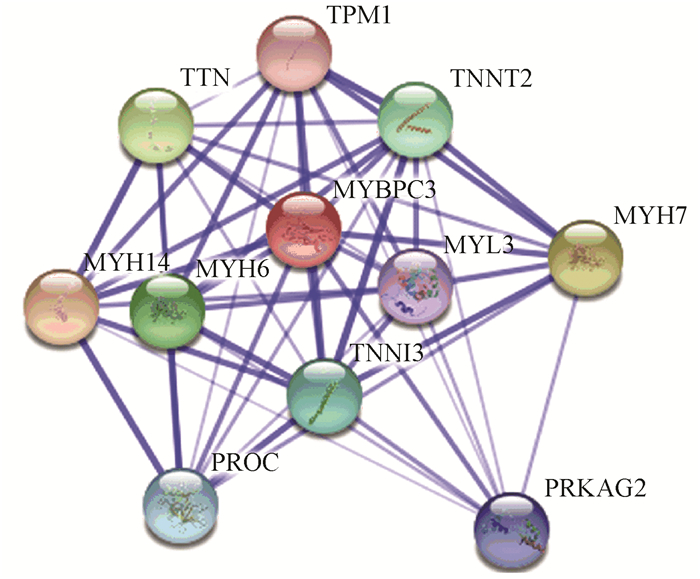
2.5 蛋白间相互作用分析利用STRING交互式数据库搜索蛋白质相互作用信息,置信度设置为0.700,数量限制为10个以内,构建cMyBP-C蛋白相互作用网络(见图 7)。cMyBP-C相互作用主要有肌球蛋白重链和轻链成分:MYH6(Myosin heavy chain 6)、MYH7、MYH14和MYL3(Myosin light chain 3),肌钙蛋白TNNT2(Troponin T2)和TNNI3,肌联蛋白TTN (Titin),原肌球蛋白TPM1(Tropomyosin 1),这些蛋白均属于肌纤维的组成成分,与cMyBP-C相互协调完成肌丝滑动的动态过程。值得关注的是,相互作用网络中还包括蛋白激酶PRKAG2(Protein kinase AMP-activated non-catalytic subunit gamma 2)以及依赖维生素K的丝氨酸蛋白酶PROC (Protein C)。
|
图 7 STRING预测cMyBP-C蛋白相互作用网络 Figure 7 Protein-protein interaction network for cMyBP-C predicted by STRING |
转录后修饰是调节蛋白质活性的重要机制。本文通过PhosphoSitePlus在线分析得出cMyBP-C含有33个磷酸化位点,这与文献报道cMyBP-C是高度磷酸化蛋白质是一致的。cMyBP-C磷酸化水平随着细胞状态不同而变化,心力衰竭终末期和肥厚型心肌病的特征之一是cMyBP-C磷酸化水平的降低[7]。去磷酸化的cMyBP-C和肌球蛋白结合,被依赖cAMP的蛋白激酶A (cAMP-dependent protein kinase, PKA)或CaMK2δ等其他激酶磷酸化后与肌动蛋白结合。在受到β-肾上腺素刺激时,磷酸化cMyBP-C加速横桥与肌动蛋白结合,从而加快横桥对细肌丝的招募,导致心脏抽搐程度增加[8],表明磷酸化的cMyBP-C是心脏收缩力度和幅度的一个关键调控因子。此外,多聚泛素化蛋白的积累是心脏疾病的一个共同特征[9],蛋白酶抑制剂治疗可以逆转MYBPC3突变体小鼠的HCM疾病表型,抑制蛋白酶体可以起到改善心脏功能和抗心肌肥厚的作用[10],本文预测到小鼠cMyBP-C蛋白存在多个泛素化修饰位点,而人cMyBP-C泛素化修饰及其降解方式与心肌病的关系有待于我们进一步探究。
MYBPC3基因突变引起扩张型心肌病,据文献报道,突变位点的不同可引起不同的症状,大致可以分为以下三类:CMH4型的特点是心室不对称肥厚,常累及室间隔,症状包括呼吸困难、晕厥、心悸、胸痛,很容易地由于运动引发[11];CMD1MM型的特点是心室扩张和收缩功能受损,导致充血性心力衰竭和心律失常[12];LVNC10型的特点是由于心肌形态发育受阻致左室心肌致密化不全、左心室肥厚、收缩功能差、小梁增深[13]。突变位点大致位置与所致表型见图 6。最近研究表明,对小鼠MYBPC3的基因治疗,可以恢复心肌cMyBP-C的水平,并能长期预防心肌病的发生发展[14]。目前CRISPR/Cas9系统已被应用于哺乳动物基因单一或群集突变的校正[15],这种方法有望很快用于纠正MYBPC3的突变,更有效地改善HCM症状。
PRKAG2基因编码单磷酸腺苷激活蛋白激酶γ2亚基,文献报道PRKAG2突变可引起心脏综合症,出现胞内糖原沉积、心室预激、传导系统疾病和左心室肥厚等各种表型[16],占肥厚性心肌病(HCM)患者比例约为0.23%~1.00%[17]。cMyBP-C和PRKAG2均为HCM的重要致病基因,尚未见报道两者间的关系研究,本文分析得出两者存在蛋白间相互作用关系,有望成为深入解析HCM疾病信号通路新的突破口。PROC是一种多功能的丝氨酸蛋白酶,已知功能主要是参与凝血系统的调节,可降解凝血因子V和VIII,具有抗细胞凋亡和抗炎症的作用[18],最新研究阐明PROC可抵抗心肌缺血/再灌注引起的急性炎症反应[19]。本文通过STRING在线预测软件分析到cMyBP-C与PROC的相互作用,两者的关系及参与的具体生理过程值得我们进一步实验研究。
目前,基因治疗是国际上的研究热点,特别是CRISPR/Cas9系统应用的深入发掘,家族性肥厚型心肌病中有40%的原因是MYBPC3基因的突变,本文对这一最重要的家族性肥厚型心肌病的易感基因的结构与功能进行生物信息学分析,为下一步开发针对该分子的基因治疗方式奠定基础。本文通过生物信息学方法分析了MYBPC3基因的保守性、cMyBP-C蛋白的亚细胞定位、组织特异性、蛋白质高级结构信息、及其相互作用蛋白,揭示了cMyBP-C是一种肌节特有的多功能蛋白分子,为完善MYBPC3基因的分子功能以及研究其在调节心肌舒缩过程中参与的具体信号通路提供一定的依据。
| [1] |
BEHRENSG V, MEARINI G, GEDICKE H C, et al. MYBPC3 in hypertrophic cardiomyopathy: from mutation identification to RNAbased correction[J]. Pflugers Arch, 2014(466): 215–223.
DOI:10.1007/s00424-013-1409-7 ( 0) 0)
|
| [2] |
CARRIER L, MEARINI G, STATHOPOULOU K, et al. Cardiac myosin-binding protein C (MYBPC3) in cardiac pathophysiology[J]. Gene, 2015, 573(2): 188–197.
DOI:10.1016/j.gene.2015.09.008 ( 0) 0)
|
| [3] |
MOSS R L, FITZSIMONS D P, RALPHE J C. Cardiac MyBP-C regulates the rate and force of contraction in mammalian myocardium[J]. Circulation Research, 2015, 116(1): 183–192.
DOI:10.1161/CIRCRESAHA.116.300561 ( 0) 0)
|
| [4] |
HARRIS S P, BARTLEY C R, HACKER T A, et al. Hypertrophic cardiomyopathy in cardiac myosin binding protein-C knockout mice[J]. Circulation Research, 2002, 90(5): 594–601.
DOI:10.1161/01.RES.0000012222.70819.64 ( 0) 0)
|
| [5] |
KENNETH S G, RANGANATH M, JULIAN E S. The contribution of cardiac myosin binding protein-c Ser282 phosphorylation to the rate of force generation and in vivo cardiac contractility[J]. Journal of Insect Physiology, 2014, 592(Pt 17): 3747–3765.
DOI:10.1113/jphysiol.2014.276022 ( 0) 0)
|
| [6] |
JACQUES A M, COPELAND O, MESSER A E, et al. Myosin binding protein C phosphorylation in normal, hypertrophic and failing human heart muscle[J]. Journal of Molecular and Cellular Cardiology, 2008, 45(2): 209–216.
DOI:10.1016/j.yjmcc.2008.05.020 ( 0) 0)
|
| [7] |
LYNCH T L, SIVAGURU M, VELAYUTHAM M, et al. Oxidative stress in dilated cardiomyopathy caused by MYBPC3 mutation cMyBP-C[J]. Oxidative Medicine and Cellular Longevity, 2015(2015): 424751.
DOI:10.1155/2015/424751 ( 0) 0)
|
| [8] |
COPELAND O, SADAYAPPAN S, MESSER A E, et al. Analysis of cardiac myosin binding protein-C phosphorylation in human heart muscle[J]. Journal of Molecular and Cellular Cardiology, 2010, 49(6): 1003–1011.
DOI:10.1016/j.yjmcc.2010.09.007 ( 0) 0)
|
| [9] |
WEEKES J, MORRISON K, MULLEN A, et al. Hyperubiquitination of proteins in dilated cardiomyopathy[J]. Journal of Proteomics, 2003, 3(2): 208–216.
DOI:10.1002/pmic.200390029 ( 0) 0)
|
| [10] |
SASKIA S, SONIA R S, BIRGIT G, et al. Proteasome inhibition slightly improves cardiac function in mice with hypertrophic cardiomyopathy[J]. Frontiers in Physiology, 2014, 16(5): 484.
DOI:10.3389/fphys.2014.00484 ( 0) 0)
|
| [11] |
MORITA H, REHM H L, MENESSES A, et al. Shared genetic causes of cardiac hypertrophy in children and adults[J]. The New England Journal of Medicine, 2008, 358(18): 1899–1908.
DOI:10.1056/NEJMoa075463 ( 0) 0)
|
| [12] |
HERSHBERGER R E, NORTON N, MORALES A, et al. Coding sequence rare variants identified in MYBPC3, MYH6, TPM1, TNNC1, and TNNI3 from 312 patients with familial or idiopathic dilated cardiomyopathy[J]. Circulation Cardiovascular Genetics, 2010, 3(2): 155–161.
DOI:10.1161/CIRCGENETICS.109.912345 ( 0) 0)
|
| [13] |
PROBST S, OECHSLIN E, SCHULER P, et al. Sarcomere gene mutations in isolated left ventricular noncompaction cardiomyopathy do not predict clinical phenotype[J]. Circulation Cardiovascular Genetics, 2011, 4(4): 367–374.
DOI:10.1161/CIRCGENETICS.110.959270 ( 0) 0)
|
| [14] |
BEHRENS G V, MEARINI G, GEDICKE H C, et al. MYBPC3 in hypertrophic cardiomyopathy: from mutation identification to RNA-based correction[J]. Pflugers Arch, 2014, 466(2): 215–223.
DOI:10.1007/s00424-013-1409-7 ( 0) 0)
|
| [15] |
HSU P D, LANDER E S, ZHANG F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262–1278.
DOI:10.1016/j.cell.2014.05.010 ( 0) 0)
|
| [16] |
PÖYHÖNEN P, HIIPPALA A, OLLILA L, et al. Cardiovascular magnetic resonance findings in patients with PRKAG2 gene mutations[J]. Journal of Cardiovascular Magnetic Resonance, 2015(17): 89.
DOI:10.1186/s12968-015-0192-3 ( 0) 0)
|
| [17] |
GRUNER C, CARE M, SIMINOVITCHK, et al. Sarcomere protein gene mutations in patients with apical hypertrophic cardiomyopathy[J]. Circulation Cardiovascular Genetics, 2011, 4(3): 288–295.
DOI:10.1161/CIRCGENETICS.110.958835 ( 0) 0)
|
| [18] |
GRIFFIN J H, ZLOKOVIC B V, MOSNIER L O. Protein C anticoagulant and cytoprotective pathways[J]. International Journal of Hematology, 2012, 95(4): 333–345.
DOI:10.1007/s12185-012-1059-0 ( 0) 0)
|
| [19] |
WILDHAGEN K C, SCHRIJVER R, BECKERS L, et al. Effects of exogenous recombinant APC in mouse models of ischemia reperfusion injury and of atherosclerosis[J]. PLoS One, 2014, 9(7): e101446.
DOI:10.1371/journal.pone.0101446 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14

