据普查资料显示,肝癌(Hepatocellular carcinoma,HCC)的年死亡率仅次于胃癌和肺癌,成为威胁人类健康的重要肿瘤疾病之一,而全球60%~80%的肝癌与乙肝病毒感染相关[1-3]。HCC表现出地理区域相关的差异特征,约占肿瘤的4%[4]。目前,应用于临床的抗肝癌药物有限,分子机制的研究发展较慢,肝癌治疗缺乏突破性,尚待深入研究重要分子及蛋白质的结构功能、表达调控及生理和病理环境下的调控机制。
伴HBV感染性肝癌的发生与发展涉及HBV和宿主基因的共同调控影响。随着分子生物学、细胞生物学等技术的应用,有关肝细胞癌发生发展中机制方面的研究取得了一定进展,主要集中在肝癌细胞增生机制,肝癌细胞凋亡机制的失活,细胞分化异常,肿瘤血管生成以及信号转导通路。RBR蛋白家族成员Parkin基因与泛素相关蛋白分解途径有关,已发现Parkin的缺失能促进肝癌的发生与发展[5-6]。另外,研究发现细胞因子,如肝细胞生长因子、血小板源性生长因子、转化生长因子等与肝癌的进程密切相关[7-9]。但是,仅仅从局部关注单个或某几个基因已经不能满足这种具有高度复杂性的调控研究[10]。立足于调控网络整体,肿瘤组织中某些基因异常表达且与其他多个基因关系密切,它们的表达可能在肿瘤的发生发展过程中扮演重要的角色,这类处于调控网络枢纽位置的基因称作枢纽基因(Hub genes)。近年来,随着基因芯片、RNA-Seq等高通量测序技术的兴起,产生了大规模组学数据[10],进一步数据分析与挖掘将有利于系统性地研究多个基因表达关联性。因此,根据基因芯片数据来研究伴HBV感染性肝癌发生与发展过程中的枢纽基因具有明显意义。
在生物信息学领域中,网络分析的应用日渐成熟。其中,作为系统生物学分析方法中极具潜力的一种新方法,权重基因共表达网络分析(Weight Gene Co-express Network Analysis,WGCNA)以其在描述解析分子作用机制和网络关系等方面具有的独特优势迅速脱颖而出。自开发以来,WGCNA已成功应用于多种生物环境,并取得了一系列的研究成果[11-14]。
本研究采用WGCNA进行研究[15-16],从大量基因中筛选与乙肝相关性肝癌密切相关的枢纽基因。结合基因本体富集分析,进行功能注释,然后对基因模块进行功能分析,研究枢纽基因与肝癌发生与发展间的关联性[17],这些工作能够为某些信号通路的调控机制提供参考,有利于构建评估HCC早期诊断预测平台,降低人群HCC发病率及死亡率。有理由相信,随着基因筛选方法的不断发展,WGCNA有望揭示HBV相关性肝癌发生与发展的分子机制,并在识别潜在的治疗靶点等领域发挥越来越大的作用。
1 材料与方法 1.1 数据与材料本文数据集来源于NCBI数据库中的数据GSE47197(http://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE47197)。质粒pcDNA3.1-SHARPIN由本实验室构建,酶切位点BamHI和XbaI;上游引物:CGCGGATCCATGG CGCCGCCAGCGG,下游引物:TGCTCTAGACTAGGTG GAAGCTGCAGCAAG,酶切鉴定后送到武汉天一辉远公司进行测序并成功验证。在大肠杆菌DH5α中扩增,Takara质粒中量提取试剂盒提取纯化,-20 ℃保存。HepG2细胞为中国科学院武汉病毒研究所李朝阳研究员惠赠,培养细胞用含10%胎牛血清(FBS)的DMEM (FBS及DMEM购自Biological Industries©)。
1.2 WGCNA 1.2.1 构建差异表达谱矩阵数据鉴于一个基因对应一个探针,而一个探针对应多个Identifier VALUE,需另外下载注释文件(其中共计28 782个Identifier VALUE实际对应16 390个基因)用于计算同一基因的Identifier VALUE平均值,通过Multi Experiment Viewer,计算每个基因的倍数差异值(取对数变换),并推断差异发生的概率p,当倍数差异值大于预设倍数,且筛选p < 0.05时即判定为差异表达基因(即该基因表达无差异的概率小于0.05)。最终分别构建了癌症(Tumor)组和癌旁(Non-tumor)组基因差异表达谱矩阵。
1.2.2 去除离群的样本离群样本定义为使聚类树形图高度发生明显偏移的样本。离群的样本对网络模块分析结果具有较大的影响,在构建网络之前,必须先检查是否存在离群样本,去除相关数据。通过取样本的平均表达值构建距离矩阵,选择类平均法对样本进行层次聚类,绘制样本聚类树形图。
1.2.3 基因模块分析作为一种系统生物学分析方法,WGCNA分析建立了幂指数邻接函数,具体方法就是对基因表达相关系数取n次幂指数加权,即用邻接系数αij作为衡量它们之间相关关系的指标:αij=power(Sij, β)=|Sij|β,使得相关系数数值的分布在加权系数β作用下逐渐符合无尺度分布。其中,算法定义了相关系数至少应达到0.8,则表达量数据就转换为调控关系。基因间相关系数经过幂函数处理,理论上能够按表达模式对基因进行分类。通过动态剪切树聚类分析获取多个共表达模块,这样每个模块里的基因具有非常类似的表达模式,它以颜色命名模块。WGCNA选择加权系数的来无限逼近Scale-free网络分布,使得网络调控关系符合“无尺度关系”,即服从幂律分布。
WGCNA的主要任务就是构建加权基因共表达网络/模块,分析的核心步骤如图 1所示:
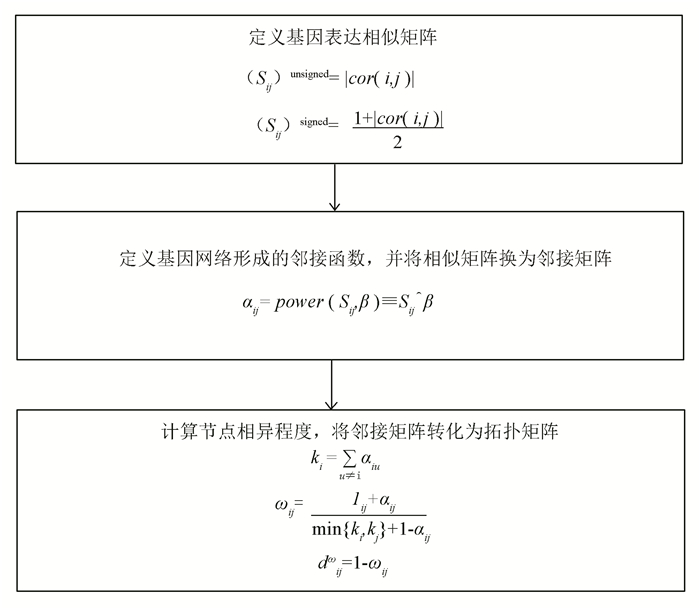
|
图 1 WGCNA算法基本步骤 Figure 1 The basic steps of WGCNA |
总之,WGCNA使用拓扑重叠矩阵(Topological Overlap Matrix,TOM)来计算基因与基因之间的关联程度,得到的模块具有生物学意义。
1.3 基因富集分析利用生物信息学在线工具GOrilla对筛选的网络模块基因进行GO富集分析,利用PANTHER进行通路分析,并以统计学方法计算p值,预测其生物学功能。
1.4 枢纽基因筛选为了直观地展示模块内部的调控关系网络,我们将以癌症组计算得到的模块为分类标准,计算癌旁组的TOM,然后利用VisANT软件对癌症组与癌旁组同一类模块进行可视化展示[18],对比模块中各基因的节点数并筛选枢纽基因。
1.5 SHARPIN对HepG2迁移影响实验观察本研究筛选出来的枢纽基因之一,SHANK家族蛋白的RH结构域交互作用蛋白(SHANK-associated RH domain interacting protein,以下简称SHARPIN),对HepG2细胞的迁移影响:(a)在细胞培养板六孔板中以每孔5×105个细胞铺板,过夜长满后,换无血清培养基培养12 h;(b)用10 μl枪头沿着直尺垂直于背后的横线划痕,用PBS小心清洗划下的细胞;(c)按实验设置分别转染各组细胞,4~6 h后换成含10% FBS的DMEM培养基于37 ℃、5% CO2的细胞培养箱中培养,分别于0/24/48 h取样,使用Olympus IX 73倒置荧光显微镜观察并拍照记录。
2 结果 2.1 数据基本信息GSE47197全基因组表达数据包括61个癌症组与63个癌旁组数据,这些数据均已经过背景校正和标准化处理。由于数据中包括的基因探针数量太大,质量低且差异不显著的数据较多,在构建基因共表达模块与网络时,为了减轻噪音和减轻计算机的运行负担,还需要筛选差异基因保留差异较大的基因。通过Multi Experiment Viewer软件,滤过差异并不显著的基因,最后保留5 968个差异基因(abFC>2,FDR < 0.05)作为本文的重点研究对象。
2.2 样本数据初筛采用层次聚类法,得到癌症组及癌旁组样本聚类图(见图 2和图 3),发现61个癌症组样本表达谱数据中存在1个离群的样本,而63个癌旁组织样本表达谱数据中存在3个离群的样本。离群样本经过剪枝去除(剪枝高度分别为115和90),最后剩余60个癌组织样本和60个癌旁组织样本分别进入后续的网络分析。
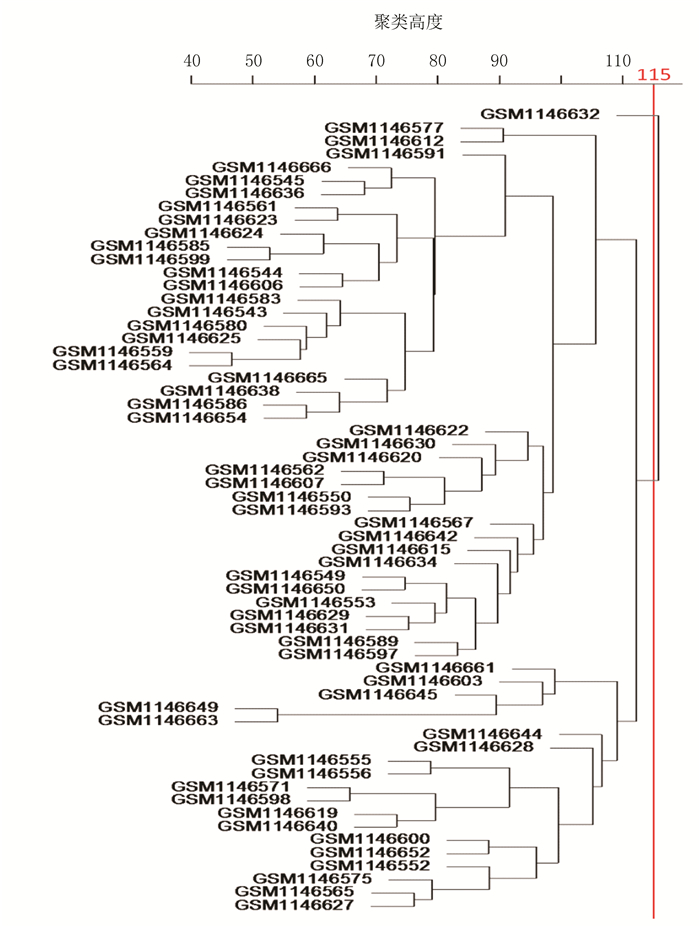
|
图 2 癌组织样本聚类树 Figure 2 Tumor samples clustering |

|
图 3 癌旁组织样本聚类树 Figure 3 Non-tumor samples clustering |
选择合适的邻接矩阵权重参数β以尽量满足无尺度分布。参数β从1~20取值,对某节点连接度的对数log (i)与该节点出现的概率的对数log (p(i))建立线性模型,参数β即系数R的平方,按照候选的参数β,返回被检测的网络参数(见图 4和图 5)。我们选取了首次接近0.90时的β值来构建基因网络/模块,针对肿瘤组织和癌旁组织样本模型,我们分别选择β=9和β=10,这样既保证了网络接近于无尺度网络同时也是使曲线趋于平滑的最小阈值,并且它也使得网络的平均链接程度不会太小,这有利于网络包含足够的信息(例如,挖掘模块)。
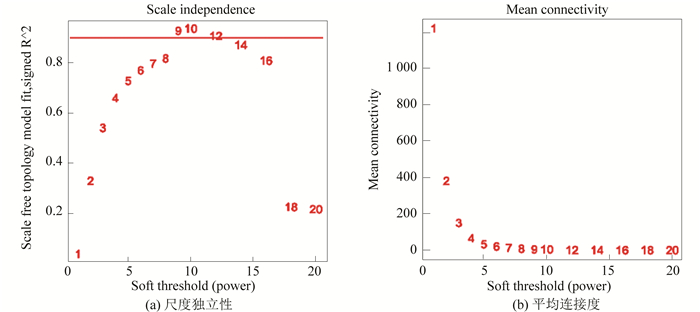
|
图 4 癌症组参数β选取参考图 Figure 4 Figures for selecting β in tumor sample 注:图 4的横轴均代表权重参数β,(a)图的纵轴代表对应的网络中log (i)与log (p (i))相关系数的平方,该系数越高(最小应达到0.8,完美无尺度网络的模型适应指数是1),表明该网络越逼近无尺度网络的分布;(b)图的纵轴代表模型中所有基因邻接系数的均值,它反映了网络的平均连接水平。 |
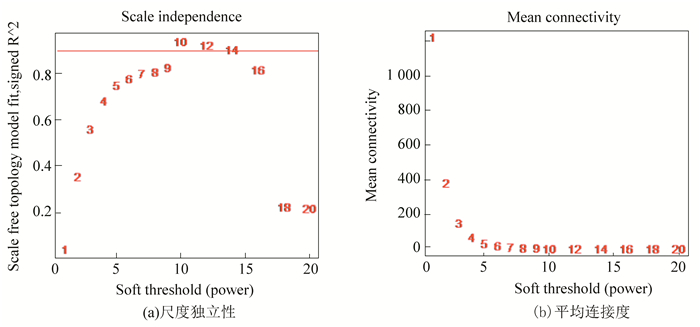
|
图 5 癌旁组参数β选取参考图 Figure 5 Figures for selecting β in non-tumor sample 注:图 5的横轴均代表权重参数β,(a)图的纵轴代表对应的网络中log (i)与log (p (i))相关系数的平方,该系数越高(最小应达到0.8,完美无尺度网络的模型适应指数是1),表明该网络越逼近无尺度网络的分布;(b)图的纵轴代表模型中所有基因邻接系数的均值,它反映了网络的平均连接水平。 |
选取β=9来计算癌症组的网络拓扑重叠TOM,利用层次聚类法得到基因系统聚类树。根据动态分层剪切树算法来挖掘基因模块。设置每个基因模块中基因数目最小为50,设定基因的聚类高度上限为0.995。基因模块分析结束后,计算每个基因模块的特征向量基因ME (Module Eigengene)对这些模块聚类作降维处理,将距离较近的模块合并成新的模块(设置height=0.25)。
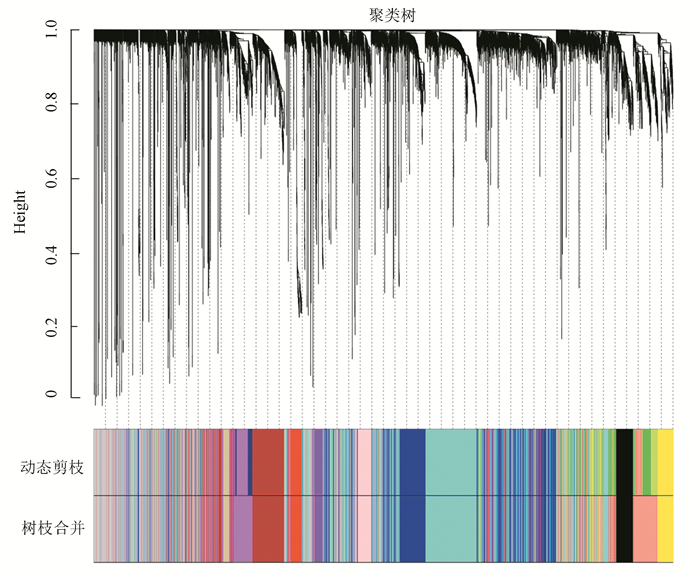
如图 6所示,观察16个模块最终降维得到13个共存在基因模块,而模块中的基因个数从74到1 161不等。另外,grey模块表示未分配到任何一个模块的基因集, 其中含有1 169个基因。
|
图 6 动态分层聚类*
Figure 6 Merged dynamic vluster
注:不同的颜色表示不同的基因模块,Dynamic Tree Cut对应最初得到的16个模块,Merged dynamic对应最终得到的合并之后的13个模块。 *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第4期doi:10.3969/j.issn.1672-5565.2016.04.02)。 |
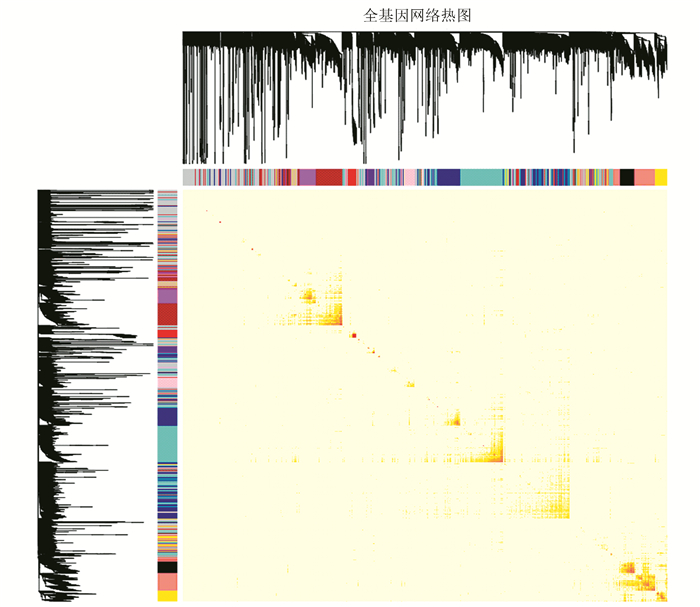
根据拓扑重叠程度,选择全部的基因制作热图(见图 7)。通过WGCNA,我们识别了癌症中相关度高的基因模块,即共表达模块代表的基因在功能上具有紧密的关系。
|
图 7 TOM图展示基因网络的基因相互关系热图*
Figure 7 TOMplot:topological overlap matrix plot
注:该图基于拓扑重叠,选择所有基因进行作图,热图中每一行及每一列均对应一个基因,颜色越深,提示基因间的相关性或者拓扑重叠越高,基因之间的关系越密切。图中的左侧和上方为基因聚类树形图集对应的基因模块。 *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第4期doi:10.3969/j.issn.1672-5565.2016.04.02)。 |
经过GO富集分析,我们得到了各个模块的详细功能(见表 1),发现它们主要参与免疫系统调节、小分子代谢过程、刺激反应、炎症反应凝血级联反应、细胞外基质受体相互作用、细胞周期、趋化因子和细胞因子介导的炎症反应信号通路、血管形成等分子功能和生物学过程及信号通路,可能与HBV相关肝癌的发生有一定的联系。
| 表 1 GOrilla GO功能富集分析结果 Table 1 The GO analysis of GOrilla |
根据富集分析结果,选择与癌症的发生与发展相关性较高的模块为主要研究对象;基于Eigengenes的PANTHER分析,发现Pink模块与Magenta模块的相关基因与T细胞的活化和通路趋化因子以及细胞因子介导的炎症反应信号通路存在显著富集关联(Enrichment倍数> 5,p值=0.000)。
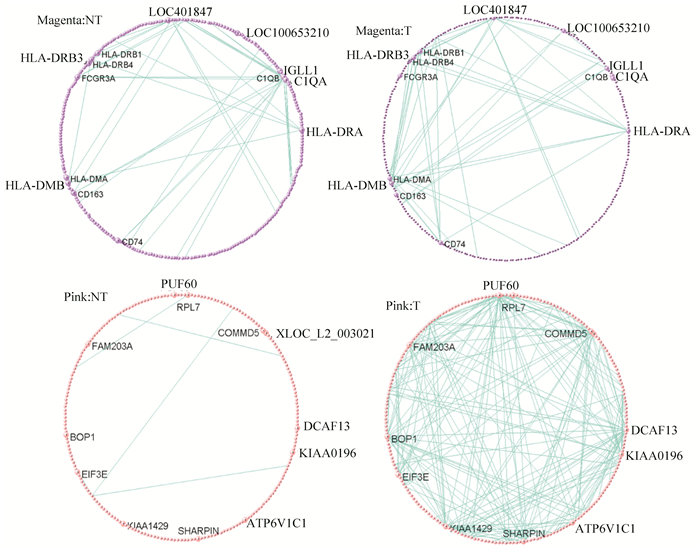
2.6 枢纽基因识别结合模块分析与富集分析的结果,将显著通路模块中的基因调控关系可视化展示,以Pink模块与Magenta模块为例(见图 8,同一模块的拓扑重叠阈值相同)。其中,Pink选择的权重值标准为0.07~0.16,Magenta选择的权重值标准为0.18~0.28。通过可视化展示,Pink模块与Magenta模块中的基因存在多个枢纽基因(见表 2)。
|
图 8 模块中的关键节点基因 Figure 8 The hub genes indicated in the module 注:左栏NT表示癌旁组织,右栏T表示癌组织,根据节点度数差值反映枢纽基因的重要程度。 |
| 表 2 模块中的关键节点枢纽基因 Table 2 The hub Genes of modules |
细胞划痕实验结果见图 9,实验结果显示:右栏图 9(b)过表达SHARPIN组与左栏图 9(a)对照组进行比较,除了刚开始转染的0小时(见图 9)无差别外,转染24小时后图 9(b)过表达SHARPIN组比图 9(a)对照组迁移更明显(见图 9);48小时后,这种差别更大,图 9(b)过表达SHARPIN组中左右二边红线边界的间距只有图 9(a)对照组间距的50%左右(见图 9)。该结果显示,SHARPIN基因能够促进细胞迁移,符合GO分析结果,提示上调SHARPIN基因促进乙肝相关性肝癌发生或发展。

|
图 9 HepG2细胞在转染质粒0h/24h/48h时的迁移情况 Figure 9 Migration of HepG2 at 0h/24h/48h post transfection |
目前学术界普遍认为HBV相关性肝癌的发生与发展的情况异常复杂,不仅与HBV相关,还与肝细胞微环境密切相关。细胞内的调控网络发生异常导致肝癌的发生,其中伴随着某些信号通路的改变,但大部分的调控关系仍处于长期探索中。
本研究首先从NCBI的GEO数据库下载HBV相关性肝癌的表达数据集GSE47197(含124个样本),利用WGCNA模拟基因簇筛选出了HBV相关性肝癌基因模块并对模块进行GO功能注释和KEGG通路富集分析,发现了与HBV相关性肝癌显著相关的基因模块内的枢纽基因,如IGF2、CD163、EIF3E,CARM1、HLA-DRB1/3/4、CD72等,注释到了细胞外基质(ECM)受体相互作用、整合素信号通路、趋化因子和细胞因子介导的炎症反应信号通路、血管形成等生物学过程。值得注意的是部分模块同时显著富集到T细胞的活化和通路趋化因子和细胞因子介导的炎症反应信号通路,提示这些通路模块中的基因与HBV相关性肝癌的发生发展有着显著的内在关联[19, 20]。基于此,这些工作有助于为肝癌发生的调控机制的研究以及肝癌的治疗诊断、靶点选择提供更多的参考信息。
显然,和全局网络中的枢纽基因相比,这些模块中的枢纽基因更具有生物学意义,其中部分枢纽基因功能已经得到行业的普遍认可,如CD74、CD163、EIF3E、HLA-DRB1/3/4等,这恰好说明通过WGCNA筛选出的枢纽基因具有生物学意义;相反地,有些基因的功能,尤其它们在肝癌中行使的生物学功能还不太清楚,如SHARPIN、BOP1、FCGR3A等[21-23]。
本研究筛选出来的枢纽基因SHARPIN,经实验验证发现能促进细胞迁移,提示该基因对于研究HBV相关性肝癌的发生与发展具有价值。前期有文献报道SHARPIN上调ERK/Akt信号通路,而细胞因子Akt和ERK参与增殖、分化、侵袭和转移。总体来看,关于SHARPIN在HBV相关性肝癌发生发展中上下游分子的相互作用等方面,目前知之甚少。我们的该项研究,对肝癌发生的调控机制以及发现HBV慢性感染导致肝癌的新型诊断标志物和(或)药物作用靶点,提供了新的视野。
| [1] |
JEMAL A, BRAY F, CENTER M M, et al. Global cancer statistics[J]. CA: A Cancer Journal for Clinicians, 2011, 61(2): 69–90.
DOI:10.3322/caac.20107 ( 0) 0)
|
| [2] |
BOSETTI C, TURATI F, LA-VECCHIA C. Hepatocellular carcinoma epidemiology[J]. Best Practice & Research Clinical Gastroenterology, 2014, 28(5): 753–770.
DOI:10.1016/j.bpg.2014.08.007 ( 0) 0)
|
| [3] |
张原青, 郭津生. 乙型肝炎病毒促进肝细胞性肝癌发生的机制[J]. 中华肝脏病杂志, 2016, 24(2): 152–156.
ZHANG Yuanqing, GUO Jinsheng. Pathogenesis of hepatitis B virus-related hepatocellular carcinoma[J]. Chinese Journal of Hepatology, 2016, 24(2): 152–156.
DOI:10.3760/cma.j.issn.1007-3418.2016.02.018 ( 0) 0)
|
| [4] |
ZENG H, ZHENG R, GUO Y, et al. Cancer survival in China, 2003-2005: A population-based study[J]. International Journal of Cancer, 2015, 136(8): 1921–1930.
DOI:10.1002/ijc.29227 ( 0) 0)
|
| [5] |
WOO M G, XUE K, LIU J, et al. Calpain-mediated processing of p53-associated parkin-like cytoplasmic protein (PARC) affects chemosensitivity of human ovarian cancer cells by promoting p53 subcellular trafficking[J]. Journal of Biological Chemistry, 2012, 287(6): 3963–3975.
DOI:10.1074/jbc.M111.314765 ( 0) 0)
|
| [6] |
WANG F, DENSION S, LAI J, et al. Parkin gene alterations in hepatocellular carcinoma[J]. Genes Chromosomes & Cancer, 2004, 40(2): 85–96.
DOI:10.1002/gcc.20020 ( 0) 0)
|
| [7] |
LAI J P, CHIEN J R, MOSER D R, et al. hSulf1 sulfatase promotes apoptosis of hepatocellular cancer cells by decreasing heparin-binding growth factor signaling[J]. Gastroenterology, 2004, 126(1): 231–248.
DOI:10.1053/j.gastro.2003.09.043 ( 0) 0)
|
| [8] |
POTAPOVA O, FAKHRAI H, BAIRD S, et al. Platelet-derived growth factor-B/v-sis confers a tumorigenic and metastatic phenotype to human T98G glioblastoma cells[J]. Cancer Research, 1996, 56(2): 280–286.
DOI:10.1139/cjpp-79-7-566 ( 0) 0)
|
| [9] |
BOMMIREDDY R, ORMSBY I, YIN M, et al. TGFβ1 Inhibits Ca2+-Calcineurin-Mediated activation in thymocytes[J]. Journal of Immunology, 2003, 170(7): 3645–3652.
DOI:10.4049/jimmunol.170.7.3645 ( 0) 0)
|
| [10] |
MINGUEZ B, HOSHIDA Y, VILLANUEVA A, et al. Gene-expression signature of vascular invasion in hepatocellular carcinoma[J]. Journal of Hepatology, 2011, 55(6): 1325–1331.
DOI:10.1016/j.jhep.2011.02.034 ( 0) 0)
|
| [11] |
PING Y, DENG Y, WANG L, et al. Identifying core gene modules in glioblastoma based on multilayer factor-mediated dysfunctional regulatory networks through integrating multi-dimensional genomic data[J]. Nucleic Acids Research, 2015, 43(4): 1997–2007.
DOI:10.1093/nar/gkv074 ( 0) 0)
|
| [12] |
LIU Z P. Reverseengineering of genome-wide gene regulatory networks from gene expression eata[J]. Current Genomics, 2015, 16(1): 3–22.
DOI:10.2174/1389202915666141110210634 ( 0) 0)
|
| [13] |
HE D, LIU Z P, HONDA M, et al. Coexpression network analysis in chronic hepatitis B and C hepatic lesion reveals distinct patterns of disease progression to hepatocellular carcinoma[J]. Journal of Molecular Cell Biology, 2012, 4(3): 140–152.
DOI:10.1093/jmcb/mjs011 ( 0) 0)
|
| [14] |
IVLIEV A E, 'T HOEN P A, SERGEEVA M G. Coexpression network analysis identifies transcriptional modules related to proastrocytic differentiation and sprouty signaling in glioma[J]. Cancer Research, 2010, 70(24): 10060–10070.
DOI:10.1158/0008-5472.CAN-10-2465 ( 0) 0)
|
| [15] |
TAMINAU J, MEGANCK S, LAZAR C, et al. Unlocking the potential of publicly available microarray data using inSilicoDb and inSilicoMerging R/Bioconductor packages[J]. BMC Bioinformatics, 2012, 13(1): 1–9.
DOI:10.1186/1471-2105-13-335 ( 0) 0)
|
| [16] |
LANGFELDER P, HORVATH S. WGCNA: an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9(1): 1–13.
DOI:10.1186/1471-2105-9-559 ( 0) 0)
|
| [17] |
WANG J G, HUANG Q, LIU Z P, et al. NOA: a novel network ontology analysis method[J]. Nucleic Acids Research, 2011, 39(13): e87.
DOI:10.1093/nar/gkr251 ( 0) 0)
|
| [18] |
HU Z, SNITKIN E S, DELISI C. Visant: Anintegrative framework for networks in systems biology[J]. Briefings in Bioinformatics, 2008, 9(4): 317–325.
DOI:10.1093/bib/bbn020 ( 0) 0)
|
| [19] |
UTSUNOMIYA T, SHIMADA M, KUDO M, et al. A comparison of the surgical outcomes among patients with HBV-positive, HCV-positive, and non-B non-C hepatocellular carcinoma: a nationwide study of 11, 950 patients[J]. Annals of Surgery, 2015, 261(3): 513–520.
DOI:10.1097/SLA.0000000000000821 ( 0) 0)
|
| [20] |
SINGAL A K, SALAMEH H, KUO Y F, et al. Meta-analysis: the impact of oral anti-viral agents on the incidence of hepatocellular carcinoma in chronic hepatitis B[J]. Alimentary Pharmacology & Therapeutics, 2013, 38(2): 98–106.
DOI:10.1111/apt.12344 ( 0) 0)
|
| [21] |
WANG Z, POTTER C S, SUNDBERG J P, et al. SHARPIN is a key regulator of immune and inflammatory responses[J]. Journal of Cellular & Molecular Medicine, 2012, 16(10): 2271–2279.
DOI:10.1111/j.1582-4934.2012.01574.x ( 0) 0)
|
| [22] |
CHUNG K Y, CHENG I K, CHING A K, et al. Block of proliferation 1 (BOP1) plays an oncogenic role in hepatocellular carcinoma by promoting epithelial-to-mesenchymal transition[J]. Hepatology, 2011, 54(1): 307–318.
DOI:10.1002/hep.v54.1 ( 0) 0)
|
| [23] |
CONESA-ZAMORA P, SANTACLARA V, GADEA-NINOLES E, et al. Association of polymorphism in FcGR3A gene and progression of low-grade precursor lesions of cervical carcinoma[J]. Human Immunology, 2009, 71(3): 314–317.
DOI:10.1016/j.humimm.2009.11.012 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


