基因组DNA序列k-mer非随机使用的已有许多研究报道。研究主要关注k-mer字频非随机使用的生物学意义[1-2]、k-mer分布的概率模型[3-4]、稀有k-mer和富含k-mer的片段及其在基因组序列上的分布[5-8]。一些工作分析了特殊k-mer片段与CpG岛序列的关系[9-11],从k-mer使用入手寻找功能位点的调控片段,预测RNA功能片段,给出基因组组装方法[12-15]。最近,一些研究者特别关注了基因组序列k-mer的频谱分布,期望揭示基因组序列的构成特性和进化。Chor[16]研究了近百个物种基因组序列k-mer频谱的分布特征,发现不同生物基因组序列或不同类型DNA序列的k-mer (k>7)频谱分布呈现单峰或多峰现象。低等生物基因组祖列一般是单峰分布,高等生物如四爪哺乳动物基因组序列呈现三峰分布,所有物种编码序列是单峰分布。研究组Bao[17-18]等分析了人类1号染色体各类序列的8-mer频谱,结果显示基因间序列和内含子序列呈三峰分布,编码序列是单峰分布,5’UTR和3’UTR序列分布介于两者之间。研究发现8-mer集合按照CG二核苷含量分成三类后,三个CG模体子集的频谱各自形成独立的单峰分布,而其它15种XY二核苷分类则没有此现象,由此揭示了产生单峰和多峰频谱的根本原因并推测了三个CG模体子集的生物学功能。为了验证基因组序列中三个CG模体子集独立进化规律的普适性,以七个物种基因组序列为研究对象,分析每个物种中三个CG模体子集频谱的分布形式,由此验证独立进化规律的普适性。同时,通过对物种间的对比,分析这种进化分离现象与物种进化之间的关系。
1 数据与方法 1.1 七种生物全基因组序列的提取与总长度七个物种狗、牛、鸡、斑马鱼、青鳉鱼、秀丽线虫、酿酒酵母的全基因组序列以及注释信息均来自UCSC Genome Browser Home (http://genome.ucsc.edu)。所有的物种没有考虑性染色体,因为性染色体与物种的进化关系还不明确。其相关信息见表 1。
| 表 1 七种生物基因组序列信息 Table 1 Sequences information of seven genomes |
8-mer模体集合共有48=65 536个。理论上,包含0个二核苷XY (X,Y=A、T、C、G)的8-mer有40 545个,记为0XY;包含1个二核苷XY的8-mer有21 468个,记为1XY;包含两个或两个以上XY二核苷的8-mer有3 523个,记为2XY。若两个核苷酸相同,则三类集合的数目分别是44 631、14 930和5 974个。按照上述约束,可将全体8-mer集合分为0XY、1XY和2XY三个模体子集。这种分类称为XY二核苷分类,这种分类一共有16种。
1.3 8-mer相对模体数对于给定的DNA序列,以8 bp作为窗口,1 bp作为步长,统计得到每个8-mer在该序列中出现的频次。8-mer相对模体数(Frequency of Appearance, FA)定义如下:若频次区组i中出现的8-mer个数为Ni,则该区组上8-mer的相对模体数为:
| $ FA=\frac{{{N}_{i}}}{{{4}^{8}}} $ | (1) |
以8-mer使用频次作为横坐标,相对模体数FA作为纵坐标,得到8-mer相对模体数随频次的分布。
1.4 序列长度标准化对于不同长度的DNA序列,为了方便比较它们的8-mer使用频次之间的关系,对序列进行标准化。对于本文研究的基因组序列,将所有基因组序列的长度标准化到109 bp。若某序列长度为Nbp,则对该序列8-mer出现的频次乘以一个权重系数λ。
| $ \lambda=\frac{{{10}^{9}}}{N} $ | (2) |
统计得到七个物种全部常染色体DNA序列的8-mer频次,按照公式(1)得到各物种8-mer相对模体数随频次的分布,结果见图 1中用“8-mer”标注的外围曲线。图 1展示的频谱分布经过了光滑处理。分析频谱发现:狗和牛的8-mer频谱有三个峰,而斑马鱼、青鳉鱼、秀丽线虫和酿酒酵母的8-mer频谱只有一个峰,鸡的频谱分布形状介于两者之间。对于不同的物种,8-mer频谱展现的分布形式有所不同。
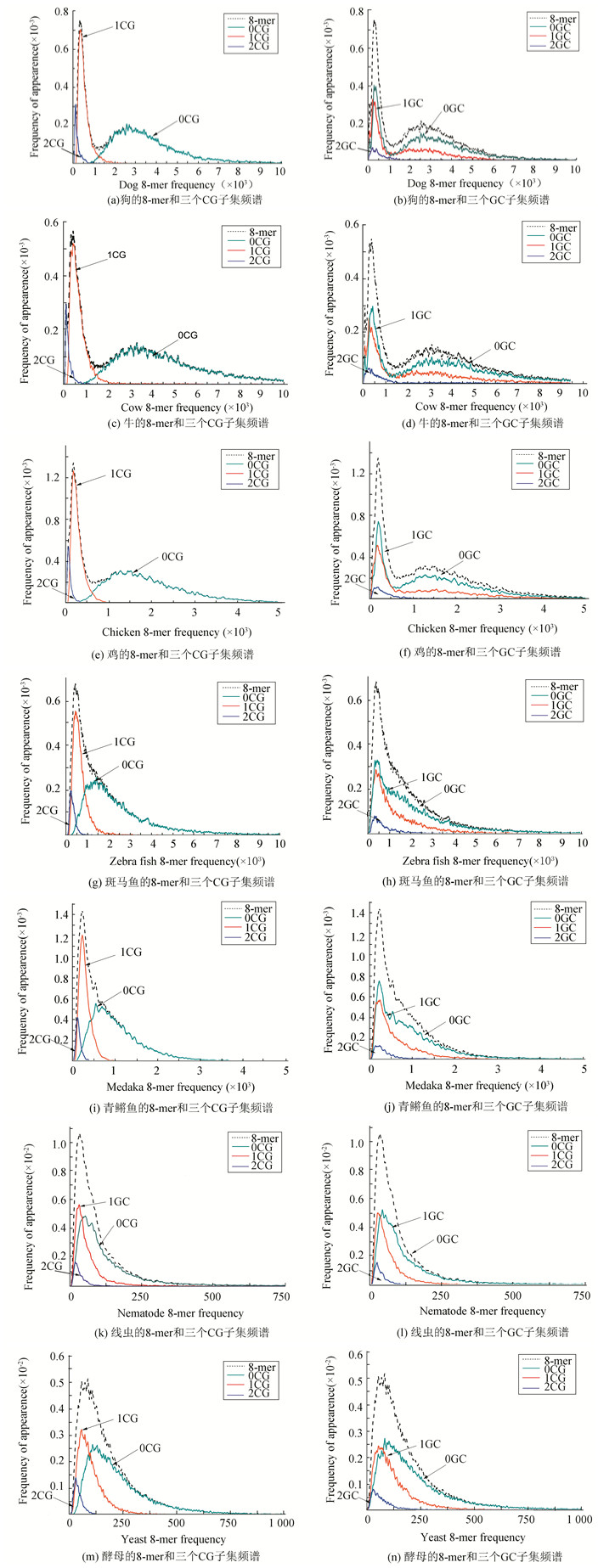
|
图 1 七个物种基因组序列8-mer相对模体数(FA)随频数的分布图谱 Figure 1 Distributions of frequency of appearances (FA) with 8-mer frequencies in seven genome sequences |
为了进一步研究不同物种8-mer频谱分布的差异和8-mer非随机使用的规律,把各基因组的8-mer做16种XY分类,部分结果见图 1。图 1中只给出了CG和GC分类下各个8-mer子集的频谱。分析16种XY分类下0XY、1XY和2XY子集的8-mer频谱,发现所有物种在CG分类下三个子集的8-mer各自形成完全独立的单峰分布,而其他15种XY分类下的三个子集均不能形成独立的单峰分布。
分析图 1,可以发现所有物种中,基因组在CG分类下0CG、1CG和2CG三个模体子集的分布具有相同的规律。首先,1CG模体出现的频次小于0CG,2CG模体出现的频次小于1CG。其次,2CG和1CG模体出现的频次具有保守性,即它们的频次分布在一个很窄的范围之内。而0CG模体的频次分布范围很广。其次,三个CG子集模体分布的最概然相对模体数具有相同的规律,即1CG的最概然相对模体数明显高于0CG和2CG。使用其他15种二核苷来分类得到的图谱中,频数分布并不是完全独立的单峰,每个子集的频数分布与全部8-mer分布形状相似。第三,三个CG子集之间的距离随着进化水平的提高而增加,1CG和2CG的峰越来越高,峰的宽度越来越窄,显示了这两个子集模体的使用保守性越来越强。第四,CG模体的独立进化现象具有物种普适性,这种进化规律在低等真核生物酵母基因组中就已经显现出来。
对于狗和牛而言,三个CG子集分布的中心与总体8-mer多峰分布中心相对应,由于三个CG子集分布中心距离很远,其叠加效果呈现三峰形式。对于单峰分布的物种,由于三个CG子集分布中心距离很近,其叠加效果呈现单峰形式。对于鸡这个物种,因为三个CG子集分布中心相对单峰分布的物种要远一些,叠加之后形成的总体分布会出现介于单峰和三峰之间的分布形状。之所以不同物种的8-mer图谱会有单峰和多峰的现象,是因为每个物种的三个CG子集分布之间的距离不同造成的。研究结果圆满解释了不同物种8-mer频数单峰和多峰分布现象产生的原因。
2.3 三个CG子集频谱分布与随机序列比较为了揭示三个CG子集的使用与进化的关系,针对每一个基因组序列,使用长度相同,碱基组分完全随机的随机序列作为对照分析。选取完全随机序列的原因是我们只关心随机序列8-mer频次分布的最概然频次位置。若将碱基含量设定为与实际基因组序列组分相同的随机序列,则k-mer分布会出现k+1个尖锐且离散的峰,比较其包络曲线的最概然频次很不直观,但效果相同。
图 2给出各物种三个CG子集频数分布与随机序列的分布。可以看出,各物种中0CG子集的频数分布中心最靠近随机分布中心,1CG次之,2CG子集频数分布中心距离随机分布中心最远。这说明0CG模体的使用频次基本上是围绕随机中心进化的,或者说0CG模体的频次分布主要是在随机压力下进化的。而1CG和2CG模体的频次使用远离随机中心,表明这两类模体是定向进化的,而且随着物种进化,其频次使用相对越来越小,且越来越保守。另外,在三类CG模体中,2CG模体的G+C含量最高,0CG模体的G+C含量最低,1CG模体的G+C含量介于两者之间。就是说G+C含量越高的模体出现的频率越低。
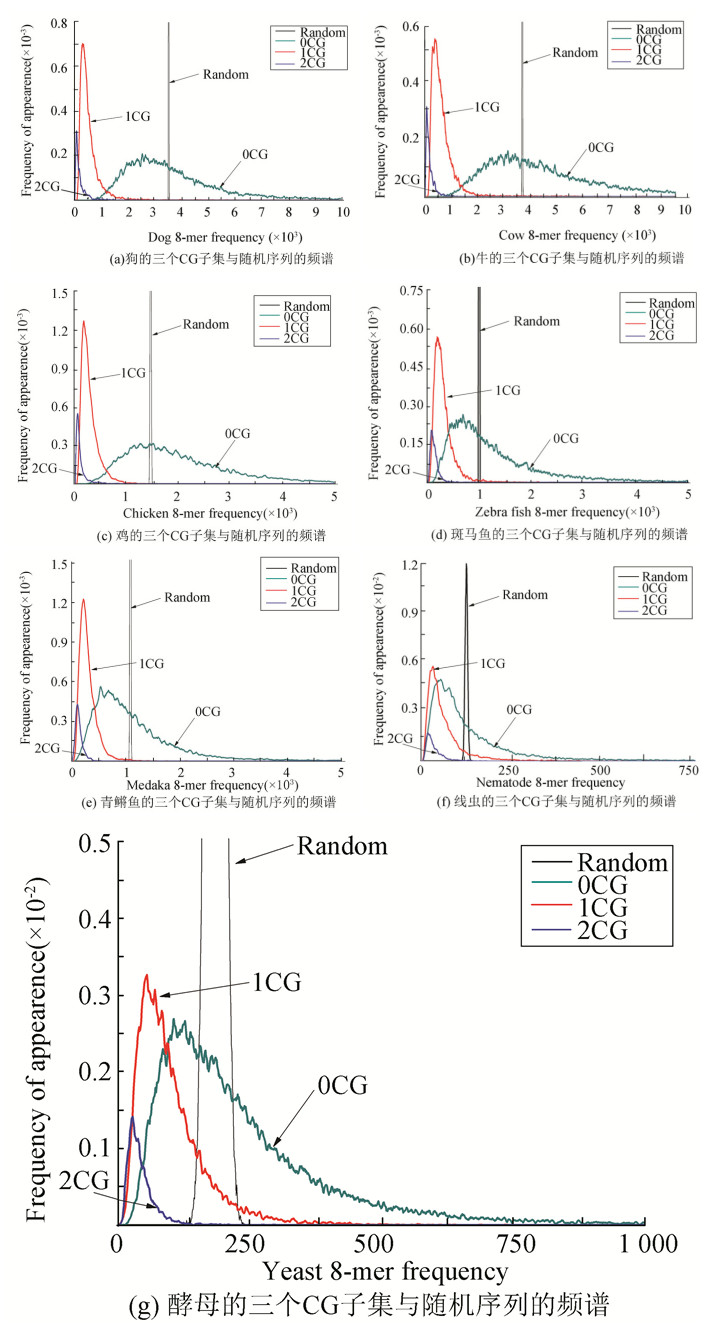
|
图 2 七个物种的三个CG子集8-mer频谱与随机序列的频谱比较 Figure 2 Comparison between three CG subsets' spectrums of seven genomes and 8-mer spectrum of random sequences |
为了分析各物种之间三个CG模体使用的进化规律,将各物种基因组序列长度标准化到109 bp后做对照分析。长度标准化后各物种三个CG模体子集的最概然频次见表 2。以随机序列8-mer最概然频次为参考点,得到三个CG模体子集最概然频次与随机序列最概然频次的比值,记为ρ, 它们与物种进化的关系见图 3。
| 表 2 基因组序列标准化后三个CG模体子集最概然频数 Table 2 Most probable frequencies of the three CG subsets spectrums in the normalized genome sequences |
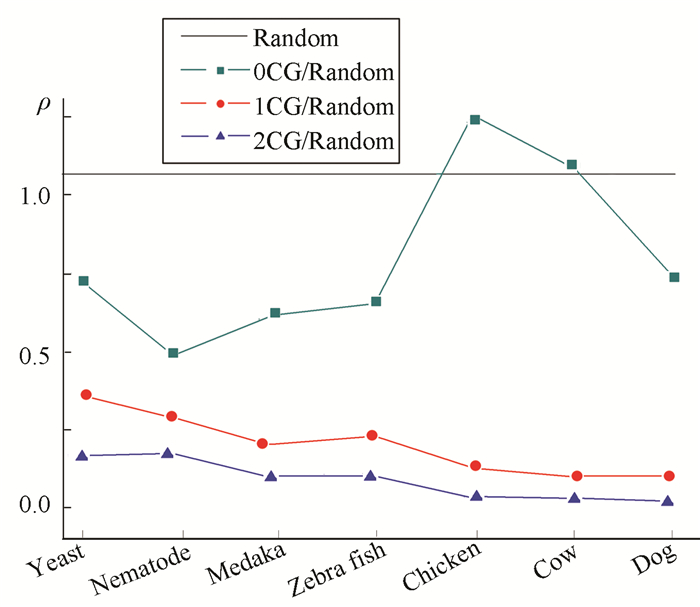
|
图 3 三个CG模体频谱的最概然频次与基因组进化之间的关系 Figure 3 Relations between the most probable frequencies of the three CG subsets spectrums and genome evolutions 注:纵坐标ρ为三个CG模体子集最概然频次与随机序列最概然频次的比值。 |
可以看出在序列标准化后,随着物种由低等向高等的进化,1CG和2CG模体的最概然频次与随机序列的最概然频次的比值逐渐减小,1CG和2CG之间频次比的差值基本保持不变。表明1CG和2CG模体使用与物种进化密切相关,1CG和2CG之间其频次使用保持同步进化,说明这两类模体使用在生物进化过程中受到的进化压力是相同的。0CG模体的最概然频次与物种进化没有明显的联系,说明0CG模体的使用主要是在随机压力下进化的。比如酵母和狗,两类物种其0CG模体的最概然频次与随机序列的比值基本相同。
显然三类CG模体应该执行着不同的生物学功能。关于三类CG模体的功能,本研究组提出了一个猜想:2CG模体是构成CpG岛序列的核心模体,也是DNA序列上各个功能位点的识别信号模体,1CG模体与核小体结合模体紧密相关,0CG模体与表观遗传多样性相关。关于2CG和1CG模体的功能,分别在人类和酵母基因组中得到初步验证。对于0CG模体的功能,Time Quante推测富含AT片段的DNA序列(也就是0CG模体)是表观基因组的重要组成部分[19],该结论从另一个角度支持了我们的猜想。
3 讨论与展望分析了七个物种基因组序列8-mer相对模体数随频次的分布规律,发现所有物种基因组序列在16种XY二核苷分类中只有在CG二核苷分类下三个CG模体集合的频次分布各自形成完全独立的单峰分布。从统计学上来讲,三个独立的单峰分布代表它们来自三个独立的总体,也就是说基因组序列是由这三类独立进化单元构成的。0CG模体的分布中心在随机中心附近,1CG与2CG模体出现的频次远低于0CG模体出现的频次,且它们的分布中心则远离随机中心。这表明0CG模体主要是在随机压力下进化的,而1CG与2CG模体是定向进化的。CG模体的独立进化现象具有物种普适性,这种进化规律在低等真核生物酵母基因组中就已经显现出来。随着物种进化,1CG和2CG模体使用保守性越来越强。0CG模体使用与物种进化无明显关系,1CG和2CG模体使用与物种进化显著相关。发现,三个CG模体分布中心的距离是产生8-mer图谱(或k-mer图谱)单峰或多峰现象的根本原因,因此这一现象也就得到了自然的解释。研究结论预示了这三类CG模体具有不同的生物学功能。
由于其它15中XY分类中各个子集均未显示出各自独立的进化现象,具有物种普适性的CG分类独立进化现象勾画出了基因组序列结构和进化的规律。在生命的早期,在随机进化压力下,形成以碱基A或T组成的原始DNA序列背景,出于功能的需要,以CG二核苷作为定向进化的中心,逐步形成含CG二核苷的功能片段,以满足越来越复杂的生物的需求。为何生物只选择CG二核苷作为进化的核心而不是其它二核苷呢?我们不得而知,但一个普遍现象是必须承认的,就是只有CG二核苷存在甲基化现象,而不是其它二核苷。两者之间应该存在必然的联系。
许多工作根据DNA序列的k-mer频次偏好来揭示物种之间的进化,他们往往关注序列中高频次出现的模体。根据以前的研究结论,高频次出现的模体属于0CG模体子集,这些模体与物种进化无明显的关系,而低频次出现的1CG和2CG模体与进化显著相关。如果用1CG和2CG模体来构造系统发生关系,得到的结果应该会更加可靠。另外,无论实验还是理论分析核小体中心序列上与组蛋白相互作用模体时,也存在这种倾向。其实,核小体结合模体往往是出现频次较低的模体。如何从大的背景噪音中将这些低频出现的功能模体提炼出来才是正确的思考方向。
| [1] |
CSÜRÖS M, NOÉ L, KUCHEROV G. Reconsidering the significance of genomic word frequencies[J]. Trends in Genetics, 2007, 23(11): 543–546.
DOI:10.1016/j.tig.2007.07.008 ( 0) 0)
|
| [2] |
D'HAESELEER P. What are DNA sequence motifs[J]. Nature Biotechnology, 2006, 24(4): 423–425.
DOI:10.1038/nbt0406-423 ( 0) 0)
|
| [3] |
TULLER T, CHOR B, NELSON N. Forbidden penta-peptides[J]. Protein Science, 2007, 16(10): 2251–2259.
DOI:10.1110/ps.073067607 ( 0) 0)
|
| [4] |
HAO Bailin, LEE H C, ZHANG Shuyu. Fractals related to long DNA sequences and complete genomes[J]. Chaos, Solitons & Fractals, 2000, 11(6): 825–836.
DOI:10.1016/S0960-0779(98)00182-9 ( 0) 0)
|
| [5] |
SUBIRANA J A, MESSEGUER X. The most frequent short sequences in non-coding DNA[J]. Nucleic Acids Research, 2010, 38(4): 1172–1181.
DOI:10.1093/nar/gkp1094 ( 0) 0)
|
| [6] |
HAMPIKIAN G, ANDERSEN T. Absent sequences: Nullomers and primes[J]. Pacific Sympusium on Biocomputing, 2007(12): 355–366.
DOI:10.1142/9789812772435_0034 ( 0) 0)
|
| [7] |
HARIHARAN R, SIMON R, PILLAI M R, et al. Comparative analysis of DNA word abundances in four yeast genomes using a novel statistical background mode[J]. PLoS One, 2013, 8(3): e58038.
DOI:10.1093/bioinformatics/btn166 ( 0) 0)
|
| [8] |
YU Hongjie. Segmented k-mer and its application on similarity analysis of mitochondrial genome sequences[J]. Gene, 2013, 518(2): 419–424.
DOI:10.1016/j.gene.2012.12.079 ( 0) 0)
|
| [9] |
CHAE H, PARK J, LEE S W, et al. Comparative analysis using k-mer and k-flank patterns provides evidence for CpG island sequence evolution in mammalian genomes[J]. Nucleic Acids Research, 2013, 41(9): 4783–4791.
DOI:10.1093/nar/gkt144 ( 0) 0)
|
| [10] |
YANG Y, NEPHEW K, KIM S. A novel k-mer mixture logistic regression for methylation susceptibility modeling of CpG dinucleotides in human gene promoters[J]. BMC Bioinformatics, 2012, 13(Suppl 3): S15.
DOI:10.1186/1471-2105-13-S3-S15 ( 0) 0)
|
| [11] |
CHIKHI R, MEDVEDEV P. Informed and automated k-mer size selection for genome assembly[J]. Bioinformatics, 2013, 30(1): 31–37.
DOI:10.1093/bioinformatics/btt310 ( 0) 0)
|
| [12] |
BINAA M, WYSSA P, SHERYL A, et al. Discovering sequences with potential regulatory characteristics[J]. Genomics, 2009, 93(4): 314–322.
DOI:10.1016/j.ygeno.2008.11.008 ( 0) 0)
|
| [13] |
BINAA M, WYSSA P, RENB W, et al. Exploring the characteristics of sequence elements in proximal promoters of human genes[J]. Genomics, 2004, 84(6): 929–940.
DOI:10.1016/j.ygeno.2004.08.013 ( 0) 0)
|
| [14] |
X IE, Xiaohui, LU Jun, KULBOKAS E J, et al. Systematic discovery of regulatory motifs in human promoters and 3'UTRs by comparison of several mammals[J]. Nature, 2005, 434(7031): 338–345.
DOI:10.1038/nature03441 ( 0) 0)
|
| [15] |
ZHANG Yi, WANG Xianhui, KANG Le. A k-mer scheme to predict piRNAs and characterize locust piRNAs[J]. Bioinformatics, 2011, 27(6): 771–776.
DOI:10.1093/bioinformatics/btr016 ( 0) 0)
|
| [16] |
CHOR B, HORN D, GOLDMAN N, et al. Genomic DNA k-mer spectra: Models and modalities[J]. Genome Biology, 2009, 10(10): R108.
DOI:10.1007/978-3-642-12683-3_37 ( 0) 0)
|
| [17] |
BAO T, LI H, ZHAO X Q, et al. Predicting nucleosome binding motif set and analyzing their distributions around functional sites of human genes[J]. Chromosome Research, 2012, 20(6): 685–698.
DOI:10.1007/s10577-012-9305-0 ( 0) 0)
|
| [18] |
周德良, 李宏, 杨小希. 人类1号染色体DNA序列8-mer的相对模体数分布及8-mer使用的进化分离[J]. 生物物理学报, 2015, 31(1): 53–64.
ZHOU Deliang, LI Hong, YANG Xiaoxi. Frequency distributions of 8-mer and the evolution diversity of 8-mer usage in human DNA sequences[J]. Acta Biophysica Sinica, 2015, 31(1): 53–64.
DOI:10.3724/SP.J.1260.2015.40050 ( 0) 0)
|
| [19] |
QUANTE T, BIRD A. Do short, frequent DNA sequence motifs mould the epigenome?[J]. Nature Reviews Molecular Cell Biology, 2016, 17(4): 257–262.
DOI:10.1038/nrm.2015.31 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


