2. 昆明理工大学信息工程与自动化学院,昆明 650500
2. School of Information Engineering and Automation,Kunming University of Science and Technology, Kunming 650500, China
随着人类基因组计划的胜利完成和后基因组时代的来临[1],DNA测序技术已成为人类探索生命秘密的重要手段之一,对生物、生命科学、医学等领域的技术发展起到了巨大的推动作用[2]。经过三十多年的努力,DNA测序技术已经取得巨大的进展,在第一代和第二代测序技术的基础上,以单分子测序为特点的第三代测序技术已经诞生。第三代测序技术虽然解决了第二代测序技术读长短、速度慢等缺点,但由于其成本和错误率偏高、通量低,目前最常用的依然是以Illumina公司的Solexa技术[3]为标志的第二代测序技术。
第二代测序技术拥有相当高的测序通量,覆盖度高。得到的reads不仅长度短,数量又极为巨大,这给序列拼接带来了巨大的挑战,而基因组测序中的一个关键的步骤就是序列拼接[4]。拼接后,还需要对所有的SNP进行分析和注释。
针对SNP的分析,目前有一些基于云端的高通量测序数据分析平台,比如Galaxy[5]。Galaxy等现行的生物信息学平台,使大量的生物信息学工具易于操作,用户上传数据后可立即开始分析。但是,当用户拥有超大数据量时,存储限制了数据的传输速度,较长的工作排队时间使其变得不切实际。除了平台解决方案,还有其他独立途径可进行SNP的多样分析。例如SeqMule[6]、HugeSeq[7]、Ngs_backbone[8]和Bcbio-nextgen[9]四款集成分析软件,可以运用自带的工具对SNP进行比对、注释、分析等。但是,由于部分软件集成某些专用工具,比如Bcbio-nextgen软件专有的比对工具NovoAlign[10],不是对所有研究人员免费开放。四款集成分析软件相比,SeqMule软件结合了5种SNP比对工具和5种SNP分析工具,其余三款分析软件只有一种或两种SNP比对工具和SNP分析工具。除此之外,只有SeqMule软件拥有可选且开源的SNP比对工具,具有更高的灵活性和可用性。
SeqMule软件是以人类遗传病研究为背景,专门针对外显子组或全基因组序列分析设计的。它采用高度灵活的各种调用格式对SNP进行完全自动化的分析和注释,支持Sun Grid Engine并行处理,可以进行测序质量的检测、孟德尔错误率检测、一致性评估,生成最终的HTML报告。相比之下,SeqMule是上述解决方案中较好的一款软件,推荐生物信息学人员使用。
1 SeqMule软件 1.1 基本介绍对测序数据进行分析的时候,除了测序平台的差异[11],仍要考虑算法间的差异。例如,5种生物信息学算法(SOAP、BWA-GATK、BWA-SNVer、GNUMAP、BWA-SAMtools)分析SNV(Single Nucleotide Variants)的一致性只有57.4%,而每种计算途径间的变异数为0.5%~5.1%[12]。在不同的测序错误率和indel标记下,校准也存在差异[13]。目前,公开发表的计算方法几乎没有提供两种或更多的比对和SNP分析方法。
分析软件的安装和配置是首要问题,而且这个问题的重要性已经被许多试图去使用它的人所证实,像Bioconductor、Bioperl和Web-based三款软件[14-16]。理论上,来自一个程序的输出结果很难被输入另一个和它类似的程序中。例如,GATK不能接受来自SOAP2的输出。此外,软件的不同步更新,可能导致软件的不兼容。虚拟机和虚拟化技术为用户解决了该问题[17-19],然而,虚拟机系统不可避免地限制了客户系统可用的计算资源,减少了软件工具的灵活性。因此,对于没有计算机背景的普通用户来说,部署软件成为了一个很大的难题。针对普通用户,迫切需要一种易于执行和整合多种工具的分析途径。
在不影响易用性、高效性和重复性的前提下,由南加州大学的王凯实验室开发了一个全能的解决方案——SeqMule,能够执行一系列自动化的命令来分析高通量测序数据。它结合了5种比对工具:BWA(包括BWA-backtrack和BWA-MEM)、Bowtie、Bowtie2、SOAP2、SNAP [20-24],5种不同SNP分析工具:GATK(包括GATKLite 和version 3)、SAMtools、VarScan 2、Freebayes、SOAPsnp[25-28]和一些配件程序:FastQC、Picard、tabix、VCFtools 30,而且可以通过修饰配置文件来获得多种组合。通过不同工具结合而设置变量形成交叉,从而获得更高的准确性、敏感性和特异性。SeqMule能提供建立在不同调用者之上的并行功能,还能够更好地分析高通量测序数据,提升分析的一致性和准确性。针对目前主流服务器(CPU:2 Intel Xeon X5650,内存48GB),只需24小时,SeqMule可从设置好的全基因组数据生成带注释的VCF文件。
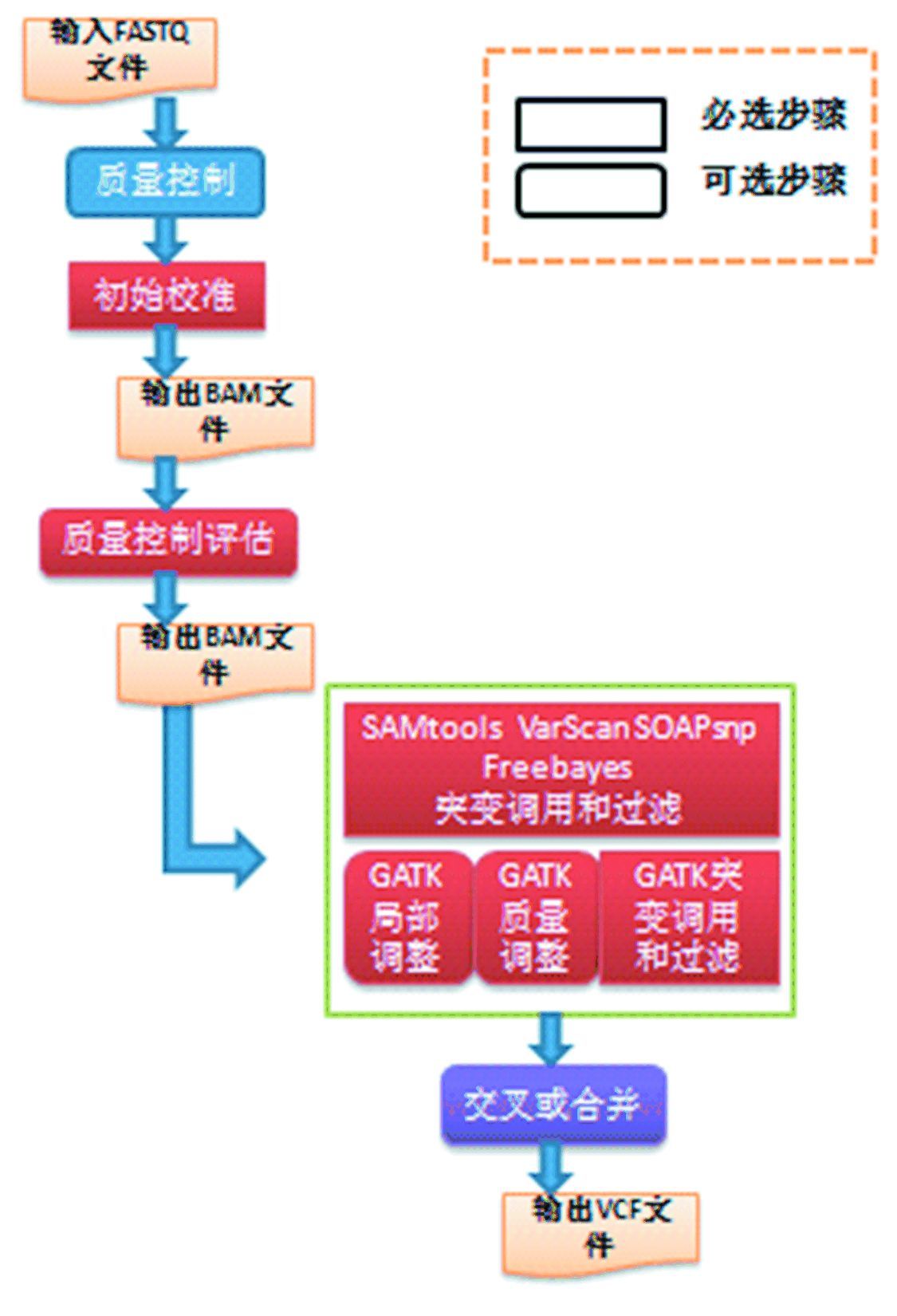
SeqMule的工作流程如图 1所示,分析过程中有很多可利用的工具。其中,先使用FastQC进行质量控制,再采用BWA-backtrack、BWA-MEM、Bowtie等工具进行初始校准,校准后可使用Picard Tools对质量控制进行评估,再使用GATK、SAMtools、SOAPsnp、VarScan工具进行突变调用和过滤,最后采用GATK CombineVariants交叉或合并。
1.2 SeqMule安装方法SeqMule可在如下网址下载:http://seqmule.openbioinformatics.org.
1) 笔者使用的是CentOS 7系统,安装SeqMule之前要先安装必要的软件和环境,相关命令如下:
sudo yum install-y gcc gcc-c++ make cmake ncurses-devel ncurses R unzip automake autoconf git-core gzip tar
|
图 1 SeqMule的工作流程图 Figure 1 Scheme of SeqMule workflow |
下载SeqMule程序 git clone https://github.com/WGLab/SeqMule.git,如果Https不支持也可以用git模式git clone git://github.com/WGLab/SeqMule.git。
3) 进入SeqMule文件夹,利用./Build freshinstall进行初始安装。
4) 安装一次后,利用./Build installexes安装missing的部分。
5) 由于GATK要单独安装,利用./Build gatk看安装命令,核心就是把GATK的jar文件拷贝到制定文件夹。
6) 把环境变量写到用户的bashrc里面,然后用source命令更新以下环境变量,这样Seqmule就可以不制定他的绝对位置来使用了,否则会出现command找不到的情况。
echo ’export PATH=$PATH:absolute_path_to
_seqmule/bin’ >> ~/.bashrc source ~/.bashrc
7) 下载Seqmule要使用到的Database,seqmule download -down hg19all。
此部分利用terminal下载很缓慢,建议使用专用下载工具下载,大概有40 G左右。将下载的文件放到SeqMule的database文件里,再利用SeqMule download-down hg19all命令进行解压缩,然后把文件再按名称放到指定文件夹。另外,笔者已将下载好的database文件夹都放置到百度云,读者可以通过shunxirm@163.com获取下载秘钥。
1.3 SeqMule软件的运行SeqMule软件运行在Linux系统平台下,命令简单且易于掌握。根据测序方法不同,SeqMule的分析方式也不同。SeqMule主要包括三种分析方式,分别是:典型的外显子组分析、快速转换的全基因组分析和基于家系的三人外显子组分析。此外,SeqMule软件可以一次性分析多个样本,大大简化了生物信息学研究人员的操作。
1.3.1 SeqMule软件的使用命令在存放需要分析的FASTQ格式文件的文件夹下,打开系统终端,输入以下命令运行SeqMule:
seqmule pipeline-a normal_R1.fastq.gz-b normal_R2.fastq.gz-prefix example-N 2-capture default-threads 4-e
其中,normal_R1.fastq.gz和normal_R2.fastq.gz是DNA经过测序仪测序后产生的FASTQ格式的压缩文件,分别是一条DNA上两条链的基因数据。参数“-prefix example”是告诉SeqMule软件你的样本名称是example;“-capture default”是让SeqMule软件使用默认的区域定义文件——hg19外显子区,对应文件可从安捷伦SureSelect工具包中下载;“-threads 4”是令SeqMule软件在运行时使用该计算机的四个线程;“-e”的意思是这个数据集是外显子组数据或者捕获的测序数据,而不是全基因组数据。
1.3.2 典型的外显子组分析命令通过测序仪对外显子组测序后,得到四个FASTQ文件的压缩文档,在终端下运行以下命令进行典型外显子组分析:
seqmule pipeline-a sample_lane1_R1.fq.gz,sample_lane2_R1.fq.gz-b sample_lane1_R2.fq.gz,sample_lane2_R2.fq.gz-capture seqmule/database/hg19-
nimblegen/nexterarapidcapture_exome_targetedregions_v1.2.bed-m-e-advanced seqmule/misc/predefined_con-
fig/bwa_gatk_HaplotypeCaller.config-quick-t 4-prefix mySample
命令中“-advanced seqmule/misc/predefined_config
/bwa_gatk_HaplotypeCaller.config”表示使用BWA和GATK这两个可选工具包进行SNP的比对和分析。参数中-quick是使软件使用更多的计算机内存来进行快速分析;“-t 4”表示分析时使用计算机的四个CPU;“-m”是合并两个数据集。
1.3.3 快速转换的全基因组分析命令通过测序仪对全基因组测序后,得到两个FASTQ文件的压缩文档,在终端下运行以下命令进行快速转换的全基因组分析:
seqmule pipeline-a sample_R1.fq.gz-b sample_R2.fq.gz-advanced seqmule/misc/predefined_c-onfig/snap_freebayes.config-quick-t 12-g-prefix mySample
命令中“-advanced seqmule/misc/predefined_config/snap_freebayes.config”表示使用SNAP和FreeBayes这两个可选工具包进行全基因组SNP的比对和分析。参数“-g”表示全基因组分析;“-t 12”是令SeqMule使用计算机的12个CPU进行比对分析,因为SNAP工具使用时非常消耗内存,因此采用多个CPU来提高软件运行速度。
1.3.4 三人外显子组分析命令对同一个家庭的三个人进行外显子组测序后,使用SeqMule软件对三人的测序数据进行分析来发现致病基因,命令如下:
seqmule pipeline-a fa_R1.fq.gz,mo_R1.fq.gz,son
_R1.fq.gz-b fa_R2.fq.gz,mo_R2.fq.gz,son_R2.fq.gz-ms-e-q-t 4-prefix father,mother,son-capture default-sge "qsub-V-cwd-pe smp XCPUX"
命令中“-sge "qsub-V-cwd-pe smp XCPUX””表示使用SG工具包来进行分析。参数中“-ms”表示针对多样本的基因突变识别,可以更加准确地分析来自同一家庭的三个人的外显子组数据。
2 使用SeqMule软件进行SNP分析 2.1 数据准备患者数据来自舜喜再生医学工程有限公司。昆明医科大学第一附属医院的两名痛风患者在云南舜喜再生医学工程有限公司抽血并提取DNA后,使用Illumina公司的Hiseq3000测序仪进行外显子组测序。测序后得到FASTQ格式文件的压缩文件,作为实验前准备数据。使用SeqMule软件进行基因的比对、拼接并进行SNP分析。
2.2 分析报告SeqMule分析完成后,生成HTML格式的详细分析报告(SeqMule Report)。分析报告网页上有分析总结、样本分析报告、分析途径、分析参数和帮助文件按钮,点开后即可查看详细信息。
样本分析结果展示了统计资料、SNV与NON-SNV韦恩图和覆盖度图。统计资料里包含基因校准数据表、基因覆盖率统计数据表和基因突变数据表。其中,表 1是SeqMule软件对该患者的基因数据进行初始校准得到的校准统计表,包含通过的质量控制读长数、失败的读长数、比对的读长数及和数据库匹配上的读长数等数据。表 2是该患者的基因覆盖率度统计数据表,包括总的长度、目标区域的平均覆盖度等数据。表 3是该患者的基因突变数据表,包含该患者的所有突变位点数、SNV突变位点数和插入/缺失位点数等数据。
| 表 1 患者1的校准统计表 Table 1 Alignment stats of the NO.1 patient |
| 表 2 患者1的覆盖率统计数据表 Table 2 Coverage stats of the NO.1 patient |
| 表 3 患者1的突变数据表 Table 3 Variant stats of the NO.1 patient |
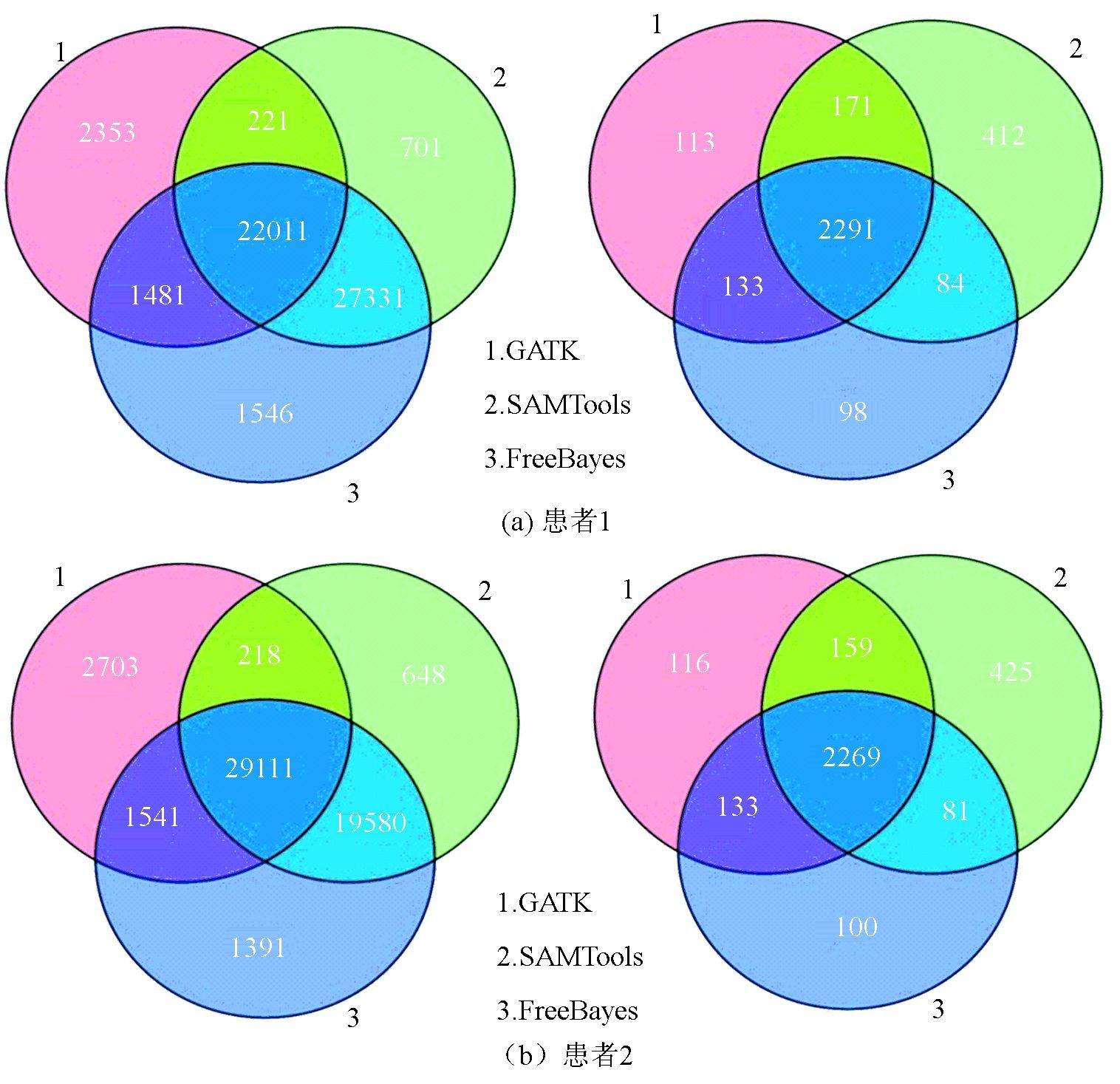
SNV与NON-SNV韦恩图是SeqMule结合3种不同的SNP分析工具得出的SNV和NON-SNV突变重叠图。图 2是两名患者的SNV与NON-SNV韦恩图。从图中可以得出,基于3种分析工具单独分析出的基因突变结果、两两之间分析出的相同突变基因的结果以及三种分析工具分析出的相同突变基因的个数。患者1的数据中,GATK、SAMtools和freebayes三种分析工具的分析结果中都出现SNV突变的位点有22 011个,NON-SNV突变的位点有2 291个。患者2的数据中,三种分析工具的分析结果中都出现SNV突变的位点有29 111个,NON-SNV突变的位点有2 269个。
|
图 2 两名患者的SNV与NON-SNV韦恩图 Figure 2 Venn Diagram (SNV and NON-SNV) of two patients |
从实验结果可以看出,SeqMule可对外显子组测序数据实现全面、简易、灵活、高效的一站式自动化分析。分析结果采用HTML报告的方式,展示出详细、美观的图表,简单易读。除了外显子组测序,SeqMule还支持对全基因组测序进行一站式自动分析,更加多元化。SeqMule解决了大部分分析软件存在的软件兼容性、配置复杂及不能访问高性能计算设施等问题,能更好地分析高通量测序数据,提升基因数据分析的一致性和准确性。该软件的5种比对方式、5种SNP分析工具和多种多样的配件程序给用户提供了众多选择,内置的并行处理能力可加快分析的进程。除了上述特点外,SeqMule使用单行命令完成复杂的任务,使其成为易于下载、安装、配置和运行的生物信息学的工具。
笔者已经用SeqMule来分析测序数据,并且获得了有意义的结果。随着新一代测序技术的快速发展和部署,我们期望SeqMule能够促进即将来临的大量测序数据分析,从而为人类遗传病研究奠定基础,并促进人类遗传病的诊断方法的完善。
| [1] |
唐旭清, 朱平. 后基因组时代生物信息学的发展趋势[J]. 生物信息学, 2008, 6(3): 142–144.
TANG Xuqing, ZHU Ping. The development trends of bioinformatics in post-genomic era[J]. China Journal of Bioinformatics, 2008, 6(3): 142–144.
( 0) 0)
|
| [2] |
陈文辉, 罗军, 赵超. 固态纳米孔:下一代DNA测序技术——原理?工艺与挑战[J]. 中国科学:生命科学, 2014(7): 649–662.
CHEN Wenjun, LUO Jun, ZHAO Chao. Solid nano pore: Next generation DNA sequencing technology-principle, techonology and challenge[J]. Science in China: Life Sciences, 2014(7): 649–662.
( 0) 0)
|
| [3] |
CAPORASO J G, LAUBER C L, WALTERS W A, et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. Isme Journal, 2012, 6(8): 1621–1624.
DOI:10.1038/ismej.2012.8 ( 0) 0)
|
| [4] |
逯雯雯, 卢志远, 王亚旭, 等. 面向新一代基因组测序技术的序列拼接算法[J]. 生物信息学, 2010, 8(3): 248–253.
LU Wenwen, LU Zhiyuan, WANG Yaxu, et al. Facing the sequence stitching algorithm of new generation genome sequencing technology[J]. China Journal of Bioinformatics, 2010, 8(3): 248–253.
( 0) 0)
|
| [5] |
AFGAN E, BAKER D, CORAOR N, et al. Galaxy CloudMan: delivering cloud compute clusters[J]. BMC Bioinformatics, 2010, 12(6): S4–S4.
( 0) 0)
|
| [6] |
GUO Y, DING X, SHEN Y, et al. SeqMule: automated pipeline for analysis of human exome/genome sequencing data[J]. Scientific Reports, 2015, 5: 14283.
DOI:10.1038/srep14283 ( 0) 0)
|
| [7] |
LAM H Y, PAN C, CLARK M J, et al. Detecting and annotating genetic variations using the HugeSeq pipeline[J]. Nature Biotechnology, 2012, 30(3): 226–229.
DOI:10.1038/nbt.2134 ( 0) 0)
|
| [8] |
BLANCA J M, PASCUAL L, ZIARSOLO P, et al. ngs_backbone: a pipeline for read cleaning, mapping and SNP calling using next generation sequence[J]. BMC Genomics, 2011, 12(1): 193–201.
DOI:10.1186/1471-2164-12-193 ( 0) 0)
|
| [9] |
GUIMERA R V. Bcbio-nextgen: Automated, distributed next-gen sequencing pipeline[J]. Embnet Journal, 2012, 18(Supplement B): 1–153.
DOI:10.14806/ej.17.B.286 ( 0) 0)
|
| [10] |
RAMOS E, LEVINSON B T, CHASNOFF S, et al. Population-based rare variant detection via pooled exome or custom hybridization capture with or without individual indexing[J]. BMC Genomics, 2012, 13(1): 1–15.
DOI:10.1186/1471-2164-13-1 ( 0) 0)
|
| [11] |
LAM H Y K, CLARK M J, CHEN R, et al. Performance comparison of whole-genome sequencing platforms[J]. Nature Biotechnology, 2012, 30(1): 78–82.
( 0) 0)
|
| [12] |
ORAWE J, JIANG T, SUN G, et al. Low concordance of multiple variant-calling pipelines: practical implications for exome and genome sequencing[J]. Genome Medicine, 2013, 5(13): 1735–1742.
( 0) 0)
|
| [13] |
RUFFALO M, LAFRAMBOISE T, KOYUTURK M. Comparative analysis of algorithms for next-generation sequencing read alignment[J]. Bioinformatics, 2011, 27(27): 2790–2796.
( 0) 0)
|
| [14] |
GENTLEMAN R C, CAREY V J, BATES D M, et al. Bioconductor: open software development for computational biology and bioinformatics[J]. Genome Biology, 2004, 5: R80.
DOI:10.1186/gb-2004-5-10-r80 ( 0) 0)
|
| [15] |
STAJICH J E, BLOCK D, BOULEZ K, et al. The Bioperl toolkit: Perl modules for the life sciences[J]. Genome Research, 2002, 12(10): 1611–1618.
DOI:10.1101/gr.361602 ( 0) 0)
|
| [16] |
CHANG X, WANG K. wANNOVAR: annotating genetic variants for personal genomes via the web[J]. Journal of Medical Genetics, 2012, 49(7): 433–436.
DOI:10.1136/jmedgenet-2012-100918 ( 0) 0)
|
| [17] |
KRAMPIS K, BOOTH T, CHAPMAN B, et al. Cloud BioLinux: pre-configured and on-demand bioinformatics computing for the genomics community[J]. BMC Bioinformatics, 2012, 13(1): 1–8.
DOI:10.1186/1471-2105-13-1 ( 0) 0)
|
| [18] |
NOCQ J, CELTON M, GENDRON P, et al. Harnessing virtual machines to simplify next-generation DNA sequencing analysis[J]. Bioinformatics, 2013, 29(17): 2075–2083.
DOI:10.1093/bioinformatics/btt352 ( 0) 0)
|
| [19] |
ANGIUOLI S V, MATALKA M, GUSSMAN A, et al. CLOVR: a virtual machine for automated and portable sequence analysis from the desktop using cloud computing[J]. BMC Bioinformatics, 2011, 12(49): 356–356.
( 0) 0)
|
| [20] |
LI H, DURBIN R. Fast and accurate short read alignment with Burrows-Wheeler transform[J]. Bioinformatics, 2009, 25(14): 1754–1760.
DOI:10.1093/bioinformatics/btp324 ( 0) 0)
|
| [21] |
LANGMEAD B, TRAPNELL C, POP M, et al. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome[J]. Genome Biology, 2009, 10(3): 1–10.
( 0) 0)
|
| [22] |
LANGMEAD B, SALZBERG S L. Fast gapped-read alignment with Bowtie 2[J]. Nature Methods, 2012, 9(4): 357–359.
DOI:10.1038/nmeth.1923 ( 0) 0)
|
| [23] |
LI R, YU C, LI Y, et al. SOAP2: an improved ultrafast tool for short read alignment[J]. Bioinformatics, 2009, 25(15): 1966–1967.
DOI:10.1093/bioinformatics/btp336 ( 0) 0)
|
| [24] |
ZAHARIA M, BOLOSKY W J, CURTIS K, et al. Faster and more accurate sequence alignment with SNAP[J]. ARXIV, 2011(1): 1–10.
( 0) 0)
|
| [25] |
MCKENNA A, HANNA M, BANRS E, et al. The genome analysis toolkit: a mapreduce framework for analyzing next-generation DNA sequencing data[J]. Genome Research, 2014, 20(9): 1297–1303.
( 0) 0)
|
| [26] |
LI H, HANDSAKER B, WYSOKER A, et al. The sequence alignment-map format and SAMtools[J]. Bioinformatics, 2009, 25(16): 2078–2079.
DOI:10.1093/bioinformatics/btp352 ( 0) 0)
|
| [27] |
KOBOLDT D C, ZHANG Q, LARSON D E, et al. VarScan 2: somatic mutation and copy number alteration discovery in cancer by exome sequencing[J]. Genome Research, 2012, 22(3): 568–576.
DOI:10.1101/gr.129684.111 ( 0) 0)
|
| [28] |
LI R, LI Y, FANG X, et al. SNP detection for massively parallel whole-genome resequencing[J]. Genome Research, 2009, 19(6): 545–552.
( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


