2.南京晓庄学院,南京 211171
2.Nanjing Xiaozhuang University,Nanjing 211171,China
2016年5月2日,Nature Biotechnology报道了韩春雨等发明的DNA指导的基因组编辑(Genome editing)系统NgAgo-gDNA [1],NgAgo是格氏嗜盐碱杆菌(Natronobacterium gregoryi)AGO蛋白(Argonaute)的简称,其本质是一种核酸内切酶。NgAgo酶根据指导DNA的定位,可以有效地对基因组目标区域进行编辑。这项研究不能仅仅看作是对现有的RNA指导的基因组编辑系统CRISPR-Cas9的技术改进,其能否商业化以替代CRISPR-Cas9也不是最重要的。NgAgo-gDNA只是一个新的开始,沿着这条研究路线,很可能开发出更先进的基因组编辑系统。一项研究的意义在于研究者的原始出发点以及在实验和分析中体现的智慧,更为重要的是它能否拓宽相关领域的研究思路并开辟新的研究方向。结合作者在多个项目中的实际工作经验,本文谈谈这项研究的意义以及未来的发展方向。
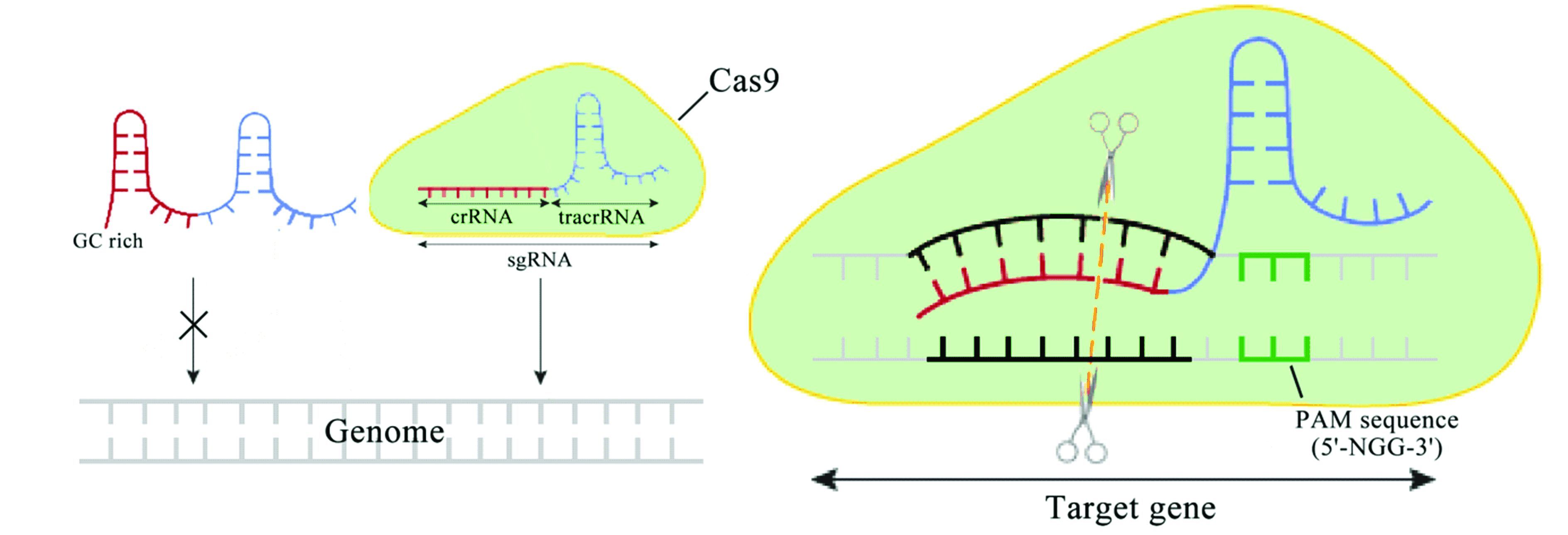
1 NgAgo-gDNA不是简单改进一个基因组编辑系统,可以简单分为负责识别(目标)靶序列的一段核酸(DNA或RNA)序列和负责切割的酶两个部分(见图 1)。CRISPR-Cas9系统中是单链RNA(Single-stranded guide RNA,简称sgRNA或gRNA)指导Cas9蛋白切割;NgAgo-gDNA系统中是5’端磷酸化的单链DNA(Single-stranded guide DNA,简称gDNA)指导AGO蛋白切割。靶序列识别的特异性是关键问题,酶的效率虽然也重要但是次要问题。NgAgo-gDNA主要的技术进步(按照重要性)包括以下几点:
|
图 1 CRISPR-Cas9简单原理 Figure 1 How CRISPR-Cas9 works 注:A:sgRNA(single-stranded guide RNA)包括至少一个crRNA(CRISPR-derived RNA,红色)和一个tracrRNA (trans-activating crRNA,蓝色)。如果crRNA中GC含量过高,会形成茎环结构,严重影响sgRNA结合到基因组目标区域;B:sgRNA(红色)指导Cas9酶在与crRNA配对的靶序列(黑色)剪切双链DNA。sgRNA要求目标基因后面有一个特征三碱基序列(即PAM序列)才能工作。 Notes:A:a sgRNA (single-stranded guide RNA) contains at least one crRNA (CRISPR-derived RNA,in red color) and one tracrRNA (trans-activating crRNA,in blue color). A stem-loop structure may form from a crRNA due to its high GC content,which could prevent the binding of sgRNA to the target region on the genome.B:a sgRNAs (in red color) guides an Cas9 enzyme to cleave double stranded DNA of the target gene (black). This system requires a featured three base (PAM) sequence to work. |
1) 5'端磷酸化的单链DNA在哺乳动物细胞中几乎不存在,这保证了NgAgo不会被内源的DNA序列误导,靶向错误的基因组位点,称作脱靶(off-target)。点评:这说明转基因技术并不是绝对安全;另外引出一个问题,古细菌内是否有单链DNA,如果没有,这个酶在古细菌中是怎么工作的?
2) gDNA一旦与NgAgo结合,就不允许其他DNA片段插进来替换,这又从另一方面保证了不脱靶。点评:工作极为严谨,考虑到了酶与底物的动态作用关系。
3) NgAgo-gDNA系统中的gDNA是24 bp长度,CRISPR-Cas9系统中的sgRNA是19 bp长度,24 bp大大提高了目标位点在基因组上的特异度。点评:21 bp以上长度的序列才能保证其在大型基因组中的唯一性,19 bp实用价值很低,PCR引物设计通常也要21 bp以上;现在各类升级版的CRISPR-Cas9系统已出现,但NgAgo-gDNA起点高于CRISPR-Cas9。
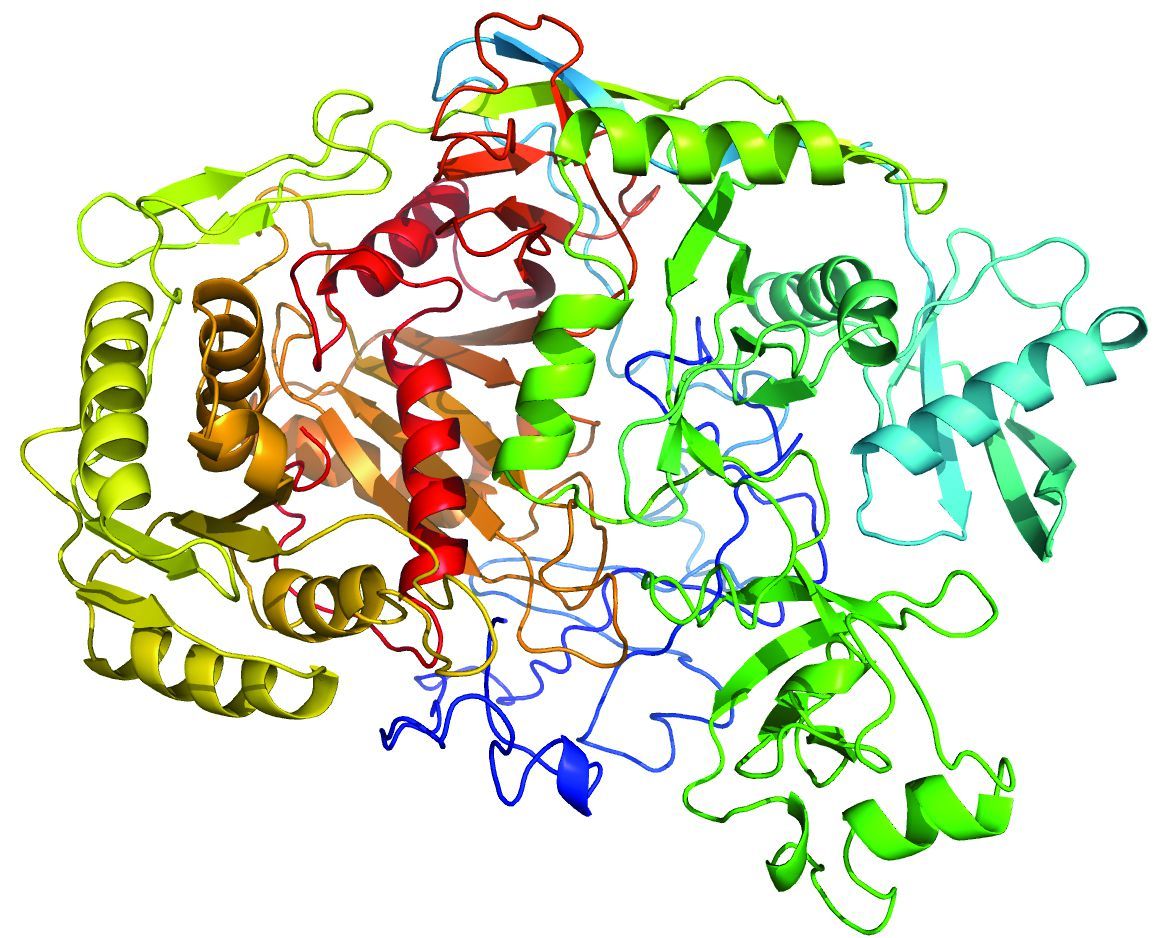
4) 在NgAgo-gDNA系统中,指导序列-靶序列错配容忍度很低,错配一个碱基即减少73%~100%的酶切效率,三个错配则完全没效果。另外,有实验证明gDNA的第8到11 bp位置最重要,这个有待NgAgo的蛋白质结构数据(见图 2)来解释。点评:前四点从多个角度最小化脱靶可能性。
|
图 2 通过串线法预测的NgAgo酶结构 Figure 2 The structure of NgAgo predicted using the threading method |
5) CRISPR-Cas9系统中的sgRNA需要由质粒转入细胞并表达,而后形成一定结构才能工作,可控性很差。举一个最典型例子,如果crRNA富含GC碱基,它会在单链内形成碱基互补配对,即茎环结构,GC碱基配对之间形成三个氢键,因此茎环很难打开,严重影响crRNA与靶序列结合(图 1a)。NgAgo-gDNA系统中的gDNA直接转入细胞,时间和浓度较CRISPR-Cas9系统更可控,但是,NgAgo酶依然要通过表达载体导入,其表达效率等问题依然存在。gDNA理论上不会产生茎环结构(这个还有待深入研究),有实验证明NgAgo-gDNA系统在富含GC碱基区域表现更好(原文献[1]中1图 4f1)。点评:向大量细胞递送gDNA不是那么简单,影响RNAi进入临床的一个主要问题就是递送(Delivery)问题。
6) Cas酶仅仅是剪开双链DNA(图 1b),NgAgo酶不仅剪开DNA,而且同时去除几个碱基,彻底让这个基因的功能丧失。点评:细胞内有一些连接酶,可能会把切断的地方连接上,使基因得到恢复。
7) CRISPR-Cas9系统要求指导序列后面有一个特征三碱基序列(即PAM序列)才能工作,限制了它的作用范围。点评:NgAgo-gDNA系统不要求PAM序列,因而扩大了可以编辑的区域,这点改进最不重要。
该研究起始于另外两个AGO蛋白(TtAgo和PfAgo),它们需要在65 ℃工作。韩春雨等首先通过生物信息学常用的比对软件PSI-BLAST,根据TtAgo和PfAgo的已知序列,搜索NCBI NR非冗余蛋白质序列数据库[2],找到了很多相似的蛋白质序列,都是来自不同物种的AGO蛋白。而后,通过一系列生物信息分析和少量实验,最终找到了可以在37 ℃工作的NgAgo酶。点评:这就是典型的大数据挖掘,这个数据还不够大,第二代测序和第三代测序数据更是海量。PSI-BLAST得到的相似蛋白质序列可能成千上万,不可能逐个去做实验,必须通过生物信息学方法进行初步筛选,初步筛选后得到的少量候选蛋白质才可能进行实验验证,文章没有介绍这个筛选过程,估计应该是经验方法,没有采用当前主流的机器学习算法。如果筛选找不到符合条件的酶,还可以走这条路线:找到温度最接近37 ℃的AGO酶,设计点突变改造。具体来说,就是将酶上每个氨基酸位点当做特征,构建数据集进行机器学习分类或拟合,再通过特征选择筛选出关键位点进行突变设计[3-8]。点评:酶的改造或设计对生物信息学依赖很大。gDNA的24 bp长度的确定(原文献[1]中图 3d),得益于巧妙地利用了质粒中增强型绿色荧光蛋白(Enhanced green fluorescent protein,简称EGFP)的亮度变化来指示酶切割效率,从20~27 bp几种长度中选择了亮度最低(即切割效率最高的)的24 bp长度。这个实验设计非常简单,仅使用了蛋白质印迹法(Western blot)精度就够了,但24 bp与25 bp结果亮度差异不大。无论是蛋白质印迹法还是定量PCR方法都受实验条件和人工操作影响较大,高通量测序可以获得更为精准的比较结果。
下一步生物信息工作可以立刻展开,南开大学阮吉寿、杨建益和高山等通过串线法(Threading)解析了NgAgo酶的结构(见图 2),沿着这条研究路线,可以深入了解gDNA指导以及靶序列切割的机制;通过序列比对以及结构比对,阮吉寿等又获得了很多有相似功能的酶,这些工作几天内即可完成,这是传统单纯使用实验手段望尘莫及的。当务之急是找到更多具有相似功能的酶,利用这个已经成熟的流程或许会有更多新的发现。点评:国内的生物信息研究团队或者个人应该抢先进行大数据挖掘,充分发挥我们国家人多的特点,保持这一领域优势,防止国外高水平实验室抢在前面。另外,实验的跟进也很重要,南开大学陈德富等根据韩春雨提供的NgAgo酶的动物表达载体构建了植物表达载体。
2 又回到了AGO蛋白CRISPR-Cas9与NgAgo-gDNA中用到的生物学机制,普遍认为是来自细菌和古细菌在长期演化过程中形成的一种适应性免疫防御机制,即识别并切割入侵的病毒或外源DNA。NgAgo-gDNA中使用了AGO蛋白,与RNA干涉(RNA interference,简称RNAi)有相似机制,这是更早获得广泛研究的机制,也认为是细胞对于外源病毒的一种防御机制。NgAgo-gDNA是gDNA指导切割外源的双链DNA;RNAi是小干扰RNA (Small interfering RNA,简称siRNA)指导切割外源的双链RNA。细胞内还有更多相似的机制,从这个角度继续挖掘,是一个很重要的研究方向。相关的基础问题有AGO酶作用的核酸复合体种类的特异性(DNA-DNA、DNA-RNA或RNA-RNA);序列特异性(互补、回文以及两端的碱基种类和修饰);细胞内还有更多的酶切割作用,例如miRNA成熟需要切割单链RNA中的茎环结构,都有什么普遍规律?AGO蛋白的故事还没有完,与AGO具有相同结构域(Domain)或模体(Motif)的DNA或RNA结合蛋白(DNA-binding or RNA-binding proteins)还有多少?有没有RNA指导的AGO酶切割双链DNA?是否存在某些生物利用AGO酶对自身基因组进行编辑?AGO从低等生物到高等生物中的广泛存在,又赋予了它进化上的巨大研究价值。例如,切割双链RNA病毒的AGO酶和切割双链DNA病毒的AGO酶的宿主是否和病毒存在共进化关系?当前,普遍认为宿主利用AGO对病毒切割是一种免疫机制,反之,病毒是否利用AGO切割宿主以整合进自己的某些片段?
这里再介绍两个RNAi相关研究的新方向,都是基于当前高通量测序技术的。第一个是新的病毒检测方法。2009年,国际马铃薯中心的Jan Kreuz首先在国际上提出small RNA高通量测序可以作为一种通用手段来检测动植物DNA或RNA病毒 [9]。这种方法具有灵敏度高、能够检测新病毒、不需要已知序列信息和不需要纯化培养等优点。康奈尔大学费章君和高山等开发了第一个基于small RNA高通量测序的病毒检测软件,可以大规模自动化检测动植物携带的病毒组[10-13]。南开大学高山在2013年国际微生物大会(WCM 2013)上提出small RNA测序可以用于临床病毒检测 [14],并通过大数据挖掘检测到六类严重危害人类健康的病毒 [15],分别是EBV、HBV、HCV、HIV、HPV和SMRV。另一个研究方向是通过RNAi中产生的siRNA双链体(siRNA duplex),寻找病毒影响细胞的机制。费章君等发现病毒siRNA片段主要集中于21、22、23和24 bp长度,其中21和22 bp来自病毒,23和24 bp来自宿主[10];高山等分析植物siRNA duplexes主要集中于21 bp而且有对称的2 bp突出(Overhang);根据昆虫small RNA测序大数据挖掘结果,高山等发现动物可能还存在以33 bp为中心的RNA降解或切割机制(未发表)。
3 更多RNA的功能需要进一步揭示CRISPR-Cas9中的crRNA发现于1987年,日本微生物学家石野良纯(Yoshizumi Ishino)在克隆大肠杆菌碱性磷酸酶同工酶(Isozymes of alkaline phosphatase,简称Iap)基因编码序列时,意外发现iap基因的3’端侧翼区(Flanking region)存在一个称作间隔串联重复(Spaced tandem repeat)的DNA片段,它包括五个包含29个保守碱基的重复片段,这些重复片段之间由32个碱基的居间序列(Intervening sequence,简称IVS)隔开。由于受技术和认识限制,很长一段时间内,基因组研究的重点集中于可以转录的区域(转录组),转录组研究的重点又集中于编码蛋白质的序列,导致基因组中重复序列(Repeat)被忽视。因此,后来才知道这个重复序列不仅表达,而且有如此重要的功能。
根据南开大学卜文俊和高山等利用最新的PacBio流程在国际上首次对昆虫进行全长转录组测序[16]的结果发现,基因组中很多过去认为的不转录的重复序列、控制序列、假基因以及各种垃圾序列(Junk DNA)都是转录的,基因组可转录区域由于受二代测序技术限制被低估了[17]。江西师范大学张帆涛、南开大学陈德富和高山在研究水稻(日本晴)转录组时发现了一些新的可变剪接模式,以及大量双向的反义转录本(Antisense transcripts)被误判为一个方向转录。南开大学刘林和高山等通过单细胞测序技术发现,大部分过去认为不表达或无功能的假基因在干细胞或肿瘤细胞中高度表达,并且很可能是有功能的。卜文俊和高山等的研究证实了通过PacBio全长转录组测序可以获得完整的成熟体、转录前体和部分原始转录本,有助于了解RNA从初始转录、加工到成熟以及编辑等方面的机制,也是认识一些非编码RNA(Non-coding RNAs)功能的强有力手段[18]。更多PacBio全长转录组测序方面的内容,参见南开大学高山等编著的《PacBio单分子测序指南》。
4 对于生物信息学研究的启示第一点,该研究的专业归属问题。该研究只用到了非常基本的分子生物学实验方法,其核心工作就是从已有数据库中寻找线索,而后对系统进行优化,这些属于生物信息学的研究内容。第二点,生命科学的研究内容包括了一些分子层面的基本作用元素,简单说就是酶的切割连接、合成降解、碱基互补、核酸与蛋白质以及蛋白质与蛋白质几个层面的相互作用。在不同物种和系统中,一些规律相同或非常相似,通过信息整合再进行实验测试,不仅大大提高效率,而且能够发现一些更高层次的共性或产生更深入的理解。第三点,生物信息学未来研究方向,必须从大数据,特别是高通量数据出发。NgAgo-gDNA系统的成功对生物信息学研究者的最大启发就是当前积累的生物数据没有充分利用,有巨大潜力可以挖掘。
5 专利保护与技术保护当前,也有一些“专家”对NgAgo-gDNA系统的原创性提出质疑,其中一个重量级的证据就是驯鹿生物科学公司(Caribou Biosciences)的专利(WO 2014/189628 A1),它保护了一种DNA指导的AGO酶系统,并且专利保护扩展到了具有一定同一性(identity)的蛋白质序列。点评:专利只是停留在纸上(很多专利是扩展保护,其实并没有相应技术),开发一个可以实用的基因组编辑系统的原创性不容置疑,现在如果能找到工作在更低温度的AGO酶(植物转基因所需)依然是原创性工作。况且,基因组方面的专利保护,涉及到基因或蛋白序列,基本上毫无可操作性。第一,基因组学研究的对象是自然界存在的天然物质(注意与计算机软硬件的人工产物不同),测序序列虽然是劳动产物,但是其包含的信息难以纳入私人产权,况且这些结果包括了大量前人公开的成果或数据(例如引物可能来自NCBI数据库)。曾经多次有人试图将人类基因组测序结果纳入专利保护,最终还是失败了。第二,即使可以将某些增量信息(例如新发现一条突变序列)纳入专利保护,也没有一个标准可以参考。举个简单例子,某人测了一条AGO蛋白,并且首次发现它有某个功能A,可以用于基因组编辑,但不能把AGO蛋白注册为他的。自然界相似的蛋白质序列数量惊人,即使能够注册了这条蛋白质序列,当然可以允许它设定一个同一性阈值扩展保护,那么这个阈值如何设,没有标准可以参考,设90%可否?对于一个非常保守的蛋白质,90%的同一性,可能从脊椎动物跨越到无脊椎动物。再举一个例子,某人发现一个蛋白,本身可能是无法实际应用的(例如要求65 ℃才能工作),另外一个人做几个点突变就可以实际应用(例如可以在常温下工作),同一性可能保持99%,如果第一个人的专利获批了,就阻止了后人的技术开发。NgAgo酶的序列来自NCBI,其工作条件37 ℃等天然属性是韩春雨等发现的,专利保护可以覆盖以37 ℃为中心一定范围内工作的AGO酶么?或保护全部与gDNA一起工作的AGO酶么?唯一能保护的就是实验或临床工作时的流程或相关技术(比如大规模细胞的递送技术)。专利保护的逻辑悖论就是,不注册专利没人知道,仿造不出来;去注册专利会导致技术泄密。在科研成果保护方面,中国不要跟随西方体制,盲目崇拜专利。对于大的垄断公司,核心技术往往首选技术保密,其次才去申请专利,没什么实用价值的再去发论文,论文发出来,大家都学会了方法,也就没法保护了。专利保护更适合大家都看得见的外观设计等非核心技术方面。另外,欧美大公司为了实现技术垄断,围绕一个技术写很多关系不大的东西,把有可能想到的实际做不出来的都保护上,目的就是阻止落后国家开发新技术。况且,专利注册消耗的精力太大,CRISPR-Cas9的发明者消耗了大量精力抢夺CRISPR-Cas9专利,才给了其他人发明新基因组编辑系统的机会。因此,本文作者建议对于我国重要的达到国际一流的技术采取专项经费支持,走技术保密路线,既不发表英文论文也不申请专利,避免与发达国家产生技术纠纷。
致谢 感谢科学网各位老师对这项工作的评论与传播,主要有孙学军、许培扬、丁广进、侯成亚、杜立智、戴德昌、王毅翔、张忆文、刘立、牛登科、陆绮、徐晓、姬扬、曾泳春、李春杰、田云川、吕洪波、王涛、姚伯元、任文龙、张钫、马志超、赵保明、史晓雷、王伟、石磊、罗教明、袁海涛、秦逸人、罗湘南、孟凡、张洋、沈律、陈方锐、黄彬彬、张超、刘建彬、黄秀清、王林平、李红雨和邵鹏等。
| [1] |
GAO F, SHEN X, JIANG F, et al. DNA-guided genome editing using the Natronobacterium gregoryi Argonaute[J]. Nature Biotechnology, 2016.
DOI:10.1038/nbt.3547 ( 0) 0)
|
| [2] |
高山, 欧剑虹, 肖凯. R语言与Bioconductor生物信息学应用. 天津: 天津科技翻译出版公司, 2014 .
GAO Shan, OU Jianhong, XIAO Kai. Using R and bioconductor in bioinformatics (in Chinese). Tianjin: Tianjin Science and Technology Translation Publishing Co, 2014 .
( 0) 0)
|
| [3] |
GAO S, ZHANG N, DUAN G, et al. Prediction of function changes associated with single-point protein mutations using support vector machines (SVMs)[J]. Human Mutation, 2009, 30(8): 1161–1166.
DOI:10.1002/humu.v30:8 ( 0) 0)
|
| [4] |
GAO S, FANG J. Predicting kinase-specific phosphorylation sites using a multitask classification framework[J]. in 2011 IEEE International Conference on Bioinformatics and Biomedicine, BIBM 2011, November 12, 2011.
DOI:10.1109/BIBM.2011.57 ( 0) 0)
|
| [5] |
GAO S, XU S, FANG Y, et al. Using multitask classification methods to investigate the kinase-specific phosphorylation sites[J]. Proteome Science, 2012, 10(Suppl 1): S7.
DOI:10.1186/1477-5956-10-S1-S7 ( 0) 0)
|
| [6] |
ZHANG N, LI B, GAO S, et al. Computational prediction and analysis of protein γ-carboxylation sites based on a random forest method[J]. Molecular Biosystems, 2012, 8(11): 2946–2955.
DOI:10.1039/c2mb25185j ( 0) 0)
|
| [7] |
FANG Y, GAO S, TAI D, et al. Identification of properties important to protein aggregation using feature selection[J]. Bmc Bioinformatics, 2013(14): 314.
( 0) 0)
|
| [8] |
ZHANG N, GAO S, CHEN L, et al. Using multitask learning methods to investigate signal peptides and signal anchors[J]. Current Bioinformatics, 2013, 8(5): 533–538.
DOI:10.2174/1574893611308050002 ( 0) 0)
|
| [9] |
Kreuze J F, PEREZ A, UNTIVEROS M, et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: a generic method for diagnosis, discovery and sequencing of viruses[J]. Virology, 2009, 388(1): 1–7.
DOI:10.1016/j.virol.2009.03.024 ( 0) 0)
|
| [10] |
LI R, GAO S, HERNANDEZ A G, et al. Deep sequencing of small RNAs in tomato for virus and viroid identification and strain differentiation[J]. PLoS ONE, 2012, 7(5): e37127.
DOI:10.1371/journal.pone.0037127 ( 0) 0)
|
| [11] |
LI R, GAO S, FEI Z, et al. Complete genome sequence of a new tobamovirus naturally infecting tomatoes in mexico[J]. Genome Announcements, 2013, 1(5): e00794-13.
( 0) 0)
|
| [12] |
PADMANABHAN C, GAO S, LI R, et al. Complete genome sequence of an emerging genotype of tobacco streak virus in the United States[J]. Genome Announcements, 2014, 2(6): e01138-14.
DOI:10.1128/genomeA.01138-14 ( 0) 0)
|
| [13] |
LI R, GAO S, BERENDSEN S, et al. Complete genome sequence of a novel genotype of squash mosaic virus[J]. Genome Announcements, 2015, 3(1): e01583-14.
DOI:10.1128/genomeA.01583-14 ( 0) 0)
|
| [14] |
GAO S, LI R, LING K, et al. A novel method to detect Virome based on small RNA deep sequencing technologies[J]. in BIT’s 3rd Annual World Congress of Microbes , WCM 2013, July 30, 2013.
DOI:10.1016/j.jcv.2014.06.013 ( 0) 0)
|
| [15] |
WANG Fang, SUN Yu, RUAN Jishou, et al. Using small RNA deep sequencing to detect human viruses[J]. BioMed Research International, 2016, 2016(2016): 9.
( 0) 0)
|
| [16] |
任毅鹏, 张佳庆, 孙瑜, 等. 基于PacBio平台的全长转录组测序[J]. 科学通报, 2016, 61(11): 1250–1254.
REN Yipeng, ZHANG Jiaqing, SUN Yu, et al. The study of full-length transcriptome sequencing on PacBio platform (in Chinese)[J]. Chinese Science Bulletin, 2016, 61(11): 1250–1254.
( 0) 0)
|
| [17] |
刘圣, 冯祖仁, 高山. 下一代测序数据的质量控制研究[J]. 军事医学, 2014(005): 377–380.
LIU Sheng, FENG Zuren, GAO Shan, et al. Study on quality control of the next-generation sequencing data[J]. Military Medicine, 2014(005): 377–380.
( 0) 0)
|
| [18] |
GAO S, REN Y, SUN Y, et al. PacBio Full-length transcriptome profiling of insect mitochondrial gene expression[J]. RNA Biology, 2016, 13(6): 635.
DOI:10.1080/15476286.2016.1197481 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


