2. 辽宁大学环境学院, 沈阳 110036;
3. 辽宁大学轻型产业学院, 沈阳 110036
2. School of Environmental Science, Liaoning University, Shenyang 110036, China;
3. College of Light Industry, Liaoning University, Shenyang 110036, China
大肠杆菌DnaK蛋白是热休克蛋白70(Hsp70,Heat shock protein 70)家族的重要成员,以分子伴侣形式发挥作用,帮助新蛋白质的正确折叠,错误折叠和聚集蛋白的重新折叠,调节蛋白的活性控制,细胞器和分泌蛋白的跨膜易位等[1-2],而因为DnaK在蛋白质内稳态网络中具有的中心作用,对DnaK的结构和功能的研究是很有意义和价值的[3]。
DnaK蛋白与其他Hsp70s相同,都由一个N-端高度保守的核苷酸结合功能域和一个C-端的底物结合功能域(SBD,substrate-binding domain)组成,两者之间可以通过变构作用进行相互调节,相互影响[4]。其中,NBD的结构类似于凝集素和己糖激酶,由4个子域组成,分别是Ⅰ-A(残基3-38;112-184),Ⅱ-A(残基185-228;310-388),Ⅰ-B(残基39-111),Ⅱ-B(残基229-309)。这些子域通过两个交叉的α-螺旋相连接,在子域的中心形成一个包围核苷酸与金属粒子的特定结合口袋,对ATP进行水解,而在NBD对ATP水解时,能够刺激增强C-端另一功能域SBD部位的底物亲和能力,从而降低SBD的底物交换率,影响Hsp70发挥伴侣活性与帮助蛋白重折叠等功能,所以研究核苷酸结合功能域对ATP的水解能力,即ATPase活性,对研究Hsp70的各种功能有重要意义[5-6]。
之前,Peter等人的研究指出,DnaK蛋白中位于NBD的Ⅱ-A和Ⅱ-B之间的一些在域间通信中起扭链作用的残基,在变构调节中发挥重要作用[7],他们选出一些重要的扭链残基(见图 1),对其进行突变后,发现其ATPase活性发生了变化。其中,突变体S203A、G228A的ATPase活性增强,突变体I202A、L227A的ATPase活性减弱,突变体G223A和WT的ATPase活性基本一致。但是,Peter等人并没有从分子水平去探讨扭链残基突变体ATPase活性变化的原因,对DnaK蛋白中扭链残基突变与ATPase活性变化之间的关系研究仍不太清楚,尚需进一步研究。另外,McKay等人研究发现牛Hsc70(Heat shock cognate 70 protein)的T13位点(同源于DnaK蛋白T11)的自身磷酸化与ATPase活性有重要关系[8],并且T13侧链的烃基对ATPase活性有重要作用[9]。此外,Chiappori 等人研究Hsp70与Hsp40相互作用时发现,DnaK蛋白中β220(214-221)部位的紧致性对ATPase活性也有重要影响,这部位结构越紧致,越容易与DnaJ蛋白的J结构域相结合,从而促进ATP的水解[15],并且β220与这些突变的扭链残基位于同一部位上,这些扭链残基突变后,可能使相连的β220部位的紧致性产生了改变,从而使其与DnaJ蛋白J结构域相互作用情况发生改变,影响DnaJ对ATP的水解刺激作用,影响ATPase活性,而我们的模拟结果也证实了这一点。
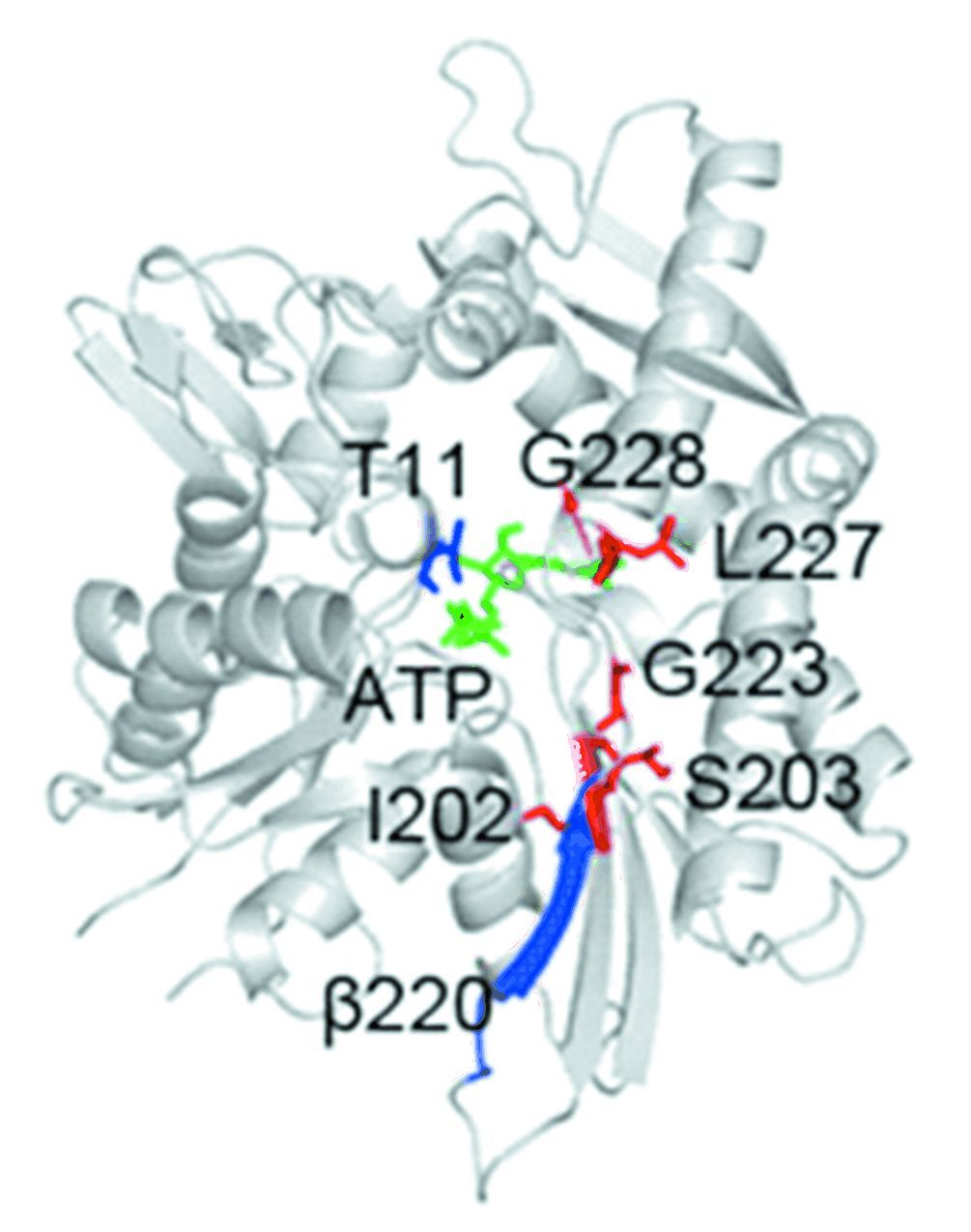
|
图 1 WT模型平衡后构象展示及各扭链残基与重要部分 Figure 1 The show of WT models and the hinge residues and important parts after equilibrium |
本研究不仅是对Peter等人生化试验数据进行分子动力学上的解释,也是对Chiappori 等人研究结果的进一步验证,对后续其他Hsp70蛋白家族中氮端NBD,特别是扭链残基的研究具有重要的借鉴与指导意义。
1 实验过程 1.1 蛋白质来源及同源建模本研究采用RCSB蛋白数据库中大肠杆菌DnaK蛋白经典核苷酸结合域模型(PDB编号1DKG: D)[10],并参考与DnaK蛋白同族的包含有小分子ATP的Hsp70与ATP结合状态模型(PDB code:4B9Q: A)为模板[11],通过同源建模软件Modeller9v8构建DnaK蛋白NBD-ATP 结合状态的WT与突变体I202A、S203A、G223A、L227A、G228A三维结构模型。所有结构模型均使用Procheck对其合理性进行了评估。
1.2 动力学模拟过程分子动力学模拟均在GROMACS 4.5.5 [11] 软件包下完成,模拟体系DnaK蛋白核苷酸结合域直接使用GROMOS96 43a1力场[12],小分子ATP使用PRODRG2服务器(http://davapc1.bioch.dundee.ac.uk/cgi-bin/prodrg)添加基于GROMACS的小分子ATP的力场[13]。然后将蛋白配体模型溶于约包含26 186个SPC216水分子的立方体盒子中,蛋白边缘与盒子边缘的最小距离为1.0 nm。采用LINCS(linear constraint solver)算法对体系中所有键长进行约束。范德华力通过LJ势(Lennard-Jones potential)方法进行估算,截断半径为1.0 nm。静电相互作用采用PME(Particle mesh Ewald)算法进行估算,截断半径为1.0 nm。体系采用NPT系综,温度和压力通过采用V-rescale 和Parrinello-Rahman 算法分别维持在300 K和1 bar,pH为7.0。体系中加入Na+或Cl-中和多余电荷,中和后的中性体系先进行2 000步的能量最小化,然后进行80 ps的限制性模拟。最后各体系在恒定的温度和压力下进行10 ns分子动力学模拟。分子的运动轨迹每4 ps保持一次,用于后续的数据分析。
1.3 蛋白对接本模拟利用PatchDock网站服务器进行蛋白-蛋白对接实验[14],DnaJ模型使用(PDB编号1XBL: A),相应结合位点根据Chiappori等人文献中确定[15],其他设置默认,然后与平衡后各模型分别进行对接,根据打分函数对结果进行相应排序,用网站上后续工具FireDock评测其中最优的10个构象结合所需的结合能[17]。
2 结果与讨论 2.1 结构稳定性RMSD ( Root mean square deviation,均方根偏差) 是评价蛋白质稳定性的一个重要参数,通过比较RMSD曲线我们发现各模型在7ns后均达到结构保持稳定的平衡状态(见图 2)。

|
图 2 NBD主链的RMSD* Figure 2 The RMSD values of the backbone of NBD 注:*彩图见电子版(http://swxxx.alljournal.cn/ch/index.aspx)(2016年第3期doi:10.3969/j.issn.1672-5565.2016.03.03 )。 |
其中除S203A外,所有突变体的RMSD波动都比较大,但活性增强突变体S203A其实在5-7ns时波动也较野生型大,平衡后构象也与野生型也有很大差别(见图 3)。这说明所有突变体与野生型相比,其构象都发生了一定变化。平衡过程中,各模型的整体RMSD值,分别如下:WT(0.24 nm),I202A(0.27 nm),S203A(0.26 nm),G223A(0.30 nm),L227A(0.32 nm),G228A(0.35 nm),并且RMSD值明显随扭链残基所在部位不同,呈现分级趋势,202位点与203位点,虽然ATPase活性变化相反,但两位点却相邻在一起,而223、227、228这三个位点RMSD值比其他都大,它们也都在另一相连部位(见图 3)。从这可以看出,突变不同部位的扭链残基,其引起的波动程度也是有差别的。而为什么活性基本不变的突变体G223A的RMSD波动也非常大,可能是其引起波动的部位与ATPase活性无关或者影响作用被抵消的原因,这需要进一步研究。
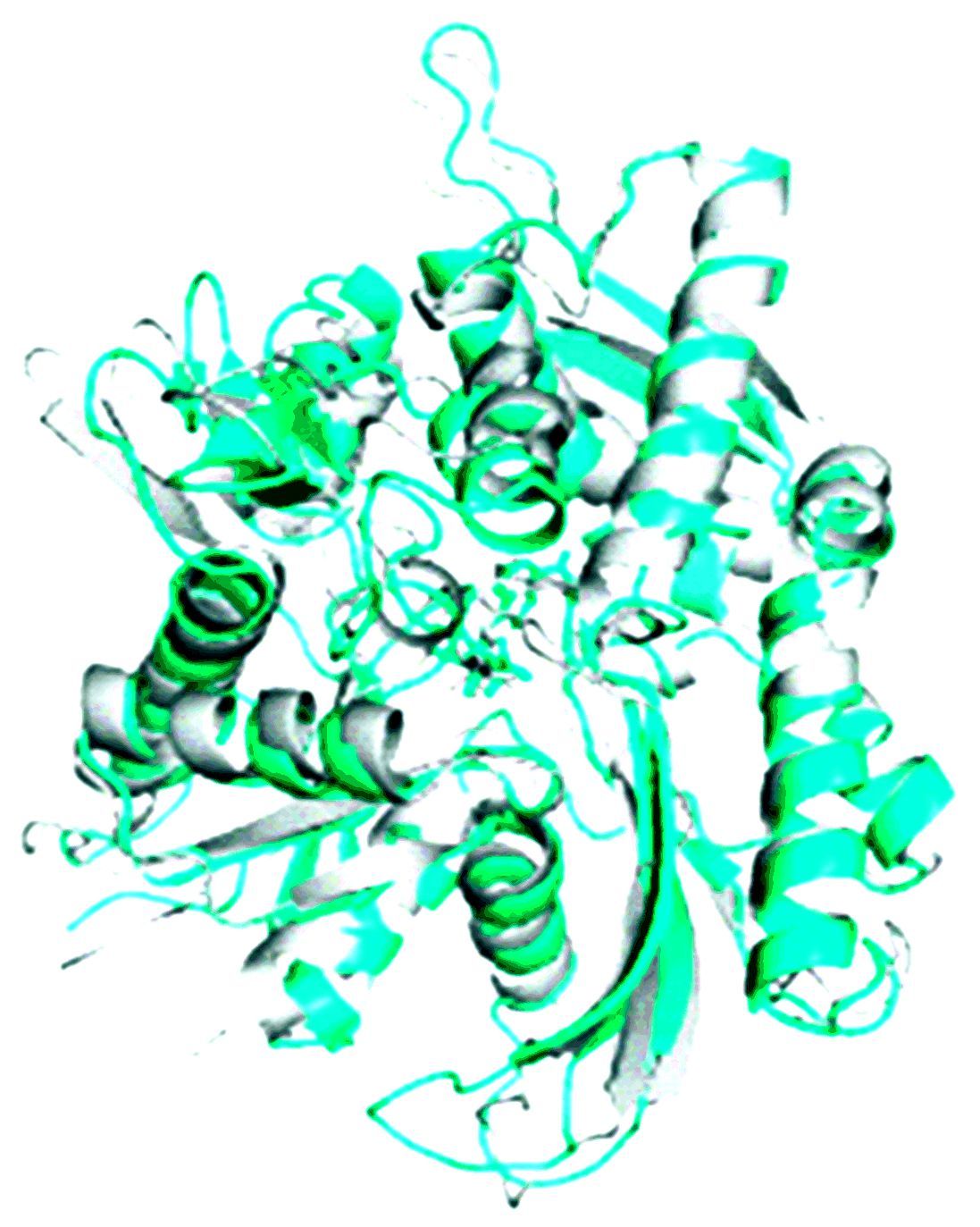
|
图 3 野生型WT与活性增强突变体S203A平衡后三维构象对比 Figure 3 The comparison of three-dimensional conformation between WT and the positive mutant S203A after equilibrium |
ATP与NBD间的相互作用是影响DnaK蛋白ATPase活性变化的直接原因之一,主要有氢键、盐桥、疏水作用,结果如下(见表 1),其中G223A突变体活性虽然与野生型差不多,但与ATP相互作用是增强的,尤其是G223A的氢键作用是远远强于其他突变体的,NBD与ATP间相互作用结果并没有什么明显的规律。
不过进一步查看NBD与ATP间氢键作用存活时间发现(见图 4),影响ATPase活性的重要残基T11与ATP之间的氢键作用具有明显规律。活性不变突变体G223A虽然整体与ATP氢键较多,但其T11:ATP只有一条(100%),与野生型的类似T11:ATP(98%),活性减弱突变体I202A没有T11:ATP的氢键,而L227A只有一条T11:ATP(92%),但存活时间比野生型弱,这可能与其活性减弱有一定关系。并且活性增强突变体S203A、G228A都具有两条强T11:ATP的氢键,S203A的两条还强于G223A突变体的,ATPase活性规律符合,这很可能是影响扭链残基突变体活性变化的重要原因。
| 表 1 模拟过程中各模型NBD与ATP间相互作用结果 Table 1 The results of interaction between NBD and ATP in all the models during MDs |

|
图 4 模拟过程中NBD与ATP间氢键存活时间 Figure 4 The Hydrogen bond existence map of NBD and ATP during MDs |
进一步根据McKay等人的研究,提取T11与ATP残基的距离发现(见图 5a),活性减弱突变体I202A、L227A的T11在整个模拟过程中,与野生型和活性增强、活性不变突变体相比,T11明显的远离了ATP,使其更难与ATP相互作用,这可能是其与ATP间氢键作用减弱的主要原因。
而继续提取T11的烃基与ATP的γ-磷酸基团间的距离(见图 5b)发现,扭链残基突变体T11距离的变化与ATPase活性间的关系更加明显,其平衡过程中的T11:ATP平均距离如下:WT(0.43 nm),活性增强突变体S203A(0.40 nm)、G228A(0.39 nm),活性减弱突变体I202A(0.59 nm)、L227A(0.45 nm),活性基本不变突变体G223A(0.42 nm)。活性增强突变体的T11烃基与ATP的γ-磷酸距离变近,更易接触,形成更多的氢键作用;活性减弱突变体间的T11烃基与ATP的γ-磷酸距离变远,更难接触,形成较少或没有氢键作用,虽然L227A的距离增大并不太明显,氢键结果也显示了这一点,只是稍微减弱,可能还有其他影响L227A的ATPase活性变化的原因;另外,活性基本不变突变体T11烃基与ATP的γ-磷酸距离稍微减小,基本不变,氢键作用强弱也不改变,对ATP的水解活性也基本不变,在一定程度上能说明Peter等人的生化实验结果。
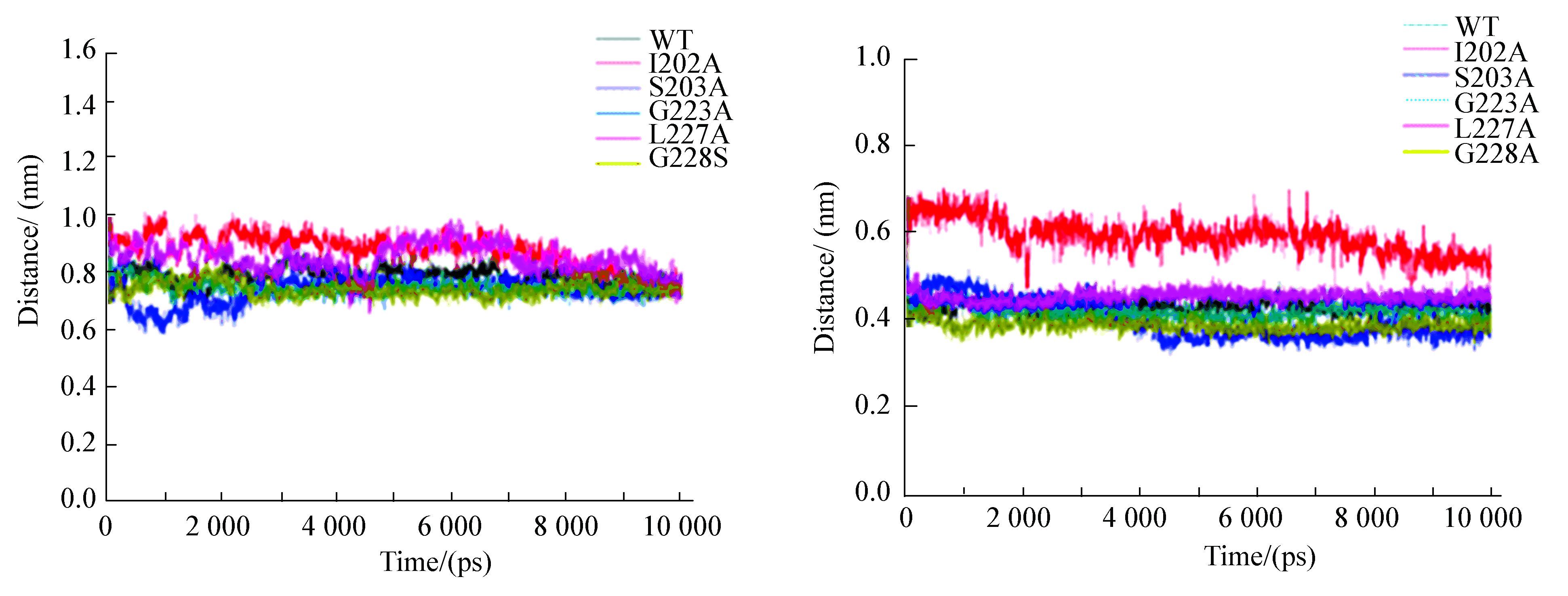
|
图 5 重要位点T11与ATP距离* Figure 5 The distance of important loci T11 and ATP 注:*彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2016年第3期doi:10.3969/j.issn.1672-5565.2016.03.03 )。 |
进一步查阅文献,Chiappori等人的研究结果表明,DnaK的β220(214-221)部分紧致性能影响与DnaJ结合,从而影响ATPase活性[15],而扭链残基都与β220部分相连,推测是不是这些扭链残基突变后,从而影响了相连的与DnaJ结合β220部分发生改变,从而造成ATPase活性发生变化。
而蛋白质结构的紧致性可以通过其Rg(Radius of gyration,回旋半径)来分析[16],Rg越大,表明其紧致性越低,通过提取各模型在模拟过程中β220的Rg发现(见图 6),活性减弱突变体I202A、L227A的Rg增大很多,I202A(0.51 nm)、L227A(0.51 nm),其β220部位紧致性降低,其中,I202A的Rg在整个模拟过程中都比WT高很多,L227A的Rg在平衡过程中急剧升高,最后体系平衡后,活性减弱突变体的Rg都保持在一个很相似的程度,这也表明,平衡之后的Rg值可能更准确。另外如图 5所示,平衡过程中,活性基本不变突变体G223A与WT的Rg基本保持一致G223A(0.48 nm)、WT(0.48 nm),而活性增强突变体S203A、G228A的Rg减小,S203A(0.47 nm)、G228A(0.47 nm),紧致性增强,可见,与突变点相连的β220部位的紧致性在ATPase活性变化中具有一定的规律,与最后ATPase活性变化结果符合,故扭链残基突变后,可能也通过引起相连的β220部位紧致性发生改变,从而影响与DnaJ的结合,影响了ATPase的活性,对T11烃基与ATP的γ-磷酸距离变化规律形成补充,共同影响造成最后ATPase活性变化结果。
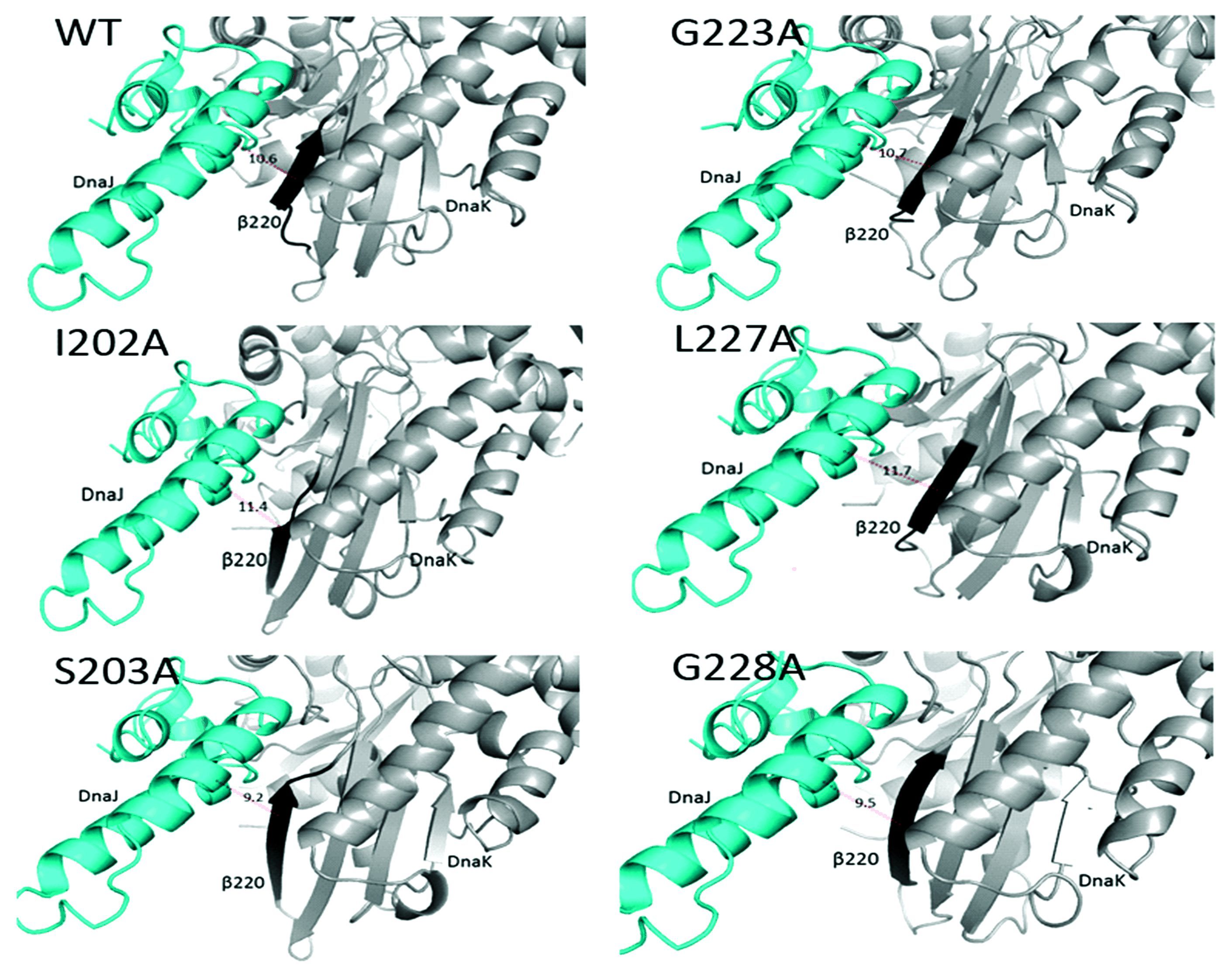
2.4 蛋白-蛋白对接与结合能分析通过PatchDock网站进行DnaK与DnaJ对接,结果见图 7,以对接后得分最高构象为例,DnaK与DnaJ对接后,野生型与活性基本不变突变体DnaK和DnaJ间的平均距离差别不大,WT(10.6 nm)、G223A(10.7 nm),活性减弱突变体与DnaJ的平均距离增大,I202A(11.4 nm)、L227A(11.7 nm),活性增强突变体与DnaJ的平均距离减小,S203A(9.2 nm)、G228A(9.5 nm),这一结果能在一定程度上反应DnaK与DnaJ蛋白作用情况,但两蛋白之间相互作用距离只是反映其相互作用强弱的一个方面,进一步对大范围各模型与DnaJ对接结果进行分析,主要是能直接反应相互作用情况的平均所需结合能的分析(见表 2)。
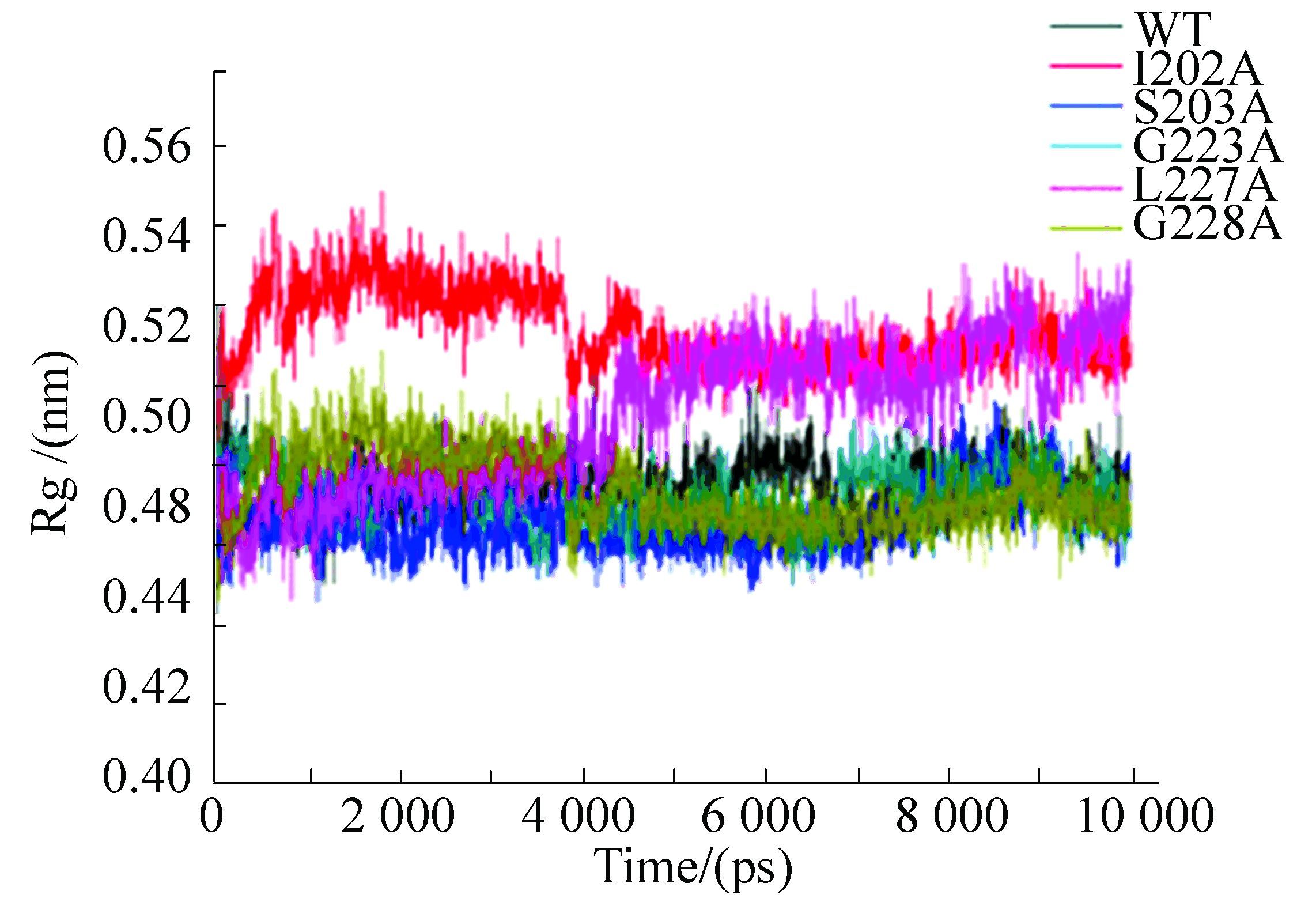
|
图 6 模拟过程中β220 Rg随时间的变化* Figure 6 ime dependence of the Rg in region β220 during MDs 注:*彩图见电子版(http://swxxx.alljournals.cn/ch/index.aspx)(2016年第3期doi:10.3969/j.issn.1672-5565.2016.03.03 )。 |
发现其中所有突变体都比野生型形成更多构象,DnaK与DnaJ接触面积增加,虽然活性减弱突变体反而倾向形成最多的构象,I202A(638个)、L227A(622个),远远多于其他突变体。不过进一步的结合所需能量结果发现,活性减弱突变体虽然倾向形成更多构象,但其结合所需能量远远高于其他突变体,更难与DnaJ结合,影响DnaJ刺激ATP水解。而活性不变突变体G223A所需结合能显示与野生型相差不多,活性增强突变体S203A、G228A所需结合能量比野生型减少,更有利于结合。
|
图 7 各DnaK模型与DnaJ蛋白对接后得分最高构象及平均距离展示 Figure 7 The highest score conformation after DnaK model docking with the DnaJ and the average distance |
| 表 2 蛋白对接结果统计 Table 2 The statistical results of protein docking |
这说明突变体的紧致性变化的确影响会其与DnaJ的结合情况,从而影响ATPase活性变化。至于为什么活性减弱突变体倾向形成最多的突变体,这可能与其β220下面部分形成更多的β片层有关。另外,也不排除β220及其相连部位也是NBD向SBD域间信号传递的重要扭链区域之一,将核苷酸信号改变通过一系列扭链区域作用传递到域间linker和SBD中[7],那么反过来,β220及其相连部位扭链的结构改变,如紧致性、β片层程度变化,也可能会对ATPase活性产生直接影响,但具体是怎样影响和传递的,本实验还没有找到明确规律,尚需要进一步的研究。
3 结 论通过分子动力学模拟的实验数据可知,活性增强突变体S203A、G228A突变引起ATPase活性区域重要残基T11的烃基与ATP的γ-磷酸基团靠近,使ATP更容易与T11发生反应,提高ATP水解速率,从而增强ATPase活性;活性基本不变突变体G223A突变没有使T11烃基与ATP的γ-磷酸基团距离发生改变,从而使ATP水解速率与野生型时一样,不改变ATPase活性;活性减弱突变体I202A突变使T11的烃基与ATP的γ-磷酸基团远离,使ATP更难与T11发生作用,从而降低ATP水解速率,降低ATPase活性,L227A虽然远离的不明显,但其与突变残基相连的β220(214-221)部位的紧致性降低,所需结合能增大,难与DnaJ结合,从而影响了其ATPase活性。另外,活性增强突变体S203A、L227A突变使相连的β220的紧致性增强,更易结合,所需结合能减小。活性基本不变突变体G223A紧致性与所需结合能结果也与ATPase活性结果相符,所以,扭链残基突变体很可能是通过突变引起T11部分与β220部分结构改变,从而影响了其ATPase活性的。
本研究为我们从分子水平上解释Peter等人的生化实验结果提供了数据支持,同时也从分子水平对McKay等人研究发现的牛Hsc70的T13位点(同源于DnaK蛋白T11)侧链的烃基对ATPase活性有重要影响在DnaK中进行了验证,另外,也对Chiappori 等人研究发现的DnaK蛋白中β220(214-221)部位的紧致性对ATPase活性也有重要影响的结果进行了进一步的验证,对后续的Hsp70家族NBD部分突变及机制的研究有重要的借鉴作用。
| [1] |
MCCLELLAN A J, TAM S, KAGANOVICH D, et al. Protein quality control: chaperones culling corrupt conformations[J]. Nature Cell Biology, 2005, 7(8): 736–741.
DOI:10.1038/ncb0805-736 ( 0) 0)
|
| [2] |
ALBANSE V, YAM A Y W, BAUGHMAN J, et al. Systems analyses reveal two chaperone networks with distinct functions in eukaryotic cells[J]. Cell, 2006, 124(1): 75–88.
DOI:10.1016/j.cell.2005.11.039 ( 0) 0)
|
| [3] |
PATURY S, MIYATA Y, GESTWICKI J E. Pharmacological targeting of the Hsp70 chaperone[J]. Current Topics in Medicinal Chemistry, 2009, 9(15): 1337.
DOI:10.2174/156802609789895674 ( 0) 0)
|
| [4] |
GOŁAŚE , MAISURADZE G G, SENET P, et al. Simulation of the opening and closing of Hsp70 chaperones by coarse-grained molecular dynamics[J]. Journal of Chemical Theory and Computation, 2012, 8(5): 1750–1764.
DOI:10.1021/ct200680g ( 0) 0)
|
| [5] |
MAYER M P, BUKAU B. Hsp70 chaperones: cellular functions and molecular mechanism[J]. Cellular and Molecular Life Sciences, 2005, 62(6): 670–684.
DOI:10.1007/s00018-004-4464-6 ( 0) 0)
|
| [6] |
HARTL F U, HAYER-HARTL M. Converging concepts of protein folding in vitro and in vivo[J]. Nature Structural & Molecular Biology, 2009, 16(6): 574–581.
( 0) 0)
|
| [7] |
UNG P M U, THOMPSON A D, CHANG L, et al. Identification of key hinge residues important for nucleotide-dependent allostery in E. coli Hsp70/DnaK[J]. PLOS Computational Biology, 2013, 9(11): e1003297.
DOI:10.1371/journal.pcbi.1003297 ( 0) 0)
|
| [8] |
CHANG L, THOMPSON A D, UNG P, et al. Mutagenesis reveals the complex relationships between ATPase rate and the chaperone activities of Escherichia coli heat shock protein 70 (Hsp70/DnaK)[J]. Journal of Biological Chemistry, 2010, 285(28): 21282–21291.
DOI:10.1074/jbc.M110.124149 ( 0) 0)
|
| [9] |
SOUSA M C, MCKAY D B. The hydroxyl of threonine 13 of the bovine 70-kDa heat shock cognate protein is essential for transducing the ATP-induced conformational change[J]. Biochemistry, 1998, 37(44): 15392–15399.
DOI:10.1021/bi981510x ( 0) 0)
|
| [10] |
HARRISON C J, HAYER-HARTL M, DI LIBERTO M, et al. Crystal structure of the nucleotide exchange factor GrpE bound to the ATPase domain of the molecular chaperone DnaK[J]. Science, 1997, 276(5311): 431–435.
DOI:10.1126/science.276.5311.431 ( 0) 0)
|
| [11] |
KITYK R, KOPP J, SINNING I, et al. Structure and dynamics of the ATP-bound open conformation of Hsp70 chaperones[J]. Molecular Cell, 2012, 48(6): 863–874.
DOI:10.1016/j.molcel.2012.09.023 ( 0) 0)
|
| [12] |
GUEX N, PEITSCH M C. SWISS-MODEL and the Swiss-Pdb Viewer: an environment for comparative protein modeling[J]. Electrophoresis, 1997, 18(15): 2714–2723.
DOI:10.1002/(ISSN)1522-2683 ( 0) 0)
|
| [13] |
SCHUTTELKOPF A W, VAN AALTEN D M F. PRODRG: a tool for high-throughput crystallography of protein-ligand complexes[J]. Acta Crystallographica Section D: Biological Crystallography, 2004, 60(8): 1355–1363.
DOI:10.1107/S0907444904011679 ( 0) 0)
|
| [14] |
SCHNEIDMAN-DUHOVNY D, INBAR Y, NUSSINOV R, et al. PatchDock and SymmDock: servers for rigid and symmetric docking[J]. Nucleic Acids Research, 2005, 33(suppl 2): W363–W367.
( 0) 0)
|
| [15] |
AHMAD A, BHATTACHARYA A, MCDONALD R A, et al. Heat shock protein 70 kDa chaperone/DnaJ cochaperone complex employs an unusual dynamic interface[J]. Proceedings of the National Academy of Sciences, 2011, 108(47): 18966–18971.
DOI:10.1073/pnas.1111220108 ( 0) 0)
|
| [16] |
PRONK S, PÁLL S, SCHULZ R, et al. GROMACS 4.5: a high-throughput and highly parallel open source molecular simulation toolki[J]. Bioinformatics, 2013, 29(7): 845–854.
DOI:10.1093/bioinformatics/btt055 ( 0) 0)
|
| [17] |
MASHIACH E, NUSSINOV R, WOLFSON H J. FiberDock: a web server for flexible induced-fit backbone refinement in molecular docking[J]. Nucleic Acids Research, 2010, 38(suppl 2): W457–W461.
( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


