热休克蛋白(Heat shock protein,Hsp)是生物体受到环境中物理、化学、生物等因素刺激时发生应激反应而在细胞中产生的一类应激蛋白[1]。按照其分子量大小的不同,热休克蛋白可以分为Hsp20(sHsp),Hsp40 (J-class protein),Hsp60,Hsp70,Hsp90和Hsp100等6个家族[2],它们在细胞中发挥着不同的生物学功能。
自1962年首次在果蝇体内发现以来[3],研究人员已从细菌,酵母,线虫,大鼠,小鼠,和人类等生物体内分别发现了热休克蛋白的存在。近年来,热休克蛋白的应用更是十分广泛,从农业到抗癌试剂的制备,热休克蛋白都扮演着重要的角色。
然而我们对热休克蛋白进化关系的认识仍旧有限,因此通过比较基因组学和生物信息学方法对热休克蛋白进行研究,将帮助我们阐明热休克蛋白的进化规律,对理解热休克蛋白的功能也具有重要的生物学意义。
Hsp90作为热休克蛋白家族中的一类应激蛋白,广泛存在于各类机体中,古老且具有高度保守性[4]。其家族之下又分为五个亚家族,分别是Hsp90A,Hsp90B,Hsp90C,TRAP和HtpG亚家族,其中Hsp90A亚家族在Hsp90家族中数量是最多的,分布于除细菌外的其他所有物种中,Hsp90C亚家族只存在于植物中,而HtpG亚家族则绝大多数存在于细菌中[5]。2006年,Chen[5]等人通过对32个物种的Hsp90家族的研究,建立了包含所有Hsp90家族信息及进化的框架。
作为一种重要的分子伴侣,Hsp90参与多种生理过程,如信号传导、蛋白质折叠降解和多种应激反应的调节[6]。研究表明Hsp90的表达与多种癌症是密切相关的,如肺癌,胰腺癌等,因此可以通过检测Hsp90的含量进而有效地监控癌症[7]。然而,随着科技的飞速发展,我们需要快速简洁的从生物信息学的角度去分析Hsp90家族的进化情况,进而研究Hsp90的功能作用,确定其进化及基因结构情况,验证Hsp90五个亚家族在物种间的分布情况以及亚细胞定位情况。
1 材料与方法 1.1 材料从HSPIR(http://pdslab.biochem.iisc.ernet.in/hspir/)数据库,收集百日咳博代氏杆菌(Bordetella pertussis),破伤风杆菌(Clostridium tetani),致病疫霉(Phytophthora infestans),四膜虫(Tetrahymena thermophile),构巢曲霉(Aspergillus nidulans),新型隐球菌(Cryptococcus neoformans),莱茵衣藻(Chlamydomonas reinhardtii),埃及斑蚊(Aedes aegypti),大豆(Glycine max),高粱(Sorghum bicolor) 和水稻(Oryza sativa)11个物种的Hsp90基因家族成员的CDS(或基因)序列和蛋白质序列。
由此得到来自上述11个物种的37条Hsp90序列,它们在物种间分布情况和Hsp90亚家族的分布情况见表 1。其中属于Hsp90A亚家族的有20条,约占了所选物种亚家族的54.05%,在除百日咳博代氏杆菌和破伤风杆菌外的其余物种中都存在;Hsp90B亚家族的数量较少但分布较广泛,在致病疫霉、新型隐球菌、莱茵衣藻、高粱、水稻和埃及斑蚊中都有发现;Hsp90C亚家族存在于莱茵衣藻、高粱和水稻中;TRAP1亚家族存在于致病疫霉、四膜虫、水稻和埃及斑蚊四个物种中;HtpG亚家族只存在于百日咳博代氏杆菌和破伤风杆菌中,其他物种中没有发现。进一步验证了Chen[5]对于Hsp90亚家族在物种间的分布情况即Hsp90A亚家族分布于除细菌外的其他所有物种中,Hsp90B和TRAP1亚家族在物种间的分布情况没有规律,Hsp90C亚家族只存在于植物中,HtpG亚家族则绝大多数存在于细菌中。
| 表 1 Hsp90亚家族在11个物种间分布情况 Table 1 The distribution of Hsp90 family in 11 species |
根据所处理数据的类型,可以将系统发育树的构建方法分为两大类。一类是基于距离的构建方法,利用所有物种或分类单元间的进化距离,依据一定的原则及算法构建对应的系统发育树。其中包括非加权分组平均法(Unweighted-pair-group method with arithmetic mean,UPGMA)、最小进化法(Minimum evolution)和邻近归并法(Neighbor-joining method)等。其中UPGMA法已经较少使用,因大多数蛋白家族起源较早,序列分化程度较大,相互之间较为远源,因此构建此类系统发育树的时候使用邻法近归并法较多。
另一类方法是基于离散特征的构建方法,利用的是具有离散特征状态的数据,如基因序列中特定位点的核苷酸。此类方法中包括最大简约法(Maximum parsimony method),最大似然法(Maximum likelihood approach)等。
1.3 亚细胞定位的概述及软件介绍亚细胞定位是某种蛋白或者蛋白的产物在细胞内具体存在的位置,即根据所给出的蛋白质序列来预测蛋白质所在的亚细胞位置[8]。蛋白质亚细胞定位的准确预测使得我们可以更快的了解该蛋白质功能。为了准确的给出Hsp90的亚细胞定位信息,选取Cell-PLoc[9],SubLoc v1.0[10],PSORT II[11]和MultiLoc[12] 四个常用的蛋白质亚细胞定位预测软件,采用集成分类器方法对Hsp90进行亚细胞定位分析,即通过上述四种软件分别对Hsp90进行亚细胞定位预测,以预测结果中所出现的频率最高的亚细胞位置作为最终的亚细胞定位结果。
2 结果和分析 2.1 Hsp90基因结构分析根据HSPIR数据库中的基因信息在NCBI数据库中搜索本文所选的37条Hsp90的基因序列,搜索结果显示,在37条基因序列中,有8条序列无注释信息,使用GSDS2.0[13](https://gsds.cbi.pku.edu.cn/)软件绘制其余29条Hsp90基因序列的外显子-内含子结构图(见图 1)。发现在Hsp90的5个亚家族中,Hsp90C亚家族的基因结构最为复杂,内含子数目较其余4个亚家族的内含子数目要多,其中高粱基因组中的Hsp90C0251Sb和Hsp90C0252Sb基因的结构最为复杂,含有18个内含子。
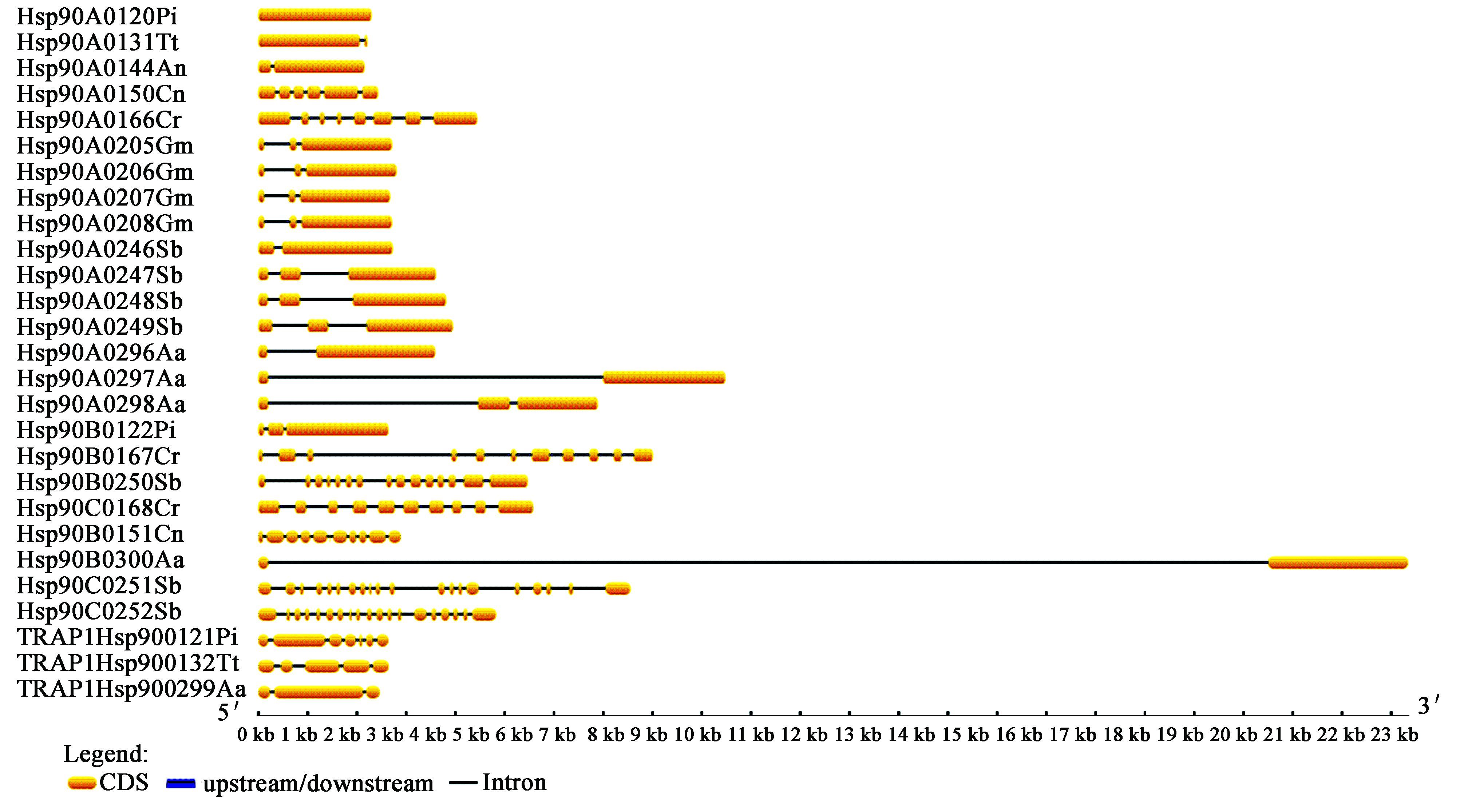
|
图 1 Hsp90家族的基因结构分析 Figure 1 The genetic structure analysis of Hsp90 family |
利用MEGA软件对所研究的11个物种的Hsp90家族的基因做CLUSTAL多序列比对,之后使用邻近归并法构建对应的系统发育树,设置自举检验(Bootstrap)的值为1 000,并对系统发育树中自举检验值小于50的树枝做归并处理,结果如图 2所示。从构建出的系统发育树可以看出,Hsp90家族形成了四个分支,分别对应亚家族Hsp90A,亚家族Hsp90C,和亚家族TRAP1,而Hsp90B和HtpG两个亚家族区分不明显,两者聚集到了同一分枝上,这可能与HtpG亚家族样本数量少有关。
2.3 Hsp90家族的亚细胞定位的结果分别使用Cell-PLoc、SubLoc v1.0、PSORT II和MultiLoc对Hsp90家族的蛋白质进行定位预测,综合四个分类器得到的结果最后确定每个蛋白质在亚细胞中的位置,如表 2所示。可以看出,Hsp90家族在细胞质、线粒体、内质网和细胞核等细胞器中都有发现。其中Hsp90A和HtpG两个亚家族在细胞质中存在的比例较高,Hsp90B亚家族集中于细胞质和内质网中,Hsp90C亚家族则集中于细胞质和线粒体中,TPAP1亚家族则更偏好定位于线粒体中。
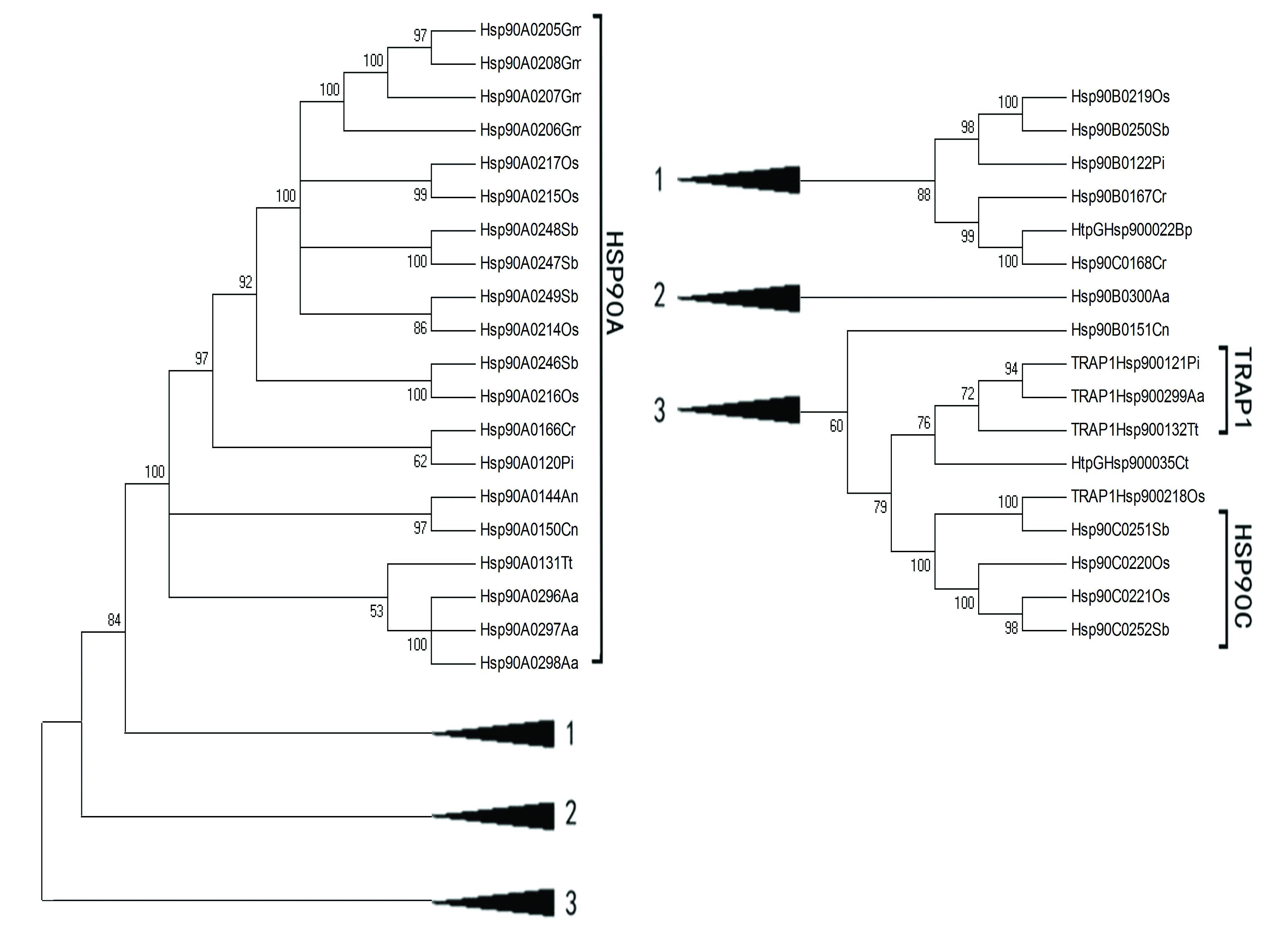
|
图 2 11个物种Hsp90家族的系统发育树 Figure 2 The Hsp90 family phylogenetic tree in 11 species 注:图中Bp代表百日咳博代氏杆菌、Ct代表破伤风杆菌、Pi代表致病疫霉、Tt代表四膜虫、An代表构巢曲霉、Cn代表新型隐球菌、Cr代表莱茵衣藻、Gm代表大豆、Sb代表高粱、Os代表水稻、Aa代表埃及斑蚊。 |
| 表 2 Hsp90家族亚细胞预测结果 Table 2 Hsp90 family subcellular prediction results |
Hsp90分布广泛,存在于各个物种间,本文通过对其不同亚家族在物种间分布进行统计,验证了其五个亚家族在物种间分布各有特点,其中Hsp90A亚家族数量最多,分布于除细菌外的其他所有物种中,Hsp90C亚家族只存在于植物中,而HtpG亚家族则绝大多数存在于细菌中。Hsp90家族的基因结构分析说明Hsp90C亚家族的结构最为复杂。构建的系统发育树进一步证明Hsp90在进化过程中具有保守性,不同物种的同一亚家族在系统发育树上聚集明显,即同一亚家族间的序列有较好的相似性。通过对Hsp90亚家族进行亚细胞定位分析,基本确定了Hsp90A,Hsp90B,Hsp90C,TRAP1和HtpG亚家族在细胞器中的位置。希望这些研究结果能够为Hsp90功能的研究提供新线索。
| [1] |
SAKATANI M, BONILLA L, DOBBS K B, et al. Changes in the transcriptome of morula-stage bovine embryos caused by heat shock: relationship to developmental acquisition of thermotolerance[J]. Reproductive Biology Endocrinology, 2013(11): 3.
DOI:10.1186/1477-7827-11-3 ( 0) 0)
|
| [2] |
RATHEESH K R, NAGARAJAN N S, ARUNRAJ S P, et al. HspIR: a manually annotated heat shock protein information resource[J]. Bioinformatics, 2012, 28(21): 2853–2855.
DOI:10.1093/bioinformatics/bts520 ( 0) 0)
|
| [3] |
RITOSSA F. A new puffing pattern induced by temperature shock and DNP in drosophila[J]. Experientia, 1962, 18(12): 571–573.
DOI:10.1007/BF02172188 ( 0) 0)
|
| [4] |
CSERMELY P, SCHNAIDER T, SÖTI C, et al. The 90-kDa molecular chaperone family: structure, function, and clinical applications[J]. Pharmacology Therapeutics, 1998, 79(2): 129–168.
DOI:10.1016/S0163-7258(98)00013-8 ( 0) 0)
|
| [5] |
CHEN B, ZHONG D, MONTEIRO A. Comparative genomics and evolution of the Hsp90 family of genes across all kingdoms of organisms[J]. BMC Genomics, 2006(7): 156.
DOI:10.1186/1471-2164-7-156 ( 0) 0)
|
| [6] |
ALI A B S, OCARROL R, OVSENEK N. Hsp90 interacts with and regulates the activity of heat shock factor 1 in Xenopus oocytes[J]. Molecular Biology, 1998, 18(9): 4949–4960.
( 0) 0)
|
| [7] |
宋晓敏.分泌型热休克蛋白90a在肿瘤发生和转移中的作用机理[D].北京:清华大学,2010.
SONG Xiaomin.Secretory type heat shock protein 90a mechanism in tumorigenesis and metastasis [D].Beijing:Tsinghua University,2010.
http://epub.cnki.net/kns/detail/detail.aspx?QueryID=1&CurRec=1&recid=&FileName=1011280316.nh&DbName=CDFD0911&DbCode=CDFD&pr= ( 0) 0)
|
| [8] |
吴泽月, 陈月辉. 蛋白质亚细胞定位预测研究进展[J]. 山东师范大学学报, 2012, 4(27): 33–37.
WU Zeyue, CHEN Yuehui. The research progress of protein subcellular localization prediction[J]. Journal of Shandong Normal University, 2012, 4(27): 33–37.
( 0) 0)
|
| [9] |
CHOU K C, SHEN H B. Cell-PLoc: a package of Web servers for predicting subcellular localization of proteins in various organisms[J]. Nature Protocols, 2008(153): 162.
DOI:10.1038/nprot.2007.494 ( 0) 0)
|
| [10] |
HUA S, SUN Z. Support vector machine approach for protein subcellular localization prediction[J]. Bioinformatics, 2001, 17(8): 721–728.
DOI:10.1093/bioinformatics/17.8.721 ( 0) 0)
|
| [11] |
MITSUTERU C N, KENTA N. Improvement of PSORT II Protein Sorting Prediction for Mammalian Proteins[J]. Genome Informatics, 2002(13): 441–442.
( 0) 0)
|
| [12] |
HÖGLUNDA, DÖNNES P, BLUM T, et al. MultiLoc: prediction of protein subcellular localization using N-terminal targeting sequences, sequence motifs and amino acid composition[J]. Bioinformatics, 2006, 22(10): 1158–1165.
DOI:10.1093/bioinformatics/btl002 ( 0) 0)
|
| [13] |
BO H, JINPU J, AN-YUAN G, et al. GSDS 2.0: an upgraded gene feature visualization server[J]. Bioinformatics, 2015, 31(8): 1296–1297.
DOI:10.1093/bioinformatics/btu817 ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


