2. 国家民委-教育部重点实验室(西南民族大学生命科学与技术学院), 成都 610041
2. Key Laboratory of Animal Genetics and Breeding of State Ethnic Affairs Commission & Ministry of Education (College of Life Science and Technology, Southwest University for Nationalities), Chengdu 610041, China
鮰爱德华菌(Edwardsiella ictaluri)属肠杆菌科(Entero bacteriaceae)爱德华菌属(Edwardsiella)的一种革兰氏阴性短杆菌,该菌主要感染鲶形目鱼类[1]。已有报道该菌曾引发黄颡鱼的红头病、斑点叉尾鮰肠型败血症(Enteric septicemia of catfish,ESC)[2-3],在其它非鲶形目鱼类也可以被感染,如在虹鳟鱼、鳖也见报道[4-5]。鮰爱德华氏菌病具有发病急、发病率和死亡率高的特点,随着养殖规模和养殖密度的加大,该菌对淡水鱼类的养殖危害日益严重,但目前控制该菌引起的相关疾病主要以抗生素为主,存在细菌耐药性增强、水体药物污染、引发食品安全等问题。因此,研发相关中草药制剂和高效疫苗越发重要。
外膜蛋白(Outer membrane proteins, OMPs)是革兰氏阴性细菌外膜的主要结构成分,是外膜A蛋白、微孔蛋白、脂蛋白和微量蛋白等蛋白的总称[6],OMPs除作为细菌的结构组成之外,对细胞物质的运输和有关物质的合成和致病力也有重要作用。研究表明外膜蛋白具有良好的免疫原性,不仅可诱导体内产生较强的细胞和体液免疫,而且不同血清型分离菌株具有交叉保护作用,因而成为疫苗研发的热点。随着基因组学、蛋白质组学、生物信息学等相关学科的发展,越来越多病原菌的外膜蛋白的基因序列得以测出,这为从病原菌中扩增目标基因,进行重组表达,快速筛选疫苗抗原提供了可能。目前,已有学者对OmpK、 OmpU、OmpS2、OmpW、OmpC等外膜蛋白的结构和免疫原性进行了研究[7-11]。但是OmpLC(Outer membrane porin protein LC)作为一种特定外膜蛋白,在国内外鲜见报道,其主要特性和功能尚不是很清楚。本文以鮰爱德华菌OmpLC基因序列为基础,对该蛋白进行一系列生物信息学分析,以期为今后进一步研究该蛋白的功能及鮰爱德华菌相关发病机制以及疫苗的研制提供理论依据。
1 材料与方法 1.1 实验材料 1.1.1 菌种鮰爱德华菌,由西南民族大学生命科学与技术学院水产养殖实验室从患病黄颡鱼肠道采样,经回感实验确认为此次黄颡鱼患病的主要病原菌。鮰爱德华菌经分子鉴定后,保存-20℃冰箱中备用。
1.1.2 主要试剂和仪器营养琼脂(Nutrient Agar;北京奥博星生物技术有限责任公司)。TaqMix、DNA Marker DL2000购自TIANGEN BIOTECH(BEIJING)CO., LTD.。Yeast Extract(OXOID)、 Tryptone(OXOID),PCR仪(Eppendorf Germany),电泳仪(BIORAD)凝胶成像系统(BioRAD Laboratories Segrate Italy),超低温冰箱,离心机(Eppendorf),恒温振荡培养箱(HZQ-F100),恒温培养箱(上海齐欣科学仪器科技有限公司)。高压灭菌锅(SANYO Electric CO., LTD Japan)。
1.2 OmpLC基因序列扩增参考GENBANK已有的OmpLC基因CDS序列,利用软件Primer Premier 5设计上游引物(ompLCF): 5'CCCGATGGAATCAAAT3' 、下游引物(ompLCR): 5'CCTGCCTG ATCGG A TA 3',委托上海生工生物工程有限公司合成。以反复低温和煮沸法粗提细菌总DNA模板。
以提取的鮰爱德华菌总DNA为模板,用引物OmpLCR、OmpLCF,对OmpLC基因进行PCR扩增。PCR反应体系:上下游引物各1 μL(引物浓度均为10 μmol/L),DNA模板3 μL,ddH2O 20 μL,Mix 25 μL,总体系为50 μL。PCR反应条件:94 ℃预变性4 min;94 ℃、30 s,51.5 ℃、30 s,72 ℃、1 min,30个循环;72 ℃延伸4 min。PCR扩增产物用1%琼脂糖凝胶电泳检测,将阳性样品胶回收后送交上海上海生工生物工程有限公司进行测序。
1.3 OmpLC基因生物信息学分析 1.3.1 核酸序列分析测序完成后获得上下序列,利用DNAstar软件包的SeqMan程序进行序列拼接,并根据测序峰图进行调整。获得完整序列后利用NCBI网站提供的Blastn和Blastx程序进行序列比对,查找相似序列,初步确定其功能。用BioEdit软件对基因序列进行ORF查找,确定CDS区,并将CDS区翻译成氨基酸序列。用CodonW1.4对编码区进行密码子偏好性分析。最后将该序列提交至GenBank。
1.3.2 蛋白质序列分析用ProtParam分析蛋白的一级结构和及其理化参数(分子量、等电点、吸光系数等);用ProtScal和DNAstar软件包Protean程序预测其亲疏水性;用TMHMM Server 2.0进行跨膜区分析;用SignalP 4.0 Server软件进行信号肽预测;用PredictProtein预测蛋白质二级结构;用InterProScan、MotifScan、CDD预测其结构功能域。用Phyre2进行三级结构预测;用DNAstar软件包中的Megalign进行分子进化分析。所用网络服务资源见表 1。
| 表 1 生物信息学分析在线网络资源 Table 1 Online database and software of bioinformatic analysis |
PCR产物经琼脂糖凝胶电泳后,结果见图 1。经测序分析OmpLC基因序列片段大小为1 133 bp,序列提交至GenBank,收录入号为JX064520。鮰爱德华菌(Edwardsiella ictaluri)OmpLC基因的cDNA从32到1 111全长1 080 bp,编码360个氨基酸。
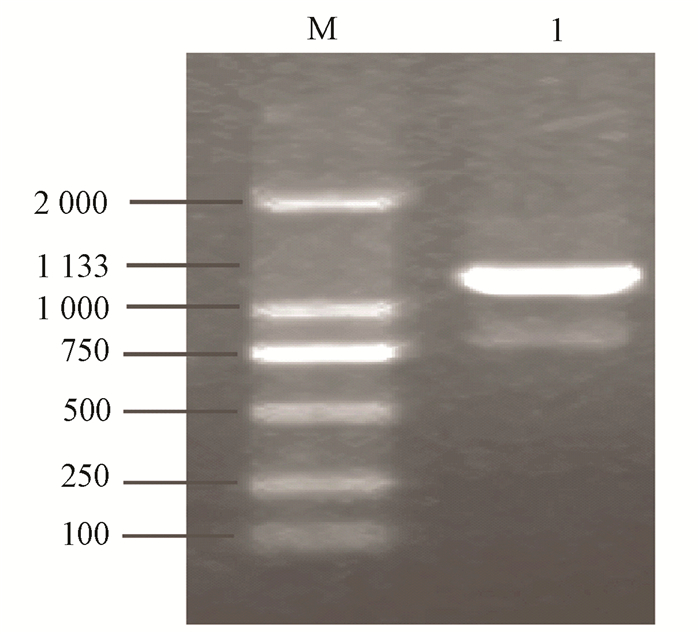
|
图 1 OmpLC PCR扩增结果 Figure 1 The PCR amplification results of OmpLC 注:M: DNA Marker 2 000 bp;1:OmpLC基因PCR扩增产物。 Notes:M:DNA Marker 2 000 bp; 1:The PCR amplification product. |
不同物种不同基因对于密码子的使用具有明显的偏好性,密码子使用的偏性与基因的时空表达、表达产物的结构及功能有密切关系。对鮰爱德华菌OmpLC基因进行偏好性分析表明:编码Phe的密码子UUC、编码Leu的密码子CUG、编码Ile的密码子AUC等20种密码子为该基因的偏好性密码子(见图 2)。
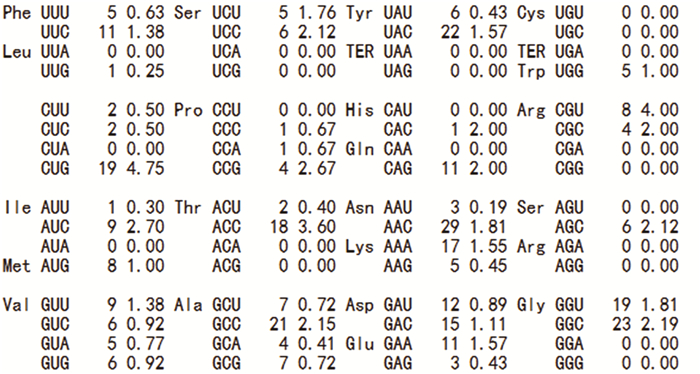
|
图 2 密码子偏好性分析 Figure 2 Analysis of codon preferences |
蛋白质的基本理化性质包括其相对分子量、氨基酸组成、等电点等。用ProtParam tool软件预测鮰爱德华菌OmpLC蛋白的理化性质。OmpLC蛋白分子量为中不含Cys,Gly、Ala、Asn的含量较多,分别为11.67%、10.83%和8.89%。相对含量较少的氨基酸为His(0.28%)(结果见表 2)。推测OmpLC蛋白分子量为39.407 kDa,理论等电点PI为4.98,带负电荷的氨基酸残基(Asp+Glu)总数为41,带正电荷的氨基酸残基(Arg+Lys)总数为34,不稳定系数为18.26。根据不稳定参数的数值低于40才是稳定的蛋白[12],鮰爱德华菌OmpLC蛋白是一种稳定性蛋白,软件预测该蛋白在体外环境下的半衰期为30 h。
| 表 2 氨基酸基本组成 Table 2 Basic composition of amino acids of OmpLC |
运用Protean程序Kyte-Doolittle算法对OmpLC编码产物进行亲水性/疏水性预测疏水性图谱:正值越大说明越疏水,负值越大说明越亲水;亲水性图谱反之。由亲疏水性图谱可知在前3-22氨基酸残基区域有较强的疏水性,参考ProtScal的分析结果,对于疏水性第10位的Val具有最大值为3.000,第51位的Asp具有最小值为-2.867,平均疏水性值为-0.449,整个蛋白表现为亲水性,由此预测该蛋白是一种亲水性蛋白(见图 3)。
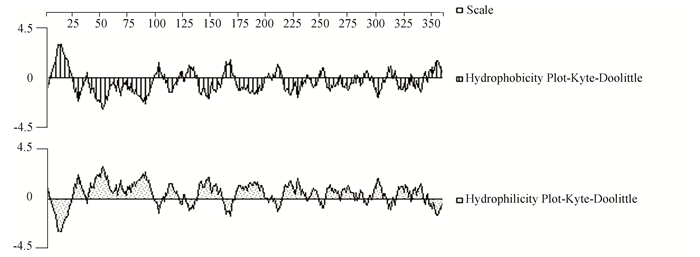
|
图 3 OmpLC编码产物亲疏水性图谱 Figure 3 Hydrophobicity analysis of OmpLC protein |
TMHMM,是一种基于隐马尔可夫模型的跨膜螺旋预测算法。利用TMHMM server 2.0在线软件对OmpLC编码产物的跨膜区域分析(见图 4),结果显示,1-6区域位于膜内,7-26区域预存在一个跨膜螺旋,27-360区域则位于膜外,N端在细胞膜内预测的概率为0.83。这与上述疏水性分析相吻合。
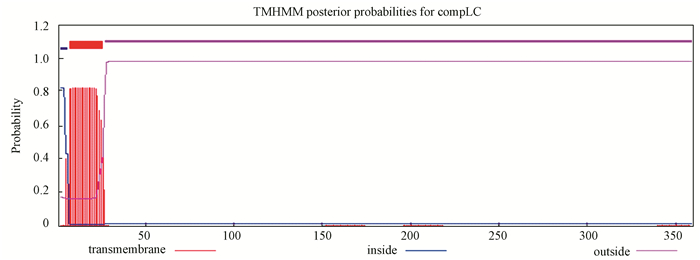
|
图 4 OmpLC编码产物跨膜区预测* Figure 4 Transmembrane region prediction of OmpLC protein *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第2期doi:10.3969/j.issn.1672-5565.2016.02.01)。 |
信号肽是分泌蛋白质前体N端15-30个氨基酸组成的多肽,引导新生蛋白的定位。本文采用的SignalP4.0是个强大的信号肽及其剪切位点检测工具,该算法基于神经网络算法,利用已知信号序列的革兰氏阴性原核生物、革兰氏阳性原核生物及真核生物的序列作为训练集,对分泌型蛋白的信号肽进行预测,并且给出C、S、Y-score计算结果[13]。其中C值是信号肽酶切位点分值,S值是信号肽分值,Y值是由C值和S值综合得出的剪切位点分值,用于更精确的确定信号肽酶切位点[12]。一个典型的信号肽,其S值在剪切位点之前高在剪切位点之后变低,Y值和C值在剪切位点达到最大值并趋近于1。本实验预测结果(见图 5)表明:C值和Y值在第23位分别获得最大值0.884、0.898,S值在第3位最大为0.949,1-22位平均S值为0.923,D值是S-mean和Y-max的平均值,为0.910,从而可以判断OmpLC编码产物存在信号肽,剪切位点位于22~23。
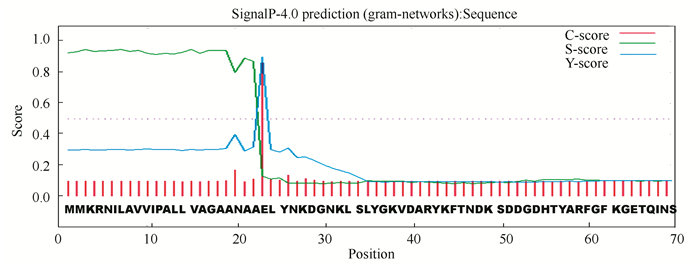
|
图 5 OmpLC编码产物信号肽预测* Figure 5 Signal peptide prediction of OmpLC protein *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第2期doi:10.3969/j.issn.1672-5565.2016.02.01)。 |
PredictProtein是欧洲分子生物学实验室提供的蛋白质序列和结构预测服务,其分析方法包括:用PROFsec分析序列的二级结构,用PROFacc分析残基溶剂可及性,PHDhtm分析潜在的跨膜拓扑结构,通过PROSITE搜索模体,借助ProDom预测功能结构域。发现该编码产物二级结构中螺旋占6.67%,折叠占45.28%,无规则卷曲占48.06%,其预测图如图 6所示,可以看出,该产物二级结构主要由折叠和环构成。
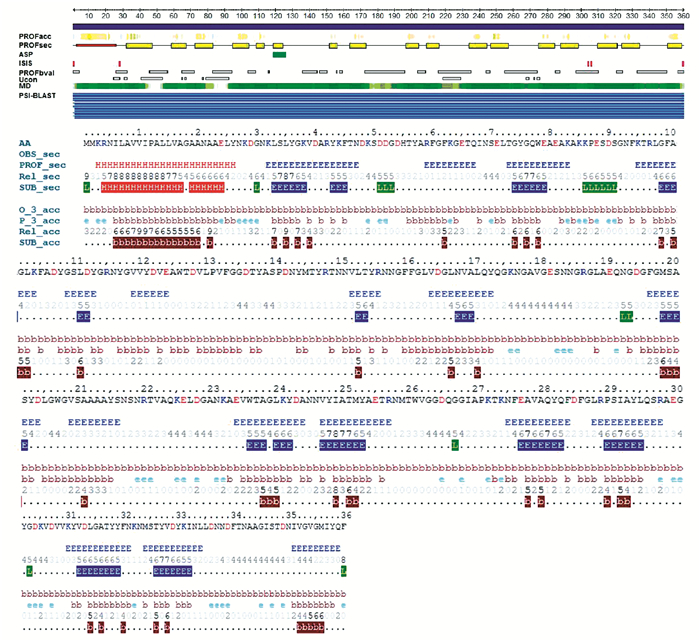
|
图 6 OmpLC编码产物二级结构预测* Figure 6 Secondary structure prediction of the OmpLC protein *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第2期doi:10.3969/j.issn.1672-5565.2016.02.01)。 |
InterPro数据库由EBI开发,整合蛋白质家族、结构域和功能位点等资源。整合UniProt、PROSITE、Pfam等12个成员数据库,检索结果准确。用CDD、InterProScan、MotifScan对OmpLC的编码产物的基序及结构域进行分析,结果均指向cl01155(OM_channels superfamily)的成员gram_neg_porins[cd00342](见图 7a,7b)。OmpLC编码蛋白由360个氨基酸组成,其中28-360个氨基酸残基为保守结构域。该结构域由多个基序(53-64、96-106、153-164、233-245、312-348等)组成(见图 7c),其中最典型的模体是第312-348位的表达式为[LIVMFY].{2}G.{2}Y.F.K.{2}[SN] [STAV][LIVMFYW]V,目标蛋白的模体为VDLGAT YYFNKNMSTY V,该家族蛋白在主要存在于细菌中,在病毒和和真核生物偶有出现(见图 7d)。

|
图 7 OmpLC编码产物结构域预测结果* Figure 7 Functional domain of OmpLC protein *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第2期doi:10.3969/j.issn.1672-5565.2016.02.01)。 |
CPHmodels是采用同源建模来预测蛋白质三级结构的一个网络服务器,同时也采用了以预测距离为基础的窜线(threading)算法。同源建模是在蛋白质结构数据库中搜索目标蛋白的同源蛋白,然后将目标蛋白和同源蛋白进行对比,根据模板结构建模,并进一步优化模型作为预测的结果[14]。对于远源同源蛋白,结构虽然有一定的相似性,但其一致性小于30%,此时很难确定序列和结构之间的关系,此时选择线串法进行蛋白结构预测效果较好。该算法是将待测序列“穿”入已知蛋白的基本骨架内,通过计算各种折叠的概率,来预测蛋白的核心结构。Phyre是一种基于profile-profile比对和线串法,对预测蛋白和模板进行1D-3D的序列谱的比对预测。因此本文采用这两种方法进行预测,CPHmodels结果显示与1osm_A的相似性达64.5 %,其3D结构图如图 8a所示。Phye2结果显示,同源蛋白为d1osma,该蛋白属于porins超家族。目标蛋白中有335个氨基酸残基(93%)以100%置信率参与建模(见图 8b)。将二者所得建模参数文件导入Swiss-Pdb V iewer 3.7,将其结构进行叠加比对(见图 8c),分析结构类似性,并利用Ramachandran参数进行检验。结果如图 7所示。黄色区域是理想的Φ角和Ψ角分布区域,而蓝色区域为不合理区域。从图 8d中可以看出,模拟得到的OmpLC蛋白的残基的二面角绝大部分在合理区域内,表明该预测结构与自然结构趋势相同,具有稳定的空间结构。
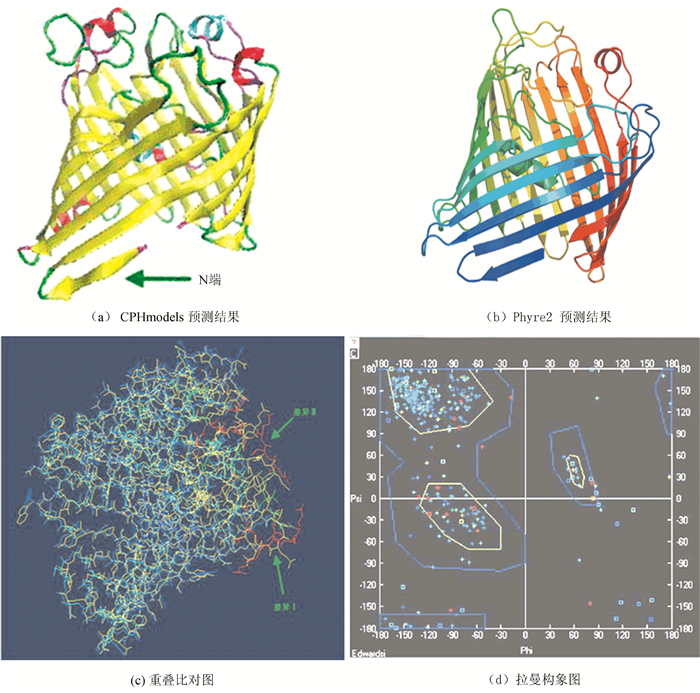
|
图 8 两种建模方法所得3D图及和重叠比较分析图* Figure 8 The three-dimensional(3D) structure model and graphic superimposition analysis of the model predicted by two modeling methods.The predicted 3D structure of OmpLC protein *彩图见电子版(http://swxxx.alljournals.cn/ch/login.aspx)(2016年第2期doi:10.3969/j.issn.1672-5565.2016.02.01)。 |
本文选用Blast p程序,利用特异性位点迭代比对算法(Position-Specific Iterated BLAST,PSI-Blast),搜索非冗余数据库(GenBank CDS translations+PDB+SwissProt+PIR+PRF),进行同源蛋白搜索。遗传距离分析结果显示,目标蛋白序列MYOMP(已用下划线标记)与OmpLC蛋白序列AEQ59632、和AEQ59639序列相似性分别达到100%和99.7%(见图 9a),且从系统进化树上可以看出MYOMP和二者聚为一类(见图 9b),因此可以判断目标蛋白为OmpLC。12个序列可以明显分为三类,首字母为m的序列1941972、378986906、EHJ79723.1属于类麦芽孔道蛋白类(Maltoporin-like),首字母为l/L的序列331641081、AAB40783.1和1FEP为配体门控通道蛋白类(ligand_gated_channel),其它的则属于革兰氏阴性孔道蛋白(gram_neg_porins)。该目标蛋白为革兰氏阴性孔道蛋白。
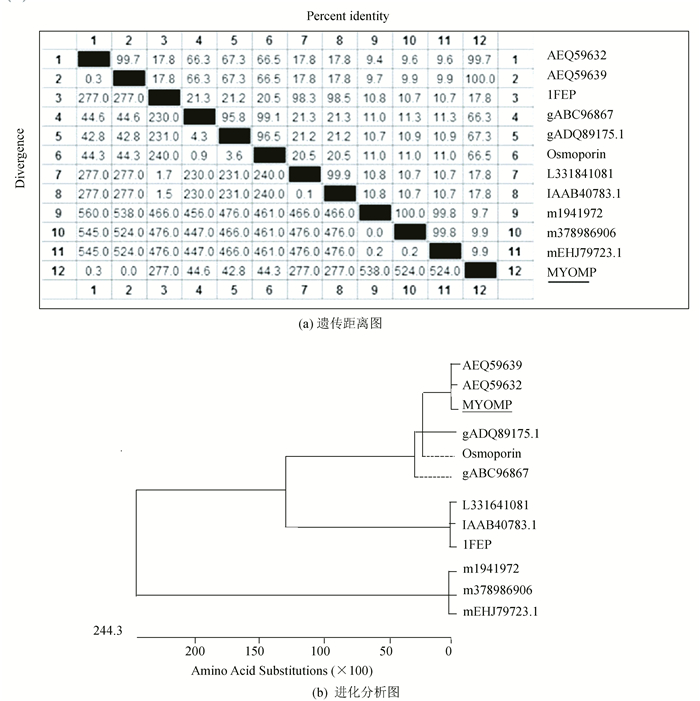
|
图 9 OmpLC蛋白序列系统进化分析 Figure 9 The phylogenetic analysis of OmpLC protein |
生物信息学是一门建立在生物科学、数学、计算机科学基础之上的交叉科学。随着生物技术的快速发和多个物种的基因组计划的相继完成,生物信息学在基因的功能发现、疾病基因诊断、蛋白质结构预测、分子药物设计等方面发挥着重要作用。目前有很多生物信息学分析软件和数据库可以从网上免费下载,有些机构还有专门的在线服务系统。生物信息学借助这些广泛的信息资源及计算机辅助软件,能够对核酸、蛋白质等序列进行快速、高效、全面的分析,可以节约实验成本、缩短研究周期、避免试验的盲目性。OmpLC基因能够编码一种特定的外膜孔蛋白,有研究表明OmpLC能够作为噬菌体ΦeiAU和ΦeiDWF的吸附受体[15],而关于其它相关功能尚还未见报道,因此选取鮰爱德华氏菌OmpLC作为目标蛋白,利用生物信息学方法分析其结构和功能。
鮰爱德华氏菌OmpLC蛋白富含Gly和Ala,且成熟蛋白N端的第一个氨基酸是Ala,它和Gly都属于稳定型残基,且不稳定系数为18.26( < 40),为一种稳定蛋白。跨膜区分析结果说明在第7-26个氨基酸残基区域存在一个跨膜螺旋,这和二级结构预测的结果较一致,但因为其1-22区域为信号肽区域,因此成熟蛋白中应该无明显跨膜螺旋。此结果与朱珊丽等[16]研究的主要外膜蛋白(MOMP)结构有所不同。这可能和外膜蛋白种类及不同菌株的特异性有关。二级结构和三级结构表明该蛋白主要由16个β-折叠和一些无规则卷曲组成,三维结构呈桶状。通过同源建模和线串法两种建模方法,得到结果较一致且理论可靠的OmpLC蛋白模型。该模型同已报道ompC、ompF的结构相似[11, 17-18]。有研究表明,ompC和ompF基因的存在对于细菌的抗酸性和相关抗生素的扩散具有重要作用[17-18],而鮰爱德华氏菌OmpLC和ompC及ompF具有相似的结构,但其是否也具有相应的功能尚不清楚,且OmpLC和ompC及ompF之间的进化关系如何亦有待于进一步研究。
革兰氏阴性孔蛋白在细菌生命过程中主要具有分子筛特性,对亲水性复合物进行过滤[19]。某些选择性孔蛋白(Porins)还具有溶质结合位点,允许特定的溶质跨膜。作为外膜蛋白的重要组成成分,孔蛋白(Porins)还可以作为噬菌体和细菌素的受体[20]。鮰爱德华菌OmpLC蛋白的结构域和同源性分析结果显示:鮰爱德华菌OmpLC蛋白属于外膜蛋白通道超家族(OM_channels superfamily)的革兰氏阴性孔蛋白(gram_neg_porins)成员,因此,鮰爱德华菌OmpLC蛋白未来可作为噬菌体ΦeiAU和ΦeiDWF的吸附受体,而OmpLC基因的表达水平高低也可成为噬菌体感染敏感性重要分子因素[20]。
由于细菌外膜蛋白(OMPs)的种类多样及其在生物体中位置的特异性,其纯化较为困难,因而限制了对其结构和功能的研究。本文对鮰爱德华氏菌的外膜蛋白——OmpLC蛋白的序列进行一系列生物信息学分析,为进一步研究鮰爱德华氏菌致病的相关发病机制、鮰爱德华氏菌OmpLC蛋白的功能、表达载体的构建、分子进化、分子定向改造、免疫原性研究和疫苗研发奠定了基础。
| [1] |
BRENNER D J, KRIEG N R, STALEY J T. Bergey's manual of systematic bacteriology. London: Springer, 2005 .
( 0) 0)
|
| [2] |
YE S G, LI H, QIAO G, et al. First case of Edwardsiella ictaluri infection in China farmed yellow catfish Pelteobagrus fulvidraco[J]. Aquaculture, 2009, 292(1-2): 6–10.
DOI:10.1016/j.aquaculture.2009.03.036 ( 0) 0)
|
| [3] |
梁万文, 陈明, 余晓丽, 等. 斑点叉尾鮰肠败血症病原菌的分离与鉴定[J]. 西南农业学报, 2007, 20(5): 1124–1129.
LIANG Wanfang, CHEN Ming, YU Xiaoli, et al. Isolation and identification of causative pathogen for enteric septicemia of catfish (ESC)[J]. South West China Journal of Agricultural Sciences, 2007, 20(5): 1124–1129.
( 0) 0)
|
| [4] |
KESKIN O, SECER S, IZGV R M, et al. Edwardsiella ictaluri infection in rainbow trout (Oncorhynchus mykiss)[J]. Turkish Journal of Veterinary and Animal Sciences, 2004, 28(4): 649–653.
( 0) 0)
|
| [5] |
肖克宇, 江为民, 舒新华, 等. 爱德华氏菌变异株C9605及对鳖的致病性研究[J]. 微生物学通报, 1998, 25(5): 262–265.
XIAO Keyu, JIANG Weimin, SHU Xinhua, et al. Studies on Edwardsiella ictaluri vatiation strain(C9605) and its pathogenicity to soft-shelled turtles[J]. Microbiology China, 1998, 25(5): 262–265.
( 0) 0)
|
| [6] |
熊静, 关瑞章, 郭松林, 等. 鱼类病原菌外膜蛋白及其免疫原性研究进展[J]. 水生生物学报, 2011, 35(1): 163–169.
XIONG Jing, GUAN Ruizhang, GUO Songlin, et al. A Review on the immunogenicity of fish pathogenic bacterial outer membrane proteins[J]. Acta Hydrobiologica Sinica, 2011, 35(1): 163–169.
DOI:10.3724/SP.J.1035.2011.00163 ( 0) 0)
|
| [7] |
LI Ningqiu, BAI Junjie, WU Shuqin, et al. An outer membrane protein, OmpK, is an effective vaccine candidate for Vibrio harveyi in Orange-spotted grouper (Epinephelus coioides)[J]. Fish & Shellfish Immunology, 2008, 25(6): 829–833.
( 0) 0)
|
| [8] |
黄辉, 毛芝娟, 陈吉刚. 哈维氏弧菌外膜蛋白OmpU的克隆、表达与免疫原性研究[J]. 华中农业大学学报, 2010, 29(3): 346–350.
HUANG Hui, MAO Zhijuan, CHEN Jigang. Cloning, expression and imImunogenicity analysis of OmpU of Vibrio harveyi ZJ 2008[J]. Joumal of Huazhong Agricultural University, 2010, 29(3): 346–350.
( 0) 0)
|
| [9] |
张亚宁, 李晓, 耿晓娜, 等. 迟缓爱德华菌HB01外膜蛋白OmpS2基因的克隆表达及其免疫原性研究[J]. 细胞与分子免疫学杂志, 2011, 27(10): 1075–1082.
ZHANG Yaning, LI Xiao, GENG Xiaona, et al. Cloning and expression of a outer membrance protein gene (OmpS2) of Edwardsiella tarda HB 01 and its immunogenicity[J]. China Journal of Cell Molecular Immunology, 2011, 27(10): 1075–1082.
( 0) 0)
|
| [10] |
刘明智, 叶星, 田园园, 等. 嗜水气单胞菌外膜蛋白W基因的表达及其免疫原性分析[J]. 微生物学通报, 2011, 38(3): 437–445.
LIU Mingzhi, YE Xing, TIAN Yuanyuan, et al. Expression and immunogenicity analysis of the outer membrane protein W gene of Aeromonas hydrophila[J]. Microbiology China, 2011, 38(3): 437–445.
( 0) 0)
|
| [11] |
LOU H B, CHEN M, BLACK S S, et al. Altered antibiotic transport in OmpC mutants isolated from a series of clinical strains of multi-drug resistant E. coli[J]. PLoS One, 2011, 6(10): 1–15.
( 0) 0)
|
| [12] |
熊伟, 杨勇琴, 张海洋, 等. 人线粒体转录终止因子1 (hMTERF 1)蛋白的生物信息学分析[J]. 生物信息学, 2015, 13(1): 23–30.
XIONG Wei, YANG Yongqin, ZHANG Haiyang, et al. Bioinformatic analysis of human mitochondrial transcription termination factor 1 (hMTERF 1)[J]. Chinese Journal of Bioinformatics, 2015, 13(1): 23–30.
( 0) 0)
|
| [13] |
PETERSEN T N, BRUNAK S, VON H G, et al. SignalP 4.0: discriminating signal peptides from transmembrane regions[J]. Nature Methods, 2011, 8(10): 785–786.
DOI:10.1038/nmeth.1701 ( 0) 0)
|
| [14] |
NIELSEN M, LUNDEGAARD C, LUND O, et al. CPHmodels-3.0-Remote homology modeling using structure-guided sequence profiles[J]. Nucleic Acids Research, 2010, 38(20103811): 576–581.
( 0) 0)
|
| [15] |
HOSSAIN M J, RAHMAN KH S, TERHUNE J S, et al. An outer membrane porin protein modulates phage susceptibility in Edwardsiella ictaluri[J]. Microbiology, 2012, 158(2): 474–487.
DOI:10.1099/mic.0.054866-0 ( 0) 0)
|
| [16] |
朱珊丽, 尤孙武, 娄崇洁, 等. 沙眼衣原体主要外膜蛋白生物信息学分析[J]. 温州医学院学报, 2009, 39(1): 5–7.
ZHU Shanli, YOU Sunwu, LOU Chongjun, et al. Bioinformatics analysis of the major outer membrane protein of Chlamydia trachomatis serovar E[J]. Journal of Wenzhou Medical College, 2009, 39(1): 5–7.
( 0) 0)
|
| [17] |
KUMAR A, HAJJAR E, RUGGERONE P, et al. Structural and dynamical properties of the porins OmpF and OmpC: insights from molecular simulations[J]. Journal of Physics: Condensed Matter, 2010, 22(45): 454125.
DOI:10.1088/0953-8984/22/45/454125 ( 0) 0)
|
| [18] |
BEKHIT A, FUKAMACHI T, SAITO H, et al. The role of OmpC and OmpF in acidic resistance in Escherichia coli[J]. Biological and Pharmaceutical Bulletin, 2011, 34(3): 330–334.
DOI:10.1248/bpb.34.330 ( 0) 0)
|
| [19] |
BENZ R, BAUER K. Permeation of hydrophilic molecules through the outer membrane of gram-negative bacteria.Review on bacterial porins[J]. European Journal of Biochemistry, 1988, 176(1): 1–19.
DOI:10.1111/ejb.1988.176.issue-1 ( 0) 0)
|
| [20] |
JAPAL B K, WALIANAL P J. Biophysics of the structure and function of porins[J]. Quarterly Reviews of Biophysics, 1990, 23(4): 367–403.
DOI:10.1017/S003358350000559X ( 0) 0)
|
 2016, Vol. 14
2016, Vol. 14


