摘要
基于加权基因共表达网络分析(Weighted gene co-expression network analysis,WGCNA)和基因富集分析(Gene set enrichment analysis,GSEA)筛选肝细胞癌的潜在生物标志物和治疗靶点。利用GEO数据库中的GEO2R在线工具对肝细胞癌和癌旁组织的基因进行差异分析,并对差异表达基因进行GO功能和KEGG通路富集分析。利用WGCNA与STRING两种算法提取关键核心(Hub)基因。然后通过GEPIA数据库中的转录数据对Hub基因的表达进行验证。Kaplan-Meier法分析各Hub基因的预后价值。此外,还使用 TIMER数据库和Spearman分析方法计算Hub基因表达与免疫细胞浸润的相关性。利用实时荧光定量RT-PCR检测6例肝细胞癌和癌旁组织中Hub基因的相对表达量。GSEA分析结果包括系统性红斑狼疮和补体和凝血级联过程。GO功能分析主要涉及淋巴细胞免疫、补体激活反应、体液免疫应答等。KEGG主要集中于补体激活、Th17细胞分化,Th1和Th2细胞分化等信号通路。研究发现C8a,C6与Mbl2基因的转录水平明显低于健康对照组,基因的表达与肝细胞癌患者的临床预后有关,同时与免疫细胞的激活也存在相关性。PCR结果显示,肝细胞癌中C8a,C6和Mbl2基因的mRNA表达水平显著低于正常组织(P<0.05)。核心基因C6,C8a和Mbl2有望成为潜在的肝细胞癌诊断生物标志物,参与补体通路,与影响肿瘤细胞免疫浸润相关,可作为临床诊断和治疗潜在治疗靶点。
Abstract
The study employs Weighted Gene Co-expression Network Analysis (WGCNA) and Gene Set Enrichment Analysis (GSEA) to identify potential biomarkers and therapeutic targets for hepatocellular carcinoma.GEO2R in the GEO database is utilized for the analysis of the differentially expressed genes in hepatocellular carcinoma and paracancer tissue, and the enrichment of DEGs in Gene Ontology (GO) function and Kyoto Encyclopedia of Genes and Genomes (KEGG) pathways is assessed. Hub genes are extracted by WGCNA and STRING. Transcriptional data of Hub genes are verified by gene expression profiling interactive analysis (GEPIA). The prognostic significance of each Hub gene is assessed by the Kaplan-Meier method. Furthermore, the correlation between Hub gene expression and immune cell infiltration is determined through the utilization of the TIMER database and Spearman analysis. The relative expression of Hub genes in six cases of hepatocellular carcinoma and adjacent tissues is measured using real-time fluorescence quantitative RT-PCR.The results of the GSEA analysis reveal the involvement of systemic lupus erythematosus and complement and coagulation cascade processes. The functional analysis of the GO primarily encompass lymphocyte immunity, complement activation response, humoral immune response, and other related aspects. KEGG primarily concentrats on complement activation, Th17, Th1 and Th2 cell differentiation, and various signaling pathways. In this study, we observe a significant decrease in the transcription levels of C8a, C6, and Mbl2 genes in comparison to healthy controls. Furthermore, we find a correlation between the expression of Hub genes and the clinical prognosis of patients with hepatocellular carcinoma, as well as their association with immune cell activation. The findings from the polymerase chain reaction (PCR) analysis indicat a significant decrease in the mRNA expression levels of the C8a, C6, and Mbl2 genes in hepatocellular carcinoma compared to normal tissues (P<0.05). These core genes, namely C6, C8a, and Mbl2, hold promise as potential diagnostic biomarkers for hepatocellular carcinoma. Additionally, they are involved in the complement pathway and have implications in tumor cell immune invasion, making them potential targets for clinical diagnosis and therapeutic interventions.
Keywords
肝细胞癌是最致命和最常见的恶性肿瘤之一,发病率全球排名第六,死亡率排名第二[1-2]。依据GLOBOCAN 2020数据库报道,新增肝癌病例为90.567 7万例,死亡病例约83.018万例[3],肝细胞癌已经成为严重影响人类健康的重要疾病之一。肝细胞癌的发病缓慢又复杂,肝硬化、自身免疫性肝炎、酒精性肝病等是主要的发病诱因,这些因素可能通过影响信号通路的调节、细胞生长凋亡、免疫调节等方面促进肿瘤的发生[4]。现阶段临床对于肝细胞癌的早期治疗主要包括免疫疗法、手术切除法、经动脉化疗栓塞肝移植、靶向治疗等[5-6]。在肝细胞癌发生过程中,若及时发现通过手术可以得到较好的治疗效果,若错过最佳治疗期,生存率则大大降低[7],因此,挖掘肝细胞癌的生物标志物至关重要。除手术治疗外,靶向治疗,如Rfr,PD-L1,VEGFR,一直是肿瘤治疗的有效手段[8],在免疫治疗方向,免疫细胞的激活与紊乱与肝细胞癌患者的预后存在相关性[9-10],寻找多靶点、多途径的治疗方案已成为攻克肝细胞癌的研究方向。
通过GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r)对肝细胞癌样本进行分析获得差异表达基因(Differentially expressed genes,DEGs)。运用STRING(https://cn.string-db.org)对DEGs进行分析,挖掘影响肝细胞癌发病的Hub基因,阐明Hub基因在肝细胞癌中的潜在作用,为肝细胞癌的临床治疗提供可靠依据,生物信息学分析已通过技术路线图进行呈现,如图1所示。

图1生物信息学分析技术路线图
Fig.1Roadmap of bioinformatics analysis technology
1 资料与方法
1.1 仪器及试剂
超微量核酸蛋白测定仪 (美国Thermo Fisher公司型号 ND-200)、荧光实时定量PCR仪 (美国Applied Biosystems公司型号ABI Verit)、-80℃超低温冰箱 (美国Thermo Fisher公司型号Forma-702)、组织研磨仪 (北京天根生化科技有限公司型号SN100235)、RNA simple总RNA提取试剂盒 (北京天根生化科技有限公司批号R7105)、ReverTra Ace qPCR RT Kit试剂盒 (日本TOYOBO公司批号812600)、SYBR Green Realtime PCR Master Mix试剂盒 (上海东洋纺生物科技公司批号83710)。
1.2 原始数据下载
基于GEO数据库获得肝细胞癌转录组数据集,以“Hepatocellular carcinoma”作为筛选关键词,筛选条件为“Expression profiling by array”,“Mus musculus”,下载GSE106769数据集,数据集信息、检测平台以及样本分类等信息见表1。
1.3 方法
1.3.1 原始数据处理
采用GEO2R在线工具分析样本GSE106769的差异基因,结合Excel软件筛选DEGs,筛选条件为P<0.05和|LogFC|≥1.5,筛选出符合标准的基因作为DEGs,利用R语言ggplot包以及Complex Heatmap包分别绘制火山图及差异基因热图。
表1样本数据采集信息
Table1Sample data collection information

1.3.2 GSEA分析
GSEA分析是对数据集基因进行分析,可以找到显著富集的基因集及其变化趋势。在 GSEA软件,根据GEO数据集将样本分成两组,基于基因表达谱和表型分组,设定最小基因集为5,最大基因集5 000,一千次重抽样,以P<0.01作为筛选条件,使用ggplot2对富集前2项结果进行可视化。
1.3.3 差异基因的富集分析
利用Matascape数据库(http://metascape.org/gp/index.html#/main/step1)将DEGs进行GO与KEGG分析,GO分析包括生物过程(Biological process,BP)、细胞组分(Cellular component,CC)、分子功能(Molecular function,MF),GO富集分析设定最小基因集为5,最大基因集为5 000,P<0.05,KEGG富集分析设定最小基因集为3,P<0.01。
1.3.4 WGCNA分析
基于438例肝细胞癌样本和临床表型数据进行WGCNA分析,WGCNA是一种能够发现具有较高生物学意义的共表达基因模块并探索基因网络与疾病之间关系的算法。在样本分组并进行基因差异分析的基础上进行WGCNA,纳入所有差异基因。使用TBtools中的WGCNA插件进行系统聚类分析,以排除孤立点样本,再进行WGCNA分析,筛选最高模块的Hub基因集合1。
1.3.5 核心基因的筛选
利用STRING数据库构建差异基因的PPI网络互作分析图,设置combined score >0.4为阈值条件,使用Cytoscape软件对其进行可视化。采用CytoHubba插件对PPI网络中的中心节点基因进行识别,筛选Hub基因,结合|LogFC|值得出Hub基因集合2,与WGCNA高分数模块Hub集合1进行重叠确定最终的Hub基因。
1.3.6 核心基因的免疫特征及预后分析
TIMER数据库(https://cistrome. shinyapps. io/timer)是基于RNA-Seq表达谱数据分析肿瘤组织中免疫细胞浸润情况的在线分析工具[9]。用 TIMER 数据库“Gene” 模块分析Hub基因与肝细胞癌浸润的免疫细胞的相关性以及在肝细胞癌免疫细胞中的表达水平。对Hub基因进行ROC曲线分析,确定核心基因的诊断阈值,评价诊断效果,采用 R-pROC包进行数据分析,ggplot2包用于可视化分析。利用GEPIA数据库(http://gepia.cancer-pku.cn)分析Hub基因对肝细胞癌患者总生存曲线的影响,并分析Hub基因在肝细胞癌中的临床表达水平。
1.3.7 样本收集
经过患者允许,收集巴彦淖尔市医院6名肝癌患者的癌组织及癌旁组织,取出组织迅速用预冷的PBS清洗至无可见血水,迅速置于液氮中,研磨后加入Trizol试剂,按照RNA提取试剂盒步骤,提取组织中的RNA,提取后-80℃保存。
1.3.8 RNA纯度分析
取 2 μL RNA 样品,使用超微量核酸蛋白测定仪检测 RNA 样本的纯度及含量。RNA 纯度以 OD260/OD280 比值范围在1.7-2.0和OD260/OD230比值大于2.0为标准。
1.3.9 逆转录
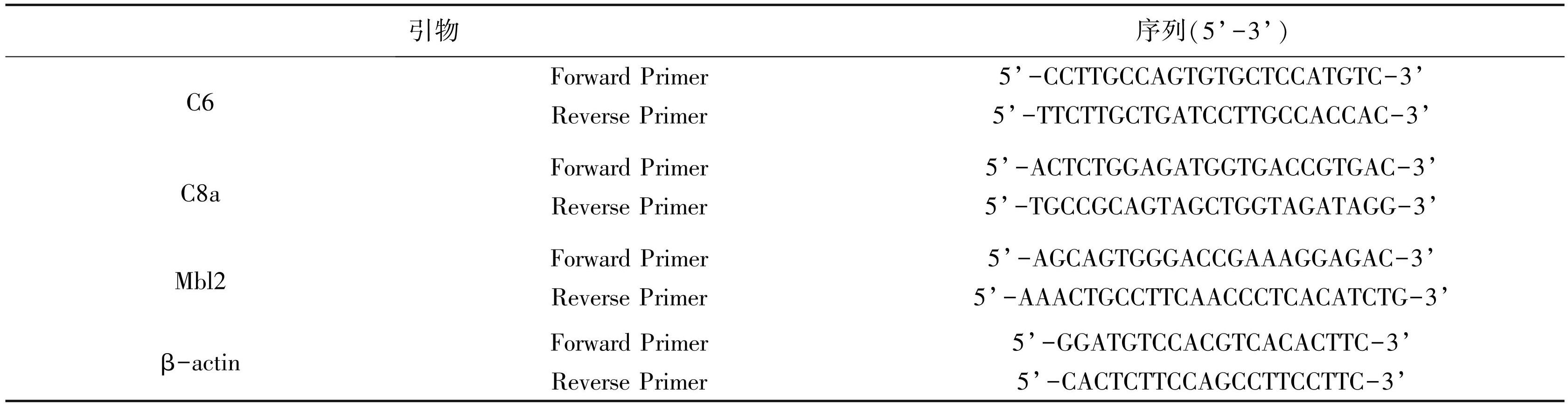
使用ReverTraAce qPCR RT Kit试剂盒进行逆转录合成cDNA,取上述cDNA进行试验,以β-actin作为内参,RT-qPCR技术检测C6,C8a,Mbl2 mRNA的表达水平。反应条件:95℃ 5 s;40个循环:95℃ 5 s,60℃ 10 s,72℃ 15 s;在60℃延伸步骤对荧光信号进行采集,反应结束后,观察融解曲线及扩增曲线,分析qPCR引物的特异性。采用Quante Studio Soft 1.3软件通过2-△△Ct法进行相对定量分析。引物序列见表2。
1.3.10 RT-qPCR 反应
引物由生工生物工程(上海)股份有限公司合成,结果见表2。
表2引物序列
Table2Primers sequence
1.4 统计学分析
实验结果的计量资料以 表示,数据使用Graphpad Prism8.0 软件和SPSS 20.0软件进行统计分析,方差齐时,两组之间比较采用独立样本的t检验,组间比较使用one way ANOVA中的 Bonferroni’s Multiple Comparison Test,方差不齐时,进行秩和检验,P<0.05为差异有统计学意义。
表示,数据使用Graphpad Prism8.0 软件和SPSS 20.0软件进行统计分析,方差齐时,两组之间比较采用独立样本的t检验,组间比较使用one way ANOVA中的 Bonferroni’s Multiple Comparison Test,方差不齐时,进行秩和检验,P<0.05为差异有统计学意义。
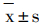 表示,数据使用Graphpad Prism8.0 软件和SPSS 20.0软件进行统计分析,方差齐时,两组之间比较采用独立样本的t检验,组间比较使用one way ANOVA中的 Bonferroni’s Multiple Comparison Test,方差不齐时,进行秩和检验,P<0.05为差异有统计学意义。
表示,数据使用Graphpad Prism8.0 软件和SPSS 20.0软件进行统计分析,方差齐时,两组之间比较采用独立样本的t检验,组间比较使用one way ANOVA中的 Bonferroni’s Multiple Comparison Test,方差不齐时,进行秩和检验,P<0.05为差异有统计学意义。
2 结果
2.1 差异基因的筛选
基于GEO数据库将 GSE106769 中3例肿瘤组织样本和3例肿瘤处理样本分为对照组和实验组进行差异基因的筛选,基于GEO2R分析模块(P<0.05)共获得770个差异基因,其中上调基因404个,下调基因366个,火山图如图2(a)所示,红色节点为表达量上调的基因,蓝色节点为表达量下调的基因。根据P<0.05,|LogFC|≥1.5筛选出的DEGs进行聚类热图分析,如图2(b)所示。
2.2 GSEA 分析
对数据集基因进行GSEA分析,GSEA-KEGG 结果显示通路富集集中于系统性红斑狼疮(Systemic lupus erythematosus)和补体和凝血级联(Complement and coagulation cascade),其中系统性红斑狼疮(FDR = 0.248 0,NES =-1.210 5),补体和凝血级联(FDR = 0.272 0,NES =-0.886 5),P<0.01,如图3所示。
图2基于GEO数据库筛选差异基因
Fig.2Screening of differential genes based on GEO database
注:(a)差异基因火山图:灰色表示差异不显著,绿色表示|LogFC|≥1.5,蓝色表示P<0.05,粉红色表示差异基因; (b)差异基因聚类热图:Group A表示肿瘤样本,Group B表示处理组样本,热图表示差异基因表达量,红色表示高表达,绿色表示低表达.(扫本文首页二维码见彩图).
2.3 富集分析
采用Metascape数据库将差异基因进行GO功能注释和KEGG富集分析。BP分析包括淋巴细胞免疫、补体激活反应、体液免疫应答、免疫球蛋白调节的免疫应答反应、B细胞介导的免疫反应等,如图4(a)所示。MF分析包括补体结合、肽结合、丝氨酸型内肽酶活性、丝氨酸水解酶活性、调理素结合、肽链内切酶活性、类固醇结合、趋化因子的活动等,如图4(b)所示。CC分析包括T细胞受体复合体、晚期内体、多泡体、质膜信号受体复合体、受体复杂、溶解液泡、溶酶体、粗面内质网,如图4(c)所示。同时,对KEGG通路的分析显示,差异基因主要富集出237条KEGG通络,其中包括Th17细胞分化、Th1和Th2细胞分化等,主要与免疫细胞有关,如图5所示。

图3GSEA富集分析
Fig.3GSEA enrichment analysis
注:红色曲线表示富集于系统性红斑狼疮途径,蓝色表示富集于补体和凝血级联途径.(扫本文首页二维码见彩图).

图4差异基因的富集分析结果
Fig.4Results of enrichment analysis of differential genes
注:(a)BP富集结果; (b)MF富集结果; (c)CC富集结果; 纵坐标表示富集到的GO-Term,横坐标表示富集到的基因数量,颜色越接近红色,表示P-value越小,颜色越接近蓝色,表示P-value越大.(扫本文首页二维码见彩图).
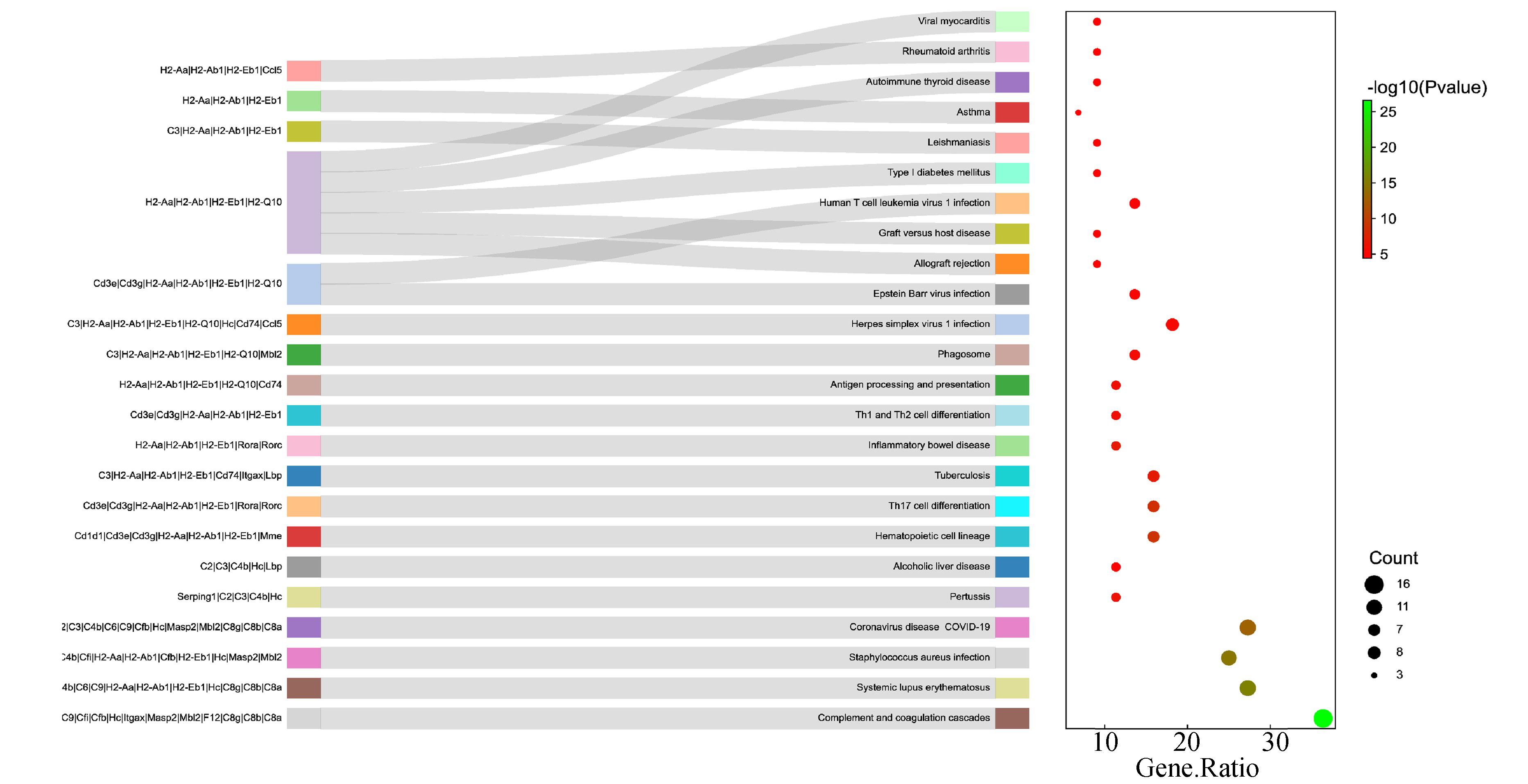
图5KEGG富集结果
Fig.5KEGG enrichment
注:桑基图的左侧表示富集某通路的基因名称,桑基图的右侧表示富集到的通路,图中横坐标表示富集到的特定KEGG通路中与基因列表相关的基因数与该通路总基因数的比例(%),图中点的大小表示富集到的基因数量,点的颜色表示P-value,越接近红色表示P-value越大,越接近绿色表示P-value越小.(扫本文首页二维码见彩图).
2.4 差异基因PPI网络构建
差异基因的PPI网络,如图6(a)所示,共有44个节点和162条边,最低交互分数设置为中等置信数大于0.4,使用cytoscape软件,进行可视化处理,如图6(b)所示。MOCDE模块差异基因的聚类分析,结果可得,差异基因共聚类得到4个聚类簇,第一子网络簇中的基因与Hub基因相同,如图6(d)所示。CytoHubba计算出Hub核心基因如图6(c)所示,分别为HC,C3,C6,C9,Cfi,Cfb,C8a,C8b,C4b和Mbl2。
2.5 WGCNA分析及关键模块的确定
利用WGCNA分析GSE106769数据集的6个临床样本,所有样本均在聚类中并没有需要剔除的样本,且阈值高度小于38,如图7(a)所示。设置软阈值0.8时,对应的阈值为7,构建基因聚类模块,如图7(b)所示,将6个临床样本770个基因聚类得到7个特征的生物模块,如图7(c)所示,模块特征的向量分析中结果可见松石绿(Turquoise)和灰色(Grey)两个颜色的模块与肝细胞癌存在正相关关系,如图7(d),图8(a)所示。因此,我们选用Turquoise“松石绿”颜色模块进行核心基因的筛选。如图8(b)所示,Turquoise颜色模块共提出核心基因51个,与上述STRING数据库所得核心基因对比,发现核心基因在Turquoise模块中完全重叠。

图6差异基因的PPI互作分析
Fig.6PPI interaction analysis of differential genes
注:(a)差异基因的 PPI 网络图;(b)交互分数设置为中等阈值的差异基因PPI网络图;(c)基于PPI网络图筛选Hub基因,颜色越接近红色表示与其互作的基因越多;(d)基于MCODE分析PPI网络不同模块.(扫本文首页二维码见彩图).

图7WGCNA分析结果
Fig.7WGCNA analysis results
注:(a)数据集GSE106769样本聚类树状图;(b)差异基因WGCNA分析,不同颜色表示不同的聚类模块; (c)各模块对应的基因数量;(d)模块基因的相关性热图,红色表示高相关性,绿色表示低相关性.(扫本文首页二维码见彩图).

图8(a)不同模块之间相关性热图:红色表示高相关性,蓝色表示低相关性,横纵坐标表示不同模块,聚类图表示不同模块的树状聚类;(b)松石绿颜色模块的核心基因
Fig.8(a) Heatmap of correlation between different modules:red color indicates high correlation, blue color indicates low correlation, horizontal and vertical coordinates indicate different modules, and the clustering diagram indicates tree clustering of different modules; (b) Hub gene of Turquoise module
2.6 核心基因的免疫特征及预后分析
综合cytohubba的Hub基因和WGCNA中“Turquoise”模块的Hub基因,取韦恩(Venny)交集重叠基因,如图9(a)所示,共有8个重叠Hub基因。Hub基因的ROC分析结果显示C6,C9,C8a,C8b和Mbl2的曲线下方的面积(Area under curve,AUC)曲线下面积大于0.7,参数分别为0.796,0.863,0.830,0.744,0.769,提示这些基因的预后价值良好,如图9(b)所示。K-M生命曲线分析可得,C3,C6,Cfi,Cfb,C8a,C8b和Mbl2基因的过表达和低表达均严重影响肝细胞癌患者的生存期,且P<0.05,如图9(c)所示。在GEPIA数据库中进行核心基因临床表达分析,结果可得,C8a,C6,C9和Mbl2在正常组织和肿瘤组织之间表达存在差异(P<0.05),如图9(d)所示(除HC基因未能在GEPIA数据库中检索外)。在KEGG富集分析中,富集的通路聚焦于Th1,Th2与Th17免疫细胞的分化,进一步分析了Hub基因与免疫细胞的浸润关系。浸润肿瘤的免疫细胞可以影响肿瘤的发展及肿瘤治疗的靶点,因此,肿瘤浸润免疫细胞的量化可以助力揭示免疫细胞在疾病发展中的重要角色。免疫浸润分析结果显示,Hub基因C8a,C8b,C6,C9,Cfi和Mbl2基因与6个肝细胞癌浸润的免疫细胞群有显著相关性,圆形图案越小P值越小,如图9(e)所示。基于AUC,K-M,表达量及免疫浸润参数,筛选到C6,C8a与Mbl2基因同时满足上述条件。因此,将C6,C8a与Mbl2组成的基因集作为肝细胞癌的特征基因集,多基因的联合的ROC的AUC为1。
2.7 RT-qPCR法测定肝细胞癌组织中C6,C8a和Mbl2的 mRNA表达水平比较
结果显示,肝细胞癌Tumor组中的C6、C8a和Mbl2的mRNA表达水平较癌旁组织组显著降低,差异具有统计学意义(P<0.05和P<0.01),见图10所示。
3 讨论
肝细胞癌为最常见的恶性肿瘤之一,发病率及死亡率位居前列[11],对健康造成重大负担[12]。肝细胞癌具有隐匿性,早期表现不明显,导致很多患者发现时就已经到了中晚期,错过了最佳的治疗窗口[13]。现阶段的临床治疗以手术、化疗、靶向用药等手段作为主导[14-15]。因此,肝细胞癌生物标志物的挖掘及治疗靶点成为攻克疾病的重中之重。
本文基于WGCNA加权分析对GEO数据库肝细胞癌的数据集进行综合分析,STRING数据库明确肝细胞癌的Hub基因,包括HC,Cfi,Cfb,C9,C8a,C8b,C4b,C6,C3和Mbl2,与WGCNA加权所得模块进行重叠,得到重叠基因Cfi,Cfb,C3,C6,C9,C8a,C8b和Mbl,对核心基因进行临床预后分析。发现C8a,C6,C9以及Mbl2与肝细胞癌的病理分期显著相关,结合ROC以及OS生存期结果,选取提示预后较好的C6,C8a和Mbl2的基因作为生物标志物,其AUC值为1,结合肝癌的特异性指标甲胎蛋白(AFP)、病理学、影像学等指标将提升肝细胞癌的诊断能力。基于肝细胞癌样本进行RT-PCR定量分析,结果C8a,C6以及Mbl2在肝癌组织中的表达显著低于正常组织,也进一步验证了结果的准确性。
C6,作为补体系统的一种迟效补体蛋白,是肝细胞合成的重要来源,与多种免疫细胞的表达有关。C6的表达与多种疾病进展和临床结局有关,其表达可成为肝细胞癌预后的独立预测指标。C8是一种血清蛋白,由3个非相同的亚基非对称地排列而成,这些亚基包括一个通过二硫键连接的α(C8a)-γ(C8g; 120930)二聚体和一个非共价结合的β链(C8b; 120960)。补体系统由20种不同的蛋白质组成,它们共同摧毁入侵者,并向其他免疫系统参与者发出攻击信号。构成补体系统的蛋白质主要由肝脏产生,并以高浓度存在于血液和组织中。

图9Hub基因在各处理组中的表达水平
Fig.9Expression level of core genes in each treatment group
注:(a)Cytohubba模块的Hub基因与WGCNA模块的基因的Venny分析;(b)Hub基因的ROC分析;(c)Hub基因的Kaplan-Meier分析;(d)基于TCGA数据库C8a,C6,Mbl2与C9的表达量分析;(e)Hub基因与免疫细胞浸润分析,气泡的大小和气泡的颜色表示Hub基因表达与不同免疫细胞之间的相关性,颜色越接近黄色,气泡越小说明其P值越小,基因表达在该免疫细胞中的差异越显著.(扫本文首页二维码见彩图).

图10C6,C8a和Mbl2在肝细胞癌和癌旁组织中表达水平(*P<0.05 ,**P<0.01)
Fig.10Expression levels of C6, C8a and Mbl2 in hepatocellular carcinoma and paracancer tissues
最丰富的补体蛋白为C3,通过“自发”切割及级联反应激活整套补体系统,作用于细胞表面,而在此反应过程中的C6,C7,C8和C9结合,形成膜攻击混合物(Membrane attack complex,MAC)。C5b,C6,C7和C8形成一个“茎”,将复合物锚定在癌细胞膜中,然后加C9蛋白以形成一个通道,以消灭癌细胞。C6和C8a,是补体系统激活途径中的关键成分,在肝细胞癌中低表达,其表达水平与补体系统在癌症免疫监视中缺失的作用有关,它们通过一系列酶促反应,最终导致细胞膜的攻击和裂解,从而消灭入侵的病原体,然而,补体系统的过度激活也可能导致炎症和组织损伤,这可能间接地与HCC的发生和发展有关。有研究发现,C6 rs9200的多态性与肝细胞癌患者的复发显著相关,成为新遗传位点[16-17],另有研究发现C6的表达与肝细胞癌分期分级相关[18]。Mbl2结构上形成由三个或更多个同源三聚体组成的寡聚复合物。这种蛋白的结构特性使得其功能相互作用能够延伸至其他蛋白。Mbl2作为补体系统中的一员,编码一种甘露糖结合凝集素(MBL)或蛋白质(MBP),这种蛋白质是急性期反应的一部分,由肝脏分泌,并参与先天免疫防御。MBL作为免疫的一线分子,能够与多种微生物表达的配体结合,从而激活补体系统并引发一系列的免疫反应,由于MBL与C1q结构相似,当MBL与肿瘤细胞结合,可能通过C1q受体介导作用,使肿瘤细胞被吞噬或溶解[19],Mbl2与C6和C8a的变化趋势一样,证实了补体在肿瘤病程中的失活。Xu等[20]的研究表明,MBL2可调控下游基因的表达从而抑制癌症的生长,抑制肝细胞癌中的促癌因子miR-942-3p的表达,从而抑制肝细胞癌细胞的增殖。
肿瘤的防范与治疗中,机体的免疫功能扮演着重要的角色。免疫机制在肿瘤的发生和发展过程中具有举足轻重的作用。免疫系统具备辨认和清除异常细胞的能力,可抑制肿瘤的增长。然而,肿瘤也能够通过多种途径抑制免疫功能,避免被免疫系统攻击。因此,对于肿瘤的防范与治疗来说,机体的免疫功能都是必须加以重点考虑的。免疫激活是癌症发生发展过程中的主要机体反应,补体系统作为先天性免疫反应中的重要部分,能够在癌症发生后诱导免疫细胞激活,促进炎症与介导免疫屏障的破坏,同时机体也会对于癌症的发生做出抗炎反应。在肝细胞癌中,补体系统的激活可以参与并调节B细胞和T细胞的活化和功能。补体系统的激活会导致C3分子的裂解和产生C3b分子,并与肝细胞癌细胞表面的抗原结合,这些免疫复合物可以作为抗原被抗原呈递细胞捕获并呈递给T细胞,从而激活T细胞的免疫应答[21]。激活的T细胞可以分化为不同的亚群,包括Th1,Th2和Th17细胞,Th1与Th17主要通过产生IFN-γ,IL-17等细胞因子,参与炎症反应,促进抗肿瘤免疫,而Th2细胞主要产生IL-4,IL-5等细胞因子,参与体液免疫应答,可能在肝细胞癌的免疫调剂中起到一定作用[22]。基于DEGs富集的通路也聚焦于各免疫细胞的应答反应,与我们的研究背景关于免疫细胞的召集结果相一致。为了进一步探究免疫浸润与肝细胞癌发生的关系,对筛选的差异基因与免疫细胞进行了浸润分析,结果显示免疫细胞在不同程度上被激活。Chen等已证实免疫细胞可作用于基因从而调控肝细胞癌的发生发展[23]。
4 结论
肝细胞癌的生物标志物为C6,C8a与Mbl2,可通过影响补体激活途径参与肝细胞癌的发展过程,同时激活免疫细胞的响应进而调控疾病的进程。

