摘要
DNA甲基化是存在于真核细胞中的一种关键的表观遗传修饰形式,它能在不改变碱基序列的前提下控制基因表达,并影响生物的发展进程。研究DNA甲基化有助于揭示发育过程中的表观遗传调控机制,为疾病诊断和精准医疗的发展提供重要支撑。Pacific Biosciences(PacBio)的单分子实时测序技术能够进行单分子甲基化检测,无需依赖化学转换过程,保留了更完整的表观遗传信息;并且能够产生平均长度达10000 bp的测序片段,有助于跨越复杂区域并提供更连贯的甲基化信息。然而,现有面向PacBio测序数据的甲基化检测可选工具仍然较少,且存在检测精度不足的瓶颈问题。因此,本研究提出一种基于深度学习技术的面向PacBio测序数据的DNA甲基化检测方法,该方法通过交叉注意力融合机制,并利用Transformer和BiGRU网络融合碱基特征与信号特征,以充分识别潜在的甲基化位点,实现DNA甲基化的高效和精准检测。文中使用GIAB标准品HG002的全基因组PacBio测序数据进行DNA甲基化检测,与当前主流工具相比,本文提出的方法在单片段水平以及基因组水平上均有最高的检测准确性。同时,该方法采用并行处理显著降低了分析时间,并在内存使用上保持了可控性,为大规模人群甲基化的高效检测提供了重要的技术支撑。
关键词
Abstract
DNA methylation is a key epigenetic modification present in eukaryotic cells that can regulate gene expression without altering the DNA sequence itself, impacting the developmental processes of organisms. Studying DNA methylation helps reveal the mechanisms of epigenetic regulation throughout development and provides crucial support for disease diagnosis and precision medicine. Pacific Biosciences (PacBio) real time sequencing technology enables single-molecule methylation detection without relying on chemical conversion processes, preserving a more complete epigenetic profile; it also produces sequencing reads with an average length of 10,000 bp, which helps span complex regions and provides more coherent methylation information. However, the existing tools for methylation detection based on PacBio data are still limited and often lack sufficient accuracy. Therefore, this study introduces a new DNA methylation detection method tailored for PacBio sequencing data using deep learning, which utilizes a cross-attention fusion mechanism and combines Transformer and BiGRU networks to integrate base and signal features, effectively identifying potential methylation sites for efficient and precise DNA methylation detection. This method was applied to the whole-genome PacBio sequencing data of the GIAB standard HG002 sample, and compared to current mainstream tools, our method demonstrated the highest detection accuracy at both the single-fragment and genome levels. Additionally, by using parallel processing, this method significantly reduces analysis time and maintains controlled memory usage, providing important technical support for efficient methylation detection in large-scale populations.
Keywords
DNA甲基化是真核细胞中的一种关键表观遗传修饰形式[1-3]。在DNA甲基转移酶的作用下,基因组CpG二核苷酸的胞嘧啶(Cytosine)的第五号碳原子上加入一个甲基基团(-CH3)[4-5]。在哺乳动物基因组中,DNA甲基化主要发生在CpG双核苷酸上,即胞嘧啶(C)与鸟嘌呤(G)之间通过磷酸二酯键相连的区域[6-7]。DNA甲基化包括多种类型,其中最常见的是5-甲基胞嘧啶(5-methylcytosine,5mC)[8],此外还有5-羟甲基胞嘧啶(5-hydroxymethylcytosine,5hmC)[9-10]、N6-甲基腺嘌呤(N6-methyladenine,6mA)[11]及7-甲基鸟嘌呤(7-methylguanine,7mG)。DNA甲基化在生物体的发育、基因表达调控、基因沉默、X染色体失活以及某些疾病的发生过程中发挥着重要作用[12-14]。研究DNA甲基化模式及其动态变化,有助于揭示发育过程中的表观遗传调控机制,探索疾病的分子机制和潜在治疗靶点,为疾病的预防和治疗提供新策略[15-16]。
面向第二代测序技术的DNA甲基化测量方法依赖于亚硫酸盐转化,该方法通过亚硫酸盐将非甲基化的胞嘧啶转化为尿嘧啶,而甲基化的胞嘧啶不发生转化[17-18]。经过处理后的DNA通过PCR扩增和测序,可以准确检测每个胞嘧啶位点的甲基化状态。然而,传统的亚硫酸盐处理会导致大量DNA断裂,复杂化了高度变异、异质表观基因组的分析,且无法分析重复基因组区域[19]。随着Pacific Biosciences(PacBio)的单分子实时测序(SMRT)[20]的发展,凭借其前所未有的读长(平均高达10 kb),开始突破短读测序在基因组分析中的局限[21-22]。PacBio测序利用DNA聚合酶在SMRT测序中的动力学变化直接检测DNA修饰,其中DNA修饰影响了DNA聚合酶的活性。PacBio推出的高精度长读长测序技术(HiFi测序技术),通过对单个分子进行多次循环共识测序生成,可实时监测聚合酶在合成带有荧光标记核苷酸的DNA链时的行为[23]。其动力学特征如脉冲宽度和脉冲间隔,与DNA的化学修饰(例如5-甲基胞嘧啶,5mC)密切相关,且无需进行亚硫酸盐处理[24]。
目前,深度学习方法已经广泛地应用于DNA甲基化检测的研究[25],例如,香港中文大学开发的基于卷积神经网络(CNN)的甲基化检测算法,利用测序信号和位置矩阵表示碱基序列,从PacBio数据中检测DNA甲基化位点[26];中南大学开发的基于注意力机制的双向门控循环网络(Bi-GRU)甲基化检测算法,通过注意力机制融合正负链信息,并结合碱基序列与测序信息进行DNA甲基化判断[27]。然而,以往的研究没有充分利用PacBio测序数据的序列信息与脉冲信号特征,且模型的预测精度较低。本文提出了一种新的基于深度学习技术的面向PacBio测序数据的DNA甲基化检测方法,该方法最大限度地利用甲基化位点的原始信息,将PacBio测序数据的原始脉冲信号以及碱基序列作为输入,通过Transformer网络对碱基特征进行编码,Bi-GRU网络对脉冲信号进行编码,然后将二者拼接后输入到Cross-Attention网络中,经过全连接网络预测DNA甲基化状态,提高检测的准确性和效率。真实数据的实验结果表明,本文提出的模型和方法在单片段和基因组水平上均有最优的甲基化检测能力,并且在检测模型不同物种间具有良好的泛化能力。
1 数据获取与预处理
1.1 数据收集
Genome in a Bottle (GIAB)是由美国国家标准与技术研究院(NIST)主办并支持的组织,致力于对基准人类基因组进行权威性表征。本研究中实验所涉及的数据来自GIAB中的德系犹太人标准品HG002正常淋巴细胞,并通过PacBio单分子实时测序技术进行测序得到,原始测序数据平均长度为20 kb,下载地址为https://console.cloud.google.com/storage/browser/brain-genomics-public/research/deepconsensus/publication/sequencing. 由于基于亚硫酸盐测序技术的基因组甲基化检测结果准确性较高[17,28],因此我们还获取了该标准品的亚硫酸盐测序数据,并通过标准检测流程识别该标准品的DNA甲基化检测结果,包括54 772 965个DNA甲基化位点,下载地址为https://labs.epi2me.io/dataindex.
1.2 原始测序数据与答案预处理
PacBio测序中的脉冲信号与发生聚合反应的核苷酸有关,包括脉冲持续时间(IPD)和脉冲宽度(PW),其中IPD表示两个连续测序碱基之间的持续时间,PW表示一个碱基被测序的持续时间。除了被测序的核苷酸,碱基修饰也会影响脉冲信号,利用修饰和未修改碱基之间脉冲信号的差异,可以开发出检测 5mC 和其他碱基修饰的方法。对于HG002样本的原始测序数据(Subreads,BAM格式),首先使用官方工具pbccs(v6.4.0,https://github.com/PacificBiosciences/ccs),并指定--hifi-kinetics参数对subreads进行数据处理,以生成高精度HiFi数据,同时提取原始测序的脉冲信号(IPD和PW)。随后,使用官方比对工具pbmm2(v1.9.0)将HiFi数据比对到人类参考基因组,使用samtools(v1.17)[29]将比对结果按照基因组坐标进行排序并建立索引,生成最终的比对结果文件(排序后的BAM格式文件)。数据处理过程中使用的人类参考基因组版本为GRCh38,该版本是目前国际上广泛采用的参考基因组。
从基于亚硫酸盐测序数据的甲基化检测结果中筛选完全甲基化位点和完全未甲基化位点分别作为训练数据答案。完全甲基化位点满足的条件为:①位点覆盖度≥5;②甲基化概率为100%;完全未甲基化位点满足的条件为:①位点覆盖度≥5;②甲基化概率为0%。
1.3 训练数据获取
在单片段水平(Read-level),选取Chr1和ChrX中的甲基化位点作为验证数据,其他染色体上的甲基化位点用做训练数据,共筛选1 200万个训练数据位点和200万个验证数据位点,训练数据和验证数据中的正负标签比例约为1∶1。类似地,在基因组水平(Genome-level),共筛选500万个训练数据位点和50万个验证数据位点,且正负标签比例同样为1∶1。
2 甲基化检测方法
基于深度学习模型的DNA甲基化检测方法主要包括:①特征提取与构建;②单片段水平甲基化位点预测;③基因组水平甲基化位点预测等3个步骤。
2.1 特征提取与构建
对于HiFi测序数据中的每条测序片段,首先将测序片段与参考基因组进行对齐,为了消除样本差异,使用标准正则化将IPD和PW进行归一化处理后分别读取正向以及反向互补链中的碱基及对应的IPD和 PW值。对于正链中的CpG位点,以CpG为中心,截取上下文各15 bp碱基序列构建碱基序列特征,同时对与这些碱基相对应的IPD和PW值进行相同的处理,构建上下文15 bp的脉冲信号特征。对于负链采用相同的方式构建对称互补的CpG位点特征。最终,采取拼接的方式将正负链的特征进行融合,具体的,碱基序列、IPD和PW均会形成1×62的特征矩阵(1为批大小(Batch),62为数据长度)。随后将IPD和PW的数据进行拼接,形成1×124的脉冲信号特征矩阵。
2.2 单片段水平甲基化位点预测
在本文中,首先使用词嵌入层将每个碱基A,C,G,T转换成维度为64的向量,构成1×62×64的特征矩阵(1为batch,62为数据长度),并将其输入到一个6层自注意力的Transformer中,其中Transformer自注意力机制的公式描述如下。对于输入的特征矩阵X∈Rn×d, 其中n表示序列长度,d 表示嵌入维度,自注意力的计算包括三个步骤:首先生成查询(Query)、键(Key)和值(Value)矩阵,如公式(1)所示:
(1)
其中 WQ,WK,WV∈Rn×d 是可学习的参数矩阵。然后计算查询和键之间的点积注意力分数,并通过 softmax 函数归一化,如公式(2)所示:
(2)
dk通常等于d,通过这种方式自注意力机制能够捕捉序列中不同位置间的全局依赖关系,获取其潜在的序列关联信息。
随后利用线性层将每个脉冲信号特征转换成维度为64的向量,构成1×124×64的特征矩阵(1为batch,124为数据长度),并将其输入到一个3层Bi-GRU网络中,其中Bi-GRU的更新主要包括如下三个步骤:
1)更新门zt与重置门rt,如公式(3)所示:
(3)
其中,σ 表示 sigmoid 激活函数,Wz,Wr是可学习的权重矩阵,ht-1是前一时刻的隐藏状态,xt是当前时刻的输入。
2)候选隐藏状态 ,如公式(4)所示:
(4)
其中⊙表示元素级别的乘法操作,Wh 是权重矩阵, 是候选的隐藏状态。
3)最终隐藏状态 ht,如公式(5)所示:
(5)
每层Bi-GRU的隐藏层大小为256,随后经过线性层将数据维度降为64,获取潜在的脉冲信号特征信息。最后将脉冲信号特征与碱基序列特征一起输入到6层交叉注意力的Transformer中,经过线性层和softmax层输出read-level的甲基化概率Pr。本文根据预测的CpG位点的甲基化概率值推断甲基化状态:若Pr > 0.5,则将该位点预测为甲基化位点,否则将该位点预测为未甲基化位点。单片段水平甲基化预测的具体流程如图1所示。
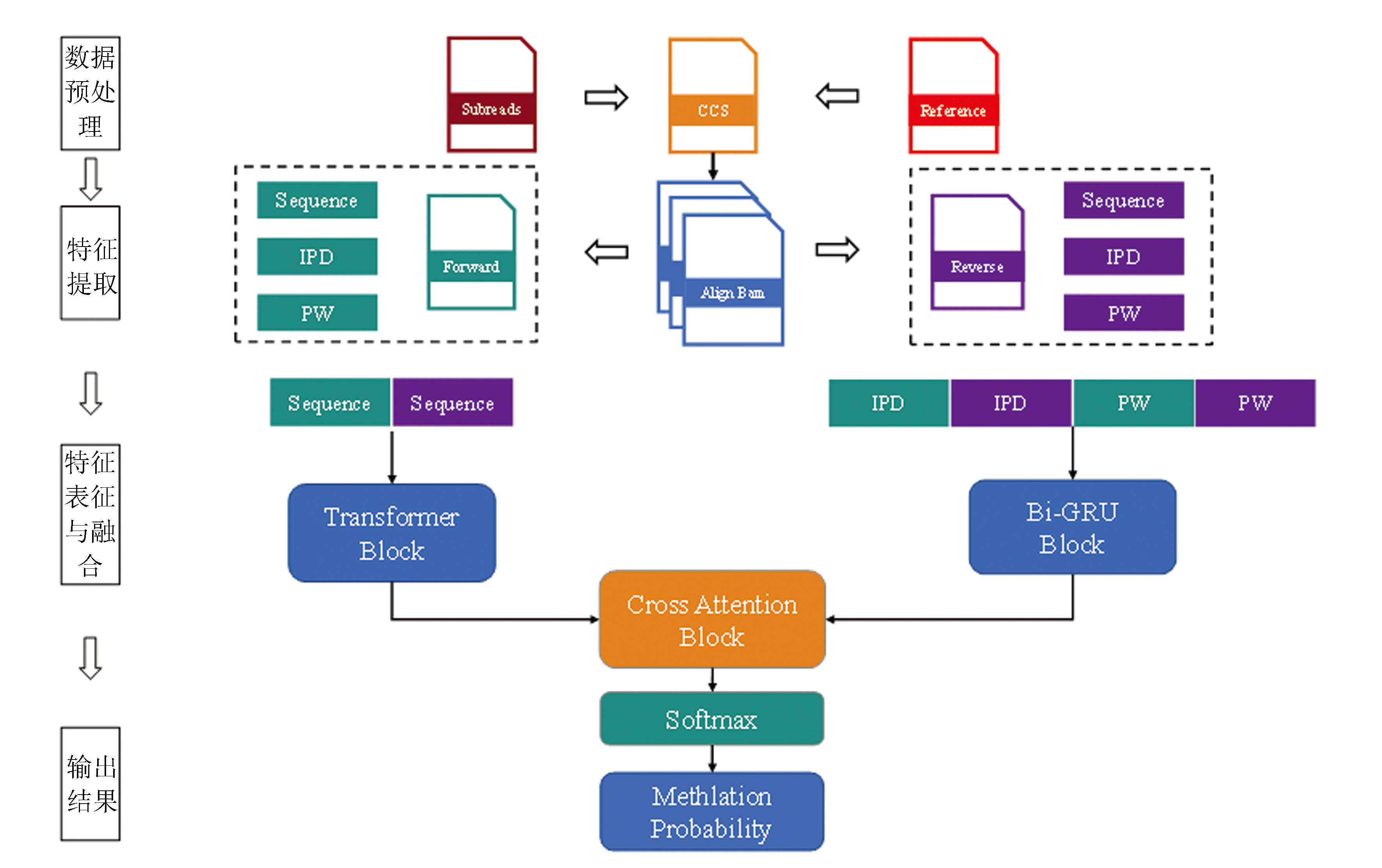
图1单片段水平甲基化预测流程
Fig.1Methylation prediction on Read-level
2.3 基因组水平甲基化位点预测
本文主要通过如下两种方法进行基因组水平的甲基化位点预测。①基于单片段计数的甲基化检测方法,该方法根据单片段的预测结果统计支持该CpG位点为甲基化位点的测序片段数量,并除以比对到当前CpG位点的所有测序片段数量,作为该CpG位点的甲基化概率Pg;②基于卷积神经网络的甲基化预测方法,该方法使用比对到同一CpG位点的所有测序片段特征信息,构建序列特征和脉冲信号特征。具体地,本文设置比对位置相同的测序片段最大数量为13,如果测序片段数量多于13则直接丢弃,如果少于13则使用0进行填充。基因组水平构建的碱基序列特征矩阵为13×62×1(13为read数量,62为数据长度,1为数据维度),采用相同的方式构建对应的IPD和PW脉冲信号特征矩阵。在基因组水平中加入了正负链信息,分别使用1,-1对正链和负链进行编码,将碱基序列、IPD,PW以及正负链的特征矩阵在第三维度进行拼接,构建13×62×4的特征矩阵(13为read数量,62为数据长度,4为数据维度)。使用卷积核为3×3,步长为2,输出维度为64,填充为2的卷积神经网络获取局部特征,使用卷积核为3×3,步长为1,填充为1的卷积神经网络以及ReLu和Batch Norm作为残差连接层,以缓解梯度消失问题,该层的输出维度为28。重复上述卷积层和残差连接层将模型维度提高至256。使用由卷积核为1×1,2×2,3×3的卷积神经网络作为感知层进一步提取甲基化特征,最后使用Flatten层整合所有数据,使用线性层和softmax层输出CpG位点的甲基化概率Pg。类似的,如果Pg > 0.5,则将该位点预测为甲基化位点,否则将该位点预测为未甲基化位点。基因组水平甲基化预测的具体流程如图2所示。

图2基于卷积神经网络的甲基化预测方法流程
Fig.2Methylation prediction based on convolutional neural network
3 深度学习模型训练
3.1 单片段水平模型训练
从HG002测序数据的比对结果中选择比对到Chr1以及ChrX的数据作为验证数据集,其他染色体按照9∶1的比例随机拆分为训练数据集和测试数据集。模型的batch为512,初始学习率为1×10-4,采用cross-entropy(交叉熵)计算最小loss损失。使用AdamW作为优化器,其参数beta设置为(0.9,0.99)同时采用L2正则化,迭代器采用ReduceLROnPlateau,并设置在验证数据集中连续两个轮次学习的性能没有提高则将学习率衰减为当前的0.1倍。为了防止模型过拟合,在交叉注意力层中加入dropout层,并将其设置为0.5,同时采用早停策略,若模型在验证集中连续三次性能下降则停止训练。
3.2 基因组水平模型训练
在基于卷积神经网络的基因组水平甲基化位点预测任务中,训练数据按照8∶2的比例划分训练数据和测试数据,数据在输入到模型之前进行正则化处理,输入模型的batch为256,初始学习率为5×10-5,优化器和损失函数与read-level模型相同,迭代器使用CosineAnnealingLR,最大学习率为5×10-4,最小学习率为5×10-5,dropout值为0.3。
4 甲基化检测结果与分析
本文使用HG002以及斑马鱼样本的PacBio HiFi测序数据,综合评估当前主流工具HK-model,Primrose以及本文提出的方法在单片段水平和基因组水平的DNA甲基化检测的性能。使用pytorch平台框架作为工具,以python3.8作为开发环境。评测指标主要包括精确率(Precision)、召回率(Recall)、准确率(Accuracy)和F1-分数(F1-score),其计算公式如下:
(6)
其中TP 表示预测为甲基化位点且实际上为甲基化位点,TN 表示预测为非甲基化位点且实际不是甲基化位点,FP 表示预测为甲基化位点且实际上不是甲基化位点,FN 表示预测为非甲基化位点且实际上是甲基化位点。
4.1 单片段水平甲基化预测结果
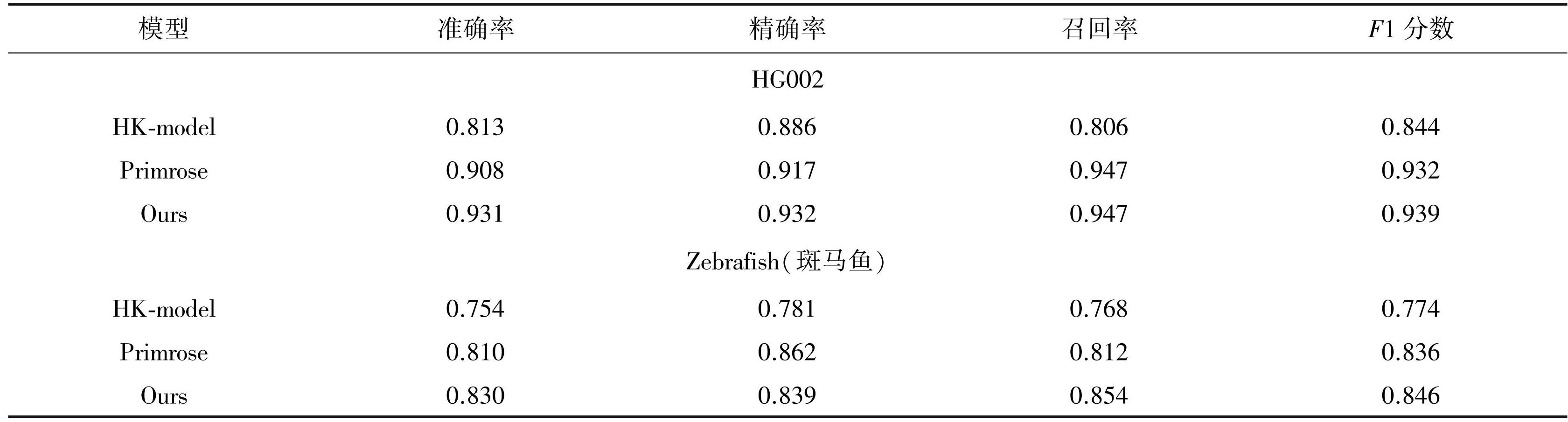
针对单片段水平甲基化预测,本文使用Transformer编码碱基特征,使用Bi-GRU编码脉冲信号特征,随后通过数据融合网络对两个特征进行融合,输出经过全连接网络输出最终的每条测序片段上甲基化预测结果。使用HG002与斑马鱼样本的PacBio测序数据分别进行甲基化预测实验,与对比工具结果进行比较。HG002上的预测结果(表1)表明,本文提出的方法在Accuracy,Precision,Recall以及F1-score等指标上达到最优,且均超过93%。具体的,相较HK-model,本文提出的方法在上述各个指标上分别提高了11.8%,4.6%,14.1%和9.5%;相较Primrose,在Recall相当的基础上,Accuracy和Precision分别提高了2.3%和1.5%。表明本文提出的方法在单片段水平的DNA甲基化预测任务上具有显著优势。
由于模型使用HG002样本部分染色体上的测序数据进行训练,使用其他染色体上的测序数据进行验证,为了进一步证明模型的有效性和泛化性,本文继续使用斑马鱼样本的测序数据对模型进行验证,结果如表1所示。结果表明所有工具的模型在处理跨物种甲基化检测的任务中,性能均有所下降,但本文提出的方法在Accuracy,Recall以及F1-score等三个指标上仍有显著优势,相较于HK-mode和Primrose,分别提高了7.6%,2%,8.6%,4.2%和7.2%,1%。因此本文所提出的方法具有良好的泛化性,可用于不同物种的甲基化检测。
表1不同工具在HG002和斑马鱼测序数据上的单片段水平甲基化检测结果
Table1The read-level results of different tools on HG002 and zebrafish data
4.2 基因组水平甲基化预测结果
本文提出了两种基因组水平的甲基化预测方法:①基于单片段计数的甲基化检测方法(Count); ②基于卷积神经网络和残差连接网络的甲基化检测方法(Model)。同样使用HG002样本与斑马鱼样本对甲基化预测结果进行验证,结果如图3所示。本文所提出的两种预测方法在不同物种上较HK-model和Primrose均有更高的F1-score。具体的,基于片段统计的方法在HG002样本上分别优于HK-model,Primrose7.3%和1.1%,在斑马鱼样本上的优势变化为9.8%和4.8%;类似的,基于深度学习模型的方法在HG002和斑马鱼上相较HK-model和Primrose的优势分别为6.9%,0.7%和7%,2%。此外,本文提出的两种方法在Precision和Recall两个指标上也均达到最优,表明本文提出的方法具有最优的甲基化检测能力和物种泛化能力。

图3不同工具在基因组水平的甲基化检测结果
Fig.3Results of different tools on the genome-level
4.3 数据下采样检测结果
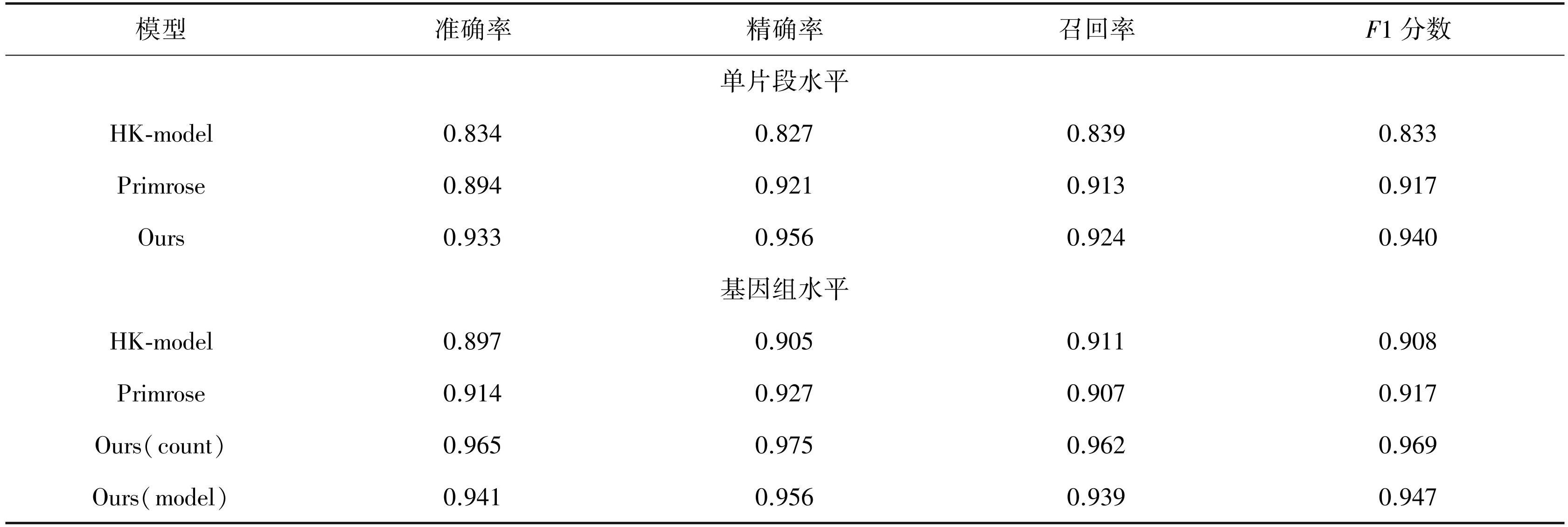
本文进一步比较了测序深度对于不同工具甲基化预测结果的影响,将HG002样本随机下采样至原测序深度的一半,重复上述实验评价不同工具的检测结果,结果如表2所示。本文提出的方法在单片段水平上,较HK-model和Primrose具有最高的Accuracy,Precision,Recall和F1-score,表明其甲基化检测的优越性能。在基因组水平,两种不同的方法在所有指标上仍显著优于其他对比工具。因此,本文提出的方法在低测序深度下,相较于其他两个工具仍表现出较高的优势,能够满足在低覆盖度下甲基化检测的需要。
表2不同工具在低深度HG002测序数据下的检测结果
Table2Results of different tools under low coverage HG002 sequencing data
4.4 运行时间及内存占用分析
文本使用10万条测序片段,与其他模型在时间效率、内存占用以及参数规模进行比较,结果如表3所示。其中运行时间为使用ccs进行HiFi数据生成之后到模型生成相应的甲基化检测结果所用的时间,包括特征提取与表示、模型训练和甲基化检测。本文提出的方法较HK-model和Primrose具有相当的运行效率且占用最小的内存,此外本文提出的方法参数量为5 910 564,略高于HK-model。综合考虑准确性和敏感性等指标,本文提出的方法针对甲基化检测任务在检测质量和性能上均具有不错的表现。
表3模型时间及内存占用分析
Table3Runtime and memory usage analysis

4.5 模型训练损失函数变化
在训练过程中为了防止过拟合影响模型性能,分别打印了单片段水平和基因组水平甲基化预测模型在训练阶段的前30轮的损失函数(Loss)的变化曲线,如图4所示。从图中可以看出模型在前期逐渐下降,单片段和基因组水平的模型分别在第6轮和第17轮迭代后趋于稳定,此时模型的各项指标达到最优。
5 讨论与结论
DNA甲基化的检测对于辅助临床诊断具有重要的应用价值,本文针对PacBio长读长测序数据,利用深度学习技术研发了DNA甲基化位点检测的新模型和新方法。本文的主要创新点包括:
1)在单片段水平甲基化检测任务中使用交叉注意力机制融合了信号特征和碱基特征,充分考虑到不同的特征信息对于甲基化结果的影响。
2)在基因组水平甲基化检测任务中使用卷积神经网络以及感知网络识别局部区域的甲基化特征并将其定位至基因组上。通过对真实人类(HG002)和斑马鱼的长读长测序数据进行甲基化的检测任务,综合评估当前主流工具HK-model,Primrose以及本文提出的方法的实验结果,表明本文提出的方法在单片段和基因组水平上均有最优的甲基化检测能力,并且在检测模型不同物种间具有不错的泛化能力。
本文提出的模型和方法仍有可以改进的地方:

图4模型训练损失函数变化
Fig.4Training loss of the model
1)参考使用k-mer的数据形式,通过k-mer可以在单片段水平中表征局部的碱基序列和特征信息,可以扩展序列上下文范围,从而更有效地识别甲基化。
2)考虑特征信息的不同组合方式,加入更多的特征信息辅助于碱基信息。
3)在碱基序列的表征中加入预训练模型,预训练的通用和复杂的生物序列特征,有助于提高模型在特定任务上的表现和准确性。
4)在基因组水平甲基化检测任务中使用LSTM或transformer等模型,不同的模型有助于发现不同的甲基化特征,提高甲基化检测的准确性。
总而言之,本文提出的基于PacBio长读长测序数据的DNA甲基化检测方法,通过深度学习技术实现了检测模型的构建,该模型在验证集中相较于目前已有的检测工具具有最好的综合性能,能够高效、准确地识别DNA甲基化,并作为一种疾病早筛的手段,具有良好的应用前景。此外,未来也可以围绕以下几方面进行探索,以进一步提升基于深度学习的DNA甲基化检测能力:
1)更多物种的泛化能力研究:尽管本研究的模型在不同物种之间表现出良好的泛化能力,未来可以扩大测试数据集,尤其是更具多样性的物种或具有复杂基因组特征的物种,以验证模型的普适性和鲁棒性。
2)结合单细胞测序技术:随着单细胞测序技术的发展,将单细胞测序数据与长读长测序数据相结合,探索单细胞层面的甲基化变化,这将为临床诊断和个性化治疗提供更加细粒度的信息。
3)模态数据融合:除了碱基序列和脉冲信号特征,未来可以引入更多生物信息学数据(如表观遗传数据、转录组数据等)来构建更为全面的多模态模型,更准确地捕捉不同层次的生物学信号,从而提高甲基化检测的准确性。
