摘要
基于局部序列相似性比对的数据库搜索系统BLAST是生物信息学领域常用工具之一。本文首先介绍数据库相似性搜索的基本概念,包括计分矩阵、空位罚分,以及灵敏度和特异度等;以血红蛋白alpha和beta亚基为例,说明BLAST搜索基本策略,包括分割种子串、确定近邻串、搜索高分对、延伸高分对、计算期望值等。讨论种子序列字长、计分矩阵、空位罚分等对搜索结果的影响。介绍blastp,blastx,blastn和tblastn四个BLAST通用程序,以及SmartBlast,Primer-Blast和Global Align等专用程序。文末简述BLAST主要用途,列举几个国际国内BLAST网站,介绍FASTA,BLAT,HMMER等其它数据库搜索程序。
Abstract
The Basic Local Alignment Search Tool (BLAST) is the sequence database search system based on local sequence alignment. It is one of the most commonly-used sequence analysis tools in bioinformatics. After giving the general concept of sequence database search, we start with the description of the main strategy of BLAST search: ① divide the seed sequence; ② find the neighborhood sequence; ③ search for high scoring pair; ④ extend the high scoring pair; ⑤ calculate the expected value E. The effect of the major parameters such as word size of the seed, the scoring matrix and the gap-penalty are discussed. In addition to the routine programs blastp, blastn, blastx and tblastn, special programs such as SmartBlast, Primer-Blast and Global Align, are also briefly described. Finally, we list the main usage of BLAST, several international and domestic BLAST web sites, and other database search tools such as FASTA, BLAT, HMMMER.
1 前言
二十世纪五十年代DNA双螺旋模型的提出,标志着生命科学进入分子生物学时代;七十年代Sanger测序技术的发明,为基因组学奠定了基础;九十年代开始的人类基因组计划,推动了生命科学进入大数据时代。近十多年来,随着二代、三代DNA测序技术的不断问世和改进,成千上万不同物种的基因组草图绘制成功,基因组、转录组、蛋白组、代谢组、表观组等组学数据爆炸性增长。众所周知,DNA、RNA和蛋白质序列,是生物大分子组学数据中最基本、最常用的数据,基于序列相似性的数据库搜索已经成为生命科学研究不可或缺的工具。
基于序列相似性的数据库搜索,顾名思义,就是以某个核酸或蛋白质序列作为查询序列(Query sequence),与核酸或蛋白质序列数据库中的序列进行比较,找出数据库中与查询序列相似性较高的序列,即目标序列(Subject sequence)。不言而喻,数据库相似性搜索的基础是序列比对(Sequence alignment)。序列比对的方法可以分为两类,一类从全长序列出发,从整体上考虑所比对序列的相似性,即整体比对,也称全局比对(Global alignment); 另一类仅考虑所比对序列部分区域的相似性,即局部比对(Local alignment)。全局比对常用来考察两个或多个序列是否具有整体相似性,并由此推断是否为同源序列;而局部比对则可以找出保守序列片段,如蛋白质序列中结构域、重复序列和功能位点等,基因上游启动子区域核酸序列调控元件、RNA发夹结构等。BLAST采用的是局部比对策略,即搜索数据库中与查询序列具有局部相似性的序列片段,若查询序列与目标序列的多个区域具有相似性,则在搜索结果中分别列出。
对于两个给定的序列,无论是整体比对还是局部比对,都可以利用动态规划(Dynamic programming)算法,找到最佳比对结果[1]。所谓最佳比对,是指基于给定的计分矩阵(Scoring matrix)和空位罚分(Gap penalty),比对结果的分值最高。这种利用动态规划进行双序列比对的方法,用于数据库搜索,找出数据库中所有与查询序列相似的目标序列,则需要将查询序列与数据库中每条序列都进行比对,计算复杂度为O(N*M)。此处,N为查询序列长度,M为数据库中所有序列总长,对数据量较大的数据库,计算量极大。BLAST采用启发式算法(Heuristic Algorithm)而非动态规划,搜索速度大为提高。
2 数据库搜索基本概念
2.1 数据库搜索和数据库检索
基于序列相似性的数据库搜索与基于关键词的数据库检索都是生物信息领域中常用工具。数据库检索,实质上是文本检索,即通过关键词匹配,从某个数据库中找出需要的条目。例如,输入作者姓名、期刊名称、文章标题等特定关键词,从美国国家生物信息技术中心(National center for biotechnology information,NCBI)生物医学文献摘要数据库PubMed中检索相关文献,就是数据库检索的典型应用。又如,输入蛋白质名称、物种名称、蛋白质功能等关键词,从蛋白质序列数据库UniProt(https://www.uniprot.org)中检索特定序列条目,则是数据库检索的另一个常用实例[2]。
基于序列相似性比对的数据库搜索,则是核酸和蛋白质序列数据库的另一个重要应用。与基于关键词的数据库检索不同,数据库相似性搜索所输入的不是文本信息,而是蛋白质或核酸一级结构序列信息;搜索对象也不是文本信息,而是数据库中的核酸或蛋白质序列信息。由于历史的原因,上述数据库检索和数据库搜索两个术语经常混用。例如,PubMed文献检索和UniProt蛋白质数据库序列高级检索都使用英文Advanced Search这一术语,既可以翻译为“高级检索”,也可以翻译为“高级搜索”。为便于叙述,除特别说明,本文将基于序列相似性比对的数据库搜索简称“数据库搜索”,而把基于关键词的数据库检索简称“数据库检索”。
2.2 序列比对和数据库搜索
数据库搜索的基础是序列比对,即序列相似性比对。序列比对通常分为两种,一种是两个序列之间的比对,找出它们的相同位点或相同区域,即双序列比对(Pairwise sequence alignment);另一种是多个序列同时进行比对(Multiple sequence alignment),找出它们之间的保守位点或保守区域。数据库相似性搜索则是从数据库中找出与查询序列具有一定相似性的目标序列,逐个进行比对。因此,数据库相似性搜索的基础是双序列比对。
数据库搜索方法和软件有多种,BLAST是较为常用的一种。BLAST是英文Basic local alignment Search Tool的缩写,按英文字面意思直译为“基本局部序列比对的搜索工具”;可以理解为“基于局部比对的序列数据库搜索工具”。具体说来,BLAST是蛋白质和核酸序列数据库搜索程序,即用一个或多个蛋白质或核酸序列为查询序列,搜索蛋白质或核酸序列数据库,找出与查询序列具有较高相似性的目标序列(Subject sequence),也称匹配序列(Match sequence)和命中序列(Hit sequence)。
2.3 计分矩阵
数据库搜索本质上为序列比对,而计分矩阵是序列比对的基础。用于核酸序列比对的计分矩阵通常有两种,即DNAMatrix和DNAFull。DNAMatrix只包含腺嘌呤A(Adenine)、鸟嘌呤G(Guanine)、胞嘧啶C(Cytosine)和胸腺嘧啶T(Thymine)四种基本核苷酸,通常匹配(Match)分值为正,错配(Mismatch)分值为负。而DNAFull计分矩阵除了上述四种基本核苷酸外,还包括嘌呤R、嘧啶Y等,分别表示不同类别核苷酸。笔者“双序列比对基础和应用实例”一文对此做了详细介绍[1]。
用于蛋白质序列比对的相似性计分矩阵有多种,BLAST系统采用PAM和BLOSUM两种计分矩阵。
PAM计分矩阵的英文全称为Point Accepted Mutation,即位点可接受突变矩阵。上世纪七十年代,美国乔治敦大学(Georgetown University)医学中心Margaret Dayhoff教授基于当时收集到的相似性高于85%的几十组蛋白质同源序列,用手工方法进行多序列比对,统计特定位点20种不同氨基酸出现的频率,并与随机背景序列的频率进行比较,用统计方法得到可接受点突变计分矩阵PAM1。将PAM1矩阵自乘,则可得到PAM2矩阵,自乘10次,则可得到PAM10矩阵,以此类推。实际使用的PAM计分矩阵是原始概率矩阵通过对数变换得到的几率矩阵。常用的有PAM30,PAM100,PAM250等。
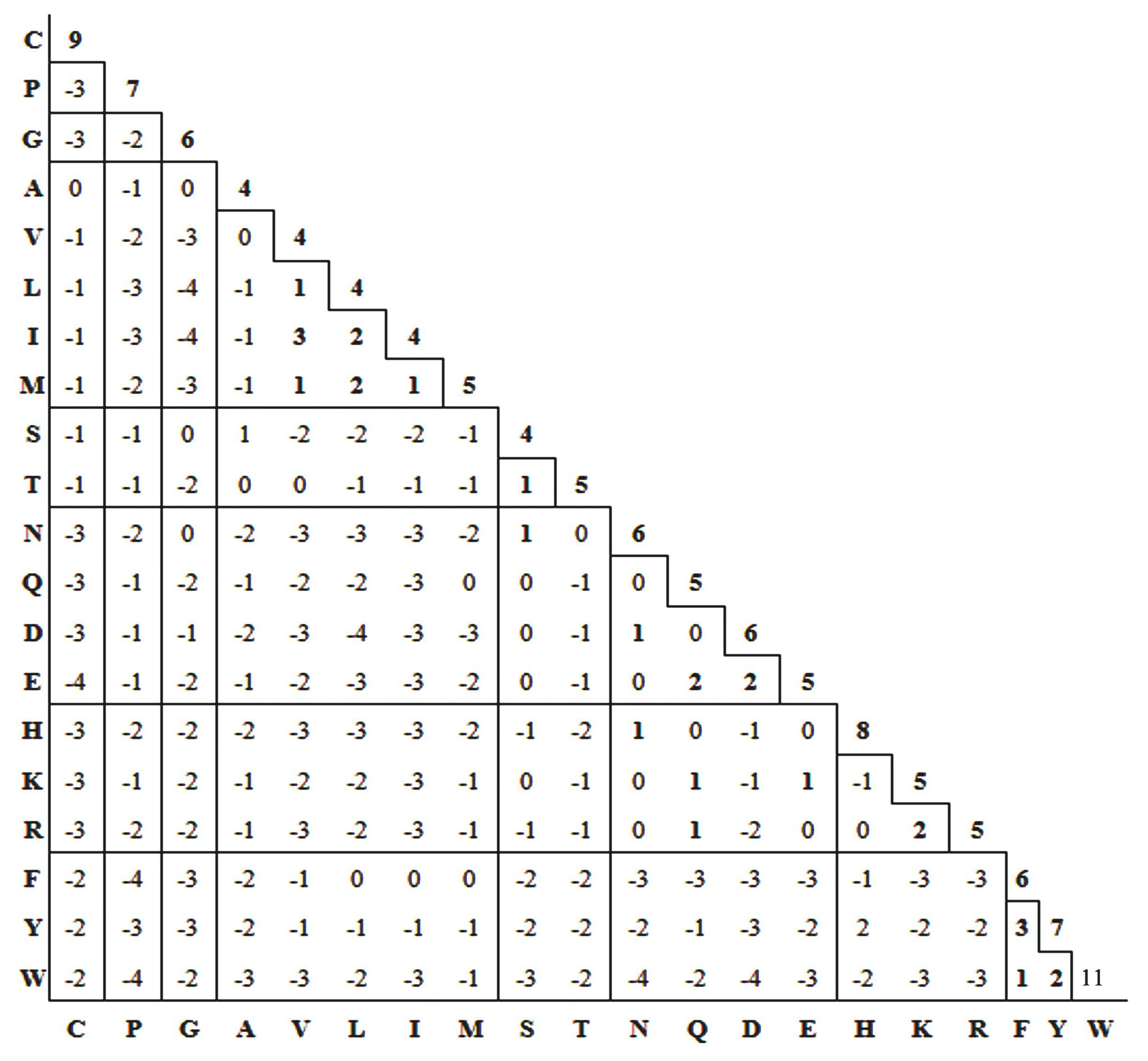
BLOSUM计分矩阵的英文全称为Blocks Substitution Matrix,由美国西雅图弗雷德·哈钦森癌症研究中心(Fred Hutchinson Cancer Research Center)Henikoff夫妇于上世纪九十年代基于蛋白质序列模块数据库BLOCKS构建[3]。构建BLOSUM矩阵时,已测定的蛋白质序列远比构建PAM矩阵时多,因此,BLOSUM数据库中的序列按不同相似性阈值分成不同数据集,如相似性阈值高于90%,60%,30%,而构建的矩阵也相应分为BLOSUM90,BLOSUM60和BLOSUM30等。NCBI BLAST数据库搜索系统的默认计分矩阵为BLOSUM62(表1)。
表1相似性计分矩阵BLOSUM62
Table1The BLOSUM62 Scoring Matrix
2.4 空位罚分
除了计分矩阵外,序列比对的另一个重要参数为空位罚分。所谓空位罚分,是指比对过程中在某个序列适当位置插入空位,使比对结果分值更高。序列比对的过程就是利用一定策略,确定何处插入空位、插入几个空位。一般说来,当两个序列进行比对时,匹配分值为正、错配分值为负,而插入空位时,分值通常为负,其大小高于错配分值。实际比对时,程序通常给定默认值,而用户可以根据具体情况进行调整。空位罚分大小设置通常采用经验值,起始空位罚分较大,而延伸空位罚分较小。所谓起始空位,是指插入的第一个空位,而延伸空位则是当插入多个空位时,第二个空位开始的其它空位。
2.5 灵敏度和特异度
判断数据库相似性搜索结果优劣的指标通常有两个,即灵敏度(Sensitivity)和特异度(Specificity)。所谓灵敏度,是指从数据库中搜索到目标序列的多少;所谓特异度,是指搜索到的目标序列与查询序列是否具有较高相似性。理想状况当然希望搜索结果既有较高的灵敏度,也有较好的特异度。用通俗的话来说,就是“该找的都找到,找到的都是要找的”。但一般说来,“鱼和熊掌不可兼得”,这两个指标很难兼顾。在实际操作中,往往顾此失彼,当调节参数使搜索结果灵敏度提高时,往往会增加假阳性(False positive),找到的并非该找的真实结果;反之,当搜索结果特异度提高时,就有可能增加假阴性(False negative),该找的没找出来。数据库搜索过程中,通过适当选择参数,可以提高搜索结果的灵敏度或特异度。例如,采用不同的搜索程序、选择不同的数据库、限定不同的物种范围、设置不同的种子序列词长、改变计分矩阵和空位罚分,都有可能改变搜索结果的灵敏度和特异度。究竟如何设置参数,需要根据具体问题和研究目的,进行实际调整,切忌生搬硬套。
3 BLAST数据库搜索基本策略
BLAST数据库搜索采用启发式算法以提高搜索速度,该算法的基本策略是找出序列中的保守片段并向两侧延伸。例如,人的血红蛋白有9种不同类型,以alpha珠蛋白(UniProt数据库序列条目名HBA_HUMAN,简称HBA)为例,BLAST搜索基本思路如下。
HBA长度为142个氨基酸残基,beta珠蛋白(UniProt数据库序列条目名HBB_HUMAN,简称HBB)长度为147个氨基酸残基。序列比对结果表明,HBA和HBB相似性约为60%,其中相同位点约占43%,其余为相似位点,即侧链基团性质相似的氨基酸,如丝氨酸S和苏氨酸T的侧链均含羟基、谷氨酸E和天冬氨酸D均带负电,等等。进一步分析可以发现,HBA和HBB之间有多个相似片段,零零星星地分布在不同区域,长度从3到5个氨基酸不等(图1)。
这些相似性片段分为两类。一类为非完全匹配,图中用下划线体表示,如LSP/LTP,DKT/EKS,共6个;另一类为完全匹配,用下划线加粗体表示,如WGKV,HGKKV,共5个。BLAST搜索的核心思想,就是要从数据库成千上万序列中,快速找出与查询序列HBA具有高分匹配的相似片段,向两边延伸得到高分对,并通过统计检验决定取舍,具体步骤如下。

图1人源血红蛋白HBA和HBB序列比对
Fig.1Sequence alignment between human hemoglobin HBA and HBB
3.1 分割种子串
根据上述寻找相似性片段的基本思路,首先按给定字长(Word size),将查询序列分割成一定长度的字符串,通常称种子串(Seed string)。如字长为3,则称三字串。以HBA为例,第1个三字串为第1到3位三个残基,即MVL;第2个三字串为第2到4位三个残基,即VLS;以此类推,一共可分割成140个三字串;其中第1-10位共10个残基,可分割成8个三字串。
3.2 确定近邻串
按上述方法分割查询序列得到种子串后,下一步则需要确定与种子序列相似性较高的其它三字串,称近邻串(Neighborhood words)。也就是说,BLAST在进行数据库搜索时,不仅需要找出与种子串完全匹配的三字串,而且也要找出与种子串具有较高相似性的近邻串。
如何确定近邻串,需要两个条件,第一个是基于计分矩阵计算匹配分值。以HBA第一个三字串MVL为例,基于BLOSUM62计分矩阵,M/M的匹配分值为5,V/V和L/L的匹配分值均为4,MVL三字串的总匹配分值为13。若以第7个三字串DKT为例,D/D的匹配分值为6,K/K和T/T的匹配分值各为5,DKT总匹配分值为16(表2)
表2HBA_HUMAN第1-10位序列片段种子串和近邻串
Table2Seeds and neighborhood words of the first 10 residues of HBA_HUMAN

我们知道,蛋白质序列由20种氨基酸组成,长度为3的三字串共203(8 000)个,其中哪些为近邻串,取决于计分阈值T(Threshold)。也就是说,任意三个氨基酸组成的三字串,按BLOSUM62计分矩阵,若匹配分值高于阈值11,则该三字串即为近邻串,如表2中第1个三字串的近邻串有3个,按分值高低排列分别为MIL,MVI和MVM;第7个三字串DKT的近邻串共11个,前5个为DRT,DET, DKS, DQT和EKT。综上所述,只要选定计分矩阵,确定计分阈值,就可找出查询序列中所有种子串及其近邻串,存放于散列表中(也称哈希表)。阈值越低,近邻串越多。从表2可以看出,不同种子串的近邻串数目不同,平均约50个;长度250 AA的蛋白质序列,所有近邻串的总数约50×250=12 500个。
3.3 搜索高分对
获得种子串和近邻串后,可构建一个搜索列表,逐个搜索数据库中每个序列,确定它们在每个序列中的位置。以HBA为例,长度为3的种子串共140个,而近邻串则有几千个。为便于叙述,我们把种子串和近邻串统称为搜索串。
如何在数据库中扫描搜索串,可以归结为一个传统算法,即如何在一个字符序列中搜索某个子串,可以理解为如何确定某个单词在一篇文章中出现的次数和位置。这种寻找长串中某个短串的经典算法有两种,即索引法(Indexing method)和有限状态自动机法(Finite state machine)。索引法的基本思路可简述如下。以搜索串长度W = 3的蛋白质序列为例,构建一个数组,共含203(8 000)个数组元素。对此数组建立索引,则可确定每个搜索串在数组中的位置。
3.4 延伸高分对
通过上述分割种子串、确定近邻串和定位高分对(High scoring pair,HSP),逐个扫描数据库中每一个序列,找到与查询序列具有高分匹配的高分对及其在序列中的位置。接下来则是将找到的高分对向两侧延伸,以增加高分对的长度。延伸的方法分为无空位延伸和有空位延伸两种。1990年,BLAST最初发表时,采用无空位延伸[4]。例如,查询序列中种子串PQG和数据库某个序列中的近邻串PEG为初始高分对,BLOSUM62分值为7+2+6=15(图2),图中粗体表示。所谓无空位延伸,是计算初始高分对上下游每个位点匹配分值,只要分值大于零(图中下划线表示),则继续延伸,直到分值为负,延伸得到的高分对为LDPQGLS。

图2无空位延伸示例
Fig.2Demonstration of un-gapped extension of high scoring pair
研究表明,上述搜索过程中最耗时的为高分对延伸。为此,1997年发表的新版BLAST作了较大改进,高分对延伸方法也与旧版不同[5]。新版BLAST在寻找高分三字串时,将阈值从13降为11。降低匹配阈值后,所得三字串更多,数据库扫描时低分匹配也更多,按理说,高分对延伸耗时也更多。基于分子生物学和分子演化的基本知识,并对大量的实例进行系统分析,发现具有较高相似性的同源序列,其高分对主要分布在主对角线上,主对角线两侧的短线段为随机匹配,有时也称为噪声。以HBA和HBB为例,若字长为3,所有高分对可用点阵图表示(图3)。图中X轴为HBA,Y轴为HBB,所用计分矩阵为BLOSUM62,滑动窗口即搜索串长度为3,近邻串阈值为11。
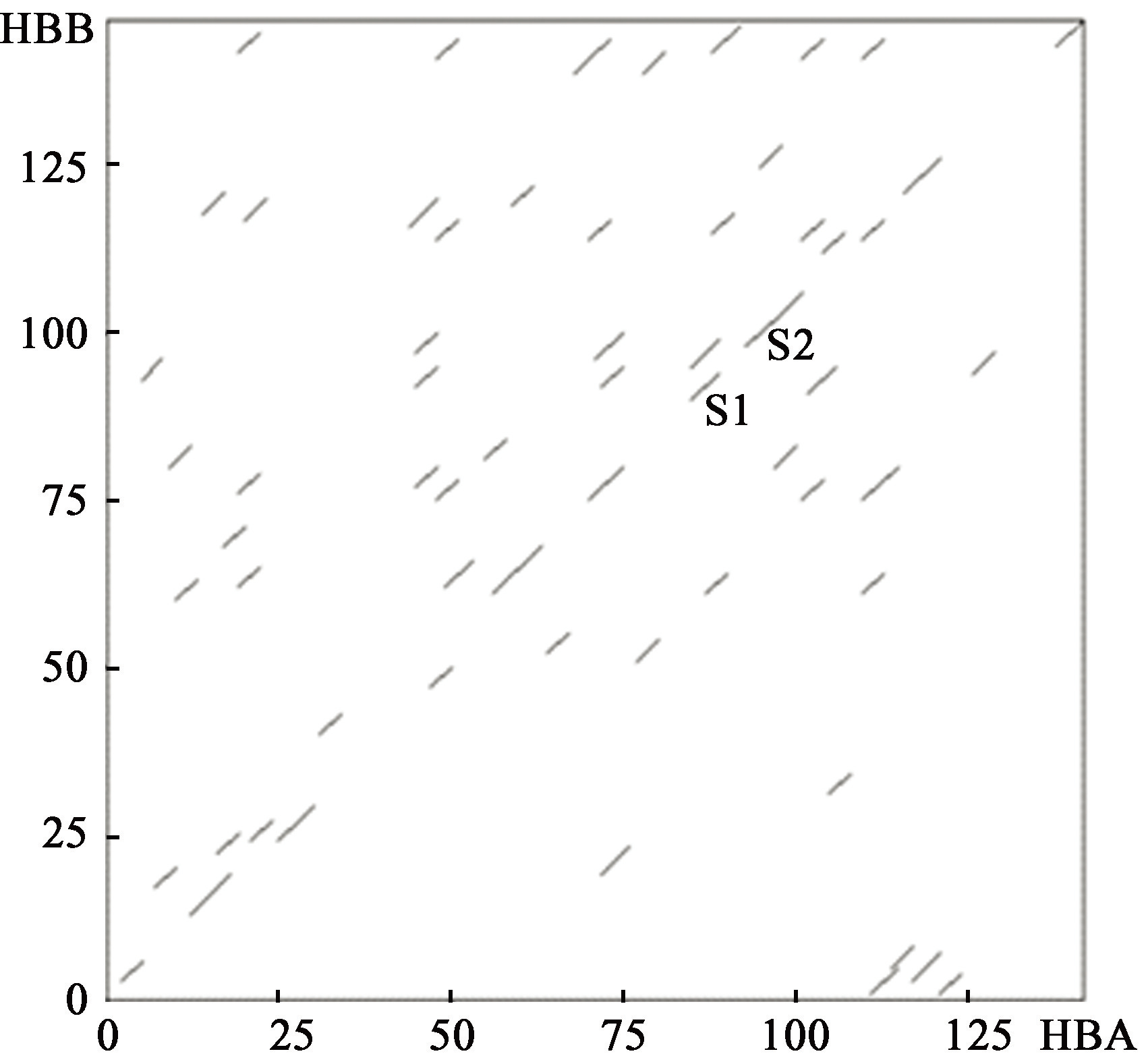
图3人血红蛋白HBA和HBB序列比对点阵图
Fig.3Dot plot between HBA and HBB
为此,新版BLAST首先将距离较近的字串合并,获得较长匹配片段。判别能否合并的标准有两个,第一个为两个被合并的字串必须处于同一方向,第二个则要求它们之间的距离不超过某个设定的范围。满足上述两个条件的区域一般只占很少一部分,绝大部分分布在主对角线上。也就是说,新版BLAST并非将所有找到的高分对都进行延伸,而是仅延伸很少一部分高分对,如图中S1和S2。由于这种策略的核心是寻找两个连续的三字串,有必要降低阈值T以保证不降低灵敏度。经过延伸得到较长高分对后,再用Smith-Waterman算法对查询序列和目标序列进行基于动态规划的局部比对,并计算比对结果的期望值。
3.5 计算期望值
所谓比对结果的期望值(Expected value,E),是指通过统计检验的方法,给出比对结果的可信度;换言之,对于某个查询序列,数据库中搜索到的每个目标序列,都有一个期望值E。
期望值E可以用公式E = kmN / eλS计算。这里,m为查询序列长度,N为数据库中所有序列长度总和,e为自然对数底2.718,S为归一化后的比对分值,K和λ为常数,其大小与计分矩阵有关。显然,E值大小与查询序列长度和数据库大小有关(表3)。
表3BLAST搜索结果期望值E含义
Table3Indication of the expected value E from BLAST search results

E值与统计检验显著性值P之间的关系可用公式P = 1-e-E表示,和统计显著性P = 0.05对应的E值为0.051,也就是说,当期望值E小于0.05时,搜索结果具有显著可靠性;E值越小,显著性也越高。期望值的计算方法涉及许多统计学基本概念,此处不予赘述。
以上我们简要介绍了BLAST数据库搜索的基本步骤。需要说明的是,为便于初学者理解,本文介绍的步骤对BLAST搜索进行了简化。美国亚利桑那大学David Mount教授编著的《生物信息学:序列和基因组分析》(Bioinformatics: Sequence and Genome Analysis)一书中详细介绍了BLAST搜索全过程[6-7]。
4 BLAST主要参数
分析BLAST搜索基本步骤可以发现,种子序列长度、计分矩阵和期望值等参数,直接影响搜索结果。实际进行BLAST搜索时,可以先使用程序给定的默认参数,在对搜索结果进行初步分析后,根据实际情况调整有关参数。下面,我们以NCBI提供的BLAST分析平台为例,介绍几个常用参数的含义。
4.1 字长
分析上述BLAST搜索步骤可以发现,第一步分割种子串,种子串的大小与搜索速度和灵敏度有关。种子串长度越小,灵敏度越高,搜索速度越慢。
用BLASTP搜索蛋白质序列数据库或用BLASTX等搜索核酸序列数据库翻译得到的蛋白质序列时,默认字长为3。若希望提高搜索敏感性,可将其改为2。用BLASTN搜索核酸序列数据库时,默认值为11,可选值为7或15;若希望提高搜索特异度,可选择MegaBLAST搜索高度相似的序列,此时默认字长为28,可选范围从16到256。字长值越大,搜索速度越快,特异度越高,但敏感性下降。
4.2 计分矩阵
NCBI基于浏览器的BLAST系统用于蛋白质序列比对的计分矩阵包括PAM系列和BLOSUM系列,默认矩阵为BLOSUM62,可选矩阵为BLOSUM90/80/50/45和PAM30/70/250。若需要提高搜索灵敏度,可选择BLOSUM45或PAM250;反之,若需要提高搜索的特异度,可选择BLOSUM90或PAM30。仔细分析BLOSUM62计分矩阵发现,20种不同氨基酸的匹配分值差别很大,尤其是主对角线上的自我匹配分值,如最高值色氨酸W为11,而丙氨酸A、亮氨酸L等5个氨基酸的自我匹配分值仅为4。这就决定了含有不同氨基酸的种子串,其近邻串的数目相差也很大。
NCBI基于浏览器的BLAST系统用于核酸序列计分矩阵按匹配(Match)和错配(Mismatch)计分,共有6种匹配/错配可选方式,即1/-1,1/-2,1/-3,1/-4,2/-3,4/-5。用MegaBLAST搜索相似性较高的序列时,默认计分为1/-2,即匹配时得1分,不匹配时得-2分。而用常规BLASTN搜索相似性较低的序列时,默认计分为2/-3,即匹配时得2分,不匹配时得-3分。若需要提高灵敏度,可选择4/-5;若需要提高特异度,则可选择1/-1。
4.3 空位罚分
空位罚分是指序列比对过程中为达到最佳匹配,插入一个或连续几个空位。空位的生物学意义可理解为,来自同一祖先的两条同源序列在演化过程中,其中一条在某些部位有单个或多个碱基的插入或删除。由于空位插入或删除的生物学机制十分复杂,目前尚无恰当的理论模型。因此,空位插入罚分选择只能依赖经验。用于蛋白质序列数据库搜索的默认空位罚分值为起始罚分11,延伸罚分1;起始罚分可选范围为7~12,延伸罚分可选范围为1和2。用于核酸序列数据库搜索的起始空位罚分为5(可选0~6),延伸罚分为2(可选2~4)。用于高度相似性核酸序列数据库搜索MegaBLASTN默认空位罚分采用线性模型,即起始和延伸罚分均为1。
4.4 可信度值
我们知道,相似性分值是用来度量序列比对相似性的重要指标,相似性分值越高,两者的相似性越大。然而,由于所用计分矩阵不同、比对结果匹配序列长度不同、数据库大小不同等诸多因素,用相似性分值评判数据库搜索结果具有一定局限性。为此,BLAST采用期望作为评判搜索结果可靠性指标。E值由公式E = m×n×P计算得到,其中,m表示数据库中所有残基总数,n表示查询序列残基数,P表示随机匹配概率。显然,E值大小不仅与随机匹配概率有关,也和查询序列长度特别是数据库大小有关。E值越低,说明随机匹配的可能性越小,所得结果越具统计显著性。NCBI BLAST服务器默认E值为0.05,若要减少输出结果的假阳性,可降低E值(表3)。
4.5 低复杂度区域屏蔽
所谓低复杂度(Low complexity)序列,使指核酸或蛋白质序列中的重复区域,如基因组序列中的Alu序列、mRNA序列中的多聚腺苷酸、蛋白质序列中简单重复序列等[5]。为避免低复杂度重复序列对搜索结果的影响,可以对查询序列中的低复杂度区域加以过滤(Filter)或屏蔽(Mask)。搜索核酸序列数据库特别是含非编码序列的基因组数据库时,通常可以选择重复序列屏蔽功能。程序根据查询序列复杂度特征,自动过滤复杂度片段,输出结果中以浅色字体显示。而当搜索蛋白质序列数据库,一般不必选择重复序列屏蔽功能。程序对查询序列进行判断,确定需要过滤的区域;用户也可设定需要过滤的序列片段,即在检索序列输入框中把需要过滤的残基改成小写字母。
5 BLAST程序
以上我们以血红蛋白为例,简单介绍了BLAST搜索蛋白质序列数据库的主要步骤,所用程序为BLASTP(P表示蛋白质Protein)。BLAST也可用于核酸序列数据库搜索,即以核酸序列为查询序列,搜索核酸序列数据库,所用程序为BLASTN。此外,BLAST系统还提供了另外两个程序:BLASTX和TBLASTN。前者将查询序列核苷酸翻译成氨基酸,搜索蛋白质序列数据库;后者将核酸序列数据库中的蛋白质编码序列翻译成氨基酸,以蛋白质序列为查询序列,搜索翻译所得氨基酸序列(图4)。
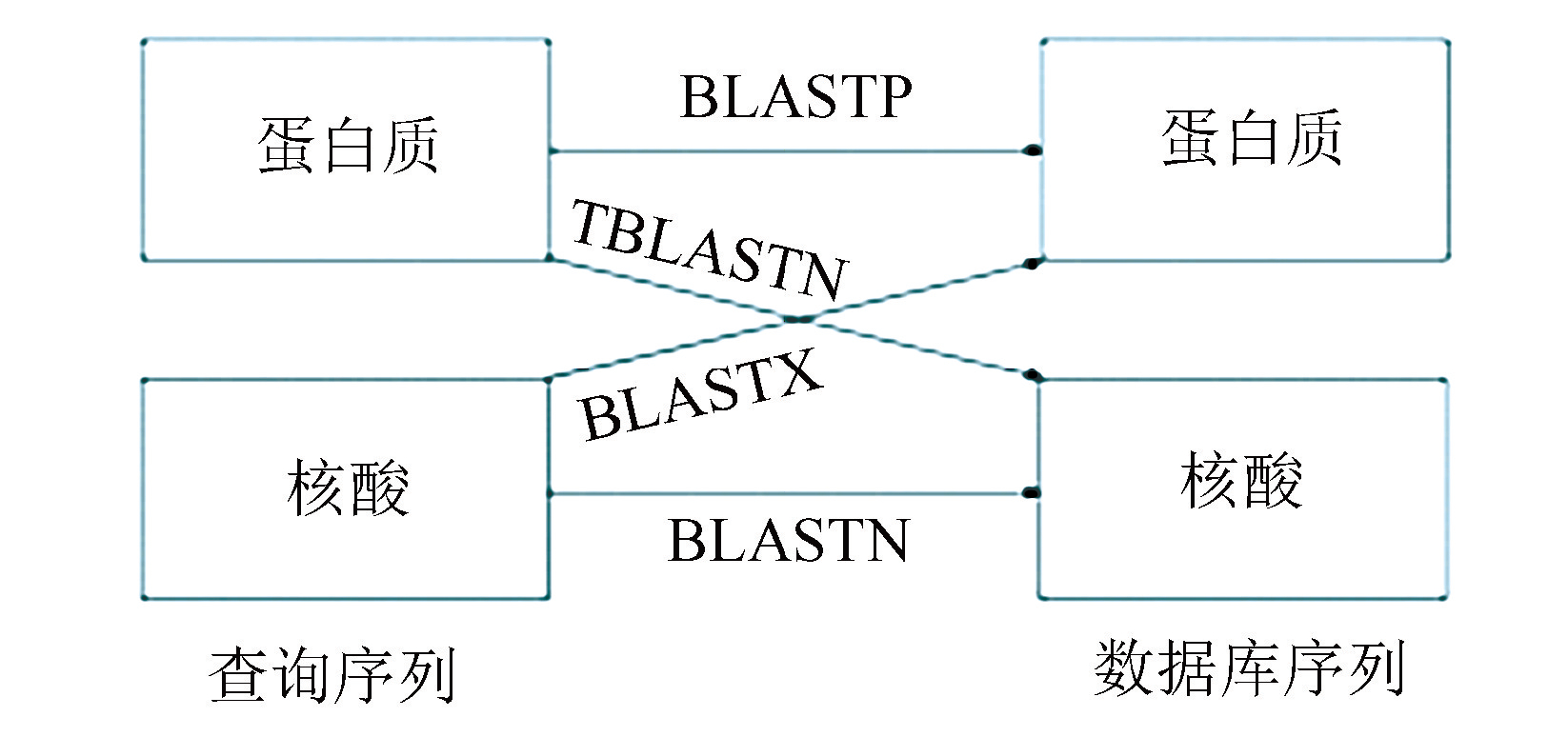
图4BLAST通用程序
Fig.4The BLAST general programs
5.1 BLASTP
蛋白质序列搜索经常用于寻找同源序列,以推断查询序列的功能和演化。例如,以人的血红蛋白alpha亚基HBA_HUMAN作为查询序列,搜索黑猩猩(Chimpanzee)、恒河猴(Rhesus monkey)等灵长类动物中的同源序列。结果表明,已经人工审阅的UniProt数据库Swiss-Prot子库中收录了36个灵长类alpha血红蛋白,与HBA_HUMAN的序列相似性均高于85%。
BLASTP也经常用于搜索查询序列中的保守结构域、重复序列片段、序列模体(Motif)在数据库序列中的相似序列。例如,以金鱼草(Antirrhinum majus)转录因子(Squamosa promotor binding protein 1,SBP1)SBP1_ANTMA第52~125位DNA结合结构域为查询序列搜索粳稻(Oryza sativa japonica)中SBP家族转录因子,共搜索到19个SBP转录因子,其全长序列长度从216 AA到1 140 AA不等,均含长度为74 AA的DNA结合结构域,与SBP1_ANTMA的序列相似性高达55%~75%,且都包含两个高度保守的锌指(Zinc finger)序列模体C3H/C和C2HC,均含两个由碱性氨基酸精氨酸R或赖氨酸K组成的核定位信号。
为提高搜索灵敏度,可采用位点特异性迭代BLAST,即Position-specific iterated BLAST,PSI-BLAST。运行过程如下:
1)按程序提供或用户选择的通用相似性计分矩阵(如BLOSUM62)进行第一轮搜索;
2)根据第一轮搜索结果所得高分匹配序列,构建位置特异性计分矩阵(Position specific scoring matrix,PSSM);
3)用所构建的PSSM计分矩阵取代通用计分矩阵进行第二次搜索,找出新的匹配;
4)根据第二次搜索结果调整PSSM计分矩阵,用于第三次搜索,找出新的匹配;
5)依此类推,直到搜索结果中没有新的匹配,或满足预定要求。
一般说来,利用PSI-BLAST进行多次迭代搜索,可以提高搜索敏感度和特异度。当然,由于进行多次搜索,所用时间增加。此外,若第一轮搜索所得结果有误,则所构建的PSSM计分矩阵也会有错,以后的多次迭代只能越走越远。
5.2 BLASTN
BLASTN用于核酸序列搜索,即用核酸序列作为查询序列,搜索核酸序列数据库;搜索步骤和BLASTP基本相同,只是种子串长度、计分矩阵和空位罚分等参数不同。核酸序列可分为两大类,一类为蛋白质编码序列,另一类为非编码序列。BLASTN多用于非编码序列的数据库搜索,包括rRNA,tRNA,microRNA,lncRNA等。例如,原核生物核糖体小亚基16S RNA长度约1 500 nt,由16S rDNA基因编码。不同物种中的16S rDNA和rRNA比较保守,可用于物种鉴定。
若查询序列为蛋白质编码序列,且已经知道编码区和起始密码子,则可将其翻译成蛋白质序列,再用BLASTP搜索蛋白质序列数据库。由于蛋白质序列由二十种不同氨基酸组成,而核酸序列由四种不同核苷酸组成,一般情况下,BLASTP搜索结果的特异度较高,假阳性率较低。
5.3 BLASTX
BLASTX用于以核酸序列为查询序列,搜索蛋白质序列数据库。查询序列为蛋白质编码序列,如转录组测序(RNA-Seq)或表达序列标签(Expressed sequence tag,EST)测序所得结果。考虑到有时不能判断编码区位于查询序列正链或反链,也无法确定起始编码位置,BLASTX自动按6条读码框将查询序列翻译成蛋白质序列,逐条进行搜索,并按翻译后所得蛋白质序列与目标序列相似性匹配给出结果。
近年来,随着高通量测序技术的普及,转录组测序已成为基因组学研究的常规方法。利用BLASTX,可以鉴定转录组测序数据中表型相关候选基因。通常,转录组测序结果提供某个样本在一定条件下高表达蛋白质编码基因序列。利用BLASTX,搜索已知功能蛋白质序列数据库,例如UniProt中人工审阅的蛋白质序列功能关系子库Swiss-Prot,查看搜索结果注释信息,可以推断某个高表达基因是否具有预期的功能。
5.4 TBLASTN
与BLASTX相反,TBLASTN以蛋白质为查询序列,搜索核酸序列数据库,如NCBI核酸参考序列数据库(Reference RNA,RefSeq_RNA)。也就是说,TBLASTN以蛋白质序列为查询序列,搜索核酸序列数据库中的核酸序列翻译所得的蛋白质序列,实际上还是蛋白质序列数据库搜索。仍以转录组测序为例,从上述Swiss-Prot蛋白质序列功能关系子库中选择某个已知功能的序列,利用TBLASTN搜索转录组测序结果中某个样本的高表达基因核酸序列数据库,分析搜索结果,用来判断所得到高表达基因中是否包含预期的功能基因。
除了上述四个程序外,BLAST系统还提供TBLASTX,将搜索序列核苷酸按六条读码框翻译成氨基酸,搜索核酸序列数据库,把数据库中的序列也按六条读码框翻译成蛋白质序列,一共进行36次搜索。显然,当数据库容量很大时,TBLASTX计算量极大,不建议使用。
5.5 专用BLAST程序
除上述BLASTP,BLASTN,BLASTX和TBLASTN四个通用程序外,NCBI BLAST网站还提供了8个专用BLAST程序(表4)。
表4BLAST专用程序
Table4The BLAST Programs with Specialized Function

表4所列8个专用程序中,SmartBLAST搜索代表性数据库(Landmark database)中参考基因组蛋白质序列,包括人、小鼠、斑马鱼、果蝇、线虫、酵母等真核生物和大肠杆菌、枯草杆菌等原核生物,结果列出5个最好匹配。
Primer-BLAST是用于引物设计的BLAST搜索程序[8]。程序运行时,首先根据用户输入的序列进行引物设计;并以所设计的引物作为查询序列搜索用户指定的数据库。程序对搜索结果自动进行分析,滤除目标序列中相似性较高的匹配,以增强所设计的引物专一性。
Global Align采用Needleman-Wunsch整体比对策略进行双序列比对。
CD-search用于搜索保守结构域数据库(Conserved domain database),找出相似性较高的序列。
VecScreen可用来找出搜索序列中是否存在测序过程中引入的载体序列。
IgBLAST是专门用于免疫球蛋白(Immunoglobulin,简称Ig)的BLAST数据库搜索程序,主要用来分析免疫球蛋白可变区(Variable region)的基因序列[9]。IgBLAST输出结果中,除包括常规的目标序列登录号、简介、分值和E值外,还分别给出V,D和J三种免疫球蛋白基因的匹配情况,同时对序列进行简单注释,包括序列中三个骨架区域(Framework regions)。
CDART是Conserved Domain Architecture的缩写,其用途是基于保守结构域数据库,找出搜索序列中所有特定的结构。
专用程序中还包括多序列比对。
6 后语
6.1 BLAST主要用途
BLAST数据库搜索的实际应用包括许多方面。例如,对于新测定的或功能未知的核酸序列,通过数据库搜索,可获得数据库中已经收录的目标序列。根据相似性程度,推断该未知序列与目标序列是否为同源序列,并进一步根据目标序列的注释信息,推测该未知序列可能属于哪个基因家族,具有哪些生物学功能。对于蛋白质序列,通过搜索已知三维结构的蛋白质序列数据库,有可能推测未知序列的空间结构。有时,数据库搜索所得结果,只是查询序列中一个或几个片段与目标序列具有较好匹配,表明它们有可能是一个序列模体或结构域。数据库搜索还可用来找出某个基因在其它物种中的直系同源基因(Orthologs),或同一物种中某个基因家族的并系同源基因(Paralogs)。当然,数据库搜索所得结果,只能用作参考。如果查询序列和目标序列的相似性程度很低,还必须通过其它方法或实验手段才能确定其是否属于同一基因家族。此外,在进行PCR扩增设计引物(Primer)时,可以通过数据库搜索,检查所设计的引物是否具有较好的特异度。
6.2 其它BLAST网站
1988年NCBI成立时,生物医学文献数据库Medline、核酸序列数据库GenBank和数据库搜索程序BLAST是“三大法宝”。BLAST之所以使用广泛,除了因为其运行速度快,可免费下载安装外,主要原因NCBI开发团队不断对该系统改进,增加新的功能,改善用户界面,目前已经成为国际上最为常用的序列相似性数据库搜索工具。用户可以直接访问NCBI的BLAST服务器,也可从NCBI免费下载BLAST软件包,自行安装并在本地运行。
除NCBI外,许多国际生物信息和基因组测序中心也提供BLAST数据库搜索服务。这些BLAST网站往往具有自己的特色。例如,欧洲生物信息学研究所(European Bioinformatics Institute,EBI)的ENSEMBL基因组数据库系统(http://www.ensembl.org),加州大学圣克鲁兹分校(Santa Cruz)的基因组浏览器(http://genome.ucsc.edu)都整合了BLAST。EBI和瑞士生物信息研究所合作开发和维护的UniProt蛋白质序列数据库(https://www.uniprot.org),也提供BLAST程序,用户可以对检索到的蛋白质序列,直接进行BLASTP蛋白质序列数据库搜索,并对搜索结果按不同分类学谱系进行过滤。美国能源部资助的联合基因组研究所构建和维护的植物基因组信息网站Phytozome(https://phytozome-next.jgi.doe.gov)整合的BLAST序列相似性数据库搜索工具,可用来搜索植物基因组、蛋白组序列。
中国科学院北京基因组研究所(国家生物信息中心)安装的BLAST程序(https://ngdc.cncb.ac.cn/blast/home)是目前国内数据库最多、数据量最大、更新最及时、操作最方便的BLAST平台,其新冠病毒基因组和蛋白组特色数据库为抗击冠疫情发挥了很大作用,北京大学生物信息中心构建的植物转录因子数据库PlantTFDB(https://planttfdb.gao-lab.org)和华中科技大学构建的动物转录因子数据库(https://guolab.wchscu.cn/AnimalTFDB4)也整合了BLAST,可搜索多种植物和动物转录因子序列。
6.3 其它数据库搜索程序
除BLAST外,数据库搜索程序还包括FASTA、BLAT、HMMER等。
FASTA于二十世纪八十年代就开始用于核酸和蛋白质序列数据库搜索,BLAST是在FASTA基础上开发的[10]。EBI数据库搜索网站(https://www.ebi.ac.uk/jdispatcher/sss)部署了FASTA系列程序。需要说明的是,FASTA通常是指序列格式,而此处所说的FASTA是指数据库搜索程序。
BLAT是BLAST-Like Alignment Tool的缩写,是在BLAST基础上,调整种子序列字长,以提高搜索速度,特别适用于搜索核酸序列中相似性较高的同源序列。EBI的ENSEMBL基因组数据库系统和加州大学的基因组浏览器(http://genome.ucsc.edu)都整合了BLAT。
和以上几种方法完全不同,HMMER(http://hmmer.org)采用隐马氏模型方法(Hidden Marcov Model),构建序列谱(Sequence profile)数据库,用于蛋白质序列数据库搜索。读者可以使用EBI安装的HMMER系统进行搜索(https://www.ebi.ac.uk/Tools/hmmer)。
6.4 结束语
以上我们简单介绍了序列相似性数据库搜索系统BLAST,限于篇幅,本文没有给出具体实例。读者在基本理解BLAST搜索策略的基础上,进行实际操作(https://blast.ncbi.nlm.nih.gov/Blast.cgi),才能熟练使用、深入理解。
笔者自2001年起,在北京大学生命科学学院和在中国农业科学院研究生院开设“实用生物信息技术”研究生课,BLAST是主要内容之一。课上除简单介绍BLAST基本概念和搜索策略外,多以实例进行演示。选修本课程的同学结合自己课题、带着问题进行实际操作,很快就能学会BLAST的使用。
致谢
感谢北京生信助力科技有限公司颜林林博士对本文初稿的修改意见,感谢复旦大学曹志伟教授无私分享她的BLAST教学课件,感谢中国科学院北京基因组研究所(国家生物信息中心)马英克博士安装维护该所BLAST系统,感谢中国科学院北京基因组研究所(国家生物信息中心)降帅博士多次在“实用生物信息技术”课上介绍BLAST。
