摘要
通过分析来自GEO数据库的儿童脓毒症转录组数据,探究了未折叠蛋白反应在该疾病发生机制中的作用。首先利用随机森林和支持向量机算法筛选了诊断模型的候选基因包括EXOSC4,EIF2AK3,CEBPB,WIPI1,EXOSC6,EXTL2和SRPRB,使用多因素逻辑回归构建诊断模型,并利用外部数据集对其进行了验证。接着,分析了这些基因与免疫细胞浸润的相关性,发现它们与中性粒细胞的浸润具有强相关性。此外,通过一致性聚类将儿童脓毒症患者分成了三个亚型,比较了它们在临床特征和炎症因子表达方面的差异。最后,通过加权基因共表达网络分析筛选出每个亚型的核心基因,并发现这三个亚型在免疫系统、代谢和细胞死亡等生物学过程上存在显著差异。药物预测结果显示不同亚型的患者可能对不同种类的药物具有不同的敏感性。总之,这项研究为儿童脓毒症的诊断和精准治疗提供了新的思路。
Abstract
By analyzing transcriptomic data of pediatric sepsis from the GEO database, the role of unfolded protein response in the pathogenesis of this disease was investigated. First, candidate genes for the diagnostic model were screened by using random forest and support vector machine recursive feature elimination algorithms, including EXOSC4, EIF2AK3, CEBPB, WIPI1, EXOSC6, EXTL2 and SRPRB. A diagnostic model was constructed by multiple logistic regression and validated with three external datasets. Next, the correlation between these genes and immune cell infiltration was analyzed, revealing a strong correlation with neutrophil infiltration. Furthermore, patients with pediatric sepsis were divided into three subtypes by consensus clustering, and their differences in clinical features and expression of inflammatory factors were compared. Finally, core genes for each subtype were selected through weighted gene co-expression network analysis, and significant differences were found among these three subtypes in biological processes such as the immune system, metabolism, and cell death. Drug prediction results showed that patients with different subtypes may have different sensitivities to different types of drugs. In summary, this study provides new ideas for the diagnosis and precision treatment of pediatric sepsis.
脓毒症是一种危及生命的多器官功能障碍综合征,其发生是由于宿主对感染反应失调所引起[1]。根据医学杂志《柳叶刀》2020年发布的研究,全球每年大约有五千万人患上脓毒症,而这种严重感染导致的死亡率超过了五分之一[2]。世界卫生组织(WHO)指出,脓毒症已成为全球范围内主要的公共卫生问题,尤其在儿童中更为突出[3]。此前有研究表明,中国脓毒症和感染性休克的发生率远高于北美和欧洲国家[4],且超过20%发生在10岁以下的儿童中[5]。而目前儿童重症脓毒症和脓毒症休克的治疗仍然是一个挑战[6],迫切需要寻找新的诊断和治疗方法来降低死亡率。因此,有必要深入探索它的生物学机制和潜在生物标志物,为早期诊断和个体化治疗提供更好的支持。
内质网(Endoplasmic reticulum,ER)是蛋白质合成、折叠和分泌的主要部位,在细胞生命活动中扮演着重要的角色。内质网应激(Endoplasmic reticulum stress,ER stress)是由于内源或外源因素导致蛋白质错误折叠或者未折叠,使得这些蛋白质在内质网积聚[7]。这种积聚从而激活未折叠蛋白反应(Unfolded protein response,UPR)通路,UPR又会激发一系列转录和翻译事件,以恢复内质网的稳定状态[8]。已有大量研究表明,UPR在脓毒症的发病机制中发挥着重要的作用[9],比如该通路基因的表达和脓毒症患者器官衰竭和内皮功能障碍的发生相关[10]。还有一些未折叠蛋白反应相关的基因已经被证实和脓毒症相关[11]。然而,未折叠蛋白反应相关的基因在儿童脓毒症的发生发展中的作用尚不清楚。
本研究旨在通过对健康个体和儿童脓毒症外周血转录组数据的系统分析,深入了解儿童脓毒症与未折叠蛋白反应通路的关系,并利用机器学习算法构建基于未折叠蛋白反应相关基因(UPRGs)的儿童脓毒症预测模型。我们采用受试者工作特征(ROC)曲线和外部数据集来评估这个模型的性能。此外,我们还通过一致性聚类分析将儿童脓毒症划分为三种亚型,对每种亚型进行了生物学功能和特异性通路的比较,并分析了它们在临床信息上的差异。最后我们采用加权基因共表达网络分析(WGCNA)获得枢纽基因,并利用CMAP数据库识别靶向每种亚型的潜在小分子药物。
1 数据和方法
1.1 数据来源与清洗
脓毒症的外周血转录组数据(GSE26378,GSE26440,GSE13904,GSE57065,GSE69063)均来源于GEO数据库(https://www.ncbi.nlm.nih.gov/geo)。其中前三个数据集为儿童脓毒症的,后两个数据集为成人的。使用了“affy”包来读取原始数据,利用rma算法对数据进行了背景校正和归一化处理,将数据集合并后进行了去批次处理。94个UPRGs从GeneCards数据库(https://www.genecards.org)中下载并使用。
1.2 差异分析和相关性分析
利用“GSVA”包对儿童脓毒症的实验组(SEP)和对照组(CT)中涉及到UPR通路相关的基因进行ssGSEA评分[12]。同时,使用了“limma”包对两组数据进行基因差异表达分析[13],选取了adj.pValue < 0.01的基因并且与UPRGs取了交集,利用了“pheatmap”包绘制了热图和火山图。
1.3 诊断模型的构建
使用了两种机器学习算法,即支持向量机递归特征消除(SVM-RFE)[14]和随机森林来筛选两个数据集中的关键基因。对于SVM-RFE,使用了“caret”包中的rfe函数,并进行了十折交叉验证。而随机森林则利用了“randomForest”包,并选择了1 000棵决策树以提高模型对训练数据的拟合程度。对这两种算法筛选出的基因交集进行了多因素逻辑回归分析,构建了诊断模型。为了验证模型的可靠性,利用了另外三个数据集进行了外部验证。通过绘制ROC曲线,对验证结果进行了展示。
1.4 免疫浸润分析
基于CIBERSORT和MCPcounter算法,利用“IOBR”包中的deconvo_cibersort方法和deconvo_mcpcounter方法,进行了免疫浸润分析[15]。使用“psych”包对结果进行了斯皮尔曼的相关性分析,并采用FDR(False Discovery Rate)对得到的p值进行了多重比较校正。
1.5 聚类分型
利用“ConsensusClusterPlus”包和UPRGs对SEP患者进行了一致性聚类分析[16]。随后生成整合的基因表达数据和临床信息(例如患者年龄)的热图,并对不同分型的患者进行了差异分析,比较它们在免疫细胞丰度上的差异。
1.6 加权基因共表达网络分析
通过R包 “WGCNA”分析了不同分型的基因表达矩阵[17],分析了每个分型的核心基因,并计算了不同分型和基因模块之间的相关性。采用“FGSEA”对不同分型的核心基因进行富集分析,结果通过“pheatmap”包进行可视化。
1.7 潜在小分子药物的鉴定
筛选出每种亚型特异性的核心基因和差异基因取交集。将交集基因输入到CMAP数据库(https://clue.io)[18],寻找与之相关联的小分子药物。
2 结果
2.1 差异分析
首先对GSE26378和GSE26440两个数据集进行了主成分分析(图1(a)),两个数据集之间存在明显的批次效应,然后利用ComBat函数对两个数据集进行去批次处理(图1(b))。为了探究CT组和SEP组在UPR通路上的差异,对两组进行了基因集变异分析(GSVA),可以看到SEP组的得分显著低于CT组,表明SEP患者UPR通路受到显著抑制(图1(c))。接着对它们进行差异表达分析,总共得到45个UPR相关的差异基因,其中15个基因(如EXOSC4,CEBPB和WIPI1)在SEP组显著上调,30个基因(包括EXOSC6,EIF2AK3和EXTL2)显著下调(图1(d),1(e))。相关性分析结果展示这些基因彼此高度相关 (图1(f)),表明UPR通路中单个基因的变化通常会导致多个基因发生级联反应。
2.2 诊断模型
利用机器学习中支持向量机和随机森林的算法筛选UPRGs,在使用随机森林算法的时候,选择其中30个基因进行进一步分析(图2(a),2(b))。两种算法都对基因进行了重要性排序,对两种方法排名前十的基因进行了进一步分析,鉴定出7个基因,从而构建了诊断模型(图2(c))。接着又计算了训练集的AUC值,值为0.984,最后利用三个数据集进行外部验证,它们的AUC值分别是0.973,0.993和0.998,证明了模型的可靠性(图2(d))。
2.3 免疫浸润
为了探究儿童脓毒症的免疫细胞浸润情况,分别利用CIBERSORT和MCPcounter算法进行计算。通过CIBERSORT计算了22种免疫细胞丰度,然后又计算了它们和模型基因的相关性,结果表明7个基因和CD8 T 细胞、CD4 T细胞和中性粒细胞显著相关(图3(a))。为了进一步验证,我们又利用MCPcounter算法计算了8种免疫细胞的丰度,并计算了它们和模型基因的相关性。同样的,它们也和T细胞还有中性粒细胞浸润显著相关(图3(b))。这些研究表明,这些基因在驱动儿童脓毒症患者的炎症反应中起着关键作用。此外,在模型权重最高的基因EXOSC4和多个免疫细胞群高度相关(图3(c))。

图1UPR通路相关的基因在儿童脓毒症中的表达情况
Fig.1Expression profile of UPR pathway-related genes in pediatric sepsis

图2预测模型的构建和验证
Fig.2Construction and validation of the prediction model
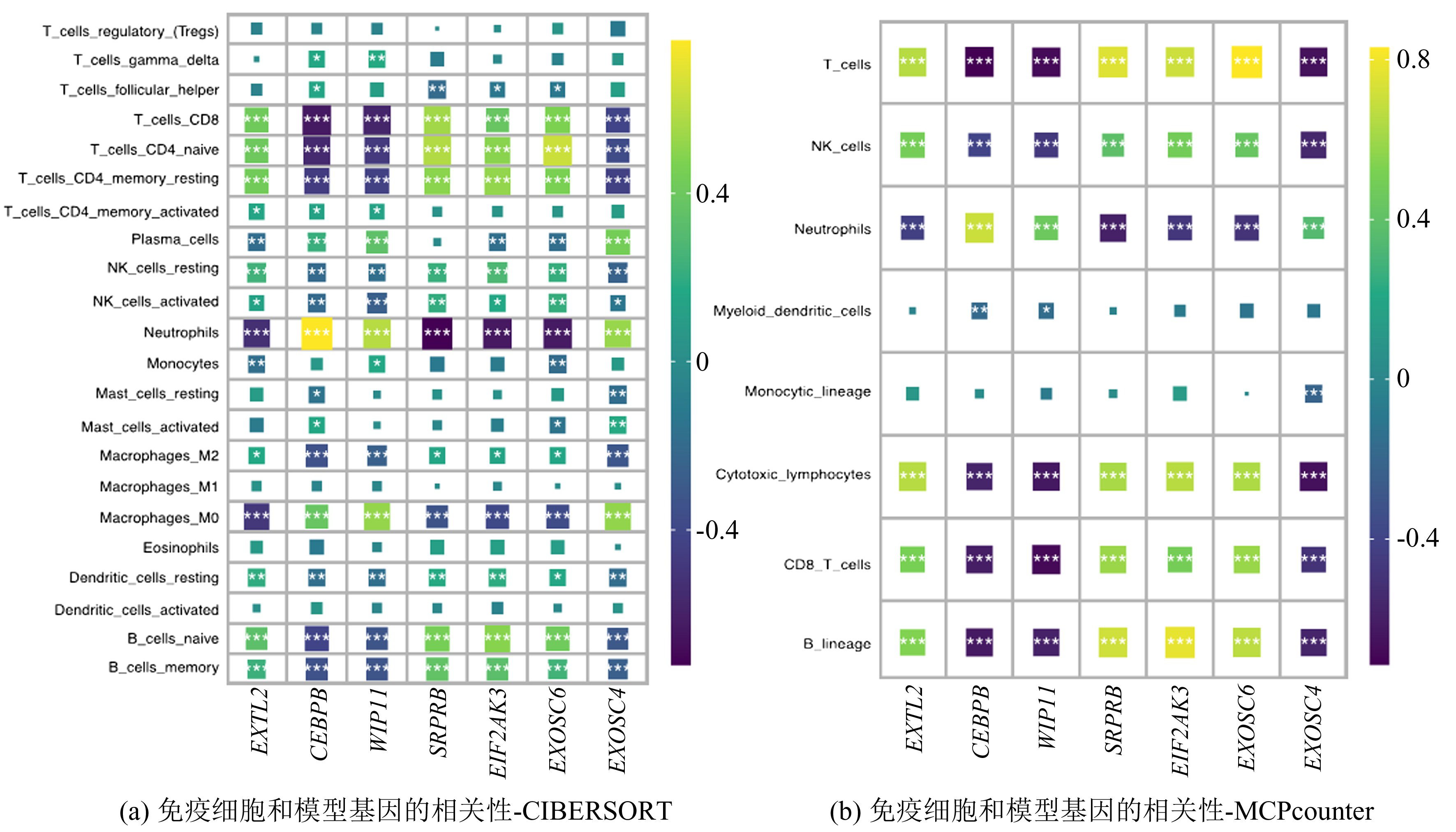

图3免疫细胞浸润分析
Fig.3Analysis of immune cell infiltration
2.4 分型
为了进一步研究儿童脓毒症之间的差异,利用UPRGs对它进行一致性聚类。根据聚类结果,SEP分成三个不同的簇(图4(a))。值得注意的是,UPRGs的表达谱和临床特征在三个簇中存在显著差异(图4(f))。根据临床特征结果,不同簇的年龄分布具有统计学意义(图4(b),4(c))。此外,三种分型免疫细胞浸润和炎症标志物的分泌也存在着显著差异(图4(d),4(e))。通过热图分析比较三组UPR通路相关基因的表达情况,发现WIPI,CEBPB和EXOSC4在第一簇中表达最高,MBTPS2,EXTL2和EXOSC6在第二簇中表达最高,PPP2R5B,GSK3A和CREB3L1在第三簇中表达最高。因此这几个基因可以被认为是每个簇的特征基因。
2.5 加权基因共表达网络分析
WGCNA结果展示不同簇与不同模块之间的相关性并不相同,第一簇和棕色模块的相关性最高,第二簇和青绿色模块最相关,第三簇和蓝色模块最相关(图5(a),5(b))。使用fgsea的进一步分析表明,第一簇内的基因主要参与免疫应答抑制信号转导,白细胞迁移、趋化,中性粒细胞免疫应答等过程。另一方面,第二簇的基因主要与抗原受体介导的信号通路,凝血调节等有关。最后,我们发现第三簇的基因集中在T细胞介导的免疫,细胞黏附等过程中(图5(c))。根据CMAP预测结果,SB-216763,camptothecin和genipin有望作为三种分型的治疗药物(图5(d))。

图4儿童脓毒症患者的不同分型以及它们之间的差异
Fig.4Different clusters of pediatric sepsis patients and their differences
3 讨论
儿童脓毒症是重症监护患者最常见的死亡原因。已有研究表明,UPR通路的紊乱可能对多种病理状态产生影响,包括感染和炎症等[9]。这条通路的激活通常具有益处,但过度或持续的激活可能引发细胞死亡并促进组织损伤[19-22]。目前尚不清楚该通路相关基因在儿童脓毒症中的发生机制中的作用。为了探究这一问题,从GEO数据库中获取了儿童脓毒症患者外周血mRNA表达数据,并对其进行了全面的生物信息学分析。GSVA结果显示,儿童脓毒症患者UPR通路受到了显著抑制。基于这一发现,推测UPRGs的变化可能在儿童脓毒症的发生和发展中起到关键作用。
根据研究,筛选出7个与UPR通路相关的关键基因,包括EXOSC4,EIF2AK3,CEBPB,WIPI1,EXOSC6,EXTL2和SRPRB。其中,CEBPB是一种重要转录因子,参与调节免疫和炎症反应的基因表达[23-25]。它被认为是脓毒症诱导慢加急性肝衰竭(ACLF)的重要介质[26]。此外,研究发现CEBPB在早期胚胎中扮演着关键的角色,可以通过介导1β-HSD1的表达从来促进肺部成熟,以应对胎儿因感染而引起的早产威胁[27]。另外,有研究报道,在肾小球肾炎中,UPR介导的CEBPB优先诱导,能够通过抑制NF-κB来减轻内质网应激的抗炎潜力[28]。WIPI1是WIPI家族的一员,该蛋白家族是参与自噬启动信号PI3P脂质传递的效应分子,并且能够招募其他因子来调控自噬体形成[29]。之前的研究表明,在脓毒症患者中,WIPI1的高表达与较差的生存率相关[30]。EIF2AK3通常称为PERK,是一种跨膜蛋白,响应内质网应激时通过磷酸化eIF2α来减缓翻译过程[31]。它与下游分子激活转录因子4(ATF4)共同组成PERK/ATF4通路,该通路的激活能够抑制铁死亡[32-33],而铁死亡介导了脓毒症引起的多器官功能障碍,包括急性肺损伤和肝损伤等[34]。SRPRB是一个人类基因,其转录本在细胞凋亡的MCF-7细胞中上调,并且通常定位于细胞质[35]。它和细胞凋亡相关,并在多种肿瘤中特异性表达[36-37]。 EXOSC4和 EXOSC6是外泌体的核心亚基,在介导mRNA降解过程中发挥作用[38]。EXTL2是EXT基因家族的成员,编码外泌体糖基转移酶。它被认为是一种抑癌基因,与多发性骨软骨瘤等疾病相关[39]。这些发现表明,儿童在发生脓毒症后,UPRGs会自发被激活,常常伴随着各种器官功能的损伤,进一步说明了这些基因在疾病诊断中的重要性。
图5不同亚型的加权基因共表达网络分析和药物预测
Fig.5Weighted gene co-expression network analysis and drug prediction of different subtypes
脓毒症患者常常伴随着两种状态,一是初始的免疫激活,二是慢性免疫抑制,这两种状态可能先后也可能同时发生[40]。在免疫激活状态阶段,如果免疫系统能够及时清除外来的病原体,机体可以恢复正常状态。而在慢性免疫抑制状态下,患者的免疫功能低下,伴随着淋巴细胞和髓系细胞的耗竭[41]。免疫系统的稳态在脓毒症病理学和生理学中起着至关重要的作用。中性粒细胞是人体抵抗病原体的第一道防线[42],我们用对诊断模型中的基因和儿童脓毒症中免疫细胞浸润进行了相关性分析,并发现它们之间存在明显的相关性。这进一步表明脓毒症已经引起了中性粒细胞的浸润。具体而言,中性粒细胞的浸润程度与CEBPB和WIPI1等呈正相关,与SRPRB和EIF2AK3等呈负相关。
通过一致性聚类分析,我们将儿童脓毒症患者分为三个亚型,并发现它们的UPRGs的表达上存在差异。第一型患者的UPRGs表达水平较低,且其中性粒细胞得分最高。进一步的炎症因子表达分析显示,第一型患者TGFB1基因的表达水平最高,而该基因被认为是预测重度肺炎患者的重要指标[43]。因此,我们推测第一型可能是重症患者。不同亚型的FGSEA结果显示,第一型主要涉及淀粉样前体蛋白降解、一氧化氮合酶生物合成、白细胞迁移和趋化过程。之前有研究表明,淀粉样前体蛋白的积累可能是脓毒症患者脑损伤的潜在机制[44-45]。值得注意的是,脓毒症也被认为是急性肺损伤的主要原因之一[46]。而急性肺损伤的发生和多种机制密切相关,其中一个共同元素是一氧化氮[47]。而且中性粒细胞的功能障碍(趋化和迁移)会导致远处器官衰竭[48-49]。这也说明第一型患者可能发生了急性肺损伤,并且有潜在的脑损伤可能。第二型主要富集在α-氨基酸代谢、蛋白质甲基化和凝血调节等过程。代谢组学研究结果显示,随着疾病的进展,脓毒症患者的氨基酸代谢产物在增加[50]。第三型则主要参与免疫球蛋白、小核RNA代谢、T细胞介导的免疫应答等通路。相较于第一型患者,第二和第三型患者都是脓毒症患者普遍存在的调节炎症和免疫的氨基酸代谢失调等问题[51],因此我们推断二三两型为轻型患者。药物预测的结果显示,SB-216763,camptothecin和genipin分别被认为是第一型、第二型和第三型患者的潜在药物。SB-216763是一种GSK-3(糖原合成激酶-3)抑制剂,GSK-3有两种亚型,GSK-3α和GSK-3β。此前已有体内和体外实验研究表明,GSK-3β能够调节炎症反应[52-53],GSK-3β抑制剂的保护作用是由于其抑制了NF-κB的激活,并且由此导致了NF-κB依赖性促炎基因表达的降低。在大鼠模型中,使用GSK-3β抑制剂可减少LPS(内毒素)给药引起的严重内毒素血症相关的器官损伤和功能障碍,并减轻LPS和肽聚糖(PepG)联合给药引起的全身炎症综合征[54]。因此,SB-216763可能对治疗一型脓毒症具有潜在益处。Camptothecin(喜树碱)是从喜树中提取的生物碱,除了具有抗肿瘤活性[55-56],还可以抑制脓毒症诱导的炎症反应[57-58]。此外,genipin(栀子苷)已被证明能够治疗脓毒症,它通过抑制Toll样受体信号传导来减轻败血症[59],通过恢复自噬来缓解脓毒症引起的肝损伤[60],以及抑制T淋巴细胞凋亡来缓解脓毒症诱导的免疫抑制[61]。
本研究存在一定的局限性。首先,使用的数据集来源于公共数据库,无法获取详细的临床信息。其次,由于缺乏临床样本,关键基因的诊断效能尚未验证。最后,由于感染因子种类繁多,儿童脓毒症的严重程度和分期各异,所鉴定的关键基因可能无法代表所有类型的儿童脓毒症。
4 结论
本研究利用机器学习方法基于未折叠蛋白反应相关基因构建了诊断模型,探究了这些基因在儿童脓毒症诊断中的潜在价值。这几个基因与多种免疫细胞浸润相关,它们在免疫微环境可能发挥重要作用。此外我们还进行了儿童脓毒症亚型划分和相关药物预测,为精准医疗提供新思路。



