摘要
DNA存储作为一种稳定性强、存储密度高、能耗低的数据存储方式,在传统存储介质逐渐无法满足数据存储需求的当下,有望解决海量数据存储困境。目前,科学家已提出多种DNA存储方案,然而由于研究人员的学科背景领域以及关注重点不同,导致这些方案的评价指标不尽相同,亟需一套能够综合评判DNA存储方案优劣的模型。本文总结了DNA存储领域的主要质量评价指标,通过专家问卷调查法统计获得了各指标的权重,基于层次分析法构建了DNA存储方案评价模型,并利用此模型对近年来具有突破性进展的DNA存储方案进行了评估分析。结果表明,Organick的随机大规模DNA存储访问技术综合得分更高,与其他方案相比在信息检索等方面有一定优势。本文对于DNA存储方案的选择提供可借鉴的框架与思路,也为DNA存储的性能提升给出对策和建议。
Abstract
DNA storage, as a data storage method with strong stability, high storage density and low energy consumption, is expected to solve the problem of massive data storage in the current situation where traditional storage media are gradually unable to meet data storage. At present, scientists have proposed various DNA storage frameworks. However, due to the different disciplinary backgrounds and focus of researchers, the evaluation indicators of these schemes are not the same. There is an urgent need for a model that can comprehensively evaluate the advantages and disadvantages of DNA storage schemes. This article summarizes the main quality evaluation indicators in the field of DNA storage, obtains the weights of each indicator through expert questionnaire survey, constructs a DNA storage scheme evaluation model based on analytic hierarchy process, and uses this model to evaluate and analyze DNA storage schemes that have made breakthroughs in recent years. The result indicates that Organick's random large-scale DNA storage access technology has certain advantages in information retrieval and other aspects compared to other solutions. This article provides a reference framework and ideas for the selection of DNA storage solutions, and also provides strategies and suggestions for improving the performance of DNA storage.
Keywords
随着当代信息技术的飞速发展,数据信息总量呈指数级增长。据统计,2011年全球生成的数据量约为1.8×1012 GB,2018年达到3.3×1013 GB,在短短7年间增长了18倍。根据上述趋势,到2025年数据量预计将超过1.75×1014 GB[1]。磁性存储、光学存储及半导体存储等传统存储技术已无法满足未来高速增长的数据存储需求,与之相比,作为承载生命信息的DNA分子介质在存储密度、稳定性及能源消耗等多方面有着独特优势[2],利用DNA碱基进行数据存储的崭新方式在大数据领域将会起到关键性作用,有望成为一种低成本、高稳定的存储解决方案[3]。
DNA存储概念在20世纪六十年代提出,但受限于DNA合成和测序技术,直到21世纪DNA存储技术才有明显发展。《Nature》、《Science》等权威期刊在近十年间都有DNA存储相关研究成果的报道。尽管在DNA存储领域已有大量研究方案和成果,但由于不同研究的优化策略只涉及DNA存储的一个或几个方面,所以多数研究对于实验结果评价的关注重点不完全相同,研究人员在进行方案遴选与对比研究时会根据实际情况选取感兴趣的指标参数进行分析,借此来说明不同方案的优缺点。但DNA存储是一个涉及编码、合成、储存、测序及解码等多步骤的项目,建立全面、系统和量化的DNA存储评价模型,对于数据综合存储性能方案的评价与选择有着重大的参考意义和价值。本文首先收集与DNA存储评价有关的指标,之后使用层次分析法建立科学完整的DNA存储评价模型,并利用该模型统计并计算了近年来在《Nature》、《Science》等期刊发表的DNA存储方案的指标分数以评估各个方案的优点与不足,从而为DNA存储技术的发展和应用提供理论指导和支持。
1 材料与方法
本节方法的实现流程如图1所示。首先从文献中收集DNA存储的评价指标,然后基于层次分析法构建DNA存储评价模型,最后选取最具代表性的DNA存储案例应用于该模型进行实证分析,从而客观评价各个方案的优缺点。
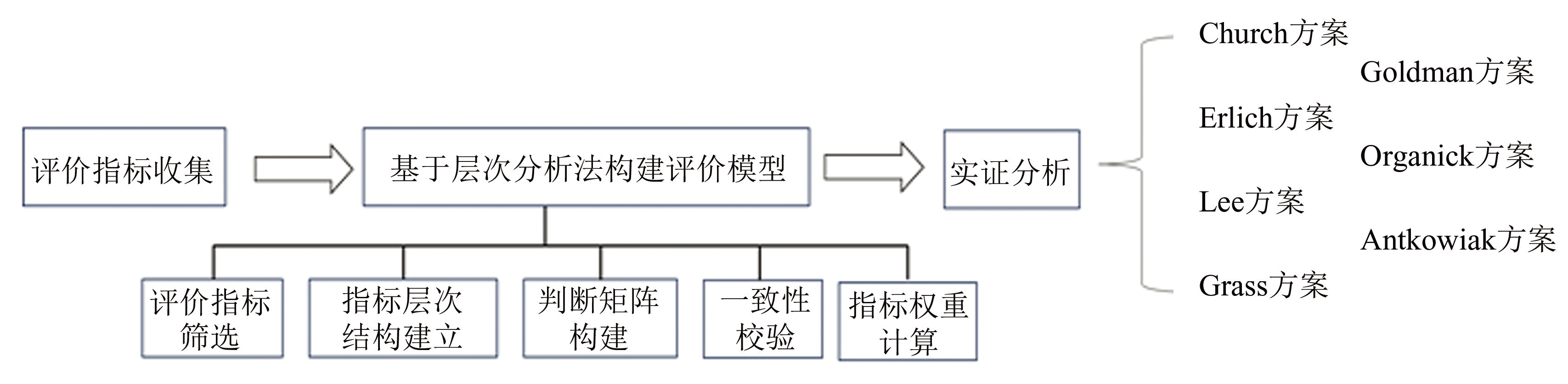
图1方法流程图
Fig.1Flowchart of the method
1.1 评价指标收集
针对DNA信息存储方案,尚未有人提出完备的评价体系,研究人员在文章中进行方案比较时会记录和计算一些重要的指标及数据,因此可选取这些文献指标作为本文初始评价指标的来源。通过对23篇DNA存储论文中涉及到的评估指标进行汇总统计和讨论,详细结果如表1所示。
1.2 基于层次分析法构建DNA存储评价模型
层次分析法由美国运筹学家Satty.T.L.于20世纪70年代提出[25],它是一种应用网络系统理论和多目标综合评价方法。该方法将定量分析与定性分析结合起来,将复杂问题划分成多个要素,进而分解为多要素的若干层次,根据决策者的经验判断各衡量目标能否实现的标准之间的相对重要程度,并合理给出每个决策方案的每个标准的权数,利用权数求出各方案的优劣次序,从而实现对于方案的定性和定量化分析。层次分析法主要包含评价指标筛选、评价指标层次结构建立、判断矩阵构建、一致性校验和指标权重计算。
1.2.1 评价指标筛选
DNA存储方案的评价指标既彼此独立又相互联系,筛选过程中应遵循以下4个原则:①系统全面性。评价指标应能够系统全面涉及DNA存储的各个步骤,包括编码、合成、保存、测序及解码等过程,全方位展现DNA存储的价值;②代表性。影响DNA存储效果的因素诸多,应选择最具代表性、最关键的因素进行权衡比较;③科学可行性。所有指标应定义明确清晰,便于专家分析论证和评估;④可量化。所有指标可通过文献中定义以及经验进行数字化描述。基于上述原则,筛选出9个主要指标,具体信息如表2所示。
表1DNA存储评价指标及来源汇总
Table1Summary of literature sources for evaluation indicators

表2筛选后的指标及定义
Table2Screening indicators and definitions

1.2.2 评价指标层次结构建立
对于筛选过后的评价指标,依据层次分析法和DNA的特性将其划分到4个一级指标中,分别为成本、信息质量、信息密度和信息检索,在一级指标下细分9个二级指标(表2),最终确定DNA存储方案评价指标层次结构,如图2所示。

图2评价指标层次结构
Fig.2Hierarchy of evaluation indicators
1.2.3 判断矩阵构建
通过专家咨询法,与6位DNA信息存储专业领域的专家讨论DNA存储方案评价指标层次结构,并使用调查问卷统计各位专家对于图2各项指标的重要性意见。
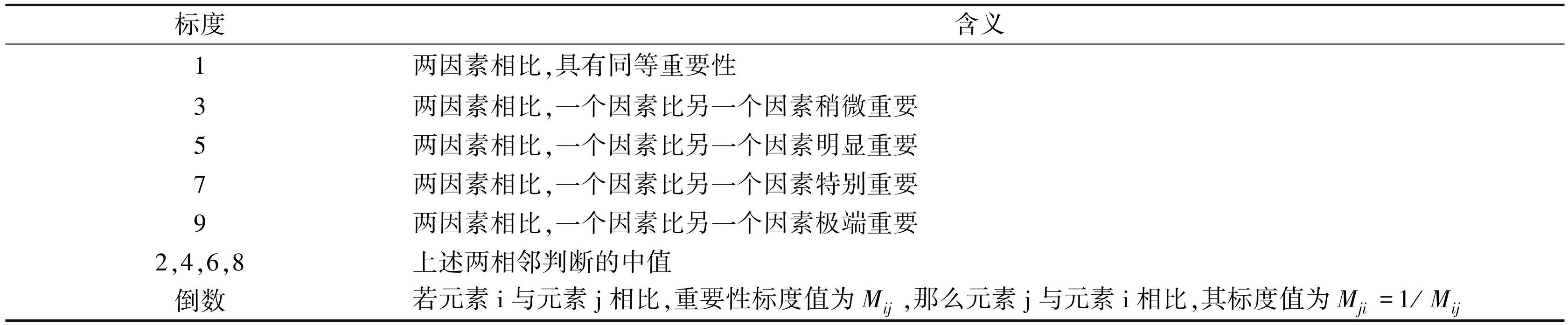
针对某一指标而言,通常使用相对重要程度aij来表示这一层中第i个元素与第j个元素的重要性。判断矩阵具体形式结构由矩阵(1)所示,标度aij含义如表3所示。
(1)
表3判断矩阵标度含义
Table3Meaning of judgment matrix scale
1.2.4 一致性校验
为了保证判断矩阵和结论的合理性,避免不同指标出现逻辑矛盾,还需要对判断矩阵进行一致性检验。一致性检验以一致性比例CR为评判标准,当CR<0.1时,表明判断矩阵的赋值中各个元素之间的关系是符合逻辑的。当CR≥0.1时,则表明各个元素之间存在不符合逻辑的情况则需要重新修正判断矩阵,计算方法由公式(2)(3)计算,λmax表示判断矩阵的最大特征值。
(2)
(3)
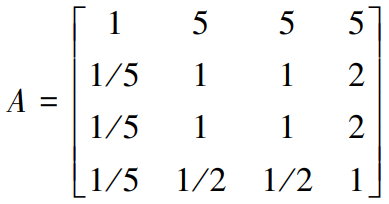
(4)
矩阵(4)是某位专家对于一级指标的判断矩阵,计算其λmax=4.0606,,,因此一致性验证通过。因此,依据特征向量从而计算出相应的各个指标权重,因此该专家调查问卷中一级指标的各层级所占权重分别为:成本0.918 3,信息密度0.147 1,信息质量0.147 1,信息检索0.087 5。
1.2.5 评价指标权重计算
汇总所有专家的调查问卷,分别按照1.2.3和1.2.4节构建判断矩阵并进行一致性校验。所有问卷都通过了一致性校验,并根据判断矩阵计算出各级指标权重,结果如表4所示。
对表4中的每个指标取六位专家的权重均值即可得到平均权重,用平均权重作为基于层次分析法构建的DNA存储方案评价模型的权重,如表5所示。
表4各专家对不同指标的权重汇总表
Table4Summary of experts' weighting assignments to various indicators
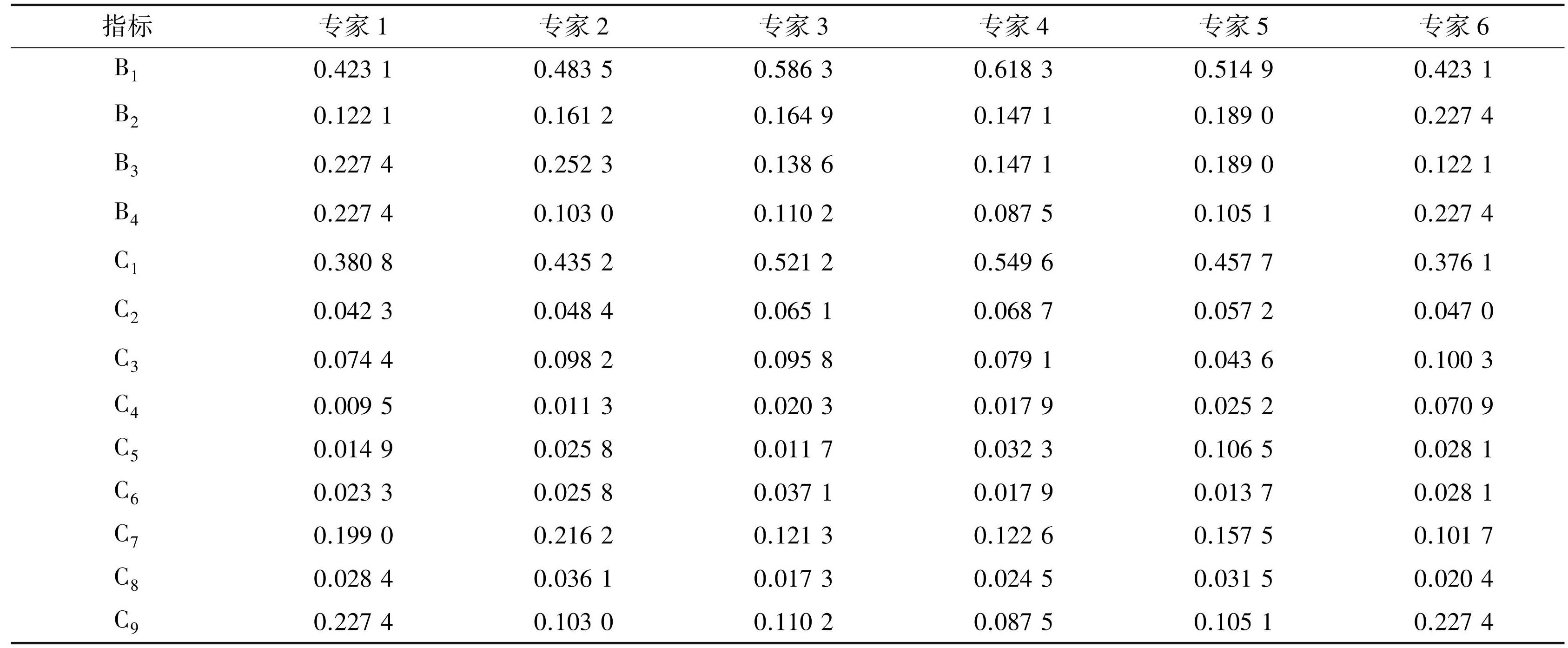
表5DNA存储方案评价模型指标权重
Table5DNA storage scheme evaluation model and index weights

1.3 实证分析方案
为了证明模型的可应用性和可扩展性,我们结合近年来DNA存储的发展前景,从《Nature》、《Science》等期刊选取了七种最具代表性和影响性的DNA存储方案应用于构建的评价模型中进行实证研究,以下是对这些方案进行简述。
1.3.1 数据方案介绍
1.3.1.1 Church团队方案[2](以下简称方案A)
2012年哈佛大学教授George Church团队提出了用于编码任意的信息的DNA信息存储架构。该团队将650 KB的文件信息转换为了二进制码流,为了避免DNA片段中均聚物的产生以及控制CG含量在合理区间,他们使用A/T表示0、C/G表示1的二进制转换模型将比特流编码为54 898个DNA序列,每个片段包含159个核苷酸,通过Agilent核苷酸文库进行合成,最后通过Illumina Hiseq单通道测序法对DNA序列测序,并使用逆二进制转换模型将恢复为原文件。
1.3.1.2 Goldman团队方案[4](以下简称方案B)
为了消除单碱基均聚物对合成或测序过程的影响,2013年Nick Goldman团队提出了一种基于霍夫曼编码的轮转编码规则,主要目的是为了建立一个容量大、维护要求低、成本低及效率高的DNA存储可行方案。该团队将739 KB的信息,包括莎士比亚的诗集、论文的PDF文件、格式为JEPG的图片和MP3音频文件,存储到153 335个长度为117个核苷酸的DNA片段中。其方案首先采用三进制哈夫曼算法对信息进行压缩编码,之后采用轮转编码的方式来解决碱基连续出现的情况,并且其通过四倍重叠移位的编码方式,使得信息位按照部分重叠的方式进行打断拼接,即相邻的100个碱基片段包含75个重叠的碱基,这样对于一个完整的DNA片段来讲,除了首尾100个碱基,其他部分都是重复出现过4次。
1.3.1.3 Grass团队方案[5](以下简称方案C)
2015年,Robert N. Grass团队为了解决在合成、测序等步骤中DNA碱基缺失、替换和插入的问题,首次将信息技术中的纠错编码机制引入至DNA存储中。他们将83 KB信息转换成4 991个158核苷酸长度的DNA序列中,并在序列中添加“前向纠错码”,将其封装在二氧化硅中保存,其理论信息密度可以达到1.78 bit/base。通过加速老化的方式来进行DNA衰老实验,结果证明,即使是在70℃的高温下,原始信息依旧得到了完整的恢复,在实验模拟仿真下DNA分子甚至可以保存数千年。
1.3.1.4 Erlich团队方案[6](以下简称方案D)
美国哥伦比亚大学Erlich研究团队创新性地使用基于LT码的喷泉码编码方式进行DNA信息编码。该团队于2017年将2.14 MB的信息,包括一个小型的计算机系统和电影,利用了LT码编码方式存储于单链DNA分子中,该方案在信息的易丢失性、弱拓展性等方面得到了改善。Erlich等在编码阶段时首先将信息源等分为块,再通过度函数和变换生成独立随机的包,采用四进制碱基转换的方式直接将信息比特映射成碱基,该算法并未将约束条件写入映射规则中,而是通过筛选机制使得最终获得的DNA序列满足GC含量和均聚物约束要求。
1.3.1.5 Organick团队方案[7](以下简称方案E)
Organick团队的方案中在对信息进行编码的过程中通过尾随机化和序列分割来进行对二进制数处理,外码采用RS码进行纠错,内码进行碱基转换,通过分配正交引物来实现通过PCR技术对信息的随机检索物,充分考虑了DNA链的均聚物、互补性和GC含量等方面的问题进行评价和筛选。对超过1 300万条DNA进行35个不同文件(大于200 MB的信息)的DNA存储,通过Twist Bioscience合成,之后采用合成和纳米孔进行测序,在信息的恢复阶段仅用5×的覆盖率就对原始两百多兆字节的信息进行了无损解码。
1.3.1.6 Lee团队方案[16](以下简称方案F)
Lee团队的方案采用了一种全新的基于酶合成的方法,主要是通过利用无模板聚合酶末端脱氧核苷酸转移酶在动力学控制条件下的作用进行实现。信息的存储主要是通过在DNA链中不同核苷酸之间的转换实现的。为了得到进行存储信息特定的DNA链,核苷酸底物被迭代添加进而产生一个或多个;同时其长度由脱嘌呤酶控制,脱嘌呤酶通过可以去除这些不必要的底物,从而控制链扩展的长度和减少错误。通过这种方案合成了携带144核苷酸长度的DNA链,并证明了通过流式纳米孔测序进行检索的可行性。
1.3.1.7 Antkowiak团队方案[9](以下简称方案G)
Antkowiak等的方案展示了一个基于定向光化学合成的DNA存储系统,该系统相比于传统的化学固相合成技术成本要低的多,该团队将100 KB的信息编码为16 383个长度为60核苷酸的DNA序列中,并在编码中增添了伪随机化算法和RS纠错编码,尽管这种合成方法比以往合成方法产生的错误要多,但是由于设计的高冗余以及编解码算法,信息成功得到了完全恢复。
1.3.2 指标数据标准化及规范化
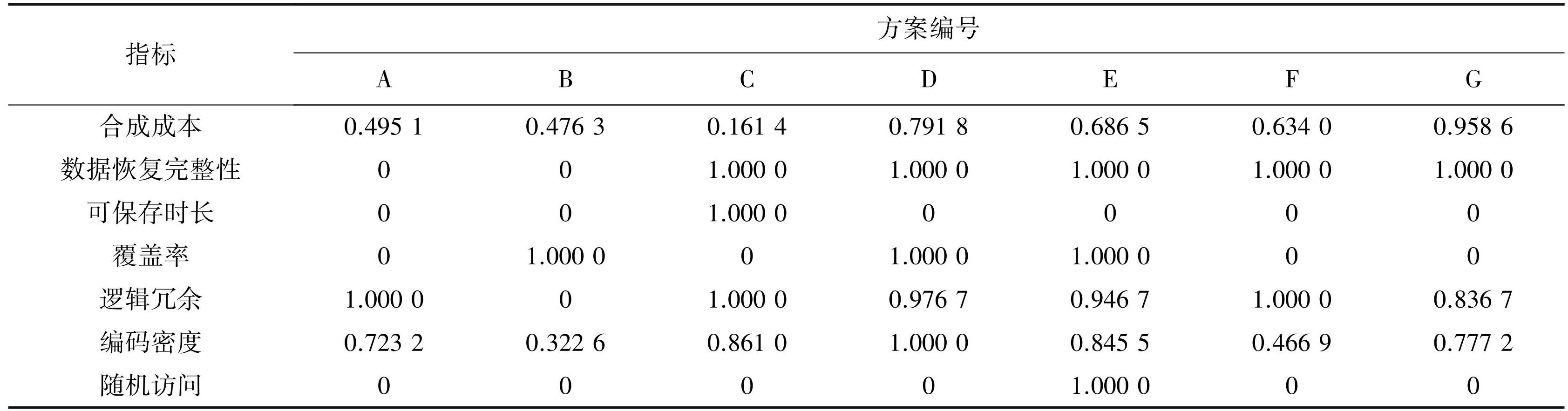
根据1.2节中构建的层次结构,从上述7种方案中提取对应的二级指标信息。对于不同指标,由于其定义和量纲不同,需要对同一类型的指标数据进行标准化以及规范化。比如对于合成成本类指标,由于其区间范围大,因为需要通过标准化将其限制在[0,1]区间;而对于数据恢复完整性,我们通过是否来进行描述,在计算时需要将其规范化为量化指标。
1)合成成本:合成成本在评价指标中为成本型指标。成本型指标的值越小,则所表示的实际成果就越大,其无量纲变化公式(5),可以通过此式确定相应的评分。
(5)
2)测序成本:此指标也为成本型指标,标准化方法与合成成本相同。
3)数据恢复完整性:对描述进行规范化,若方案完全无错误的恢复了存入的数据则评分为1,反之为0。
4)可保存时长:对描述进行规范化,目前可保存时长主要和保存存储的介质有关,按照存储介质的先进性进行排序评分,最先进评分为1,最落后评分为0。
5)覆盖率:对描述进行规范化,若覆盖率等于100%说明测序深度为1x,为低覆盖率,评分记为1,否则为高覆盖率,评分为0。
6)逻辑冗余:此指标为成本型指标,标准化与合成成本相同。
7)随机访问:对描述进行规范化,如果方案可以实现对于数据的选择性访问,则评分为1,反之为0。
8)编码密度:此指标为效益型指标,效益性指标该指标的值越大,所表示的实际成果就越大,因此应用的标准化公式为(6)。
(6)
9)物理密度:此指标为效益型指标,标准化方法与编码密度相同。
1.3.3 基于DNA存储评价模型计算得分
将1.3.2的指标数据与对应的指标权重相乘求和,得到某方案的指标得分。统计计算上述7种方案的一级指标分数以及总分数,讨论不同方案的优势以及可供借鉴的方案思想。
2 结果
2.1 数据汇总以及标准规范化
统计汇总上述方案在合成成本、数据恢复完整性、可保存时长、覆盖率、逻辑冗余、编码密度及随机访问的信息,具体见表6。将表6数据按照1.3.2节的公式进行标准规范化,具体结果如表7所示。
表6各方案原始指标数据
Table6Detailed indicators of schemes
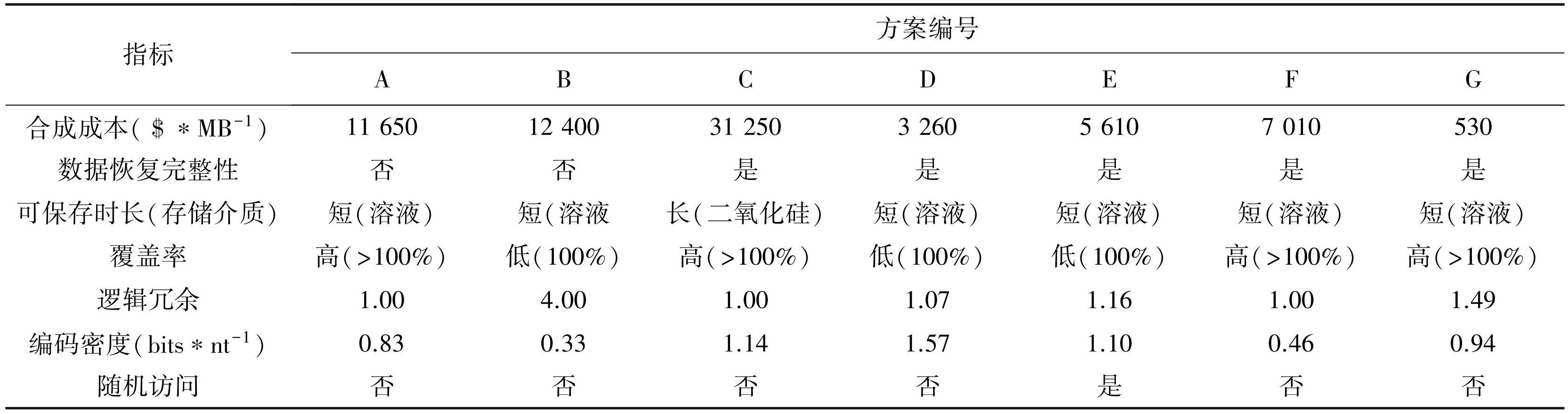
表7各方案指标标准化
Table7Numerization of detailed indicators for schemes
2.2 不同方案一级指标及总分数
将表7的标准化指标与表5对应的指标权重相乘并求和,可以得到基于层次结构模型的不同指标的评分,如图3所示。可以看到,不同方案各有其优势,从成本类指标来看,得分最高的是方案G(Antkowiak团队方案),从信息密度来看指标来看,信息密度最高的是方案D(Erlich团队)采用的喷泉码编码,编码密度达到1.57 bit/nt,从信息质量指标来看,方案D(Erlich团队)和方案E(Organick团队)信息质量都很高,从信息检索指标来看,方案E(Organick团队)是唯一一个能够进行随机访问数据的方案。综合四个一级指标,最优的方案是Organick团队的方案。该方案创新性地为其信息随机存取方案设计了一个引物库,引物序列通过算法进行筛选,直至满足GC含量、均聚物长度、序列互补性等多个方面高标准的要求。然后对二进制存储信息进行伪随机化和片段分割,通过增加RS码的冗余来加强信息的质量,之后为每段信息链结合引物从而实现信息的随机访问。其解码阶段,该方案根据相似度、地址及包括含噪声的片段进行迭代聚类。之后通过轨迹重建,从聚类中恢复原始序列。最后通过解码RS外码和反转随机化来实现对于非聚类中的某些错误恢复从而实现成功解码。其合成的序列由Twist Bioscience合成,测序通过Illumina的NextSeq来实现,在读取每个DNA序列的平均覆盖率仅为5次的情况下,无错误的恢复了200 MB的信息,其中包含35个不同大小类型的文件。
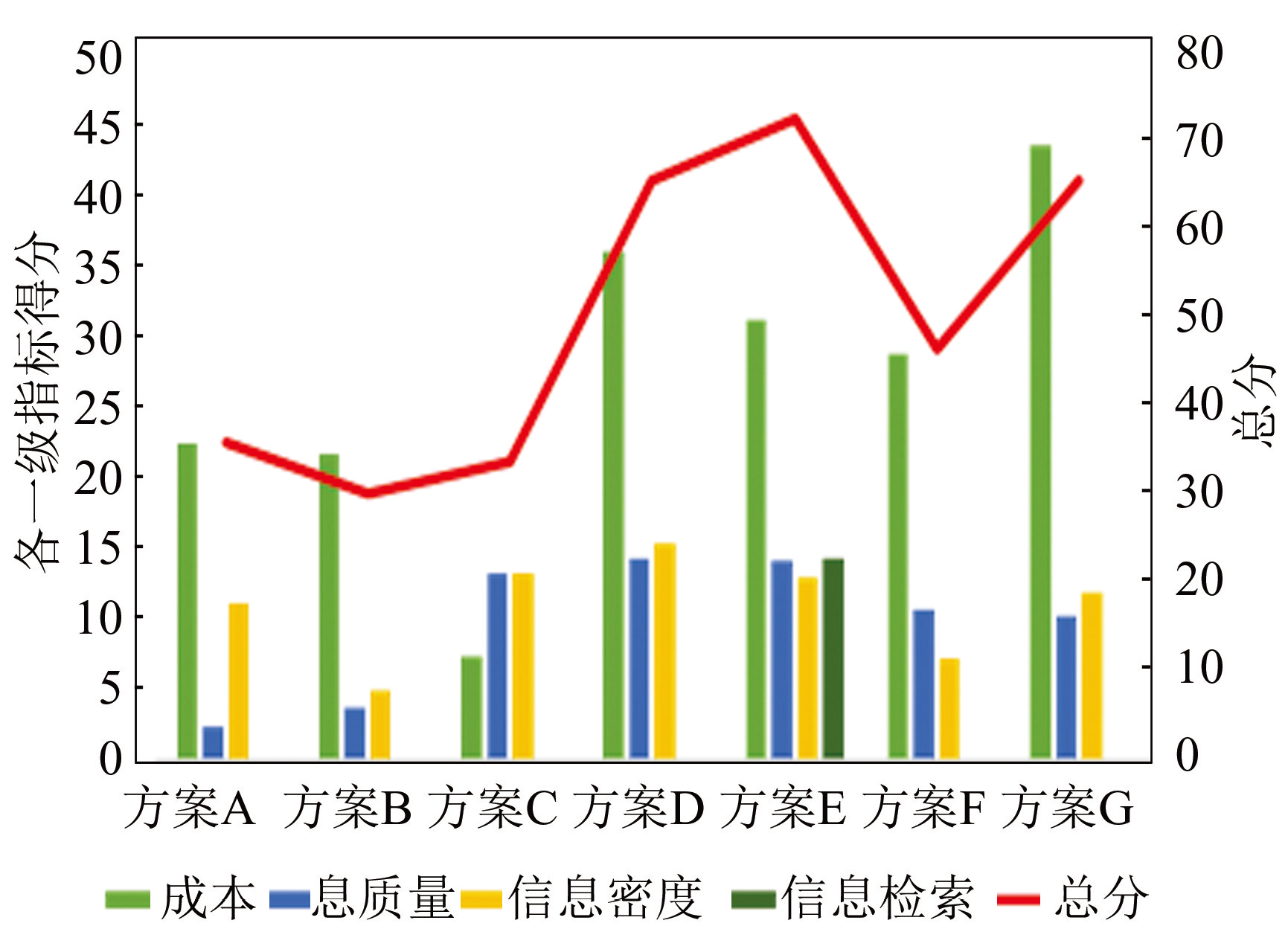
图3不同方案的一级指标分数以及方案总分
Fig.3Primary indicators scores and total scores for different methods
3 讨论
在基于层次分析法构建的DNA评价指标中,成本指标占比权重最大,这是因为合成DNA所需成本处于较高水平,是目前限制DNA存储信息的一个关键难题,尽管在过去几十年科学家在大规模寡核苷酸合成方面取得了极大进步,但是合成DNA的成本和难度仍然远远高于DNA测序。目前DNA合成主要方法依旧是化学合成,该方法缺点是合成DNA长度受限且对环境不友好,因此酶法合成及基于微阵列的并行DNA序列合成研究或许将推动大规模核苷酸合成技术的进步。其次占比较大的指标分别是编码密度和随机访问。编码密度根据所选的碱基转换方式不同而有所差异,目前较为常用的是四进制转换方式,即直接将{00,01,10,11}映射为{A,C,G,T},理论编码密度可以达到2 bit/base。但由于DNA在合成、保存及测序时不可避免地发生碱基错误或者序列丢失,所以还需要添加纠错码、地址码以便正确恢复数据,因此编码密度可以衡量DNA在编码时的序列质量。随机访问作为一种新兴的指标类型,能够使访问DNA中的信息像硬盘一样按照索引访问而不需要恢复全部信息,大大提升了DNA存储信息的便捷性,并降低了相关成本和错误率,有望成为DNA存储的里程碑式的指标。
Antkowiak团队方案在合成上采用了定向光无掩膜阵列技术,与CstomArray商业化的电极阵列技术和Twist Biosciences的喷印合成法相比,光定向合成可以通过更低的成本来实现信息存储。其他方案采用了喷墨打印法和电化学合成法,喷墨打印法与之相比合成稳定性较好,错误率较低,不需添加太多纠错冗余,信息密度更高,但同时喷墨打印合成的单条寡核苷酸产量极低;电化学合成法相比光定向合成法,合成通量较高且同时也存在合成稳定性差,错误率高的缺点,并且喷墨打印和电化学合成相比均需要较多的实际消耗量,增加了合成成本。Erlich团队采用的喷泉码编码,编码密度达到1.57 bit/nt,喷泉码在信息容量上接近香农极限,同时又具有高鲁棒性来抵抗数据错误,并且具有独立随机性,此外在解码恢复信息阶段,由于其较低的编译码复杂度,只需较低的计算资源就可以用很高的概率恢复信息。Erlich团队和Organick团队构建的DNA存储方案的信息质量都很高,首先其方案均采用了喷泉码的编码,并且都具有较低的逻辑冗余,除此之外在低覆盖率的情况下也能够完全无错地恢复存储的信息。
4 总结
展望未来,在DNA信息存储走向软硬件一体化系统的趋势下,不同程序对应的DNA存储阶段的流程方案的侧重点可能也有所不同,因此需要综合系统的评价工具对其进行科学评估,并从多种方案中遴选出适合的方案。为了能够综合比较不同DNA存储方案的优缺点,本文在总结筛选评价DNA存储指标的基础上,从成本、信息质量、信息密度、信息检索4个方面综合提出9个二级指标,通过专家调查问卷法确定了评价指标体系中的权重,基于层次分析法构建DNA存储方案评价模型和各指标的量化方案,最后结合7个样本方案进行实证研究,评价结果分析表明在所选样本中方案最优的是Organick团队的存储方案。本文所提出的基于层次分析法的DNA信息存储方案评价模型从DNA存储的多方面多步骤出发,综合考量了多个指标,可以为一体化系统设备的建立提供建议和参考,节约成本及计算资源。
