摘要
近年来,针对癌症的大量研究,积累了海量的多组学数据,这些数据为高效鉴定癌症驱动基因提供了可能;本研究提出了一种基于深度图信息最大化的异亲图卷积网络(Heterophilic deep graph information maximization convolutional network, HDGICN)模型用于识别癌症驱动基因,HDGICN首先整合了图信息最大化和个性化PageRank算法对异亲生物分子网络的基因节点进行特征增强,然后通过整合了双重残差结构的分层混合图卷积来学习异亲生物分子网络上的基因特征,最后根据预测得分来识别癌症驱动基因;实验结果显示,在三个异亲生物分子网络上,HDGICN的受试者工作特性曲线下面积(Area under receiver operating characteristic, AUROC)和精确率-召回率曲线下面积(Area under the precision-recall curve, AUPRC)均优于其他传统方法,消融实验结果进一步表明本方法有助于提升预测性能。HDGICN方法在异亲生物分子网络上能够有效识别出癌症驱动基因,可以为实现癌症的精准治疗和生物标志物的发现提供重要帮助。
Abstract
In recent years, extensive research efforts on cancer has accumulated a massive amount of multi-omics data, providing opportunities for efficient identification of cancer driver genes. In this study, we propose a novel Heterophilic Deep Graph Information Maximization Convolutional Network (HDGICN) model for cancer driver gene identification. HDGICN integrates graph information maximization and Personalized PageRank algorithm to enhance gene node features in heterophilic molecular networks. Subsequently, a hierarchical mixture of graph convolutions with dual residual structures is employed to learn gene features on heterophilic molecular networks. Finally, cancer driver genes are identified based on prediction scores. Experimental results on three heterophilic molecular networks demonstrate that HDGICN outperforms traditional methods in both Area Under Receiver Operating Characteristic (AUROC) and Area Under the Precision-Recall Curve (AUPRC). Further ablation experiments validate the method's effectiveness in improving predictive performance. HDGICN proves effective in identifying cancer driver genes on heterophilic molecular networks, offering valuable support for precision cancer treatment and biomarker discovery.
癌症是一组由遗传突变引起的细胞异常和失控增殖的疾病的总称[1]。这些突变被称为驱动突变,使得癌细胞相对于其周围正常细胞具有选择性优势,进而能够在组织内迅速生长和扩散[2]。与这些驱动突变相关联的基因被称为驱动基因[3]。鉴定癌症驱动基因对于推进精准治疗和发现生物标志物具有重大意义[4]。近年来,大规模的癌症研究项目,例如癌症基因组图谱项目(The cancer genome atlas,TCGA)和国际癌症基因组图谱项目(The international cancer genome consortium,ICGC),为鉴定癌症驱动基因提供了有价值的多组学数据[5-6]。这些数据集提供了各种类型癌症的全面分子特征信息,为癌症基因组学研究人员开发计算方法提供了可能[7]。
在早期的研究中,识别癌症驱动基因的方法可分为三类:基于突变频率的方法、基于网络的方法和基于机器学习的方法。基于突变频率的方法如MuSic[8]和OncodriveCLUST[9]利用突变率、通路分析和突变聚类来检测癌症驱动基因,具有良好的癌症驱动基因识别效率。然而,它们在识别带有罕见突变的驱动基因方面能力有限。相比之下,基于网络的方法,如DriverNet[10],DawnRank[11],Hotnet[12],Hotnet2[13],SCS[14]和PRODIGY[15],则通过整合突变、基因表达和网络数据,根据基因相互作用和网络特性来识别癌症驱动基因,具有良好的可解释性。然而,这些方法很大程度上依赖于完整的生物分子网络,因此在网络不完整的情况下性能会受到很大影响[16]。随后,研究人员开发出了基于机器学习的方法来识别癌症驱动基因。例如, sysSVM2[17] 将癌症遗传改变与基因系统级特性相结合,以预测个体患者的驱动因素。DriverML[18]结合了统计学方法,根据蛋白功能计算不同突变类型的得分,并应用监督式机器学习技术进行癌症驱动基因的识别。尽管这些机器学习方法在识别癌症驱动基因方面取得了显著进展,但它们忽视了网络结构信息在癌症驱动基因识别过程中的关键作用。
近年来,基于图卷积网络的深度学习方法已成为识别癌症驱动基因的有效途径,部分解决了前述挑战。例如, EMOGI[19]整合了生物分子网络和基因的多组学特征,利用图卷积网络(GCN)[20]来预测癌症驱动基因。随后,MTGCN[21]进一步改进了EMOGI,采用基于Chebyshev GCN[22]的多任务学习框架来识别癌症驱动基因。这些深度学习方法在识别癌症驱动基因方面表现出色,但它们忽视了生物分子网络的异亲性。异亲生物分子网络被定义为涵盖不同类型生物分子之间多种交互类型的网络,这些交互类型包括蛋白质-蛋白质相互作用(PPI)、蛋白质-DNA相互作用和蛋白质-RNA相互作用等,同时在这些相互作用网络的内部,节点与节点之间的链接也可能具有不同的含义,比如PPI网络中蛋白质分子之间的链接可以是功能关联也可以是物理相互作用[23-25]。过去的研究主要集中在同亲性假设上,即将单一基因-基因网络中具有链接的相邻节点归类到相同的类别,但癌症驱动基因通常通过参与不同类型的相互作用来促进癌症的发展,因此基于同亲性假设的方法会降低癌症驱动基因的识别效率[3,26-27]。为了解决此类问题,HGDC[23]利用个性化PageRank(PPR)[28]扩展GCN以适应异亲生物分子网络的方法,提升了模型对癌症驱动基因的识别能力。然而,该方法并未充分考虑到PPR和GCN在捕捉异亲生物分子网络全局结构特征方面的能力有限,某些远程相关的基因特征仍然可能会被忽视,导致模型对异亲生物分子网络的学习能力不足。此外,随着GCN层数的增加,模型复杂性也会上升,这将引起信息在多个卷积层之间传递时丢失部分有价值的原始特征信息,从而使得模型预测癌症驱动基因的性能降低。
为了解决上述问题,本研究提出了一种面向异亲生物分子网络而设计的图卷积模型,命名为基于深度图信息最大化的异亲图卷积网络(Heterophilic deep graph information maximization convolutional network,HDGICN)。具体而言,它将DeepGraphInfomax(DGI)[29]捕捉全局结构特征的能力与PPR算法的异亲图适应性相结合以丰富原始基因特征,使得HDGICN在进行学习时能够充分捕捉异亲生物分子网络中的全局结构信息。接着,本研究在HDGICN下游节点预测任务中引入一种带有双重残差结构的分层混合图卷积方法,以期通过双重残差连接解决因模型复杂性增加而导致的基因节点原始特征信息丢失问题。这些整合使得HDGICN在受试者工作特性曲线下面积(Area under receiver operating characteristic,AUROC)和精确率-召回率曲线下面积(Area under the precision-recall curve,AUPRC)方面优于其他传统方法,能准确识别异亲生物分子网络中的癌症驱动基因。
1 材料与方法
1.1 数据获取与整理
本研究使用与HGDC[23]相同的三个异亲生物分子网络和三个基因特征矩阵来识别癌症驱动基因,为了文章的完整性,本文对这些数据的构建过程进行详细介绍。
首先,三个异亲生物分子网络(GGNet,PathNet,PPNet)分别是从ENCORI(starBase v2.0)[30]中获得的含有11 183个节点和621 988条边的RNA交互网络(GGNet),从Wu等[31]构建的人类功能蛋白质网络中保留KEGG[32]和Reactome[33]通路后得到的含有7 695个节点和92 710条边的通路网络(PathNet)以及从STRING v11[25]中根据前5%置信度的相互作用构建的含有11 395个节点和285 843条边的蛋白质相互作用网络(PPNet)。这三种网络均已被Zhang等[23] 通过计算图中的同亲比(同类邻居节点与总邻居节点的比值)的方法证实是异亲生物分子网络。在三个异亲生物分子网络中,每个基因的标签(即是否是癌症驱动基因),是基于Schulte-Sasse等[19]提供的两个列表来确定的,其中一个列表包含了796个被确认为癌症驱动基因的基因名称,而另一个列表包含了2 187个被确认为非癌症驱动基因的基因名称。被确认为癌症驱动基因的被标记为正样本,而非癌症驱动基因被标记为负样本,未包含在正样本或负样本中的基因被视为无标签数据。具体而言,正样本包括NCG 6.0数据库[34]中的711个驱动基因和使用DigSEE[35]从PubMed中提取的85个高保真癌症驱动基因。负样本是通过递归地移除与癌症相关的基因而提取的,这些被剔除的基因包括NCG 6.0和COSMIC遗传基因集[36]中的基因、KEGG数据库中与癌症途径相关的基因、OMIM疾病数据库[37]中与癌症相关的基因以及MutSigdb[38]中与癌症相关的基因。
其次,异亲生物分子网络中每个基因的特征信息由癌症特异性多组学数据计算而来的生物分子特征和由基因全局特征计算而来的系统级特征组成。具体而言,为了获得癌症特异性生物分子特征(即与不同癌症有着特异性关联的生物分子特征),可先从TCGA数据库[5]获取29 446例患者中16种癌症类型(包括膀胱癌、乳腺癌、宫颈鳞状细胞癌、结直肠癌、食管鳞状细胞癌、头颈部鳞状细胞癌、肾透明细胞癌、肾乳头状细胞癌、肝细胞癌、肺腺癌、肺鳞状细胞癌、前列腺癌、直肠癌、胃腺癌、甲状腺癌、子宫内膜癌)的基因突变、DNA甲基化和基因表达数据,以及正常组织的基因表达和DNA甲基化数据。接着对于每种癌症类型,按照EMOGI[19]所提到的方法,通过非沉默的单核苷酸变异数量除以外显子基因长度得到基因的癌症特异性突变频率;使用所有癌症样本中的每个癌症类型与正常样本对应类型之间甲基化信号的差异值除以该类型癌症中的所有样本数量得到基因的癌症特异性DNA甲基化水平;计算基因在肿瘤样本中与相应正常样本的表达值的log2倍变化(Fold change),然后在每个癌症类型的所有样本中取平均得出基因的癌症特异性差异表达水平。之后将这16种癌症类型的特异性基因突变频率、特异性DNA甲基化水平、特异性基因差异表达水平拼接,即可得到基因的48维癌症特异性生物分子特征。接着根据sysSVM2所提供的数据和方法可获得描述基因全局特性的系统级特征,它与癌症特异性多组学数据没有直接关系,但却能将癌症基因与其他人类基因区分开来[17]。具体而言,可直接从sysSVM2中取得基因是否与癌症驱动基因同源、基因在癌症细胞系中的比例、基因在癌症发生中是否必须、基因在人体组织中的表达数量、基因编码的蛋白质所属的复合物数量、miRNA靶向调控该基因的数量共计六种系统级特征,之后再计算基因在生物分子网络中的度、是否是中心节点、网络介数和网络聚类系数得到另外四种系统级特征,在使用典型驱动基因和其余基因的可用数据的中位数估算缺失的系统级特征后,可得到10维系统级特征。最后将10维系统级特征与48维生物分子特征拼接可得到基因的58维特征向量,最后将每个网络中每个基因的58维特征向量进行组合可构建出三个异亲生物分子网络的基因特征矩阵。
1.2 HDGICN总览
如图1所示HDGICN由特征增强模块和下游节点预测任务组成。在特征增强模块中,本研究首先通过将基因的特征矩阵X与异亲生物分子网络A的结构特征相结合,利用PPR生成了辅助网络Aaux。然后,基因的特征矩阵X与异亲生物分子网络A构成了一个DGI的输入数据,而基因的特征矩阵X与辅助网络Aaux构成了另一个DGI的输入数据,之后两个DGI分别根据各自输出的损失值Lrep1和Lrep2优化自身的基因特征学习能力,当两个DGI达到最优效果时,将输出的增强特征矩阵相加取平均值之后再与原基因特征矩阵X相加构成增强特征矩阵Xenh。接着本研究将增强后的每个基因特征结合生物分子网络结构和辅助网络结构输入到下游节点预测任务中,在经过一层全连接(FC1)、三层带有双重残差结构的图卷积(GCNConv)和三层带有双重残差结构的图采样聚合卷积(SAGEConv)[39]计算后,将每个层次计算结果通过全连接层(即图中的FC2至FC5)映射为一维的向量,再经过可学习的加权求和,得到最终的输出结果Y,也就是预测得分。
图1HDGICN总体架构图
Fig.1Architecture of HDGICN
1.3 特征增强模块
前人的研究表明,个性化PageRank(PPR)是一种提高图卷积网络在异亲生物分子网络上预测性能的有效方法[23]。在HDGICN的特征增强模块中,本研究使用PPR算法创建一个辅助网络,这个辅助网络捕捉了基因之间的额外结构相似性,从而能使HDGICN捕捉到更多不同的生物分子相互作用。DGI是一种无监督的图表示学习算法,用于学习图数据中节点的表征。它的目的是通过最大化图数据中节点的信息量来学习节点的特征表示,通常用于图卷积网络中的特征增强模块,通过捕捉图数据的全局结构信息,提高模型对图数据的理解和表征能力。在本研究中,特征增强模块将PPR与DGI相结合以获取全面的异亲生物分子网络的结构信息,从而增强基因特征矩阵,使得HDGICN拥有更强的异亲适应性。
1.3.1 图信息最大化
本研究设计的DGI计算过程如图2所示,通过构建一个对比学习任务来最大化图数据中节点的信息量。对比学习任务的目标是让模型将相似的基因节点对彼此靠近,将不相似的基因节点对彼此分开。具体来说,本研究首先将生物分子网络(这里指异亲生物分子网络或其辅助网络)和基因特征矩阵X∈(其中 N 是节点数目,F是特征维度)输入到编码器1(Encoder1)中以生成正样本的特征编码,同时将他们输入到随机扰乱模块(Corruption)再经过编码器2(Encoder2)后生成负样本的特征编码,然后联合使用总结摘要模块(Summary)和判别器(Discriminator)来计算正样本与负样本的互信息(互信息衡量了目标节点与其负样本节点之间的相似性和关联程度),最后利用Adam梯度下降优化算法,根据互信息的值来更新编码器网络的参数,使得目标节点与其负样本节点之间的互信息最大化。
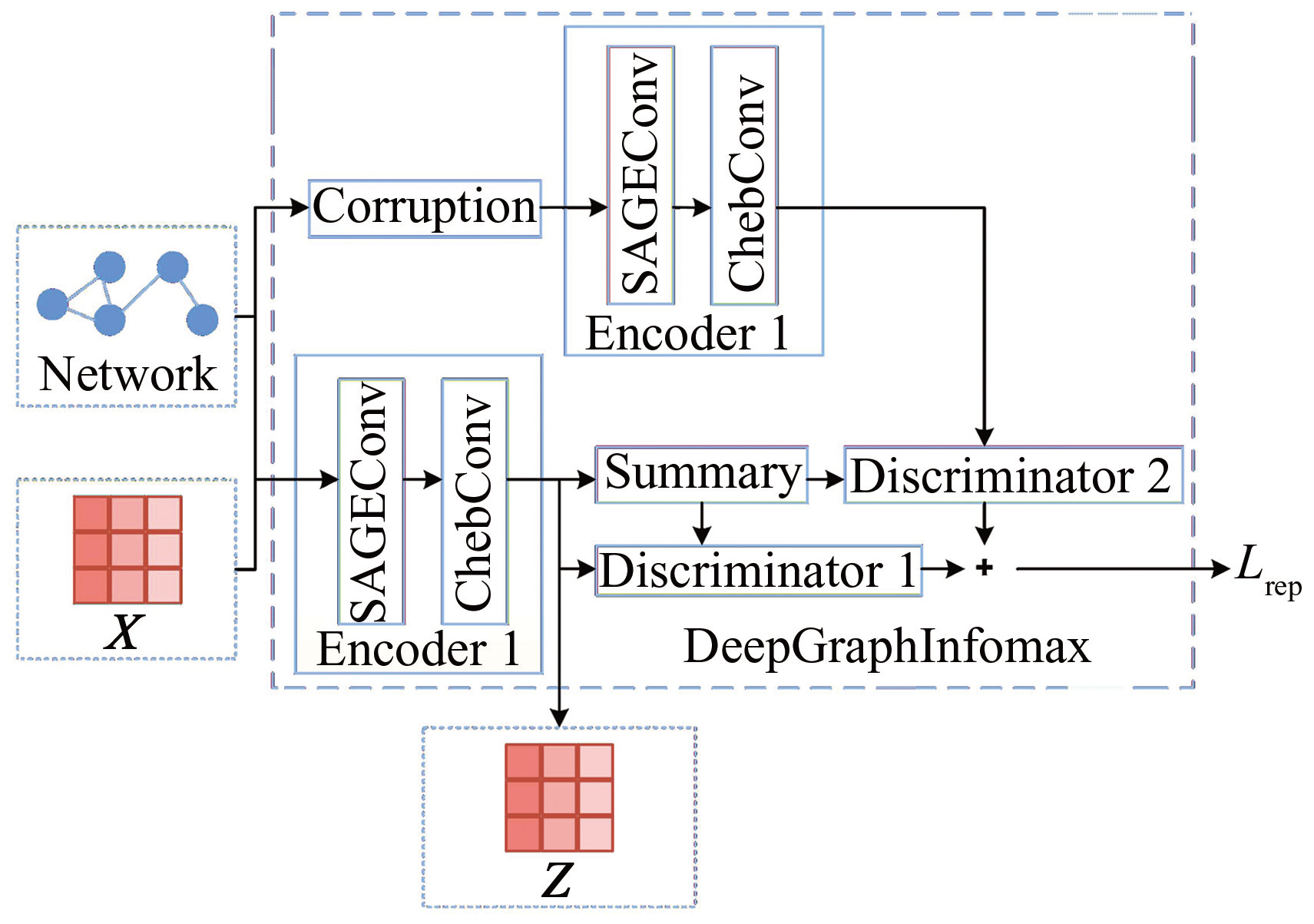
图2利用DeepGraphInfomax获得增强特征矩阵的过程
Fig.2Process of obtaining the enhanced matrix using DeepGraphInfomax
1.3.2 编码器
对比学习任务中的编码器主要由SAGEConv和ChebConv组成,用于捕捉网络中的复杂结构,例如分子间的各种相互作用和不同的功能关联。一方面SAGEConv通过进行邻居采样和聚合来计算基因节点表示以捕捉大规模网络中的局部信息[39]。另一方面,基于切比雪夫多项式的图卷积(ChebConv)是一种具有强空间不变性的卷积方法[21],在编码器中它被用于增强SAGEConv捕捉的局部基因节点信息,进一步提升模型对异亲图结构的学习能力。在编码器中,SAGEConv的计算公式如下:
(1)
(2)
其中,x′i和xi分别表示基因节点i的嵌入表示向量和特征向量,表示节点集N(i)中相邻基因节点的平均聚集,W1和W2都是可学习权重,hi表示对x′i进行归一化得到的归一化基因节点特征向量。编码器的第二层,即ChebConv,可以表示为
(3)
其中,代表ChebConv学习到的特征表示,参数K控制Chebyshev过滤器的大小,Θ是一个权重矩阵。而Z(k)是一个递归计算方法,其公式可表示为
(4)
(5)
(6)
其中,指从前一层(也就是SAGEConv)得到的基因特征表示。指缩放归一化的拉普拉斯矩阵,这里的λmax为拉普拉斯矩阵L的最大特征值,I为一个单位矩阵。
对比学习任务中的Corruption模块的计算方法为,其中π表示随机排列顺序,X(π)表示根据排列顺序π对X进行重新排序,得到负样本特征矩阵。Summary沿第一个维度计算正样本特征编码的平均值,其结果是一个与基因节点特征相同维度的58维特征向量。这个向量可以看作是网络中所有基因节点的平均表示,概括了整个网络的特征。随后本研究将与正样本特征编码一同输入判别器1,将与负样本特征编码一同输入到判别器2,以计算正样本与摘要信息的相似概率得分和计算负样本与摘要信息的相似概率得分,其过程可以用公式表示为
(7)
其中,p表示基因节点是样本与摘要信息的相似概率得分,表示基因节点样本表示,既可以是负样本基因节点特征编码,也可以是正样本基因节点的特征编码。W是一个权重矩阵。
为了最大化正样本与负样本之间的互信息,本研究设计了一个目标函数,其计算公式为
(8)
其中,Lrep表示两者概率分布之间的差异值。表示所有正样本基因节点与摘要信息的相似概率分数组成的向量,表示所有负样本基因节点与摘要信息的相似概率分数组成的向量。本研究通过Adam梯度下降算法优化该目标函数,实现正样本与负样本的互信息最大化。最终,本研究将最优情况下的正样本特征编码作为DGI的输出。
1.4 下游节点预测任务
如图1所示,在HDGICN的下游节点预测任务中,本研究使用SAGEConv和GCNConv学习异亲生物分子网络和辅助网络增强特征空间中的基因表示。每个SAGEConv的设置与公式(1)和公式(2)一致,用于捕获异亲生物分子网络的增强特征空间局部信息。而每个GCNConv用于整合辅助网络中相邻节点的信息,更新基因节点表示,其计算过程由下式定义:
(9)
其中,表示由邻接矩阵导出的对角矩阵,而邻接矩阵为辅助网络邻接矩阵Aaux加上单位矩阵I所得,是第l层的输出,Θ(l)是第l层的可学习的权重矩阵。为了避免多层图卷积模块的叠加引起增强特征的原始信息流失,本研究在图卷积模块之间引入了一种特殊的双重残差连接结构计算每层异亲生物分子网络和辅助网络的综合信息,再通过全连接层将每层的综合信息加权求和得到每个基因节点的预测得分,最后通过使用Adam梯度下降法来优化交叉熵损失函数从而实现模型的优化。
1.4.1 双重残差结构
HDGICN在将基因表示传播到下一层之前引入了残差连接方案。具体来说,在每一层中,分别将SAGEConv和GCNConv的输出和与第一层全连接层通过残差连接相加,得到新的表示和。接下来,将该层的和拼接成,作为一个综合的信息表示。然后,每一层的综合信息再通过残差连接与第一层的综合信息相加,从而创建一个独特的双重残差结构,促进消息传播。该过程的计算公式可表示为
(10)
(11)
(12)
其中H′enh表示第一个全连接层的输出,Henh代表增强基因特征的节点表示,ReLU(·)是一个激活函数,W和b分别表示全连接层的权重和偏差项。表示经过残差连接后的第l+1层综合信息,表示第一层综合信息。为了区别于加法,本文使用代表节点表示的按元素相加,使用‖代表节点表示的拼接操作。
接着,本研究分别对H′enh和(l=1,2,3)应用全连接层将其维度降至1维,再通过加权求和得到最终预测值。这一过程可用公式表述如下:
(13)
其中,σ(·)表示sigmoid激活函数,α(l)表示第l层自定义初始值的可学习权重,W(l)和b(l)分别表示第l层全连接层的权重和偏置项,K表示SAGEConv和GCNConv的层数。
1.4.2 损失函数
在下游节点预测任务中,本研究使用交叉熵损失函数计算预测值与真实值之间的损失,其计算公式如下:
(14)
其中,Lpred表示预测值与真实值之间的损失,n表示基因节点的数量,yi表示基因节点i的真实标签,表示该基因节点的标签预测值。
1.5 开发及运行环境
本研究所有方法都是基于Python3.9以及Pytorch2.0编写的,在VSCode上进行的代码调试和运行,操作系统使用Windows10,CPU为英特尔酷睿i3-12100F,GPU为NVIDIA GeForce RTX 3060,内存和显存分别为16 GB和12 GB。
2 实验与结果
2.1 参数设置
为了达到最佳性能,本研究在经过广泛实验调整后,对HDGICN模型的特征增强模块和下游节点预测任务进行了参数优化。具体来说,在特征增强模块中,参数主要有编码器隐藏层大小h1、Chebyshev滤波器参数K、学习率lr1、训练轮数e1;而在下游节点预测任务中,参数主要有隐藏层大小h2、dropout率d、学习率lr2、权重衰减wd以及训练轮数e2。本文基于常用参数设置经验设置参数取值范围,具体而言,考虑这些参数在h1,h2∈(48,58,100,200,300,400)、K∈(2,3)、lr1,lr2∈(0.01,0.001,0.0001)、e1,e2∈(100,300,500,1000,1500)、d∈(0.2,0.3,0.5)、wd∈(0.0005,0.00005)范围内的不同组合。当达到最佳性能时,HDGIN模型的参数取值为:h1=58、h2=200、K=2、lr1,lr2=0.001、e1=1000、e2=300、d=0.5、wd=0.00005。为了进行公平对比,本文用于对比的模型均遵循前人的参数设置[23]。
2.2 方法性能比较
为了评估HDGICN的性能,本研究在三个数据集(GGNet,PathNet和PPNet)上使用经典的机器学习模型、常见的深度学习模型和HDGICN模型进行癌症驱动基因预测。具体而言,本研究在数据集上使用了十次五折交叉验证法,通过在测试集上计算十次五折的平均AUROC和平均AUPRC来比较它们的性能,AUROC和AUPRC均是评估二分类任务性能的常用指标,且在类别不平衡条件下仍能提供可靠的性能评估。如图3(a)和图3(b)所示,本研究将HDGICN与逻辑回归(LR)、决策树(DT)、支持向量机(SVM)、K近邻算法(KNN)进行了对比,可以看出HDGICN模型预测效果明显比经典机器学习模型更好,相比于各数据集上表现最好的机器学习模型,HDGICN的AUROC在GGNet上提升了0.125 4,在PathNet上提升了0.1313,在PPNet上提升了0.1313,HDGICN的AUPRC在GGNet上提升了0.0552,在PathNet上提升了0.0726,在PPNet上提升了0.0730。
此外,本研究将HDGICN与本领域内常见的深度学习模型进行了比较,由于本研究的数据集与HGDC[23]一致,为了公平对比,本文使用了HGDC报告的深度学习模型运行结果。对比结果如图3(c)和图3(d)所示, HDGICN相比于性能排名第二的HGDC,其AUROC在GGNet,PathNet,PPNet上分别提升了0.0143,0.0022,0.0081,其AUPRC在GGNet,PathNet,PPNet上分别提升了0.0166,0.0119,0.0186,可见HDGICN相对于性能第二的HGDC有着更好的性能,但总体而言相差不大,这或许是因为HGDC与HDGICN采用了类似的方法来适应异亲生物分子网络。具体而言,它们都利用了PPR算法进行随机游走生成辅助网络,然后将其与原始网络一同用于模型训练,以提高图神经网络模型对异亲数据的适应性。然而,HDGICN引入了一种图信息最大化的方法来解决HGDC中捕获相关远程基因节点能力不足的问题,从而使其在异亲适应性方面表现更优。这体现在HDGICN在三个数据集上的AUROC和AUPRC差距最多不超过0.009和0.0469,而HGDC的最大差距为0.0127和0.0516。总体而言,HDGICN在这三个数据集上的AUROC和AUPRC结果相对于HGDC更接近,最大差距分别减小了0.0037和0.0389。最后,需要注意的是由于正负样本数量的不平衡,各个模型可能会更倾向于将正样本误分类为样本数量较多的负样本,导致模型出现偏斜,这也是HDGICN与HGDC性能收敛于相似水平的原因之一,即它限制了模型的性能提升潜力。对于其他深度学习模型,它们在GGNet上的AUROC和AUPRC与HDGICN相比效果差距较大,但在PathNet和PPNet上的表现却接近HDGICN,而HDGICN在三个异亲生物分子网络上都能实现最优且相近的效果,这进一步说明了HDGICN在适应异亲生物分子网络方面具有更强的能力。相比而言,每个机器学习模型也能在三个数据集上取得相近的效果,但性能远比有异亲生物分子网络适应性的深度学习模型差,这是因为机器学习模型不依靠生物分子网络而仅仅依靠生物特征进行训练,从而使得它们无法学习到异亲生物分子网络的结构特征,最终导致它们无法拥有更好的拟合能力。

图3方法性能对比
Fig.3Methods performance comparison
注:(a)经典机器学习模型的平均ROC曲线下面积对比;(b)经典机器学习模型的平均PR曲线下面积对比;(c)常见深度学习模型的平均ROC曲线下面积对比;(d)常见深度学习模型的平均PR曲线下面积对比.
2.3 消融实验
为了验证HDGICN的各个模块是否有助于提升模型性能,本研究设计了以下HDGICN的变种模型进行对比:
1)无特征增强模块(No feature enhancement,NFE):该变种模型将不使用特征增强模块进行模型训练。
2)无残差结构(No residual structure,NRS):该变种模型将不使用任何残差结构进行模型训练。
3)只使用逻辑回归作为下游预测任务(Logistic regression for prediction,LRP):该变种模型的下游预测任务将只使用逻辑回归输出每个基因的预测得分。
如表1所示,与完整的HDGICN相比,NRS的AUROC平均下降了约1.35%,AUPRC平均下降了约1.1%。同时,LRP的AUROC平均下降了约3.42%,AUPRC平均下降了约4.49%。这些结果表明,NRS和LRP的性能明显较差,突显了HDGICN的残差连接结构和完整的下游预测任务对提升模型性能的重要性。值得注意的是,在GGNet上,NFE的性能与完整的HDGICN模型相差不大,其AUROC只有0.09%的差异,AUPRC差异为0.21%。然而在PathNet和PPNet上,NFE的AUROC比完整模型平均低了1.32%,AUPRC比完整模型平均低了1.21%。这表明NFE的泛化能力相对较弱,无法适应所有异亲生物分子网络,说明特征增强模块在提高模型泛化能力方面有着一定作用。由此可见,HDGICN的各个模块都为其最佳性能做出了积极的贡献。
表1消融实验
Table1Ablation experiments
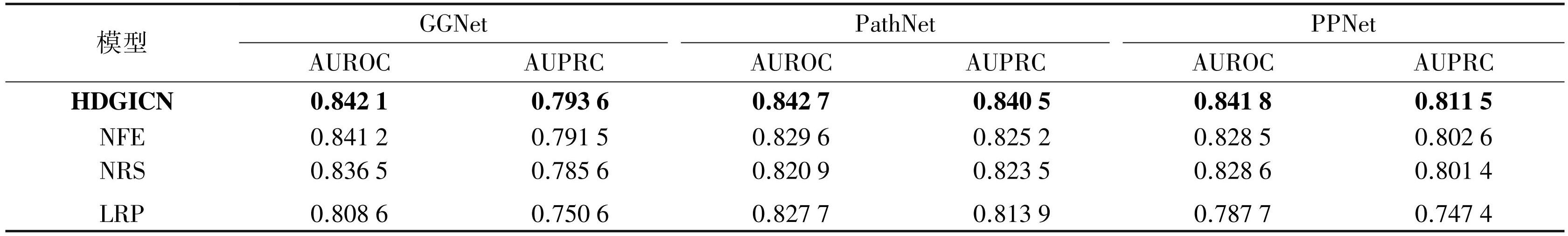
注:图表内加粗的数据为最佳性能。
2.4 预测的癌症驱动基因
在本节中,本研究运用HDGICN模型进行了新的癌症驱动基因的预测工作,覆盖了GGNet,PathNet和PPNet三个数据集。具体而言,本研究在保持所有参数不变的情况下,对这三个数据集进行了100轮的模型训练和预测操作。在每轮训练结束后,记录相应的预测得分。最后,本研究对每个数据集的无标签数据进行了100轮预测得分的平均处理,从而生成了三个独立的预测结果表。如图4(a)所示,本研究计算了三个数据集的预测得分表中分别排名前100的基因的交集,并将每个数据集合的基因与最新的NCG7.1数据库[40]进行了对比,发现在两个及以上相交的数据集合中的基因是候选癌症驱动基因的比例相对更高。为了验证HDGICN预测新癌症驱动基因的能力,本研究在这些交集中选取预测得分最高的前30个基因作为分析对象。首先,本研究计算了这30个基因在三个异亲生物分子网络中与已知的癌症驱动基因的直接交互数量,其结果如图4(b)所示,本研究发现这30个基因除了FLG外的所有基因都与癌症驱动基因有着直接相互作用,且在GGNet上与癌症驱动基因交互最多,说明HDGICN能够根据异亲生物分子网络中基因节点之间的关联预测新癌症驱动基因。为了证实这30个基因与癌症的相关性,本研究接着将这30个基因与NCG7.1数据库进行比对,发现其中有24个基因已被认定为候选癌症驱动基因(包括FLG),而候选癌症驱动基因在NCG7.1中已被证实与癌症有关。为了探究其余6个非候选癌症驱动基因(TTN,RYR2,LRP2,PCLO,UBR4,RYR3)与癌症的关联以及FLG为什么在数据集中与癌症驱动基因无直接交互的情况下被分类为驱动基因,本文分析了它们的癌症特异性生物特征。由于PPNet完全包含这七个基因,而GGNet和PathNet不完全包含这七个基因,所以为了获取最全的基因特征信息,如图4(c)所示,本研究首先将PPNet上预测得分排名前100的无标签基因的癌症特异性特征以热力图的形式来展示,并标出了这7个基因在热力图中的位置。随后通过观察发现这7个基因在16种癌症的特异性突变频率上相较于其它基因更高,其中RYR2在16种癌症组织中特异性DNA甲基化水平上相较于其他基因更高,说明HDGICN将这7个基因分类为癌症驱动基因的原因是其癌症特征较为显著。
最后,为了进一步证实6个非癌症候选基因与癌症的关联,本研究对它们使用Metascape[41]进行了癌症表达注释, Metascape主要通过The Human Protein Atlas(ProteinAtlas)[42]提供的免疫组织化学表达数据进行注释。在注释信息中,本研究发现TTN在多种癌症组织中表现出弱到中度的细胞质染色,包括皮肤癌、肝癌和黑色素瘤,此外,HAN等[43]最近的研究表明TTN基因突变是甲状腺癌(THCA)的独立风险因素。RYR2在大多数子宫内膜癌中表现出弱到中度的细胞质免疫活性,并在少数结直肠癌、肝癌和甲状腺癌中也有表达,最近的研究还验证了RYR2在食管鳞状细胞癌(ESCC)中的功能相关性[44]。PCLO在大多数恶性肿瘤中表现出强烈的细胞质和膜染色,且在恶性黑色素瘤中呈弱到中度的阳性表达,此外有研究表明PCLO在弥漫性大B细胞淋巴瘤(DLBCL)的发生中发挥了功能作用[45]。LRP2在大多数肾癌中表现出强烈的细胞质染色,且有研究表明LRP2突变可以作为肿瘤免疫治疗的生物标志物[46]。对于RYR3,虽然在ProteinAtlas的蛋白质表达分析中没有找到其与癌症相关的证据,但通过观察在ProteinAtlas中展示的TCGA癌症组织数据发现其在胶质瘤中富集。UBR4在前列腺癌、黑色素瘤、尿路上皮癌、胃癌、胰腺癌、肝癌和女性生殖器官癌中表现出中度到强烈的免疫反应。由于数据的及时性,以上基因的一些蛋白质表达信息没有在Metascape提供的注释表中进行标注,但仍然可以在ProteinAtlas中通过搜索蛋白质的表达信息进行验证。这些发现为将这六个基因与癌症联系起来提供了初步假设,但将他们正式确定为癌症驱动基因还需要进一步生物实验验证。
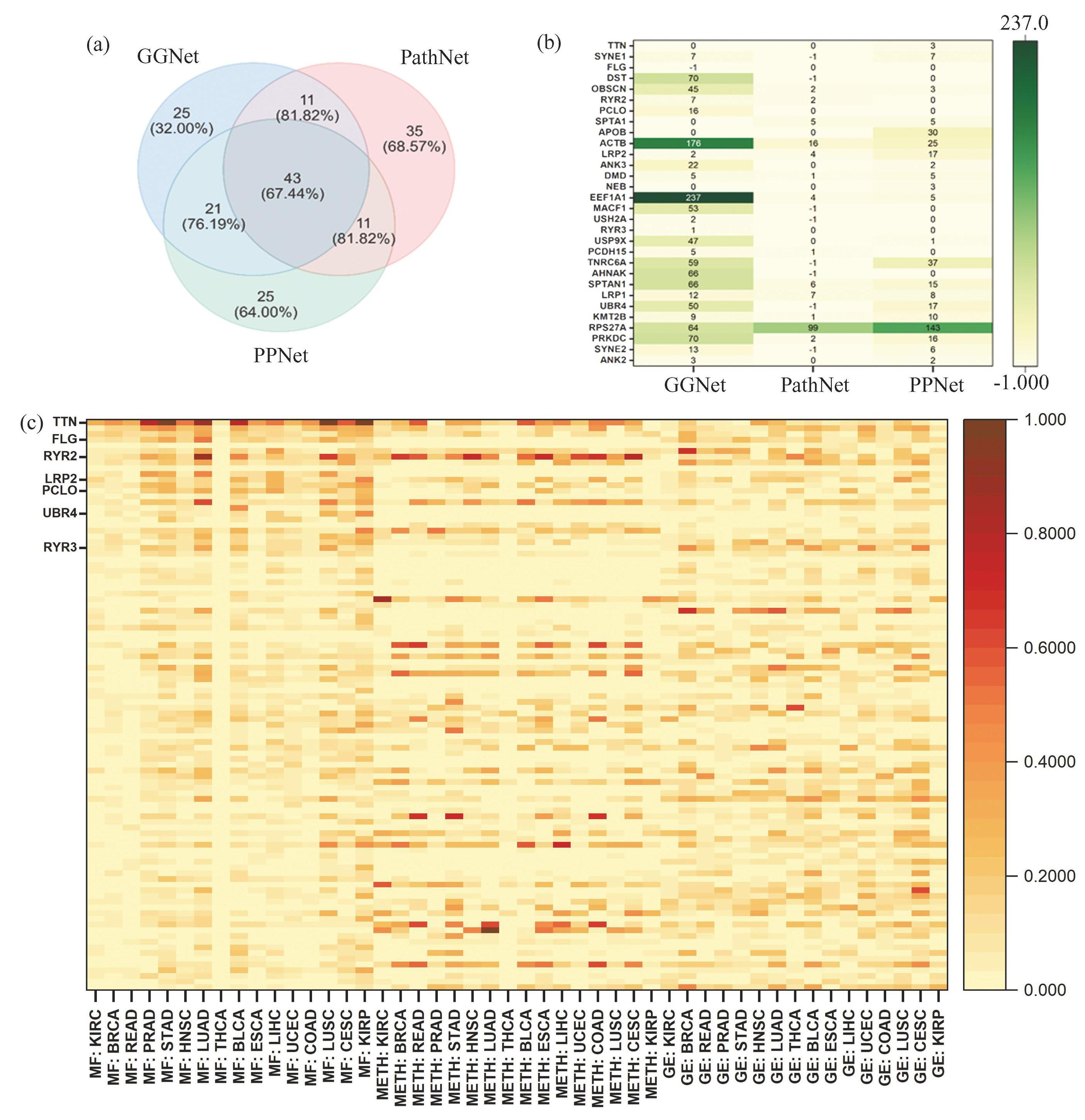
图4预测的癌症驱动基因特征分析
Fig.4Characterization of predicted cancer driver genes
注:(a)三个数据集的预测得分表中排名前100的基因交集维恩图,括号中的百分比表示该区域的基因是癌症候选驱动基因的比例; (b)预测得分排名前100的基因中,取在三个数据集上有交集的前30个基因,计算它们与已知癌症驱动基因的直接交互数量所生成的热力图。其中的数字代表基因与已知癌症驱动基因的直接交互数量,-1代表该基因在该数据集中不存在,0表示该基因与已知癌症驱动基因无直接交互; (c)在PPNet数据集上预测分数排名前100的基因的癌症特异性特征热力图,横坐标中MF表示基因突变频率,METH表示DNA甲基化水平,GE表示基因差异表达水平.
2.5 数据可用性
HDGICN的代码和数据均可在https://github.com/InterXander/HDGICN上获取。
3 讨论
癌症驱动基因的鉴定对于精准治疗和生物标志物的发现非常重要。大规模的癌症研究项目提供了宝贵的多组学数据,推动了计算方法的发展。然而,当前的方法在数据全局特征学习、模型泛化和稳健性方面存在局限。因此,需要一种新的计算方法来增强癌症驱动基因的鉴定。本研究设计并开发出了HDGICN,这是一种用于识别癌症驱动基因的新方法。 HDGICN在三个异亲生物分子网络(GGNet,PathNet和PPNet)上进行训练,利用特征增强模块生成辅助网络,并捕获全局结构信息以增强原始特征。在预测过程中,本研究采用了混合图卷积网络的方式,结合了SAGEConv和GCNConv的优点,并利用双重残差结构提升了模型性能,最后通过降维加权求和的方式综合模型中所有卷积层的输出信息来预测基因节点是否是癌症驱动基因。如图3所示,本研究进行了全面的比较实验,证明了与现有方法相比,HDGICN在预测癌症驱动基因方面具有优越的性能。表1中的消融实验证实了HDGICN的优越性能得益于特征增强模块学习全局结构信息的能力和下游任务的总结能力。最后,本研究利用HDGICN预测了新的癌症驱动基因,并分析了其被鉴定为癌症驱动基因的原因,经过调查研究后发现其中的基因已从多方面被证实与癌症有关,这将为癌症的精准治疗和生物标志物的发现提供重要帮助。虽然HDGICN方法显示出了优越的性能,但也需要承认由于不平衡的数据样本而导致的局限性,使得其预测性能还有较大的提升空间。再者,由于模型的网络结构的泛化性不足、生物分子网络数据质量的变化等原因,可能会出现一个基因在一个生物分子网络中被识别为驱动基因,但是在其他的生物分子网络中不能被识别为驱动基因的情况。而要避免这种情况,则需要尽可能完善模型的网络结构、丰富数据。未来的工作将集中在解决这些挑战,进一步提高HDGICN在预测癌症驱动基因方面的准确性和可靠性。
4 结论
本文引入了一种新的用于识别癌症驱动基因的方法HDGICN。通过全面的比较实验,本文证明了它相比其他方法有着更优越的性能,且它的各个模块在训练过程中均有助于提升性能。本文进一步将HDGICN应用于预测新的癌症驱动基因,通过对其生成的基因列表进行分析证实了它们与癌症的关联。HDGICN将在癌症驱动基因鉴定领域发挥重要作用并对精准治疗和生物标志物的发现提供重要帮助。

