摘要
细胞类型注释是单细胞RNA测序(scRNA-Seq)分析的基本任务。为了解决处理稀疏数据时出现的性能下降以及计算复杂度较高的问题,本文提出了一种基于深度学习模型Transformer的scRNA-seq数据的细胞类型识别和注释工具,scTransformer。模型包含四个模块,即基因嵌入、位置编码、变换编码器和分类层;基因嵌入过程将K个高变异基因(HVG)(K=2000)处理为N个子向量;以未分配率、F1分数、准确度、kappa分数、AUR指标作为评判标准,系统评估模型和其他9种工具的性能。结果表明:在数据集内,scTransformer的准确度达到96.59%,高于其他工具,未分配率达到了0.18%;可能因为样本的不均衡,其平均F1分数为93.46%,低于CHETAH,Clustifyr和SciBet;在跨平台相同组织间测试和完全不同组织间测试中(胰腺、血液),scTransformer的准确率、F1分数和kappa系数均是最好的(>0.99);在小鼠大脑、胰腺、肺组织中,scTransformer的AUR和未分配率仅次于Seurat工具和Clustifyr工具。scTransformer源代码和数据位于https://github.com/nanjingyuanbao/scTransformer。综上,本文提出并系统评估了一种新的基于Transformer的细胞类型注释工具。
Abstract
Cell type annotation is an essential task for single-cell RNA sequencing (scRNA-Seq) analysis. In this paper, we propose a tool for cell type identification of scRNA-Seq data, which is based on the Transformer, in order to overcome the performance degradation and high computational complexity that occur when dealing with sparse data. The model includes four modules, which are Gene Embedding, Position Encoding, Transformer Encoder and Classification Layer; The Gene Embedding process processes K Highly Variants Genes (HVGs) (K=2000) into N sub-vectors; Unassigned rate, F1 score, accuracy, kappa score and AUR metrics are used as evaluation criteria to systematically assess the performance of the model and the other nine tools. The results show that within the dataset, scTransformer achieve 96.59% accuracy, which is higher than the other tools, and the unassigned rate reaches 0.18%. Probably due to the imbalance of the samples, its average F1 score is 93.46%, which is lower than that of CHETAH, Clustifyr and SciBet; In the cross-platform same-organisation inter-tissue test and the completely different-organisation inter-tissue test (pancreas, blood), scTransformer has the best accuracy, F1 score and kappa coefficient (>0.99); In mouse brain, pancreas and lung tissues, scTransformer's AUR and unassigned rate are second only to those of the Seurat tool and the Clustifyr tool. scTransformer source code and data are available at https://github.com/nanjingyuanbao/scTransformer. In conclusion, this paper presents and systematically evaluates a new Transformer-based cell type annotation tool.
Keywords
高通量单细胞测序技术在过去十年中发展迅速[1]。测序数据样本的规模从几十个增加到数千个和数百万个,并且出现了许多新的测序平台,例如Drop-seq,inDrop和10X Genomics Chromium[2-4]。细胞类型的鉴定在单细胞RNA测序数据的分析中起着重要作用,精确注释的单细胞测序数据可以使生物学家能够进行进一步的下游分析,提高对疾病细胞机制的理解[5]。
在scRNA-seq开发之前,细胞分类的传统方法依赖于显微镜、组织学和病理学标准[6]。在免疫学领域,细胞表面标记物被广泛用于区分细胞亚型,以达到多种目的[7]。虽然这种方法在细胞分离(如荧光激活细胞分选)的实际应用中是可取的,但这些标记物可能无法反映混合细胞群在转录组和表型水平上的整体异质性[8-9]。无监督和有监督聚类方法已被用于根据样本中相似的转录特征确定细胞群[10-11],而且经常根据典型标记物的平均表达水平对聚类中的细胞进行集体标记[12]。
利用scRNA-seq数据进行细胞类型鉴定。目前的策略主要分为两类,第一类是在监督下根据基因表达谱的相似性将细胞聚类成簇,而细胞簇的注释则是通过人工给每个簇分配标签来实现的,这类方法包括Scanpy[13],Seurat[14],SIMLR[15],SC3[10]等。事实证明,这种方法在识别新细胞群方面很有价值[10,16-19]。然而,这一注释步骤繁琐且耗时,因为它涉及到对群集特异性标记基因的人工检查,并需要对已知细胞标记的先验知识。此外,人工注释通常不是基于标准化的细胞标记本体[20],在不同实验中无法重复[21]。第二类,半监督和无监督方法。scRNA-seq可提供所有基因的信息,在高维基因表达空间中比较细胞时,细胞间的距离变得更加均匀,难以区分群体间的差异,这限制了无监督方法的快速注释能力和可重复性[22-23]。半监督和无监督方法通过比较单个细胞与单细胞RNA-seq图谱参考数据库之间的相似性,确定潜在的细胞身份[24-25]。无监督方法在基础算法和先验知识(如细胞类型标记基因列表)的组合上有所不同[26]。例如,AranD等[27]提出的SingleR,通过计算参考数据集中每个细胞基因表达谱的相关性来识别细胞类型。DeKanter等[28]提出,CHETAH利用了scRNA-seq参考数据的分类树。在KiselevV.Y等[29]提出的scmap方法中,细胞是根据其与参考细胞类型的相似性,基于各种相关性度量进行分类的。Alquicira-Hernandez等[30]提出的scPred方法将识别特征与解决基因表达矩阵的方差结构相结合。Tan Y等[31]提出了使用随机森林分类器构建细胞分类模型的SingleCellNet。这些方法对数据稀疏性很敏感,可能忽略基因表达上的结构特征使性能下降[32-33]。
前面提到,现有的单细胞类型识别技术在处理数据稀疏性严重的转录组数据时,模型的性能可能会下降。为了解决该类问题,可以借助于深度学习模型Transformer,它在计算机视觉和自然语言处理等方面的学习任务中表现了突破性的性能[34-37],其优势在于能够以注意方式捕捉全局上下文信息,从而建立对目标的长程依赖。不过,基于Transformer的模型的计算复杂度较高,尤其应用于scRNA-seq数据[38-39]。因此,本文通过结合补丁概念,改进面向scRNA-seq数据的Transformer模型,降低其时间复杂度。结果,本文开发了基于Transformer的监督式细胞类型识别方法scTransformer,通过不同细胞类型注释情况下的系统比较,发现scTransformer的性能优于其他工具。
1 数据准备
1.1 数据收集
表1从不同的人类和小鼠组织收集的真实scRNA-seq数据集
Table1Real scRNA-seq datasets collected from different human and mouse tissues
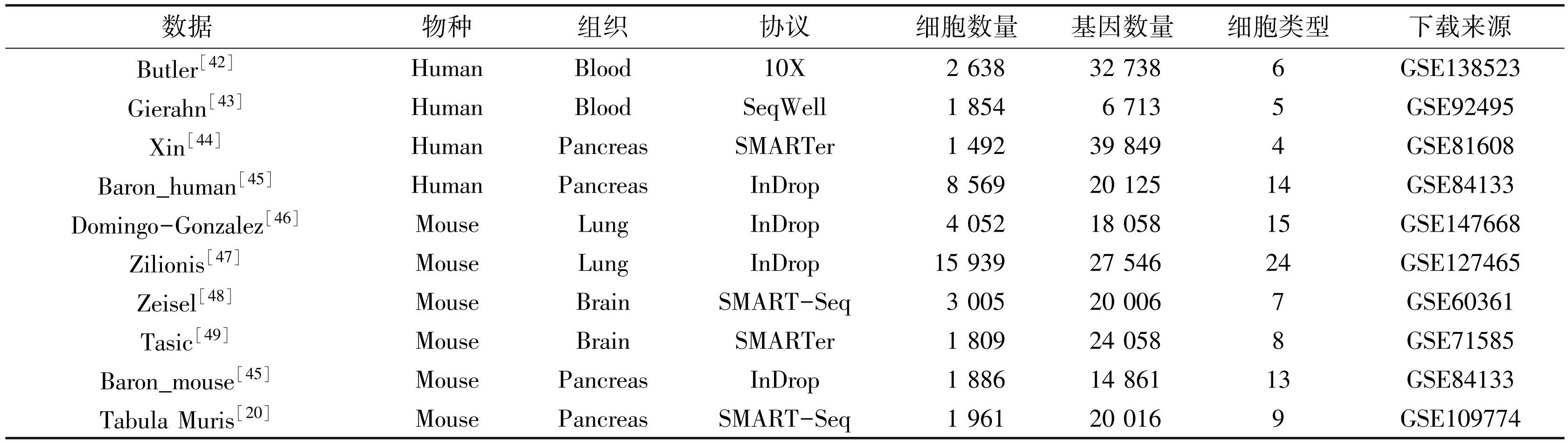
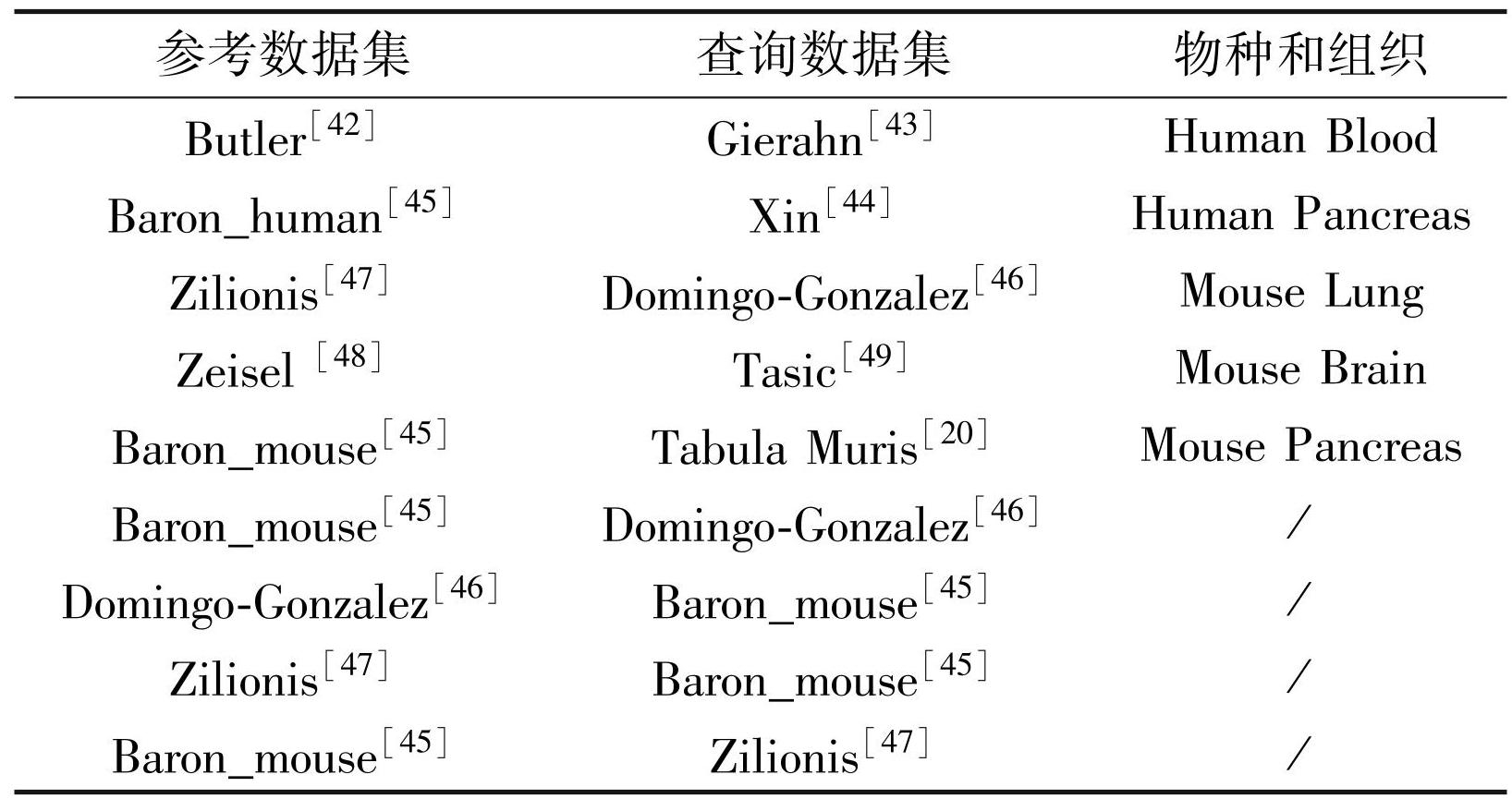
表2所有数据都使用训练-测试 scRNA-seq 对进行性能比较
Table2Performance comparison using train-test scRNA-seq pairs for all data
1.2 数据预处理
由于scRNA-seq数据具有较高的噪声,因此有必要在训练模型之前对scRNA-seq数据进行预处理。数据预处理过程主要有两个步骤。一是质量控制。首先要过滤基因表达值全为零的细胞,以及在任何细胞中都检测不到表达值的基因。此外,还需要过滤掉训练数据集中未标记的细胞。完成上述步骤后对原始数据进行归一化处理,以消除批次效应。本研究采用Seurat[14]方法进行数据归一化:每个细胞的特征计数除以该细胞的总计数,再乘以比例因子(默认为10 000)。接着对数值进行自然对数转换,最后选出前2 000个高变量基因[14]。
2 方法
2.1 scTransformer模型
scTransformer的工作流程如图1所示,scTransformer的参数如表3。
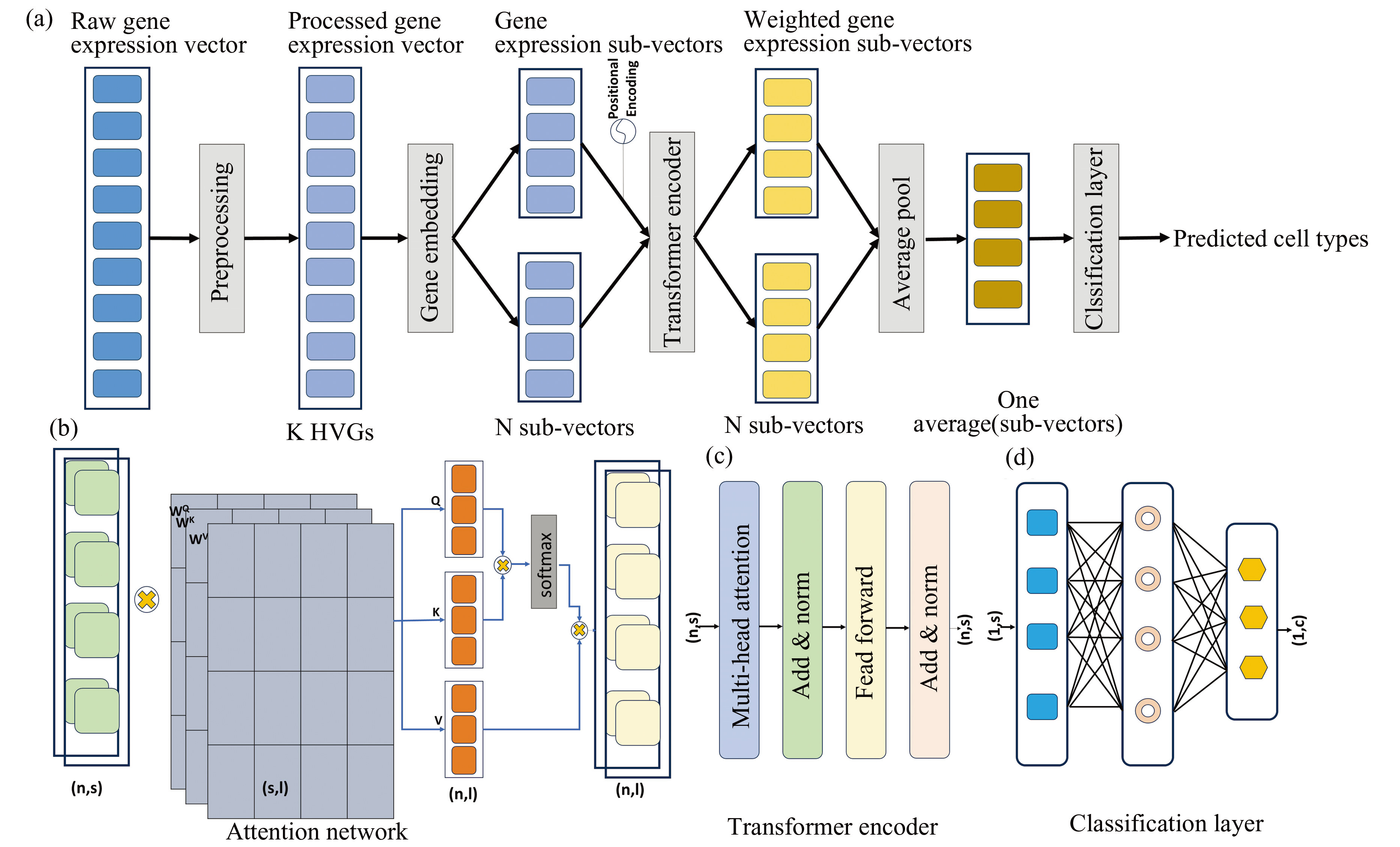
图1scTransformer结构示意图
Fig.1Schematic structure of scTransformer
注:(a)scTransformer基于深度学习框架,包含四个模块,即基因嵌入(Gene Embedding)、位置编码(Positional Encoding)、变换编码器(Transformer Encoder)和分类层(Classification Layer);(b)注意力网络,将维度为(n,s)的n个预处理子向量,通过注意力算法进行投影;(c)变换器编码器层,注意网络并行运行H次。前馈网络捕捉子向量间的所有非线性特征;(d)分类器层,两个线性连接网络, ReLU作为激活函数.
表3模型训练参数
Table3Model training parameters

2.1.1 基因嵌入
变换器编码器将一组实值向量作为输入。然而,在scRNA-seq数据中,输入是由单个基因的表达值组成的。在CV中,图像被直接分割成斑块作为变换编码器的输入,并在各种CV基准测试中取得了具有竞争力的性能[50]。受这项工作的启发,本文将每个细胞的基因表达向量划分为等长的子向量作为模型的输入。这样可以减少将单个基因表达值投射到单个细胞向量中产生的噪音,从而提高性能并节省训练时间。对于细胞i,Xi=(Xi,1,Xi,2,...,Xi,k)表示其基因表达向量,包括k个高变异基因。细胞i的第j个子向量表示为Xi,j=(Xi,j*s+0,Xi,j*s+1,...,Xi,j*s+s-j),其中j∈(0,...,k|s)。
2.1.2 位置编码
Xi,j被一次性输入模型,从而导致基因表达向量的阶次信息丢失。为了解决这个问题,本文用正弦函数来表示奇数子向量,用余弦函数来表示偶数子向量,计算公式如下:
(1)
(2)
其中, l代表在Xi,j中的位置,2i代表偶数子向量,2i+1代表奇数子向量。通过对基因表达向量应用正弦和余弦函数,可以有效地将位置信息嵌入到基因表达向量中,然后利用这些信息做出更明智的预测。Xij加上通过位置编码处理的Xij变为Xposi作为TransformerEncoder层的输入,计算公式如下:
(3)
2.1.3 变换编码器的功能
(4)
(5)
其中headh由公式(6)计算得出:
(6)
经过多头注意力后,通过残差连接和层归一化过程,将Xposi转化为Xattention作为前馈网络的输入,具体如下:
(7)
自注意力网络有助于捕捉子向量之间元素的依赖关系。然而,它可能不足以捕捉子向量之间的所有非线性特征。为此,本研究增加了前馈网络。前馈网络由两个线性层组成,它们之间有一个整流线性激活函数,由公式(8)计算得到:
(8)
2.1.4 分类层的功能
在分类层中,平均汇集层用于计算所有子向量的平均值:
(9)
然后,本研究使用两个线性连接网络和ReLU[53]作为激活函数:
(10)
2.2 评估指标
假设测试数据集中有m个细胞和k种细胞类型。对于每种细胞类型c,将属于该细胞类型的样本称为阳性样本,不属于该细胞类型的样本称为阴性样本(TP和FP表示正确和错误分类的阳性样本,TN和FN表示正确和错误分类的阴性样本)。本研究选择以下五个指标来评估多类分类问题。
2.2.1 准确性
(11)
2.2.2 未分配率
未分配率[57]专门用于计算预测中分配为“未分配”的细胞比例。它最初出现在评估细胞类型标注方法性能的综述文章中[22,58],被称为“未标注”。其计算公式为公式(12)。Count是一个函数,用于计算查询数据集所有细胞中被分配为“未分配”的细胞数量。
(12)
2.2.3 AUR指标
AUR指标来自文献[59]。同一组织的现有scRNA-seq数据集可能既有相同的细胞类型,也有不同的细胞类型。当使用一个scRNA-seq数据集作为参考时,不能保证它包含了另一个查询数据集中的所有细胞类型。因此,在确保对参考数据集和查询数据集的相同细胞类型进行准确预测的同时,还需要确保由参考数据集训练的模型在遇到两者包含的不同细胞类型时能准确识别“未标注”。因此,引入了一个新指标AUR,以同时衡量相同细胞类型之间的预测准确率和不同细胞类型之间的未分配率。在模型对比过程中,首先要去除查询数据中未标记的细胞,然后假设查询数据中仍有m个细胞。假设参考数据集和查询数据集分别包含k1和k2种细胞类型,其中有kc种相同的细胞类型;又假设查询数据中有m1个单元格与参考数据的单元格类型相同,其余单元格集包含m2个单元格。对于来自常见细胞类型(kc)的m1个细胞,计算准确率。对于非常见细胞类型的m2细胞(k2-kc),使用未分配率进行评估,因为它们应被标记为“未分配”。AUR是两个指标的加权和,权重是各部分单元格对应的单元格比例,计算公式为公式(13)。当kc等于零时,AUR退化为“未分配”率。当kc与k2相同时,AUR退化为准确率。为了使AUR在评估过程中更加可信,要尽量统一参考数据和查询数据中的细胞类型名称。
(13)
2.2.4 F1分数
F1分数是精确度和召回率的调和平均值。对于二元分类问题,F1分数的计算如下:
(14)
其中Precision和Recall分别由公式(15)和公式(16)计算得到:
(15)
(16)
至于多类分类问题,本文选择计算宏F1分数,其计算公式如下:
(17)
其中macro-P和marco_R分别由公式(18)和公式(19)计算得到:
(18)
(19)
2.2.5 kappa系数
kappa系数是多分类问题的一致性检验指标。假设参考数据集中有k种细胞类型,每种细胞类型中包含的真实样本数为a1,a2,......,ak。此外,每种细胞类型中的预测样本数为b1,b2,......,bk。因此,kappa系数计算公式如下:
(20)
其中pe和p0分别由公式(21)和公式(22)计算得到:
(21)
(22)
3 结果
scTransformer是一种基于Transformer的单细胞分配框架,可将基因表达向量分割成子向量。scRNA-seq数据集的预处理基于Seurat[14]方法。在数据集内部分析中,需要先过滤掉并非在所有细胞中都表达的基因,对基因表达数据进行归一化和对数变换,并选出前2 000个高变基因。对于数据集之间的任务,首先合并预处理后的参考数据集和查询数据集,然后选择前2 000个高变基因,最后将合并后的数据集拆分为参考数据集和查询数据集。预处理后的细胞基因表达向量被分成若干个子向量。最后,在参考数据集上训练scTransformer,并用它来注释查询数据集中的细胞。
使用scTransformer进行细胞类型分配的标注过程中,如果无法准确确定细胞类型,就会将其标记为“未分配”,以避免错误的分配并促进新细胞类型的识别。即使参考数据集和查询数据集包含的细胞类型数量不同,scTransformer也能准确标注细胞类型。为了证明scTransformer的能力和可扩展性,本文对来自不同物种和组织的多个scRNA-seq数据集进行了分析,将其与其他计算方法进行了比较,这些都是基于监督分类或相关性的方法,包括SingleR[60],Seurat[14],scmap[29],Clustifyr[61],CHETAH[28],SciBet[62],scLearn[57],scPred[30],SingleCellNet[31]。
3.1 数据内测试有效性
为了评估scTransformer的有效性,首先使用在不同物种、器官、组织、单细胞测序技术和细胞数量的10个数据集上预测细胞类型,所用数据如表1。
采用了5倍交叉验证策略,并通过准确性、宏观F1得分、未分配率作为指标来评估模型的性能。使用10个数据集上的每个细胞进行分类,图2(a)显示在Baron human[45],Tabula Muris[20],Baron mouse[45],Zilionis[47],Zeisel[48]这些数据上,scTransformer的准确度都表现最高, scTransformer在所有数据表现的平均准确度为96.59%。本研究还计算了各个识别工具在10个数据集上的平均F1分数,由图2(b)数据计算得出scTransformer的平均F1分数是93.46%,低于CHETAH[28],Clustifyr[61],SciBet[62]的93.78%,94.26%,96.35%,这可能是样本内的不平衡性引起的。有些模型会标记未被识别到的细胞,图3计算得scTransformer在所有数据的平均未分配律为0.18%,明显低于Clustifyr[61],scLearn[57],scmap[29],scPred[30],SingleCellNet[31],而SingleR[60],Seurat[14],SciBet[62]无法直接将未识别细胞标记为“未分配”。如图4所示,scTransformer能够准确预测几乎所有细胞类型,在Baron mouse[45]和Domingo-Gonzalez[46]这些数据集上经统计学显著检验发现宏观F1分数优于比较方法(P<0.05)。
图2scTransformer在不同数据集的性能比较
Fig.2Performance comparison of scTransformer across different dataset
注:(a)平均准确度比较的热图和箱线图;(b)平均F1分数比较的热图和箱线图.
图5展现了十个数据集上不同方法的运行时间。总体而言,与其他先进的方法相比,scTransformer的计算效率更高。对scTransformer运行时间的评估是在配备2.30 GHz、i7-12700 H处理器和16.0 GB内存的计算机上进行的。值得注意,scTransformer的计算效率在处理大规模 scRNA-seq数据时相比于其他方法较为显著,从而说明了其节省时间和内存的特点,且scTransformer对于那些使用传统计算机配置的用户较为友好。图6设置了不同的条件来比较模型的性能,以不同物种组织里的平均准确度为评判标准。因为基因表达向量切分输入的逻辑不太清晰,所以将基因表达向量不切分作为对比实验,结果表明将向量切分输入确实可以提高模型性能。本文参考相关文献[38-39,51-52],并设置注意力头数消融实验,结果表明将heads设置为64时模型的性能表现最好。
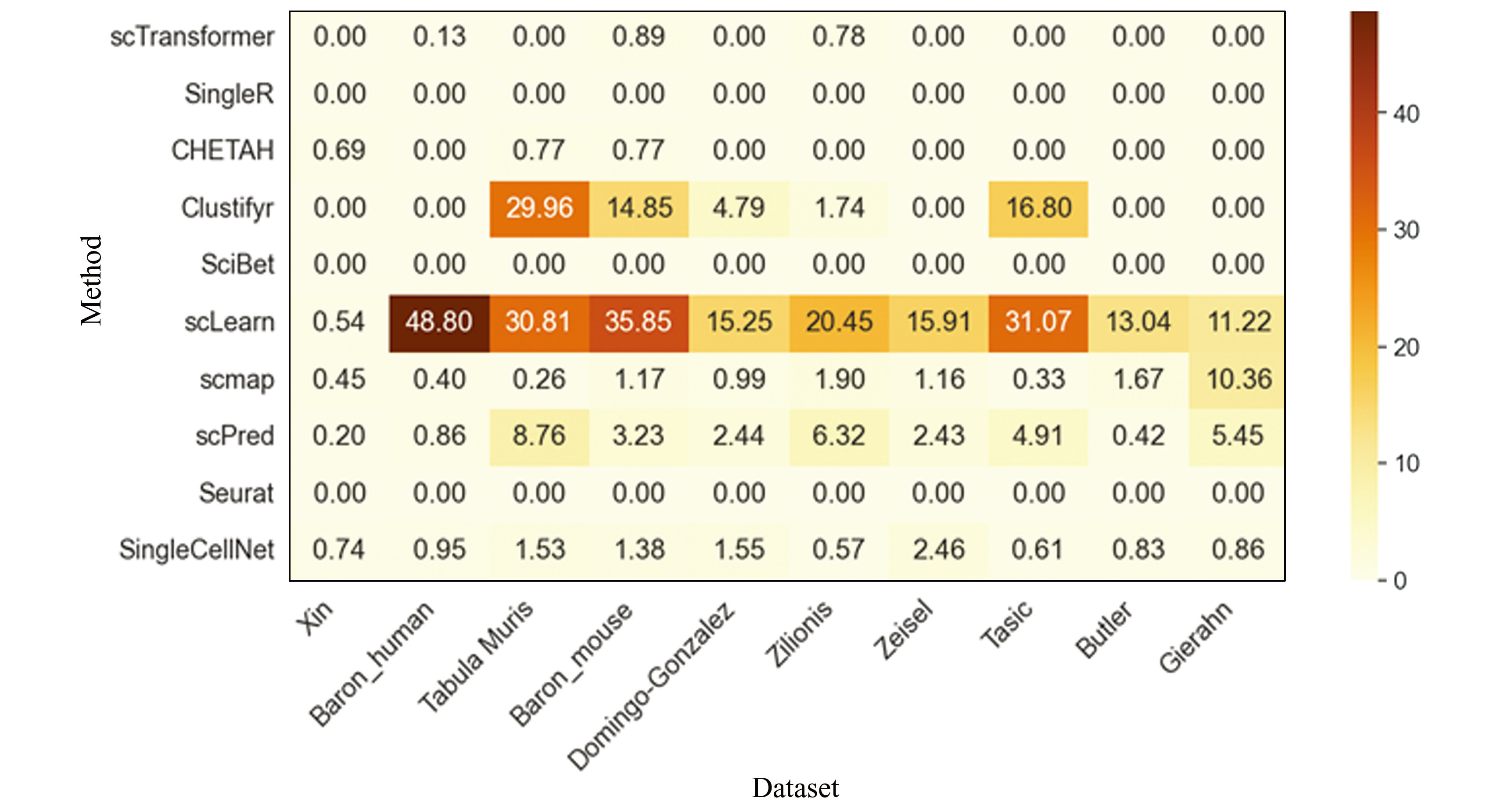
图3未分配率(百分比)的热图
Fig.3Heatmap of unassigned rates

图4箱线图展示数据集上不同细胞类型的F1分数
Fig.4Box plot showing F1 scores for different cell types on the dataset
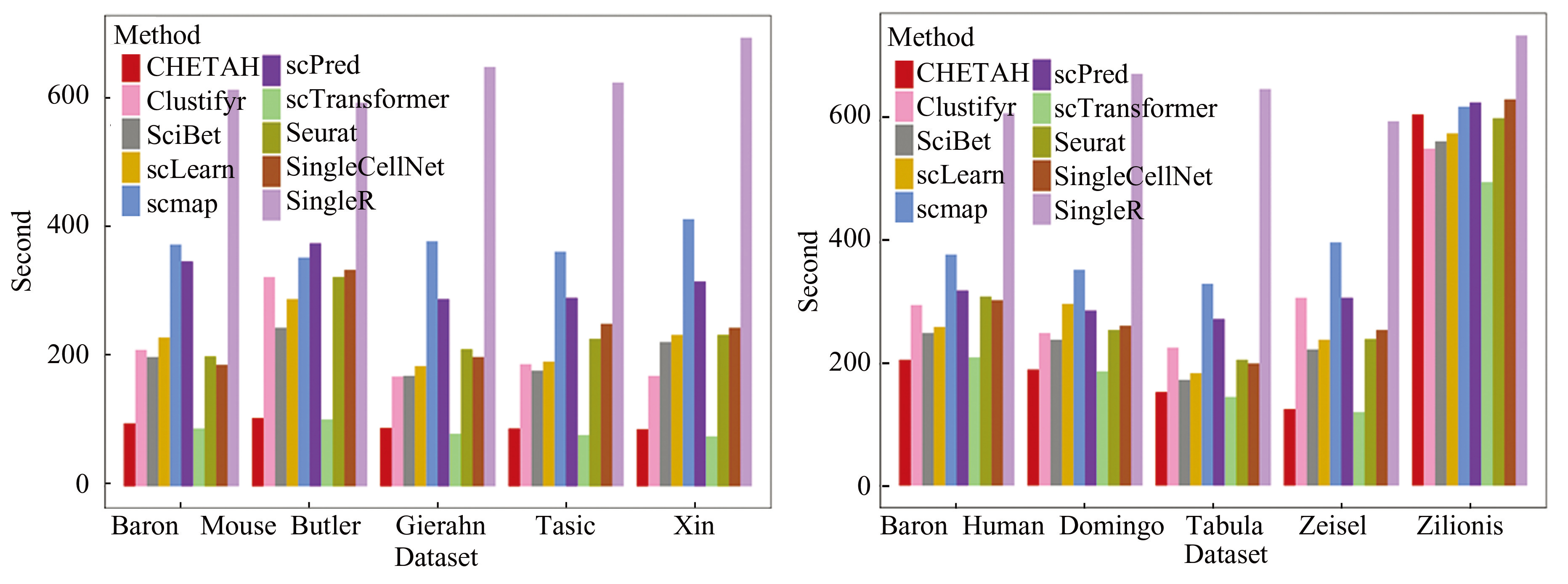
图5不同方法在10个数据集的运行时间
Fig.5Running time of different methods on 10 datasets
3.2 跨平台相同组织数据间测试有效性
3.2.1 查询数据集中的细胞类型全部包含在参考数据集的细胞类型中的情形
在这种情况下,查询数据中的所有细胞都应被赋予正确的细胞类型标签,而被注释为"未赋值"的细胞比例应尽可能小。本研究收集了人类胰腺的scRNA-seq数据集,这些数据集已被充分研究。在这种情况下,使用Baron[45]数据集作为训练数据集,因为它包含的细胞类型比其他胰腺scRNA-seq数据要多得多。Xin[48]的scRNA-seq数据集只包含与Baron[45]相同的胰腺四种主要细胞类型,包括α细胞、β细胞、δ细胞和γ细胞。因此,使用Xin[48]作为查询数据集来比较scTransformer和其他方法的细胞类型标注性能。
图6不同条件下模型的性能比较
Fig.6Comparison of model performance under different conditions
图7(a)代表了scTransformer和其他九种注释工具在Xin数据集上的UMAP图可视化,与图7(b)所代表的Xin[48]数据集的真实分类对比,scTransformer只是将少部分的α细胞错误地标记为巨噬细胞和胰管细胞,图7(c)则更能直观地比较包括scTransformer在内的10种细胞类型识别工具在Xin[48]数据集上的模型评估指标,具体包括根据细胞识别工具注释得到的预测标签与细胞原有的真实标签计算得出的准确度、F1分数、kappa分数,最终得到,scTransformer的准确率、F1分数和kappa系数表现均大于0.99,与之相对应,在模型性能上表现第二好的是scmap[29]方法,其他注释工具的表现性能则更差。总之,与所有其他方法相比,scTransformer预测的细胞类型标签更接近图7(b)中所示Xin[48]的真实细胞类型标签。
本研究使用来自于人类血液组织的Butler[42]数据集和Gierahn[43]数据集进行测试。Gierahn[43]数据集所含的原始细胞分类包含在Butler[42]数据集中,包括B细胞、CD4T细胞、CD8T细胞、树突状细胞、自然杀伤细胞,以Butler[42]数据集作为参考数据集,Gierahn[43]作为查询数据集,在10种细胞类型识别方法上进行综合比较,最终得出,scTransformer在血液数据集上的准确率,F1分数和kappa系数表现均大于0.99,均优于其他方法。
图710种细胞类型识别工具在训练-测试对Xin-baron上的比较
Fig.7Comparison of the performance of ten cell type annotation methods on predicting the cell types of Xin by baron
注:(a)10种细胞类别识别工具在Xin数据集上的UMAP可视化;(b)Xin数据集原有的真实分类UMAP可视化;(c)10种细胞类别识别工具的在Xin数据集上的准确度、宏观F1分数、kappa分数比较.
3.2.2 查询数据集和参考数据集包含相同和不同的细胞类型的情形
细胞类型鉴定的准确性体现在两个方面,一是常见细胞类型的鉴定准确性,二是非常见细胞类型的“未分配”细胞率,可以用AUR指标来评估模型,AUR用于计算查询数据集和参考数据集常见细胞类型识别准确率与非常见细胞类型未分配率之间的加权平均值。
首先使用Tasic[49]的小鼠大脑数据作为训练数据集,并使用Zeisel[48]数据作为测试数据。如图8所示,scTransformer的AUR大于0.99,高于其他注释工具,AUR值表现第二的是Clustifyr[61]方法。接着,使用Tabula Muris[20]的小鼠胰腺数据集作为测试数据集,然后把Baron_mouse[45]的小鼠胰腺数据集作为训练数据集,得到的scTransformer的AUR指标大于0.99,低于最好的方法Seurat[14]和Clustifyr[61],scTransformer,Seurat[14],Clustifyr[61]注释结果可以由图9的三个桑基图看出。最后,使用Domingo-Gonzalez[46]的小鼠肺部数据集作为查询数据集,使用Zilionis[47]的小鼠肺部数据集作为参考数据集,得到scTransformer的AUR值为0.63,仅次于最好的方法Seurat[14],其AUR值为0.64。图10的(a)图和(b)图显示小鼠肺部数据中标记为 T 细胞的细胞重注释为 T1 亚型,大多数被标记为中性粒细胞的细胞被鉴定为 N1 亚型,少部分被鉴定为N4和N6亚型,这些说明了scTransformer在识别一些稀有细胞时也有不错的效果。

图8scTransformer与九种细胞类型注释方法的 AUR比较
Fig.8AUR comparison of scTransformer with nine cell type annotation methods
注:(a)Zeisel 预测 Tasic 的准确率和未分配率的点阵图,靠近右上角性能好;(b)通过Zeisel预测Tasic时,scTransformer与其他九种方法的AUR比较;(c)Zilionis预测Domingo-Gonzalez的准确率和未分配率的点阵图,靠近右上角性能好;(d)通过Zilionis预测Domingo-Gonzalez时,scTransformer与其他九种方法的AUR比较;(e)Baron_mouse预测Tabula Muris的准确率和未分配率的点阵图,靠近右上角性能好;(f)通过Baron_mouse预测Tabula Muris时,scTransformer与其他九种方法的AUR比较.
3.3 不同组织数据间的测试
scTransformer可以准确识别查询数据集中与参考数据集不同的细胞类型,为了进一步比较预测的准确性,本研究特别使用了多个来自完全不同的人体或小鼠组织的训练-测试数据集对进行了scTransformer预测。鉴于相同的细胞类型,如T细胞和B细胞,可能存在于不同的组织中。本文仍然使用AUR作为性能评价指标,AUR中未分配率的比例应该更高。在本节中,利用本文中出现的三个scRNA-seq数据集设计了四对不同组织的训练-测试对。
如图11,scTransformer能够更好地同时兼顾常见细胞类型的准确性和非常见细胞类型的未分配率。总之,可以看出scTransformer具有准确预测不同组织细胞类型的能力,并有助于鉴定不同组织中尚未发现的新细胞类型。
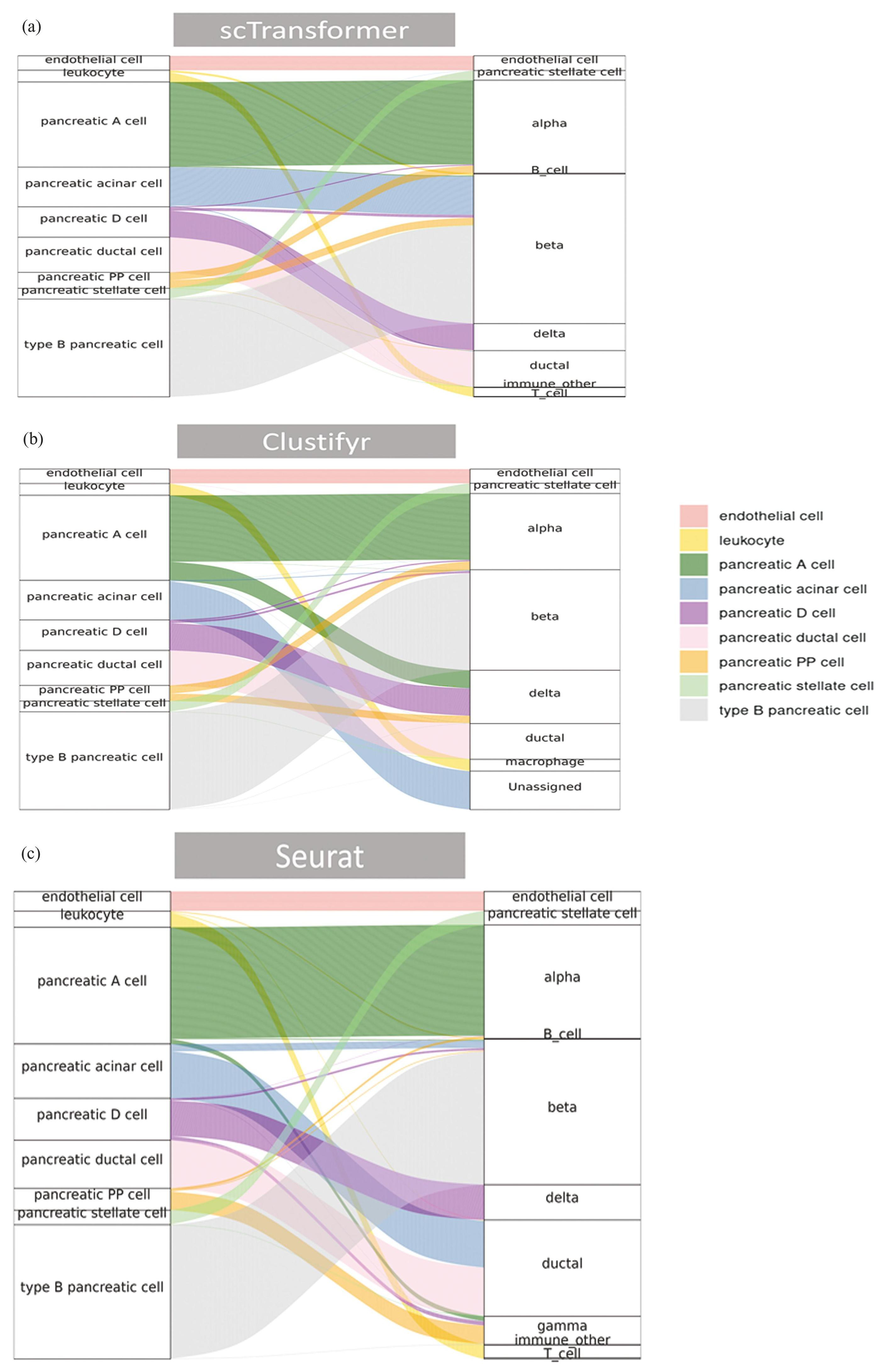
图9scTransformer在小鼠胰腺细胞的注释结果
Fig.9Annotation results of scTransformer in mouse pancreas cells
注:(a)基于scTransformer通过Baron_mouse预测Tabula Muris的桑基图;(b)基于Clustifyr通过Baron_mouse预测Tabula Muris的桑基图;(c)基于Seurat通过Baron_mouse预测Tabula Muris的桑基图.

图10scTransformer在小鼠肺部细胞的注释结果
Fig.10Annotation results of scTransformer in mouse lung cells
注:(a)Domingo-Gonzalez的原始细胞注释;(b)Domingo-Gonzalez的scTransformer重注释.
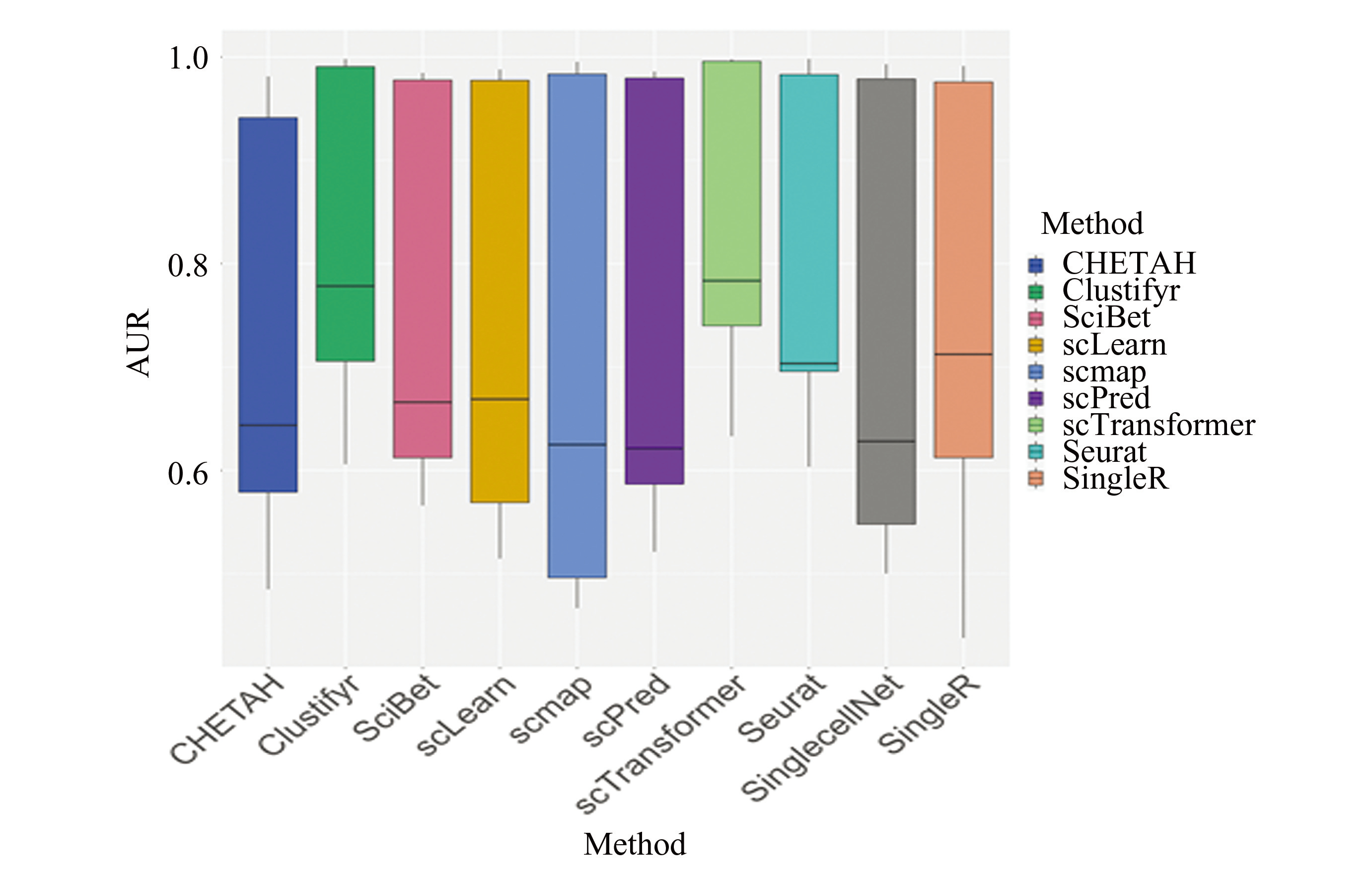
图11箱线图显示了十种方法使用的所有训练-测试对的 AUR
Fig.11Box plot showing AUR for all training-test pairs used by the ten methods
4 总结与讨论
本文提出了一个用于细胞类型注释的综合性高性能框架, scTransformer。scTransformer可以为不同组织、物种和测序平台等多种来源的scRNA-seq数据集提供可靠的细胞类型信息。与Seurat[14],SingleR[60],scmap[29],scLearn[57],Clustifyr[61],SinglecellNet[31],CHETAH[28],SciBet[62]和scPred[30]等一些流行的工具相比,scTransformer在性能上始终保持优势。首先,scTransformer在数据集内任务上的表现优异。其次,scTransformer在跨平台的相同或不同组织的单细胞分配等数据集间任务上的表现也优于其他方法,这表明它具有强大的泛化能力和鲁棒性。最后,scTransformer在识别稀有细胞类型上的表现也很出色。
scTransformer也存在一些可以进一步改进的不足之处。首先,基因嵌入过程是将单个细胞的基因表达转化为子向量输入Transformer编码器,但这一过程并不容易解释,子向量之间的关系也很难辨别。其次,在输入scRNA-seq数据之前,scTransformer需要使用Seurat方法选择高变基因。这给性能评估带来了潜在的偏差,因为选择高变基因的参数会影响结果。尽管一些成熟的细胞类型标注工具已经取得了显著的成果,但跨数据集的性能仍有改进的余地。此外,如何有效利用日益丰富的已注释或未注释scRNA-seq数据集进行单细胞类型注释也值得研究。为了克服这些局限性,有两种策略值得研究。一种策略是探索使用预训练模型来有效利用公共单细胞数据集[39]。另一种策略是利用迁移学习技术来解决跨数据集任务中数据集之间的分布差异问题[63]。单细胞测序技术已成为解决生物学问题的有效工具,并得到越来越多的利用。然而,在没有大量已知细胞类型信息的情况下准确识别非模式物种的细胞类型仍是一项挑战[64]。鉴于scTransformer在细胞类型注释方面的强大功能,值得探索开发新版本来应对该挑战,以及解决scRNA-seq分析中的细胞伪定时和细胞间通讯问题。



