摘要
基因组变异作为生物遗传多样性产生的核心,对解析生命演化、揭示物种内个体差异、探究疾病机制等方面有重要影响,而参考基因组作为遗传研究中的参考框架,其序列表征能力会直接影响遗传变异的准确识别。当前广泛应用的人类参考基因组主要由西方人群样本组成,对中国人群特异性遗传变异解析能力不足,亟需构建有中国人遗传特性的新参考基因组,以促进对中国人群遗传和进化机制的深入研究。本研究提出一种基于人群基因组变异的参考基因组改造方法,利用单核苷酸变异(SNV)、短插入删除变异(Indel)以及结构变异(SV)三种类型的东亚人群变异数据对GRCh38版本人类参考基因组进行改造,经过多层筛选、修订,建立了一系列包含不同变异频率、变异类型的中国人参考基因组。通过选取不同地域的中国人样本测序数据对所改造的中国人参考基因组进行序列比对测试,选取变异频率超过2/3,1/2,1/2的东亚人SV,Indel和SNV变异改造GRCh38参考基因组时分别获得了最佳比对效果。最终整合上述对应变异频率下的全部变异改造参考基因组时,得到了最优的中国人参考基因组。本研究所建立的中国人参考基因组将有望提升大规模中国人群基因组变异识别的能力,为后续中国人参考基因组构建工作提供有效方法。方法详见:https://github.com/azheasir/Chinese-specific-reference-genome-construction。
Abstract
Genomic variation is at the core of genetic diversity and has a significant impact on the analysis of evolution, the revelation of individual differences within a species, and the investigation of disease mechanisms. The ability to characterize the reference genome sequence is crucial for genetic research as it directly affects the accurate identification of genetic variants. Currently, the human reference genome is based on samples from Western populations, which may not accurately represent the genomic variants in Chinese populations. Therefore, constructing a new reference genome with Chinese genetic characteristics is urgently needed to facilitate in-depth research on the genetic and evolutionary mechanisms of Chinese populations. The objective of this study is to propose a method for modifying the GRCh38 version of the human reference genome based on population genomic variants. This method employs three types of East Asian population variants: single nucleotide variants (SNVs), short insertion-deletion variants (Indels), and structural variants (SVs). After screening and revisions, a series of Chinese reference genomes with different allele frequencies and variant types were established. Sequencing data from various regions in China were used to benchmark the modified Chinese reference genomes. The reference genome, which respectively selected high-frequency SVs, Indels, and SNVs from East Asian populations with frequencies of over 2/3, 1/2, and 1/2, achieved the best read mapping results. The optimal Chinese reference genome was obtained by incorporating all the above variants into GRCh38. The Chinese reference genome established in this study is expected to enhance the ability to identify large-scale genome variants in the Chinese population and provide an effective method for subsequent Chinese reference genome construction. Further details on the methodology can be found at: https://github.com/azheasir/Chinese-specific-reference-genome-construction.
基因组变异主要表现为个体间DNA序列的差异,包括单核苷酸变异(SNV)、短插入删除变异(Indel)以及结构变异(SV)[1]。SNV变异也称为点突变,是基因组序列单个核苷酸替换[2],可影响基因功能进而改变个体表型。SNV是人类基因组中最常见的变异类型,通常每个人类个体含有300~400万个SNV[3]。Indel是指发生一至多个核苷酸的插入或删除[4],可能导致编码区序列错位,从而产生功能迥异的蛋白。在人类基因组中Indel数量仅次于SNV,通常每个人类个体含有60~100万个Indel[5]。SV指的是大范围DNA序列的变化(通常超过50个核苷酸),主要包括插入、删除、倒置等类型[6],SV是基因组中核苷酸变化总量最大的变异类型,对基因组结构及功能有重大影响[7]。在人类基因组中,平均每个个体包含约2万个SV[8]。
基因组变异是生物遗传多样性产生的核心[9],它在解析生命演化、揭示物种内个体差异、探究疾病机制及药物研发方面发挥着至关重要的作用[10]。当前,基因组变异的发现通常依赖于高通量测序数据与人类参考基因组的比对分析[11],以识别DNA序列间的差异。因此,高质量的变异发现需精确的测序与数据解析,同时也取决于参考基因组的准确性。目前国际上广泛采用GRCh38版人类参考基因组作为人类基因组变异发现的标准参考基因组[12-14],它已促进了多个大规模人类基因组计划项目的实施,对生物医学前沿科学研究产生了重要影响[15]。
参考基因组在遗传研究中起到物理坐标系和参考框架的作用,其质量将直接影响遗传变异的准确识别与注释,已经成为基因组变异识别与解析的关键[16]。然而,GRCh38版本人类参考基因组主要采用的是国际人类基因组计划(HGP)所收集的欧裔和非裔人类基因组样本[17],对于具有人口规模巨大的亚裔,特别是中国人群,该基因组遗传多样性和基因组结构的表征性显著不足[18],常常会导致中国人群特异性基因组变异的误判和漏检。为了更加精准、全面地检测中国人群基因组变异,亟需构建具有中国人遗传特性的新参考基因组,以促进对中国人群遗传和进化机制的深入研究,为针对中国人群的疾病诊断与治疗提供必要支持,进而推动我国健康发展。
为了实现这一研究目标,本文主要研究内容包括4个环节(图1):①从国际公开数据集中收集包括SNV,Indel,SV三种类型的大规模东亚人群基因组变异数据;②依据变异在人群中的分布频率,按照不同类型、不同变异频率梯度将这些变异加入到GRCh38人类参考基因组中;③收集国际公开的中国人全基因组测序数据,并与改造前后人类参考基因组进行比对;④构建比对性能评测统计量,筛选适合中国人群基因组测序数据解析的最佳参考基因组。实验结果表明,在由6个人构成的东亚人基因组变异集合中,将变异频率超过2/3,1/2,1/2的共性SV,Indel和SNV变异加入到GRCh38参考基因组中,参与测序的不同地域中国人基因组样本测序数据达到了最多的碱基比对总数。此外,SV相比Indel和SNV变异对于测序数据比对效果的提升更为显著,表明数量较少的SV变异对于参考基因组的改造具有更加显著的提升效果。本项研究提出了一种基于大规模人群变异数据的中国人参考基因组构建方法(https://github.com/azheasir/Chinese-specific-reference-genome-construction),并利用该方法构建了一套能够更全面表征中国人遗传多样性和基因组结构的参考基因组序列,通过提升中国人基因组测序数据解析的精度助力高质量中国人特有遗传变异的发现,为以中国人群为核心的疾病预防与治疗等重要领域的发展提供有力支撑。
1 数据获取与预处理
中国人参考基因组构建是以原有的人类参考基因组为基础的,因此首先从NCBI(https://ftp.ncbi.nlm.nih.gov)上获取GRCh38参考基因组作为修改的对象。GRCh38参考基因组是目前国际上广泛采用的参考基因组[19],有着优质的参考序列质量,在许多领域的研究中均起到关键作用,因此选择GRCh38参考基因组还可以在后续的比对中更充分的体现出构建的中国人参考基因组在中国人基因比对方面的优越性。

图1中国人参考基因组构建流程
Fig.1Workflow of Chinese reference genome construction
随后,从人类基因组结构变异联盟(HGSVC)在第二阶段工作中构建发布的人群结构变异集合中(http://ftp.1000genomes.ebi.ac.uk/vol1/ftp/data_collections/HGSVC2/release/v1.0)获取关于SNV,Indel,SV三种不同变异类型的群体变异数据样本集。本样本集采用最新的长读长测序与基于单体型组装的变异检测新技术,所涵盖的变异种类、数量、准确性达到了空前的水平,其中包含了丰富且全面的中国人特有变异,可显著提升GRCh38参考基因组的变异识别能力。为了利用中国人变异数据对原参考基因组进行改造,需要从取自世界各地的群体变异数据样本集中提取具有中国人共性变异的东亚地区的样本,并且为了在后续的参考基因组构建中,构建包含不同频率梯度的变异的参考基因组,需要利用每个变异在群体中的变异等位基因数量(Allele count,即AC),所有等位基因数量(Allele number,即AN)[20],因此在预处理中需要计算每条变异信息的AC,AN。研究中采用bcftools工具的view功能,在群体变异数据样本集中选择指定的东亚样本(HG00512,HG00513,NA18939,HG00864,NA18534和HG01596)变异集合,同时计算变异集合内变异的等位基因数量,并将结果保存到新的VCF文件中作为后续基因组改造的基础。数据获取与预处理的流程如图2所示。

图2原始数据获取与预处理流程
Fig.2Workflow of raw data acquisition and pre-processing
2 参考基因组构建
在构建中国人参考基因组过程中,需要使用东亚人样本携带的基因组变异对参考基因组进行校正,从而识别和纠正现有参考基因组中与东亚人存在普遍差异的部分。变异频率表征了基因组变异在人群中出现的概率,是判定变异在人群中分布情况的重要指标[21]。例如,如果某个突变位点在中国人群样本中的变异频率很高,代表其在现有的人类参考基因组中的等位基因测定结果与多数中国人群样本测定结果差异较大,因此很有可能是人类参考基因组在该变异位点对中国人群的刻画不够精准。使用这些普遍分布在东亚人群中的基因组变异校正参考基因组,能够构造出更符合中国人群的中国人参考基因组。准确筛选用于校正的基因组变异,是构造高质量中国人参考基因组的核心。利用每个变异在群体中的变异等位基因数量(AC),所有等位基因数量(AN),通过公式(1)计算变异频率(Allele frequency,即AF):
(1)
通过AF对东亚人群的样本中的变异信息进行分类,将AF的梯度依次分别设置为:AF≥0,AF≥1/6,AF≥1/3,AF≥1/2,······AF≥5/6,≥1,并根据AF的不同取值梯度依次提取中国人群变异检测集合中的 SNV,Indel,SV 变异信息,从而获取不同变异频率下中国人群的变异分布与特征,作为构建不同变异频率下中国人参考基因组的基础。在获取不同变异频率下中国人群的变异分布与特征后,使用simuG工具[22]进行基因组校正与新参考基因组的生成。它可以针对不同的变异类型提供高效精准的遗传变异的模拟,构造含有变异的校正后基因组。在研究中,我们利用它处理提取后的SNV,Indel,SV变异数据,结合参考基因组的序列信息,来对参考基因组进行修改,得到对应的新参考基因组。通过参考基因组修改,能够获得分别包含3种变异类型、7种变异频率梯度的共21个不同的新参考基因组,为后续通过比较得到比对效果最好的中国人参考基因组提供了数据保障。参考基因组构建总流程如图3所示。
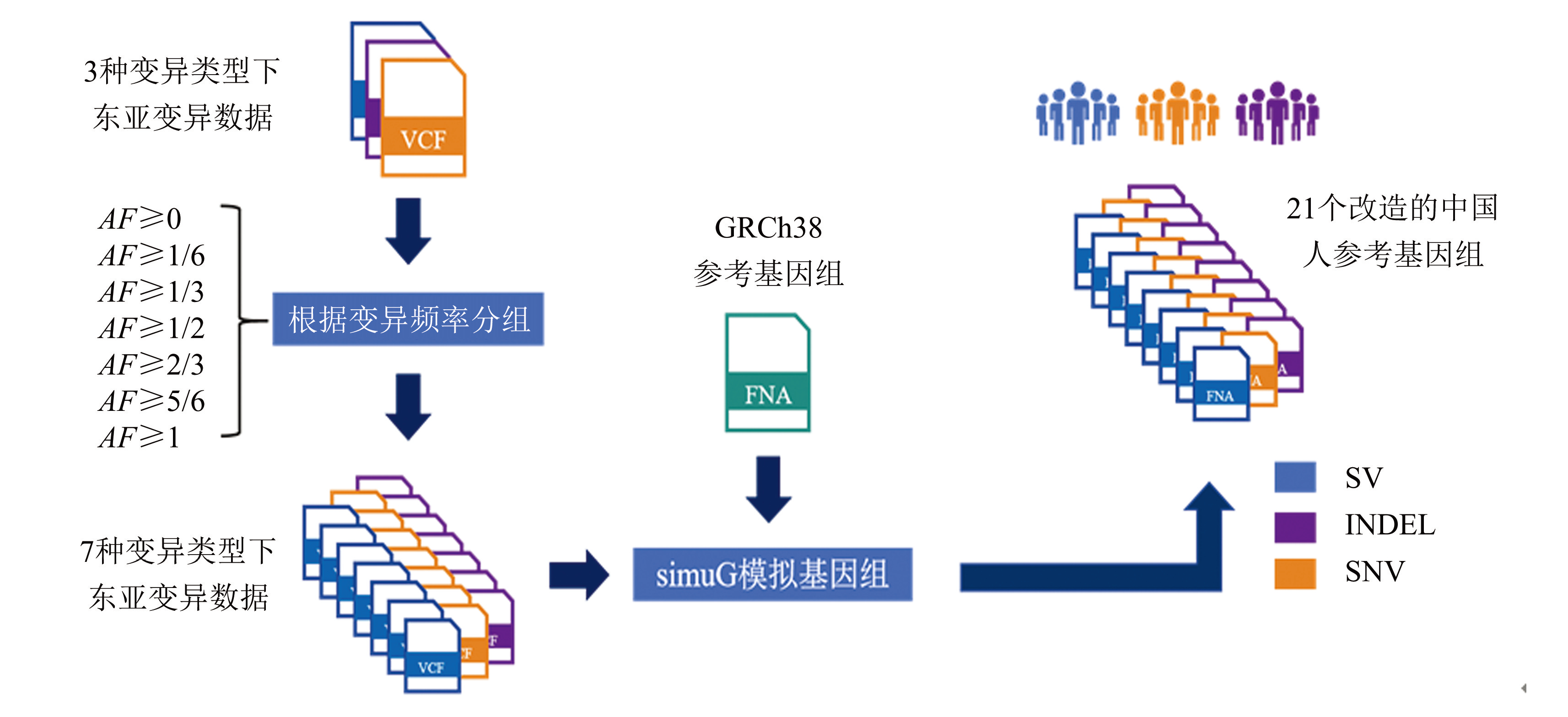
图3参考基因组构建流程
Fig.3Workflow of reference genome construction
3 中国人测序数据比对
为了全面的检验新构建的参考基因组对中国人基因的比对效果,选取了北方汉族、南方汉族、傣族三类基因组样本各五例(北方汉族样本:NA18525,NA18644,NA18757,NA18747,NA18561;南方汉族样本:HG00458,HG00717,HG00476,HG00534,HG00716;傣族样本:HG00759,HG01799,HG01028,HG02389,HG01812),从IGSR(www.internationalgenome.org)上获取到它们的测序数据(FASTQ文件)。BWA(Burrows-wheeler aligner)是一种常用的生物信息学工具,用于将DNA测序数据(如FASTQ文件)与参考基因组进行比对,使用BWA分别将15个中国人测序数据与新构建的21例参考基因组以及原有的GRCh38参考基因组进行比对,通过比对结果评测不同参考基因组的质量。比对的具体步骤如下(图4):
1)构建索引:利用“bwa index”指令对各参考基因组进行索引构建。这个过程将参考基因组划分为一系列较小的片段,以加快后续的比对速度。
2)序列比对:BWA会使用Burrows-wheeler变换[23](一种数据压缩算法)来加速比对过程。比对的目标是找到最佳的比对位置,使得测序片段与参考基因组的一致性最大化。研究中我们采用“bwa mem”指令将15个中国人测序数据分别与有索引的参考基因组进行比对,将比对结果保存在SAM文件中。
3)比对文件处理:通过samtools处理比对结果文件,samtools能够将SAM文件高效转化为BAM文件,并且能够对BAM文件进行分组[24]。由于后续的分析工作主要基于比对结果,而BAM文件是最常用的比对结果文件,且它相较于SAM是二进制文件,节省存储内存,因此利用samtools将SAM文件转换为BAM文件成为必要步骤。在转换过程中,通常使用“samtools view”命令实现SAM到BAM的格式转换。此外,为了优化后续处理流程的性能,还需对BAM文件进行排序,这一步骤对于确保数据处理的准确性和效率至关重要。采用“samtools sort”将转换获得的BAM文件按照默认的比对位置进行排序,得到比对后的最终结果。最后,为了后续能够快速访问BAM文件中的比对信息,需要对其建立索引,使用samtools的index命令为BAM文件建立其索引。
4)统计比对数据:通过qualimap进行测序片段比对结果的评估,它的BamQc模块通过分析BAM文件中的比对信息来计算各种统计指标[25]。研究中利用bamqc指令对处理好的BAM文件进行分析,得到包含各项比对结果的HTML报告,在报告中,可以直观的统计包括比对上的碱基数等所需的比对信息,从而利用其进行参考基因组比对结果的评测。
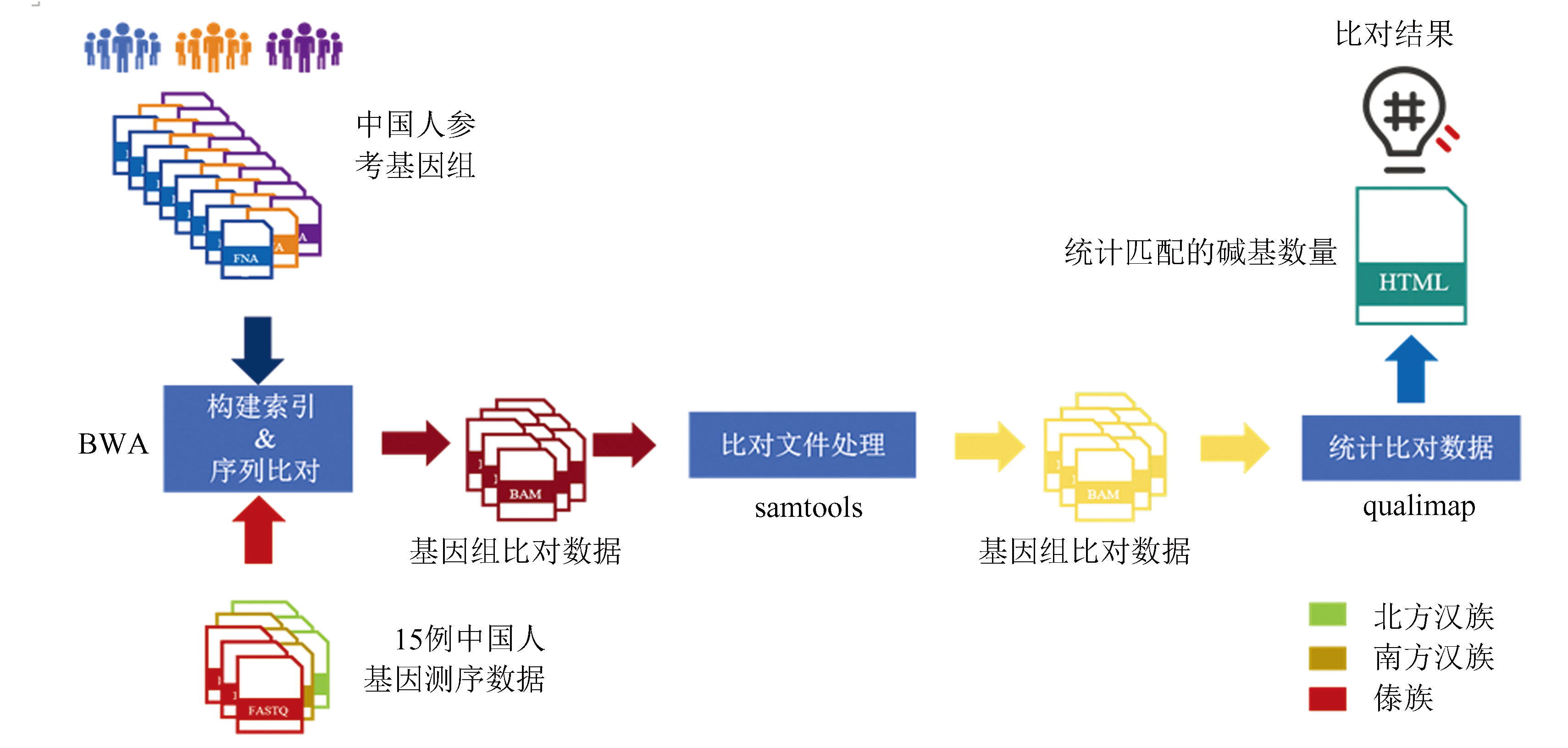
图4测序数据比对与评测流程
Fig.4Workflow of mapping sequencing data and performance benchmarking
4 参考基因组结果评测
研究中选择用成功映射到参考基因组的测序片段的碱基数目总和(Mapped base)反映比对结果,因为比对上的碱基数越多说明比对的质量越高,进一步反映了参考基因组在中国人样本的比对更为精准。将15例中国人测序数据与新构建的21例中国人参考基因组以及原有的GRCh38参考基因组进行比对,通过统计比对结果得到了mapped base数据,之后根据公式(2)对mapped base进行了归一化处理得到mapped ratio:
(2)
其中Xnormalized代表归一化后得到的mapped ratio,Xmax代表mapped reads数据中的最大值,Xmin代表mapped reads数据中的最小值,统计不同变异类型、不同地域样本的mapped ratio分布如图5所示(其中GRCh38+AF≥0组在对各样本的比对效果均最低,归一化后的值均为0,为实验结果的直观性该组的结果未展示在图中)。
图5表明,修改过的参考基因组除第一梯度(AF≥0)、第二梯度(AF≥1/6)外,比对结果均优于原参考基因组,说明根据东亚样本变异信息对参考基因组的修改可以有效提升对中国人基因样本的比对质量,从而证明了构建中国人参考基因组的方法的可行性。当利用全部变异信息(AF≥0)进行修改参考基因组时,对所有基因样本的比对结果均远不如原参考基因组,呈现这一实验结果的原因是利用全部变异信息修改参考基因组会引入个体特有的低频变异,导致构建新的参考基因组不具有普适性,不能作为代表中国人的参考基因组。在比较不同变异频率对原参考基因组修改效果时,利用变异信息中的结构变异(SV,图5(a)),短插入删除变异(Indel,图5(b)),单核苷酸变异(SNV,图5(c))对原参考基因组进行修改得到的新参考基因组的比对效果最好的梯度分别为AF≥2/3,AF≥1/2,AF≥1/2。这为之后的中国人参考基因组构建提供了经验,即应对变异数据进行适当的筛选,用筛选出的共性变异信息对参考基因组进行修改,才能够保证改造后的参考基因组的质量。
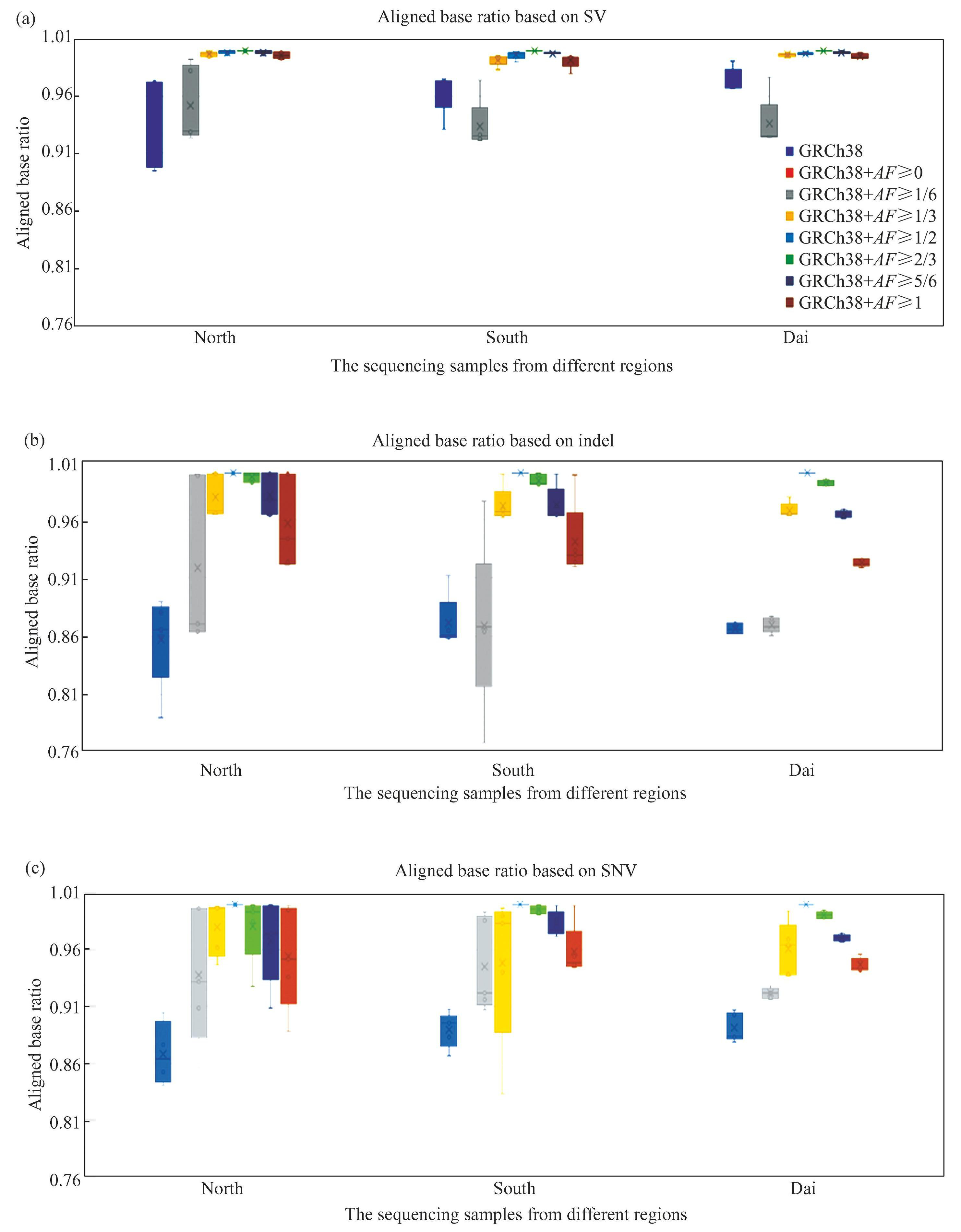
图5不同测序样本在各修改的参考基因组上比对性能统计
Fig.5Statistical chart of mapping results for each sample on each modified reference genome
注:(a)针对结构变异修改的比对性能统计;(b)针对短插入删除变异修改的比对性能统计;(c)针对单核苷酸变异修改的比对性能统计.
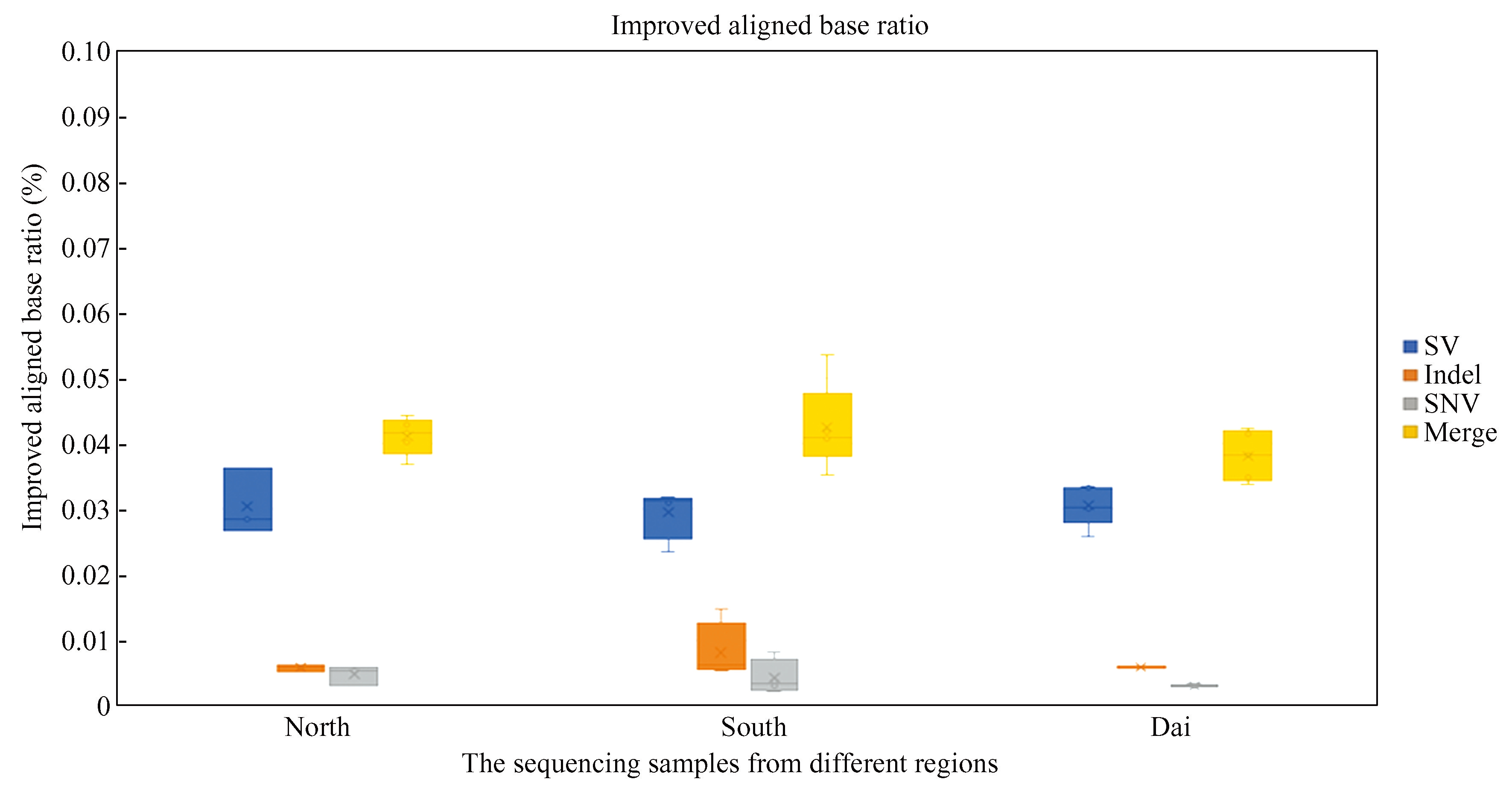
图6不同类型变异对修改的参考基因组比对性能统计
Fig.6Statistics on the effect of different types of variations on the constructed reference genome
随后,分析了不同变异类型在修改参考基因组中的影响,当对不同变异类型均选择最佳变异频率梯度时,图6表明利用结构变异进行修改对参考基因组比对效果的提升最明显,平均提升了0.03%,而SNV和Indel基本提升了不到0.01%,其原因是结构变异(SV)尽管相较于单核苷酸变异(SNV)、短插入删除变异(Indel)数量较少,但其对基因组上核苷酸序列的影响范围更大,因此未来的人类参考基因组构建应更加重视结构变异对参考基因组质量的影响。此外,通过横向比较中国不同地域基因样本在构造的新参考基因组下的比对效果,我们发现在一系列构造的新参考基因组中,傣族人群中不同样本个体之间的比对效果的提升范围均明显小于南方汉族,北方汉族人群。
最后,选择最佳变异频率梯度,即使用变异信息中频率梯度分别为AF≥2/3,AF≥1/2,AF≥1/2的结构变异(SV),短插入删除变异(Indel),单核苷酸变异(SNV)同时对原参考基因组进行修改得到最终的中国人参考基因组,统计利用不同种类的变异信息对参考基因组进行修改得到的中国人参考基因组、以及最终的中国人参考基因组对中国人样本的比对效果如图6。图中直观地表明同时用合适梯度的多种变异类型进行修改的参考基因组对中国人样本比对效果是最为优异的,该基因组可大幅提升对中国人基因样本的比对效率与质量,这也证明了在中国人参考基因组改造时同时利用多种变异类型的变异信息对原参考基因组修改的效果优于单独利用某一类型的变异进行修改。
5 结论
1)提出一种利用大规模人群基因组变异构建中国人参考基因组的方法,通过筛选、提取东亚人群不同类型的变异数据插入到现有参考基因组中,得到更适合中国人群遗传特性的参考基因组,为后续中国人参考基因组构建工作提供有效指导。
2)利用提出的构造中国人参考基因组的方法在GRCh38人类参考基因组上扩充了中国人特异性变异信息,选取的变异信息范围有针对性,包含大量中国人的共性变异数据。构造出的中国人参考基因组更符合中国人遗传特性,能更加精准、全面地检测中国人群基因组变异,为高质量中国人特有遗传变异的发现提供必要支持。
3)以不同变异频率作为梯度对变异信息分组,筛选出不同变异类型在中国人样本比对方面最优秀的梯度;通过分析不同类型的变异对改造基因组效果的影响,发现添加中国人共性结构变异(SV),对改造的新参考基因组在中国人样本比对方面的提升最大,这些结论为后续中国人参考基因组改造工作提供了重要的经验规律。
