摘要
单细胞转录组测序(scRNA-seq)已被广泛应用于基础医学研究中,分析和挖掘scRNA-seq数据有助于深入理解病变组织的细胞组成结构和功能,揭示复杂疾病过程和阐明药物作用机制,进而推动精准医学的发展。然而,如何基于海量的scRNA-seq数据对患者疾病表型进行预测,并筛选关键特征是单细胞技术临床转化的关键问题。本文综述了基于单细胞转录组数据进行患者疾病表型预测的相关方法,并对原理、算法、优缺点进行归纳和讨论,最后对相关研究的发展和应用进行了展望。
Abstract
Single-cell RNA sequencing (scRNA-seq) has been widely applied in basic medical research. Analyzing and mining scRNA-seq data facilitates an in-depth understanding of the cellular composition and function of diseased tissues, reveals complex disease processes, elucidates drug mechanisms of action, and promotes the development of precision medicine. However, how to predict patient disease phenotypes based on the massive amounts of scRNA-seq data and identify key features is a critical issue for the clinical translation of single-cell technologies. This article reviews relevant methods for predicting patient disease phenotypes using single-cell transcriptomic data, and summarizes their principles, algorithms, advantages and disadvantages. Finally, recommendations and perspectives on the application of related research are provided.
疾病表型指的是疾病在个体病程中每个阶段可能呈现出的可观察的特征或症状[1]。疾病表型是疾病本质的直接反映,通过对疾病表型的深入分析和研究,我们能够更好地揭示疾病的内在机制,并为疾病的预防、诊断和治疗提供重要依据。基因组学的发展加快了基础医学研究的步伐,并为确定疾病的生物标志物和治疗目标提供了有力的参考依据,目前相关研究已经获得了大量与人类疾病相关的分子基础知识[2]。但是对于复杂的疾病如肿瘤,其组织和微环境具有高度的异质性,导致不同的患者在治疗方案和治疗效果上存在较大的差异[3]。复杂疾病的个性化治疗和精准用药为改善患者治疗情况及预后等提供可能[4]。然而,目前仍存在两个主要挑战:①预测个体对不同治疗的反应和确定药物敏感性的分子生物标记物; ②传统的整体组织块RNA测序技术(Bulk RNA sequencing)已经被广泛应用于探索潜在的生物标志物,但是它只关注所选取样本组织细胞的平均表达,无法精确检测出各个细胞类型占比和细胞内基因表达的变化[5]。单细胞转录组测序(Single-cell RNA sequencing,scRNA-seq)技术可以在单个细胞尺度上捕获和定量多种组学的生物信息,鉴定整个组织器官中的细胞类型和细胞的表达变化,已成为深入理解组织的功能结构、揭示复杂疾病过程和阐明药物作用机制的有力工具[6-7]。
近年来,随着高通量测序技术的不断发展,研究人员得以获取大量病人样本的深度分子特征,这些特征涵盖了与疾病表型密切相关的基因、分子通路以及细胞类型等信息[8-10]。而机器学习(Machine learning,ML)等计算方法是从高维数据集中提取疾病相关模式的有效工具,在确定遗传疾病候选基因方面取得了巨大成功[11],许多学者已经尝试使用机器学习方法对scRNA-seq数据进行建模,以完成患者疾病表型预测任务[12]。本文对基于单细胞转录组测序数据进行患者疾病表型预测的相关方法进行全面综述,对其原理、算法实现、优缺点以及可解释性等方面进行深入剖析,旨在为相关领域的学者提供有价值的信息和研究思路。
1 单细胞转录组数据进行表型预测的挑战
scRNA-seq数据为精准预测患者疾病表型提供了宝贵信息。然而,如何基于海量的scRNA-seq数据准确地对患者疾病表型状态进行建模,并自动化挖掘相关亚群的关键基因仍然十分具有挑战[13]:①从不同患者(样本)收集的细胞基因表达谱数据在数据量级存在很大差异,个体测序得到的细胞数量从几百到几万不等,为标准模型的建立带来了困难;②scRNA-seq得到的细胞表达谱数据通常是十分嘈杂高维的信息,且不同样本的细胞占比和基因表达模式均存在差异,增加了数据整合和模型建立的难度;③尽管scRNA-seq可以获得大量单细胞分辨率的表达谱数据,但是由于测序成本较高,单个数据集在样本级别上通常只包含几十到几百条数据,导致模型过拟合当前数据集的特点,泛化能力受限。而不同数据集之间由于细胞类型组成、技术平台、处理流程等因素的影响,存在技术性的批次效应,也对模型迁移能力提出了挑战;④相关的模型需要一定的可解释性,以便研究人员理解模型决策流程,为进一步研究病理机制提供指导依据。
现有的基于单细胞转录组测序数据进行疾病表型预测的方法按照分辨率大致可以分为两类(图1):①样本级别; ②单细胞级别。前者根据领域内生物学知识和相关工具在样本级别获得单细胞的特征值,然后根据提取的特征值使用不同的分类模型对患者疾病表型进行分类;后者通常直接将每个细胞的表达谱数据作为输入,通过设计复杂的网络结构直接从原始数据端到端的对患者的疾病表型进行预测。
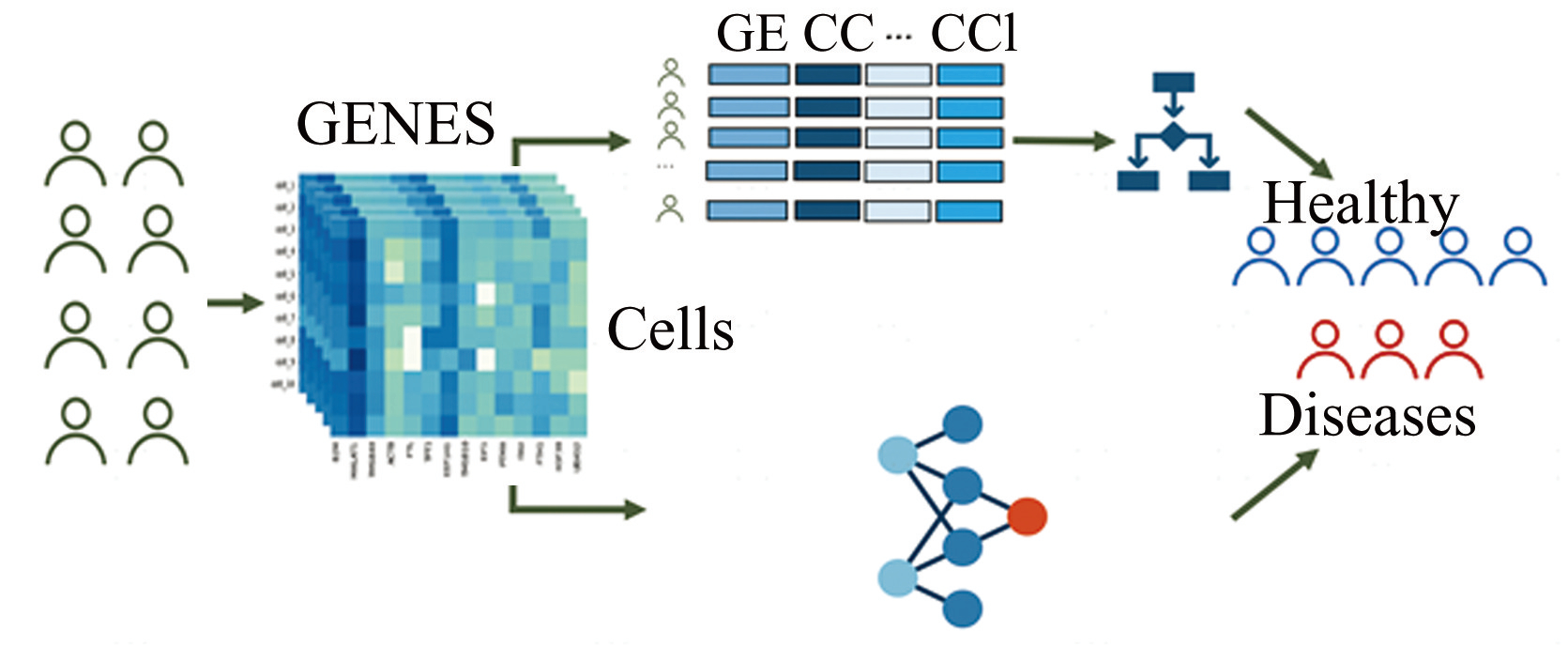
图1两类基于scRNA-seq数据预测疾病表型的方法
Fig.1Two types of methods for predicting disease phenotypes based on scRNA-seq data
1.1 基于样本级别进行疾病表型预测
由于scRNA-seq数据的复杂性,直接从不同量级、嘈杂高维的细胞表达谱数据构建表型预测模型较为困难。从样本分辨率进行疾病表型预测的方法,希望从单细胞层级复杂的数据结构中提取出样本层级的单细胞特征来表征患者潜在的疾病状态,并将这些特征输入到分类器中对患者疾病表型进行分类,达到疾病表型预测的目的。这类方法的优势在于可以直接应用成熟的生物信息学工具和机器学习算法进行有生物学意义的特征提取和分类模型建立;此外,还可以直接联合医学影像、临床特征等多源的样本级别的元数据,构建多模态的疾病表型预测模型。
1.2 样本级别进行疾病表型预测方法
从样本分辨率进行疾病表型预测的方法可细分为两类。一类直接从scRNA-seq数据中提取有生物学意义的单细胞特征,这类方法通常需要依赖领域内的先验知识以及先前的注释结果;另一类则通过概率生成模型或者深度学习模型来学习单细胞表达谱中潜在的表达模式,并将学习到的嵌入向量在样本级别汇聚为特征来进行疾病表型分类。这两类方法通过特征提取将单细胞分辨率的表达谱信息汇聚成样本级别的生物学特征,每个样本都会根据提取的特征得到一组固定维度的特征向量用于样本疾病表型预测,这就将复杂的疾病表型预测问题转化为标准的表格类数据分类问题。因此,根据具体的疾病表型预测需求,可以运用许多成熟的统计与机器学习工具、算法来进行二分类或多分类求解[14]。例如,Cao等[15]提取了细胞类型占比、细胞亚群下的特异性基因表达值、细胞类型特异性通路活性值、细胞类型特异性CCI评分、全局基因表达值和空间信息指标六个类别的样本单细胞特征,将这些特征作为输入,通过随机森林(Random forest,RF)模型来构建疾病表型预测模型,并在包括炎症、肺健康、黑色素瘤、新冠等多个公开数据集上验证了单细胞特征的有效性,实现了对相关疾病的预测;He等[13]将每个患者的细胞数据建模为高斯分布的混合模型,通过每个混合成分的均值和协方差来表征一个潜在的细胞亚群,并通过均值和协方差来估计一个患者样本的每个细胞亚群的丰度,将这些丰度作为一组患者样本的特征,同时使用了二次型分类器来对患者是否患有狼疮和患者的种族两个任务进行分类预测,得到了较好的结果;Zeng等[16]通过患者所有细胞的scRNA-seq数据来近似细胞类型和疾病表型的联合概率分布,并将生成的联合概率分布直接作为患者的预测特征输入到支持向量机(Support vector machine,SVM)中实现了对疾病状态和疾病严重程度的预测。
这类方法的核心差异在于生物学特征的提取与分类器的选择。具体而言,基于scRNA-seq数据提取的特征质量直接决定了疾病表型预测模型的上限性能,而不同分类器作用则是在逼近这个上限性能。高质量的生物学特征不仅有助于提升模型的预测精度,还能够更细致地描绘疾病的发生发展机制。目前,scRNA-seq领域已有许多成熟有效的单细胞特征提取工具与方法可以迁移到疾病表型预测任务中,本文总结了常见的用于疾病表型预测的单细胞特征见表1。
表1可用于疾病表型分类的单细胞特征
Table1Single-cell signatures that can be used to classify disease phenotypes

1.3 样本级别的特征筛选与可解释性分析
筛选与疾病表型高度相关的生物学特征是建立疾病表型预测模型的另一个重要目标。基于特征构造提取样本单细胞特征,并在样本级别进行患者疾病表型预测的方法,在模型可解释性方面也表现突出。由于构造的单细胞特征往往具有明确的生物学意义且使用的分类器通常较为简单,研究人员可以通过多种手段来分析提取的生物学特征对疾病表型预测的贡献程度,并探究哪些生物学特征更有可能预警疾病状态,为后续的病理性研究指明方向。
最简单的线性分类器可以直接观察每个自变量的系数作为模型特征的权重,根据权重大小可以直观、准确的判断哪些特征对模型预测的贡献度更高[28],即哪些生物学表达最有可能对疾病表型产生影响。基于决策树的模型,如随机森林[29]、自适应提升算法(Adaptive boosting,adaboost)[30]、极值梯度提升算法(eXtreme gradient boosting,XGBoost)[31]等,可以通过统计每个特征在决策树的分裂节点上的使用次数,以及每个特征分裂带来的信息增益大小,来直观地判断不同特征对最终模型预测结果的重要性。具体来说,在RF模型中,一个特征被使用的次数越多,以及平均信息增益越大,就表示这个特征对最终的模型预测结果的影响越大。Cao等[15]将单细胞特征出现的频率视为每个特征的特征重要性分数,并将与一个细胞类型相关的所有特征的特征重要性得分相加,然后除以该特定细胞类型构建的特征总数量,记为每种细胞类型的特征重要性得分。这使得评估不同类型的生物学特征与细胞类型对疾病表型的影响成为可能。除此以外,通过一些脱离具体模型的算法也可以对模型进行可解释分析,例如SHAP(SHapley additive exPlanations)[32]是一种统一的机器学习模型可解释框架,它以博弈论的观点计算每个特征对模型预测结果的边际贡献来解释每个独立的特征对模型的贡献度,可以很好地解释包括多层感知机(MLP,Multilayer perceptron)、随机森林在内许多机器学习模型。Lemsara等[33]使用SHAP值作为MLP模型的特征重要性来研究单细胞特征(细胞类型占比与差异基因表达均值)与COVID-19疾病严重程度间的相关性。
2 基于单细胞级别进行疾病表型预测
复杂疾病往往涉及多种细胞类型中多个不同表达的基因[34]。在兼顾样本内部细胞异质性的同时,从目标细胞群与微环境中其他细胞的相互作用进行特征解析,使基于单细胞测序技术进行疾病表型预测具有较大的优势。目前,人们已经开发了多种统计方法尝试评估疾病表型与单个细胞关联,发掘与疾病有关的细胞亚群与细胞过程[35-36]。但是这些方法尚不足以直接从单细胞转录组数据中精确预测疾病的表型。
从单细胞分辨率进行疾病表型预测的方法能够充分利用单细胞的原始表达谱数据,自动化挖掘与疾病表型相关的潜在表达模式,进一步提高疾病表型的预测精度。此外,这类方法通常不依赖于传统流程的细胞注释结果,因此能够避免细胞注释过程中可能产生的误差或偏差,端到端的预测患者疾病表型状态。
2.1 单细胞分辨率表型预测方法
从单细胞分辨率直接对患者表型预测通常需要对原始基因表达矩阵进行降维或特征分解,再输入到机器学习分类器中进行分类。例如,Alquicira等[37]提出的scPred方法,该方法首先对基因表达矩阵进行奇异值分解以获得基因表达值的正交线性组合,然后通过双尾Wilcoxon检验,筛选出在不同类别下具有显著差异的主成分,并将其输入到SVM中进行疾病表型分类。这类方法的劣势在于无法充分挖掘scRNA-seq高维数据中复杂的非线性模式。除此以外,传统的机器学习方法往往需要一次性将训练数据加载到内存中,这使得其在处理大规模的单细胞分辨率数据时受到了限制。
另一种主流的预测方法主要基于神经网络。神经网络可以接受单细胞复杂数据结构的输入,端到端的学习单细胞原始表达谱数据,在细胞层级上对疾病表型进行预测,并根据单细胞疾病表型推断患者疾病表型。这类方法通常不需要显式地进行单细胞特征工程的构建,而是通过多层非线性变换,自动从原始表达谱数据中学习有效的特征表示,挖掘与疾病表型相关的复杂表达模式[38]。此外通过迁移学习等技术,可以基于已经在大规模单细胞数据集上预训练好的单细胞大模型[39-40]对现有单细胞表达谱数据进行特征提取和模型微调,这有助于学习到scRNA-seq数据内在的高级抽象表示,提高模型在不同数据集上的泛化能力和预测精度,并降低不同应用方向的训练硬件资源壁垒。通过对构建好的神经网络模型进行可解释性分析,可以发现之前不为人知或意义不明但实际上对疾病预测有重要贡献的生物标志物,这也为后续的病理信息挖掘和疾病治疗机制研究提供了方向。研究人员可以聚焦这些新发现的重要生物学因素进行深入研究,揭示它们与疾病的内在联系。
常用的神经网络结构有深度神经网络(Deep neural network,DNN)、卷积神经网络(Convolutional neural network,CNN)、递归神经网络(Recurrent neural network,RNN)、生成对抗网络(Generative adversarial network,GAN)、自动编码器(AutoEncoder,AE)、图神经网络(Graph neural network,GNN)、注意力机制(Attention)等,许多网络结构及其变种已经与scRNA-seq结合,并且可以有效地应用到患者及疾病表型预测任务中。例如,Asimomitis等[41]利用前馈神经网络和单细胞原始表达谱数据对急性髓样白血病的单细胞表型进行了建模预测;Xiong等[42]使用了自动编码器来学习每一个细胞表达谱数据在低维空间的嵌入,同时还引入了细胞原型的概念,随机初始化细胞原型向量并在嵌入过程中通过尽可能的缩小每个细胞与相应细胞原型的距离,最大化不同细胞原型间的距离来学习到有意义的细胞亚群代表。通过自动编码器提取的嵌入向量与细胞原型,使用一个基于神经网络的自适应相关性评分器来控制每个细胞与对应细胞原型的相似性对患者表型分类的贡献度,从而为每个患者提供了个性化的精确诊断;Mao等[12]提出了一个ScRAT框架,由三部分组成:首先通过样本混合来进行数据增强,并在扩充后的样本中为每个患者选取固定大小的细胞子集作为模型输入;然后通过多头注意力机制来动态的学习每个细胞的嵌入向量;最后经过一个池化层和全连接层完成对患者表型的分类。Ravindra等[43]则使用图注意力网络来获取scRNA-seq数据的图表示,其中每一个细胞被看作是一个节点,细胞的基因表达谱数据被看作为节点特征,细胞间的相似性作为两个节点的边,这就将初始的疾病表型分类问题转化为一个图节点分类问题,通过训练图注意力网络来预测每个细胞的疾病状态。这类方法主要优势在于使用了不同的网络结构替代了传统的手工特征提取,能够从不同尺度自动化地提取出scRNA-seq中潜在的疾病表达模式。这有助于研究人员深入挖掘基因表达谱中的潜在信息,发现新的致病机制与规律。
2.2 单细胞分辨率模型的特征解析
神经网络能够充分利用scRNA-seq中每个细胞的表达谱数据,并已在患者疾病表型预测任务上展现了优越的性能。然而,由于神经网络是一个黑盒模型,即便我们能够获取其每一网络层级的参数,也很难直观理解模型是如何根据输入的数据得到预测结果,这为研究人员理解影响疾病表型的生物学因素带来了阻碍。目前研究人员通常使用基于梯度或特征的方法和基于扰动的方法[44]对神经网络进行可解释分析,这两类方法都通过对训练好的模型进行后验解释来试图理解预测器的决策过程。
基于梯度或特征的方法通过梯度或者网络隐藏层的参数值来估计输入数据的特征重要性。例如可以通过反向传播来计算预测目标相较于输入特征的梯度,或者使用隐藏层的参数值来衡量每个输入的重要性得分。Ravindra等[43]基于图注意力网络第一层的注意力权重矩阵中参数的绝对值绘制了输入数据的基因热图来分析哪些输入基因是使得模型能够区分健康细胞和多发性硬化细胞的关键特征。基于扰动的方法则是通过改变输入数据的部分特征值,即对原始数据进行一些扰动,来观察预测结果的变化情况,添加的扰动可以是直接遮挡部分特征,也可以是有意义的扰动,添加扰动后,预测结果的变化越大则代表与扰动关联的特征对于相关疾病表型的重要性越高。如Asimomitis等[41]在筛选对急性髓样白血病分类重要的基因时使用的HRT[45]技术,就是通过用零样本替换原始样本,并观察分类结果的变化情况来检验对应基因的重要性。
3 总结与展望
单细胞测序技术目前在解析组织异质性、揭示病理组织环境中细胞间的关系、追踪疾病发生发展等研究中正发挥越来越重要的作用[46-47]。近年来,基于算法模型进行挖掘生物学表达模式的方法已经在疾病表型预测任务中取得一定成果。
以每个患者为单位,基于单细胞特征提取的传统机器学习方法依赖于邻域内的相关知识,提取的生物学特征的好坏直接影响了疾病表型预测的效果。同时,由于提取的单细胞特征天然的具有实际的生物学意义且所使用的传统机器学习算法大多可以直接地进行特征重要性的解释,研究人员可以直观地了解哪些生物学特征与目标疾病表型更相关。然而,scRNA-seq数据的丰富性主要体现在细胞层级而不是样本层级,通常的疾病表型预测数据集包含的样本数很少,统计能力十分有限,这极度影响了模型的性能,降低了模型的迁移和泛化能力,很难保证模型在其他数据集和实际应用中的准确率;同时,即使相同的细胞类型内的单细胞之间也存在较大的异质性,提取患者整体的生物学特征意味着舍弃了大部分单细胞级别的生物学信息,可能会降低疾病表型的预测效果;此外,大部分单细胞特征的生成还依赖于细胞注释和其他单细胞工具,这也会对疾病表型预测模型的可用性产生影响。
以每个细胞为单位构建神经网络自动化挖掘生物学表达模式的深度学习方法则直接使用细胞表达谱作为输入,一定程度上缓解了样本不足的问题。通过不同网络结构的设计,其能够充分挖掘潜在的生物学表达,实现高精度的疾病表型预测效果。但是神经网络作为一种黑盒模型,不能直观地反应模型的决策过程,可解释较差。此外,在大量的单细胞数据上进行训练对计算资源也提出了一定要求。
尽管目前的方法具有良好的应用价值,但仍存在以下几个方面的问题:①基于样本进行特征提取的方法容易忽视样本单细胞间的潜在信息,从细胞级别建模的方法又缺少了显性的生物学知识,如何将两者的优势结合起来(如在细胞级别的建模过程中融入单细胞注释产生的生物学特征)仍然存在困难;②scRNA-seq的数据质量受多方面影响,不同制备仪器平台、不同试剂盒版本的数据存在较大非生物学差异,这对模型的性能与泛化能力提出了挑战; ③如何在构建多模态的疾病表型预测模型时将单细胞数据与其他组学或临床信息相结合,并充分利用各种组学的生物学特征进行模型预测;④如何完全理解模型的决策过程,了解疾病表型的致病机制仍然存在困难,这依赖于可解释性机器学习的发展。
机器学习在单细胞组学中应用的发展可以大大改善患者的诊断、治疗和预后的研究,本文提供了一个关于使用单细胞转录组数据进行疾病表型预测的一个广泛的概述,期望可以对后续的算法开发和临床应用提供一定的参考。
