摘要
色素沉着绒毛结节性滑膜炎(Pigmented villonodular synovitis, PVNS)是一种可累及关节的腱鞘巨细胞瘤,类风湿性关节炎(Rheumatoid arthritis, RA)作为一种慢性自身免疫性疾病,二者都极少发生在颞下颌关节(Temporomandibular joint, TMJ),给临床医师诊治造成一定困难。本研究探讨了TMJ PVNS和TMJ RA之间共病基因的功能、免疫学差异及潜在治疗靶点。根据基因芯片(GSE3698)原始矩阵数据在探针水平分析基因表达谱。使用R语言编程分析并可视化结果。筛查2种疾病共同的差异表达基因(Differentially expressed genes, DEGs),分析其分子功能和生物学过程。基于STRING数据库,将DEGs导入Cytoscape软件构建蛋白质互作网络。使用机器学习算法来识别Hub基因,并预测Hub基因的诊断效率以及与免疫浸润细胞之间的相关性。共计发现76个DEGs,与加权基因共表达网络分析获得的与临床特征相关性最高的334个基因进行交叉,最终产生22个TMJ PVNS和TMJ RA的共同基因。通过机器学习确定了PLIN,PPAP2A和TYROBP为2种疾病共同的Hub基因,并使用集成受试者工作特征曲线和列线图显示了良好的诊断效能。单样本基因集富集分析显示出此3个Hub基因与28个免疫浸润细胞有实质性关联。本研究首次发现PLIN,PPAP2A和TYROBP与TMJ PVNS和TMJ RA的发生和发展有关。它们有望成为TMJ PVNS和TMJ RA诊断和治疗的新靶点和研究方向,从而为未来提高患者的诊断和治疗水平以及临床预后提供新的理论参考。
Abstract
Pigmented villous nodular synovitis (PVNS) is a tenosynovial giant cell tumor that can involve joints; and rheumatoid arthritis (RA) is a chronic autoimmune disease. Both of them rarely occur in the temporomandibular joint (TMJ), which makes it challenging for clinicians to diagnose and cure. This study explores the co-morbidity genes between TMJ PVNS and TMJ RA, along with their functions, immunological differences, and potential therapeutic targets. The gene expression profile is analyzed at the probe level according to the raw matrix data of gene chip (GSE3698). R language programming is used to analyze and visualize the results. The common differentially expressed genes (DEGs) are screened between these two conditions. The molecular functions and biological processes of the common genes are analyzed. Based on the STRING database, DEGs are imported into Cytoscape software to establish a protein-protein interaction network. Machine learning algorithms are utilized to identify hub genes and predict the diagnostic efficiency of hub genes as well as their correlation among immune infiltrating cells accordingly. A total of 76 DEGs are found out, and they are intersected with the 334 genes acquired by weighted gene coexpression network analysis (WGCNA), to eventually produce 22 common DEGs between TMJ PVNS and TMJ RA. PLIN, PPAP2A, and TYROBP are identified as three common hub genes between TMJ PVNS and TMJ RA that demonstrated good diagnostic performance using summary receiver operating characteristic curve and nomogram. Single sample gene set enrichment analysis shows three common hub genes substantially associated with 28 immune infiltrating cells. This investigation concludes that PLIN, PPAP2A, and TYROBP are associated with the occurrence and development of TMJ PVNS and TMJ RA. They are expected to become new targets and research directions for the diagnosis and treatment, thus providing new opportunities and references for improving the diagnosis and treatment level and clinical prognosis of patients with TMJ PVNS and TMJ RA in the future.
色素沉着绒毛结节性滑膜炎(Pigmented villous nodular synovitis,PVNS)又称弥漫性腱鞘巨细胞瘤,是一种罕见的关节疾病,发病率约为百万分之1.8[1]。PVNS是一种典型的单关节疾病,最常累及膝关节,其次是髋关节、踝关节、肩关节和肘关节[2]。其组织学特征包括纤维基质增生、巨噬细胞浸润和含铁血黄素沉积[3]。类风湿性关节炎(Rheumatoid arthritis,RA)作为一种慢性自身免疫性疾病,以大关节进行性、对称性炎症为特征,最终导致关节软骨破坏、骨侵蚀、关节畸形和功能丧失[4]。其病理基础由成纤维细胞、T和B淋巴细胞、中性粒细胞和单核细胞转运到增生的滑膜组织中引起[5]。发生于颞下颌关节(Temporomandibular joint,TMJ)的PVNS和RA极为少见,故目前对TMJ PVNS和TMJ RA发病机制的认识较为局限。另一方面,二者均表现出滑膜细胞过度增殖导致滑膜增生的炎症环境,且镜下也均可见病变滑膜组织中巨噬细胞和成纤维细胞积聚[3,6]。然而对PVNS缺乏关注导致PVNS和RA共存的发病率增加[7-8],正确诊断二者的能力将直接决定患者预后,因此迫切需要开发识别这2种疾病的生物标志物。
本研究基于基因表达谱数据,利用生物信息学方法系统分析TMJ PVNS和RA的差异表达基因,并进行加权基因共表达网络分析以发现与它们临床特征最相关的模块,明确这2种疾病之间的共同基因、分子功能和生物学过程。进一步通过机器学习算法预测TMJ PVNS和RA的共表达Hub基因及其诊断效率以及与免疫浸润细胞之间的相关性。
1 资料与方法
1.1 数据来源、类型和数据预处理
GSE3698数据集[9]从Gene Expression Omnibus数据库(https://www.ncbi.nlm.nih.gov/geo/)获取,该数据集基于GPL3050(Human Unigene3.1 cDNA Array 37.5K v1.0)基因组注释平台。从GSE3698数据集提取11个PVNS样本和18个RA样本。使用R/Bioconductor的“affy”与“hgu133plus2.db”软件包对原始微阵列数据转换成可以分析的表达矩阵,即校正和归一化背景噪声(非特异性杂交斑点),剔除无已知注释、映射多个或未映射任何基因ID的探针集。
1.2 基因差异表达分析
使用“limma”软件包贝叶斯线性模型筛选差异表达基因(Differentially expressed genes,DEGs)[10]。以log2差异倍数(Fold change,FC)的绝对值≥1(|log2FC| ≥ 1)且P < 0.05为校正阈值,分别使用“ggplot2”和“pheatmap” R包选择上调和下调的DEGs绘制火山图和热图[3,10]。
1.3 WGCNA筛选关键模块基因
加权基因共表达网络分析(Weighted gene co-expression network analysis,WGCNA)旨在寻找协同表达的基因模块,并进一步探索基因网络与关注表型之间的关联模式,以及网络中的Hub基因[11]。使用平均FPKM=0.5的筛选标准,根据无标度网络概念确定加权系数。选择height = 0.25(对应0.75的相关性)进行切割,并合并模块。将基因网络模块中的最小基因计数设置为40。最后,对于与临床特征相关的主要模块,计算基因显著性(Gene significance,GS)和模块成员(Module membership,MM)。
1.4 共表达基因功能分析
使用“randomcoloR”和“venn”软件包将DEGs和选定的关键模块基因取交集以识别共表达基因。通过“ClusterProfiler”,“ggnewscale”和“DOSE”软件包对TMJ PVNS和TMJ RA的共表达基因进行GO功能和KEGG信号通路富集分析。计数 > 1且P < 0.05的GO条目和KEGG通路具有统计学意义。
1.5 构建PPI网络与筛选Hub基因
检索蛋白质-蛋白质交互作用(Protein-protein interaction,PPI)在线数据库STRING(version 11.5,https://cn.string-db.org),用于构建共表达基因的PPI网络(交互置信度截断值设置为0.40),随后将基因簇导入Cytoscape(version 3.10.0,https://cytoscape.org)在线工具以可视化网络。通过CytoNCA插件,应用介数(Betweenness)筛选出PPI调控网络中TMJ PVNS和TMJ RA共同的Hub基因。
1.6 利用机器学习识别Hub基因
鉴于机器学习方法可以提高基因筛选的准确性[12],故本研究分别利用基于Lasso回归模型和随机森林(Random forest,RF)算法的机器学习模型再次筛选前次已获得的Hub基因,并进行交叉核对,2种算法的重叠基因被认为是TMJ PVNS和TMJ RA共同的Hub基因。
1.7 Hub基因诊断效能的评价
结合已确定的Hub基因表达谱数据,运用Logistic回归分析构建列线图预测模型。使用R包“pROC”分析并绘制SROC图,计算曲线下面积(the area under the SROC curve,AUC)以评估各Hub基因的诊断效能。在AUC > 0.5的情况下,AUC越接近于1,说明诊断性能越好[13-14]。
1.8 Hub基因的生物学过程及免疫浸润分析
单样本基因集富集分析(Single sample gene set enrichment analysis,ssGSEA)是一种研究数据集中Hub基因绝对富集程度的方法[15]。使用“GSEABase”和“GSVA”软件包进行ssGSEA,以研究TMJ PVNS和RA样本之间免疫细胞表达的差异以及Hub基因的免疫浸润情况。
1.9 Hub基因表达差异的评价
使用“PerformanceAnalytics”和“Circlize”软件包进行Hub基因与浸润免疫细胞之间Spearman相关性分析,显著性阈值范围-0.4~0.8,“corrplot”软件包用于结果可视化。
2 结果
2.1 DEGs的识别
与RA滑膜组织相比,我们在PVNS滑膜组织中共筛选鉴定出76个DEGs,包括19个下调的DEGs和57个上调的DEGs,火山图如图1(a)所示。图1(b)显示了前30个DEGs的聚类热图。
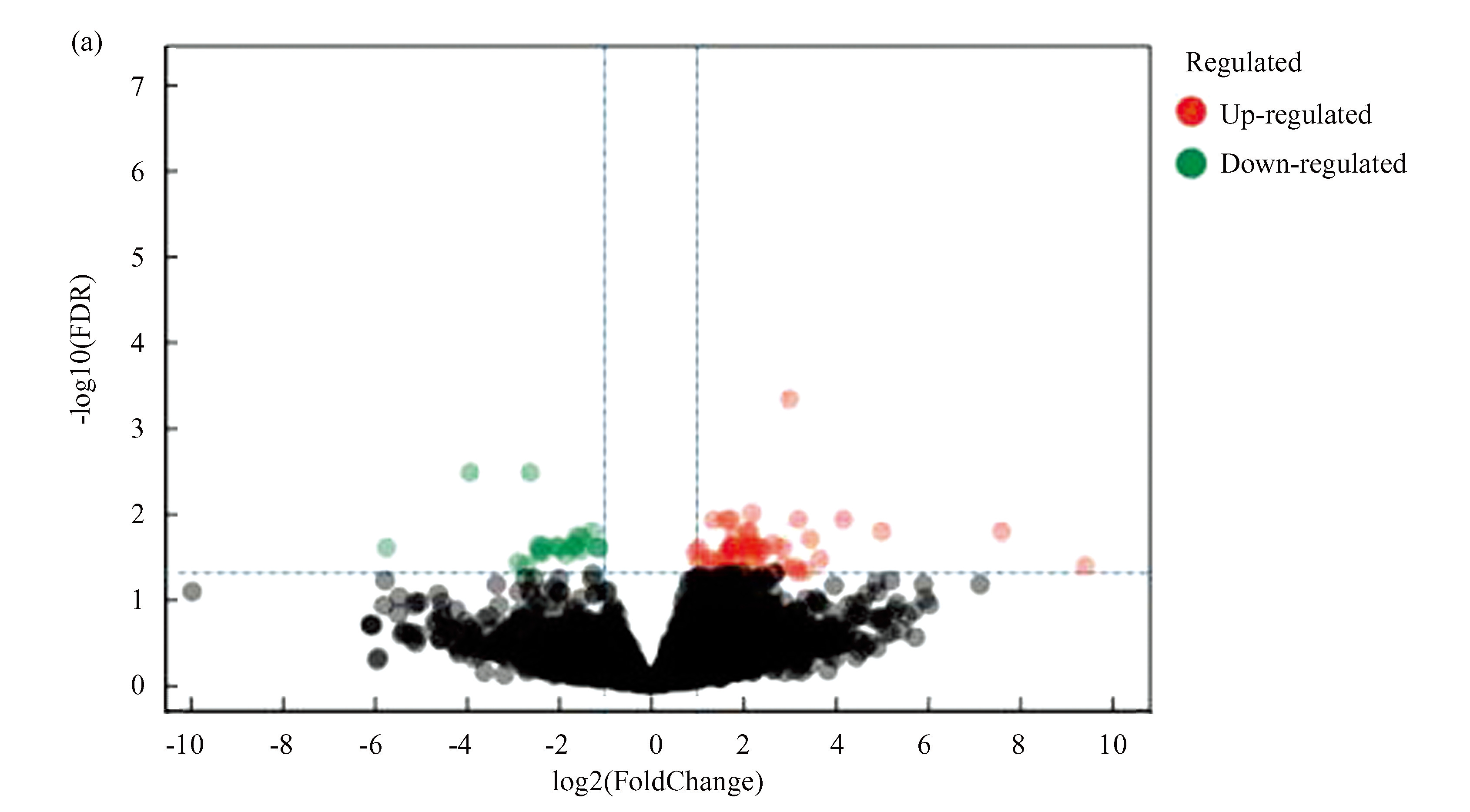
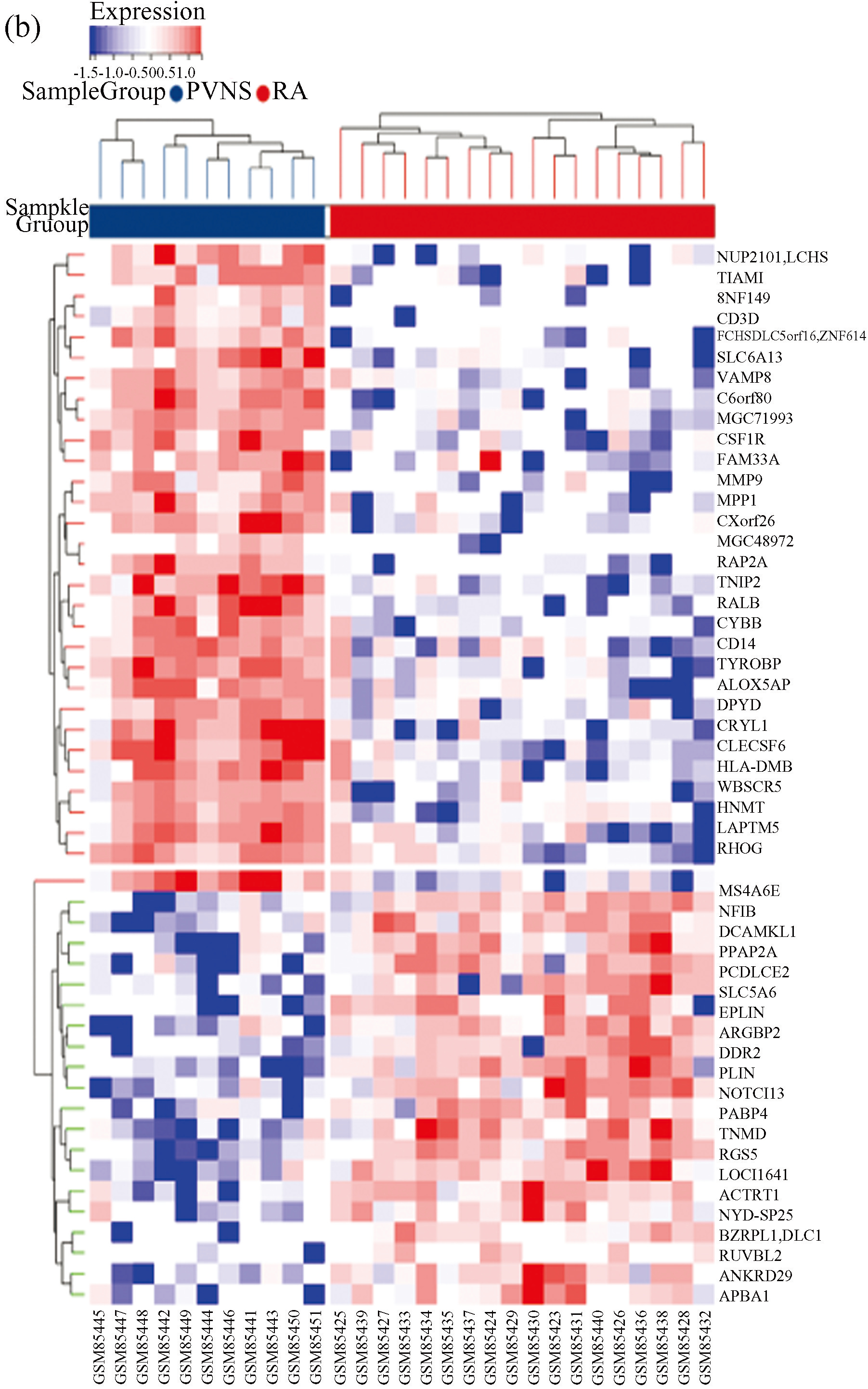
图1GSE3698数据集的火山图和热图
Fig.1Volcano map and heat map of dataset of GSE3698
注:(a)火山图;(b)热图.
2.2 关键模块基因的获取
WGCNA算法构建基因模块的过程中采用梯度法检测不同软阈值(1~20)时不同基因模块的独立性及平均连接度。当软阈值等于6时,各基因模块的平均连接度更高,因此,6为最适软阈值(图2(a))。通过动态树切割和层次聚类获得基因共表达模块(图2(b))。相关性分析显示前4个基因共表达模块中天青色(Turquoise)是PVNS最重要的模块(图2(c))。最后,将76个DEGs与WGCNA分析获得的334个关键模块基因取交集,产生22个PVNS和RA的共同基因(图2(d))。
2.3 功能富集分析和PPI网络
为了研究TMJ PVNS和RA可能的共病机制,我们使用DAVID数据库对22个共同基因进行GO和KEGG通路富集分析。功能富集分析显示,前3位显著富集的生物途径分别为:① GO-BP:免疫效应过程的调节,抗原处理和通过MHC-II类外源肽抗原的呈递以及免疫反应;② GO-CC:分泌性颗粒膜、溶酶体膜和溶解液泡膜;③ GO-MF:肽结合、中间结合和免疫受体活性(图3(a))。KEGG富集分析表明,吞噬体可能在TMJ PVNS和RA病理过程中发挥重要作用(图3(b))。将前次所得22个TMJ PVNS和RA的共同基因导入STRING及Cytoscape数据库,用于建立PPI网络和检验TMJ PVNS和RA之间共同基因的互作关系(图3(c))。
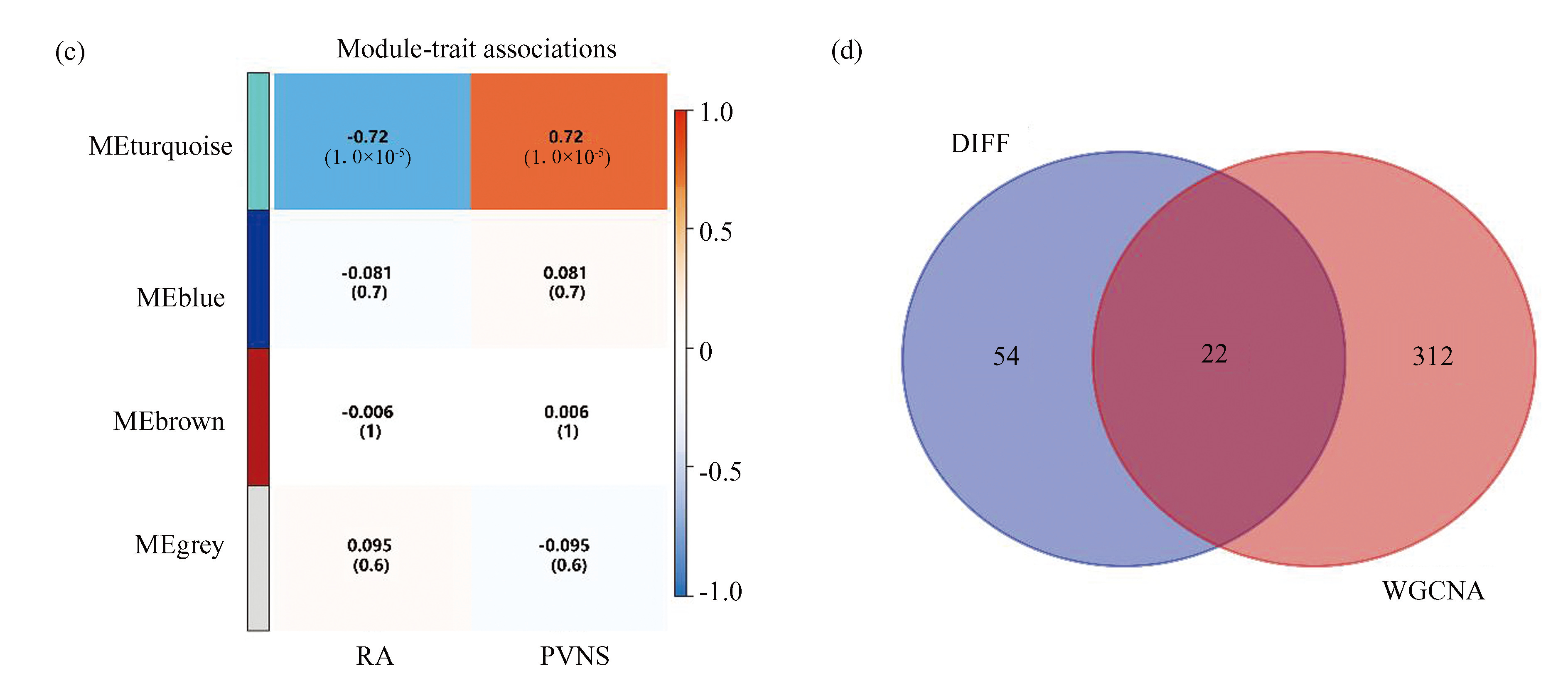
图2WGCNA算法获得DEGs和选定关键模块基因的共同基因
Fig.2Common genes obtained from the intersection of DEGs and selected key module genes using WGCNA method
注:(a)软阈值的确定;(b)动态剪切树交联图;(c)通过将PVNS和RA的临床特征联系起来,获得了4个不同颜色的模块;(d)WGCNA选定基因与前次DEGs产生基因集的韦恩图.(扫本文首页二维码见彩图).
2.4 基于机器学习算法识别Hub基因
1)Lasso回归模型确定了22个共同Hub基因中的8个,包括PLIN,PPAP2A,HLADRA,KIAA1949,RGS5,ALOX5AP,TYROBP和SLC2A5(图4(a)~4(b));2)RF算法则确定出如下13个Hub基因:CLECSF6,FABP4,TYROBP,LAPTM5,PPAP2A,PLIN,CAPG,FCGR2B,VAMP8,CD14,NFIB,IFI30和NOTCH3(图4(c)~4(d))。通过重叠2种机器学习算法选择的基因,PLIN,PPAP2A和TYROBP被最终确定为TMJ PVNS和TMJ RA共同的Hub基因(图4(e))。

图3TMJ PVNS和RA之间共同基因的功能富集分析和PPI网络
Fig.3Functional enrichment analysis and PPI network for examining the interrelationships of common genes between TMJ PVNS and RA
注:(a)前10个共同基因的GO分析结果,包括BP,CC和MF;(b)KEEG富集分析揭示了与TMJ PVNS和RA均密切相关的信号通路;(c)PPI网络表明,圆圈越大,其log2FC值就越大,颜色越红,其显著性也就越大.(扫本文首页二维码见彩图).
图42种不同机器学习算法识别Hub基因
Fig.4Identification of Hub genes based on two different machine learning algorithms
注:(a~b)Lasso回归模型;(c~d)随机森林算法;(e)TMJ PVNS和RA之间共同Hub基因的韦恩图.
2.5 Hub基因的诊断效能
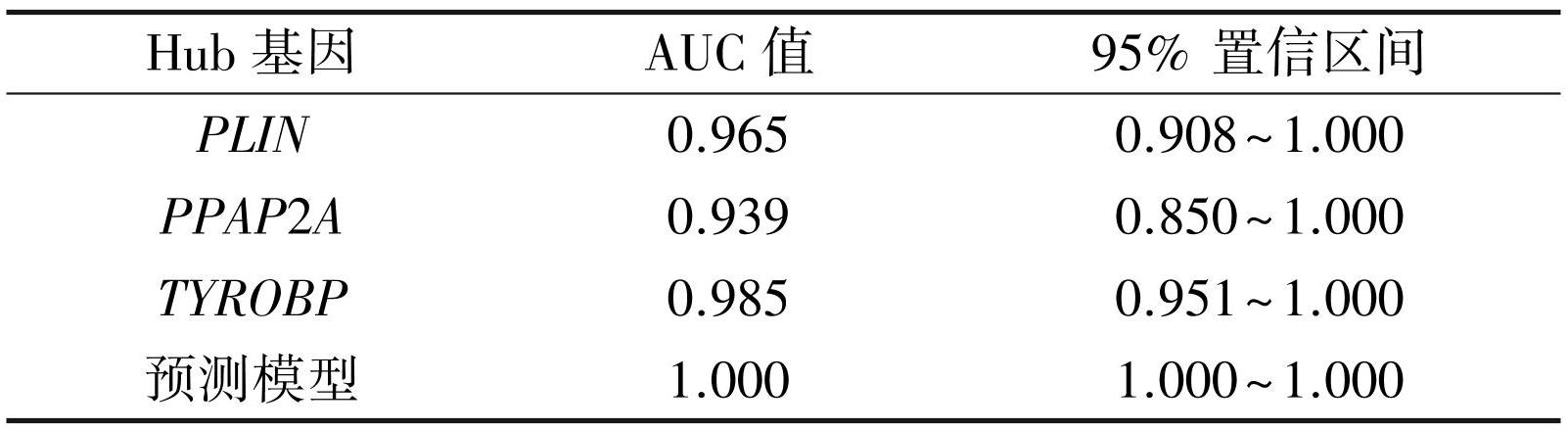
列线图用于估计3个Hub基因(PLIN,PPAP2A和TYROBP)的诊断性能(图5)。3个Hub基因及预测模型的SROC分析结果如表1所示。基于AUC > 0.9时的高诊断价值[16],我们确定PLIN,PPAP2A和TYROBP及其预测模型的诊断效能较好(表1)。

图5TMJ PVNS和TMJ RA的列线图预测模型
Fig.5Nomogram prediction model of TMJ PVNS and TMJ RA
2.6 PVNS和RA中Hub基因的ssGSEA
ssGSEA用于深入探究3个Hub基因的生物学功能。结果如下:①PLIN主要影响B细胞受体信号通路(图6(a));② PPAP2A显著影响氨基糖和核苷酸糖代谢途径(图6(b));③ TYROBP则主要影响溶酶体(图6(c))。
表1评价Hub基因及其预测模型的SROC分析
Table1SROC analysis for appraising Hub gene and its prediction model
2.7 免疫浸润分析
T细胞、自然杀伤(Natural killer,NK)细胞、巨噬细胞、活化的树突状细胞(Dendritic cells,DCs)和活化的CD8+ T细胞与TMJ PVNS呈显著正相关(P<0.001)。浆细胞样树突状细胞(Plasmacytoid DCs,pDCs)、单核细胞、髓源性抑制细胞(Myeloid-derived suppressor cells,MDSCs)、未成熟DCs、效应记忆CD8+ T细胞与TMJ PVNS呈显著正相关(P<0.01)。Th2细胞也与TMJ PVNS呈正相关(P<0.05)。而NK细胞CD56dim亚群、中性粒细胞和记忆性B细胞、效应记忆CD4+ T细胞则是与TMJ RA呈正相关(P>0.05)(图7(a))。
28 个免疫浸润细胞与3个Hub基因的关系如图7(b)所示:① PLIN与NK细胞CD56dim亚群和记忆性B细胞呈正相关(P<0.001);与T细胞和活化DCs呈负相关(P<0.001); ② PPAP2A 与NK细胞CD56dim亚群呈正相关(P<0.001); ③ TYROBP与调节性T细胞,pDCs,NK细胞,MDSCs,巨噬细胞,效应记忆CD8+ T细胞和活化DCs呈正相关(P<0.001)。TYROBP和中性粒细胞、NK细胞CD56dim亚群呈负相关(P<0.001)(图7(b))。
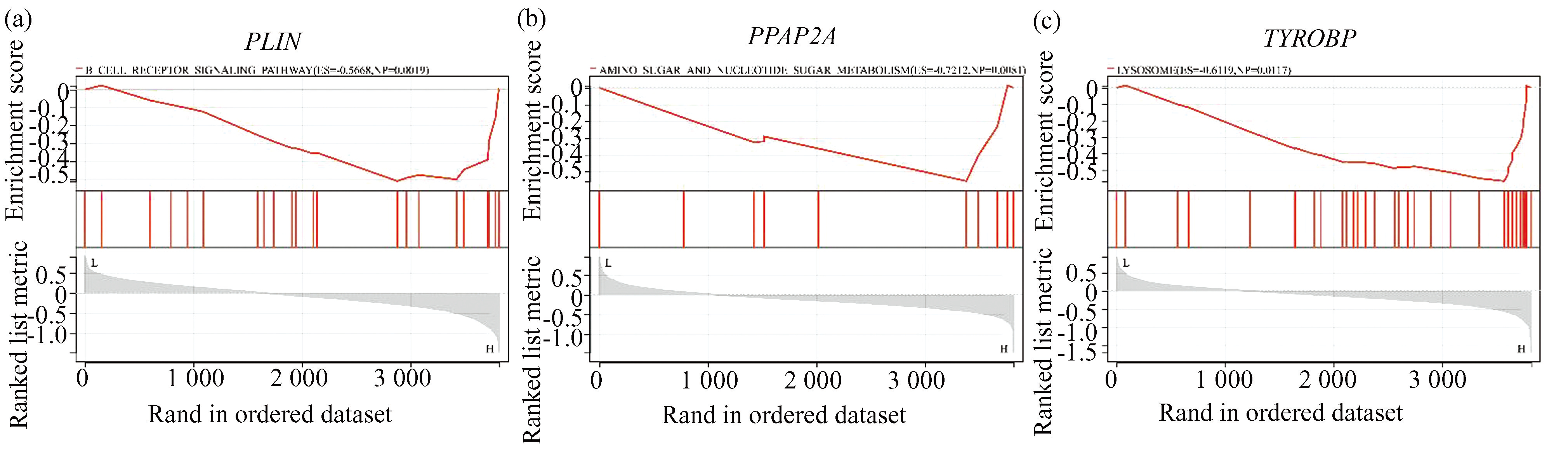
图63个TMJ PVNS和TMJ RA Hub基因的ssGSEA
Fig.6ssGSEA of three Hub genes of TMJ PVNS and TMJ RA
注:(a)PLIN;(b)PPAP2A;(c)TYROBP.
图7TMJ PVNS和TMJ RA样本中免疫细胞浸润的评估和可视化
Fig.7Assessment and visualization of immune cell infiltration in TMJ PVNS as well as TMJ RA
注:(a)28个免疫浸润细胞的表达差异;(b)Hub基因与浸润免疫细胞之间的相关性。低P值为绿色,而高P值为橘红色。(注:ns,无显著差异P < 1,# P < 0.2,* P < 0.05,** P < 0.01,*** P<0.001).(扫本文首页二维码见彩图).
2.8 Hub基因在PVNS和RA中的表达差异
PLIN和PPAP2A呈正相关性,而与TYROBP呈负相关性(图8(a)),TYROBP在TMJ PVNS中的表达水平明显高于RA(图8(b)),而PPAP2A和PLIN在TMJ PVNS中的表达水平则明显低于RA(图8(c)~(d))。总体研究结果表明,本研究检测的Hub基因在TMJ PVNS和TMJ RA的诊断中是有价值的。
3 讨论
TMJ PVNS是一种非常罕见的肿瘤样增生性疾病。组织病理学以吞噬细胞内大量沉积的含铁血黄素为特点。核磁共振影像中也由于含铁血黄素是一种磁性物质,而在T1和T2加权图像上可形成斑点状或广泛的低信号区,且在快速场回波序列图像上表现尤为明显[3]。TMJ PVNS常侵及颅中窝(43%),甚至侵蚀硬脑膜;当病变破坏中耳区骨质时可导致听力损失(18%)[17]。TMJ PVNS的进展通常是潜伏性的,一般在临床症状发作多年前就开始了,且患者在停止治疗后容易复发,这将给患者带来巨大精神和心理负担。
既往分子生物学研究表明,PVNS可能是由CSF1基因在1p13位点及COL6A3基因在2q35位点发生染色体易位引起的[18-19];免疫组织化学研究表明,白细胞介素(Interleukin,IL)-1β,IL-6,肿瘤坏死因子(Tumor necrosis factor,TNF)-α、基质金属蛋白酶(Matrix metalloproteinases,MMP)-9,MMP-13和Ki-67在TMJ PVNS组织中高度表达,TNF-α刺激MMPs的产生,这可能导致TMJ PVNS中的软骨和骨破坏[20-21]。Bhatnagar等[22]也发现在PVNS中,巨噬细胞、组织细胞和浆细胞浸润聚集会刺激并加重炎症反应。固有免疫系统细胞如单核细胞、巨噬细胞和DCs,通过其吞噬作用、抗原呈递和细胞因子产生的功能,在RA发生和发展中发挥重要作用,最终也会导致骨和软骨的破坏[23-24]。
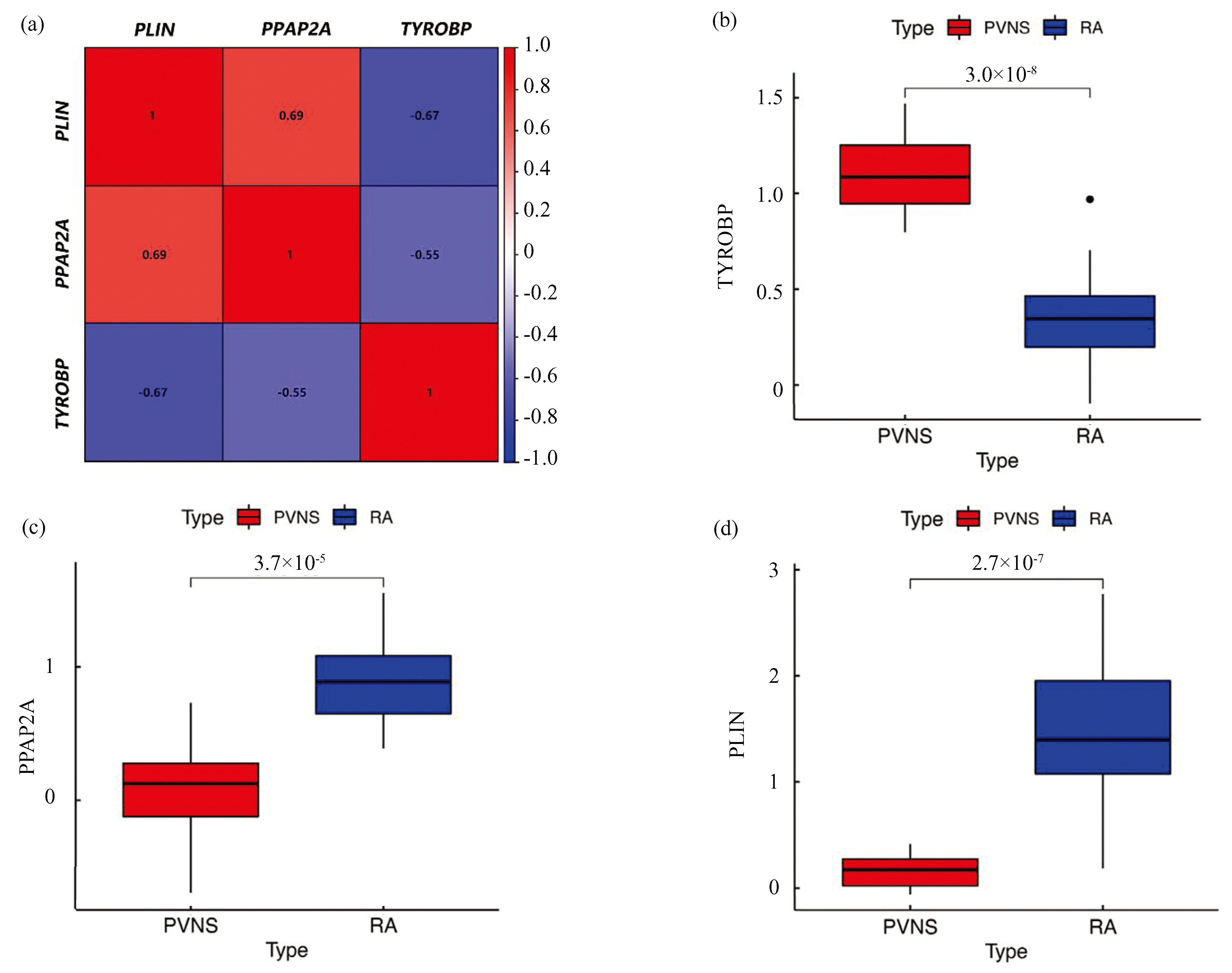
图8Hub基因在TMJ PVNS和TMJ RA中的表达差异
Fig.8Differential expression of Hub genes in TMJ PVNS as well as TMJ RA
注:(a)3个Hub基因相关性的热图;(b)PVNS中高表达的Hub基因;(c~d)RA中高表达的Hub基因.
本研究通过免疫浸润分析评估GSE3698数据集中28个免疫浸润细胞的差异表达。发现巨噬细胞、浆细胞、DCs、单核细胞等与PVNS呈正相关;NK细胞、中性粒细胞、巨噬细胞等与RA显著正相关。TMJ PVNS和TMJ RA均受到免疫浸润的影响。之前的研究表明,RA的细胞学表现与PVNS中增殖的单核滑膜细胞非常相似,增殖的滑膜细胞可刺激CD68+巨噬细胞积聚以加重炎症反应,故认为滑膜细胞增殖是RA和PVNS发病机制中的共同特征[21,25-26]。比较TMJ PVNS和TMJ RA中增殖滑膜细胞的免疫表型发现,相同的细胞群参与了增殖过程。在局限性和弥漫性PVNS中可见巨噬细胞和成纤维细胞过度增殖;且与弥漫性PVNS相比,成纤维细胞样滑膜细胞的数量在局限性PVNS中显著增加[3,27]。PVNS和RA中同样也可检测到伴随滑膜细胞的大量CD68+/163+巨噬细胞,不同的是这些细胞在RA中常集中在滑膜衬里层,而在PVNS中则分布较为分散[28]。
本研究通过2种机器学习方法筛选TMJ PVNS和TMJ RA共同的Hub基因。Lasso广义线性模型的数据需求量低,因此应用程度很广。这解决了用小样本筛选准确结果的问题[12]。RF算法对多元共线性不敏感,对于缺失数据和非平衡数据结果比较稳健,可以很好地预测多达几千个解释变量的作用[29]。因此,我们使用Lasso和RF分析筛选了二者的基因重叠,PLIN,PPAP2A和TYROBP被鉴定为PVNS和RA的Hub基因且均具有较高的诊断效能(AUC>0.9)。
4 结论
1)机器学习在分析、筛选、预测TMJ PVNS和TMJ RA共病的生物标志物中发挥着重要作用。
2)PLIN,PPAP2A和TYROBP是TMJ PVNS和TMJ RA共同的Hub基因。
3)这些异常表达的Hub基因和免疫细胞浸润密切相关,并可能在TMJ PVNS和TMJ RA的病理过程中起着关键作用。
4)PLIN,PPAP2A和TYROBP有望成为TMJ PVNS和TMJ RA的潜在诊断、预后标志物以及免疫治疗的靶点。



