摘要
运用生物信息学方法预测分析襄阳地区分离的柯萨奇病毒A组6型(Coxsackievirus A6, CV-A6)病毒蛋白(Viral protein, VP)中VP1的主要特性及表位。应用ProtParam,SOPMA,Phyre 2,DNAstar v8.1.3,Mega11,ABCpred,ElliPro,NetMHCpan-4.1,NetMHCIIpan-4.0等软件和在线网站预测分析CV-A6 VP1包括理化性质、结构特征、序列特点,亲缘关系在内的主要特性及B,T细胞抗原表位。结果发现该分离株VP1为碱性、不稳定的亲水性蛋白,其二级结构以无规则卷曲为主,核苷酸(氨基酸)同源性不一,有12个氨基酸突变位点,属于D3a亚型。该蛋白还存在多个潜在及优势B,T细胞抗原表位。CV-A6襄阳分离株与国内广西、广东地区CV-A6共进化共循环,具有一定的免疫潜能,可为分子流行病学监测以及多价疫苗的研制提供科学依据与参考。
Abstract
To predict and analyze the main characteristics and epitopes of VP1 in the viral protein (VP) of Coxsackievirus A6 (CV-A6) isolated from Xiangyang area using bioinformatics methods. The CV-A6 VP1's main characteristics including physicochemical properties, structural features, sequence characteristics, affinity and B/T cell epitopes are predicted and analyzed by software and online sites such as ProtParam, SOPMA, Phyre 2, DNAstar v8.1.3, Mega11, ABCpred, ElliPro, NetMHCpan-4.1, NetMHCIIpan-4.0. The isolate, VP1, is a basic, unstable, hydrophilic protein which has the secondary structure mainly consists of random coil, disparate nucleotide (amino acid) homology, 12 amino acid mutation sites, and belongs to the D3a isoform. There are also multiple potential and dominant B/T cell epitopes for VP1. The CV-A6 Xiangyang isolate co-evolved and co-circulated with CV-A6 in Guangxi and Guangdong, China, and has certain immune potential, which can provide scientific basis and reference for molecular epidemiological surveillance and the development of multivalent vaccines.
Keywords
手足口病(Hand,foot and mouth disease,HFMD)是由多种肠道病毒感染引起的急性传染性疾病,其主要临床症状表现为发热、手足臀部疱疹、口腔粘膜溃疡等,多发于5岁以下婴幼儿[1]。因我国多省份大规模爆发HFMD,并有死亡病例的报道,HFMD于2008年被列为《中华人民共和国传染病防治法》规定的丙类传染病进行管理。引发HFMD的肠道病毒多达二十几种,而近年来,部分地区CV-A6甚至超越肠道病毒A组71型(Enterovirus A71,EV-A71),柯萨奇病毒A组16型(Coxsackievirus A16,CV-A16)和柯萨奇病毒A组10型(Coxsackievirus A10,CV-A10)成为引起HFMD的最主要病原体[2]。CV-A6属于微小核糖核酸病毒科肠道病毒属A种,其基因组为单股正链核糖核酸,包含一个开放阅读框,编码3个前体蛋白P1~P3,其中P1又裂解成四种VP:VP1,VP2,VP3和VP4[3]。VP1在最外层,比例最高,是主要的表面可及蛋白。同时,VP1在病毒感染过程中起着重要作用,有许多重要的中和表位和毒力决定簇,可以区分和捕获细胞受体并协助病毒进入宿主细胞[4]。生物信息学是一门多学科交叉的新兴学科,能通过计算机挖掘生物相关数据背后所包含的生物学意义,其形成和发展对生物医药领域产生重大影响。本文采用生物信息学方法对CV-A6 VP1的主要特性以及B,T细胞抗原表位进行预测分析,为该病原体的生物学特性研究以及多价疫苗的研制奠定了理论基础。
1 材料与方法
1.1 材料
本研究选用的CV-A6临床分离株(CV-A6-HEV69/XY/CHN/2017)由中国医药集团武汉生物制品研究所于2017年从中国湖北省襄阳市HFMD患者标本中分离获得[5],其相关信息已上传至美国国立生物信息中心数据库(National center for biotechnology information,NCBI)中的GenBank公共数据库。该毒株的基因序列共有7 435个碱基对(GenBank登录号为:MW410845.1),编码2 201个氨基酸(GenBank登录号为:QSV27511.1),其VP1氨基酸序列位于第566~870位。
1.2 方法
1.2.1 CV-A6 VP1主要特性预测
通过Expasy[6]提供的在线分析工具ProtParam分析CV-A6 VP1的理化性质;运用SOPMA[7]网站在线分析CV-A6 VP1的二级结构;通过Phyre2[8]网站预测CV-A6 VP1的三级结构;利用ProSA-web[9]和PDBsum[10]服务器分析模型的整体质量与立体化学结构的合理性;应用DNAstar v8.1.3[11]软件提供的Megalign模块对CV-A6 VP1区核苷酸和氨基酸同源性及氨基酸位点变异进行分析;运用Mega11[12]软件通过邻接法(Neighbor-Joining)、Kimura2-parameter模型、Bootstrap检验(重复1 000次)完成基础进化树的构建,并将其输出为“nwk”格式的进化树文件上传至Interactive Tree Of Life v5[13]网站的“My trees”中进行美化。不同的常见肠道病毒原型株及国内外参考株VP1区序列均来自NCBI的GenBank。
1.2.2 CV-A6 VP1抗原表位预测分析
基于上文蛋白二级结构预测结果,借助DNAstar v8.1.3软件提供的Protean模块分析CV-A6 VP1各氨基酸性质,同时结合机器学习法ABCpred[14]、随机森林法Bepipred 2.0[15]、支持向量机法BCPREDS[16]综合预测潜在线性B细胞抗原表位;通过ElliPro[17],DiscoTope-2.0[18]综合预测潜在构象性B细胞抗原表位,并用ChimeraX[19]将其可视化;运用NetMHCpan-4.1,NetMHCIIpan-4.0工具[20]并选择相应参数进行潜在细胞毒性T淋巴细胞(Cytotoxic T lymphocyte,CTL)、潜在辅助性T细胞(Helper T cell,Th)抗原表位的预测。其抗原肽的长度分别设为9,15,等位基因选择中国常见及确定的人类白细胞抗原(Human leukocyte antigen,HLA)等位基因表(Common alleles and well documented alleles,CWD)2.2版[21]中频率大于1%的HLA-A型、HLA-B型、HLA-C型和HLA-DR型等位基因;通过Consurf[22],VaxiJen v.2.0[23],AllerTOP v.2.0[24],ToxinPred2[25],IEDB immunogenicity[26]对上述预测所得的潜在B,T细胞抗原表位分别进行保守性、抗原性、致敏性、毒性、免疫原性分析,最终可得优势B,T细胞抗原表位。
2 结果
2.1 CV-A6 VP1主要特性
ProtParam分析结果显示,该蛋白由305个氨基酸组成,各氨基酸占比见图1,分子式为C1472H2276N414O460S12,分子量为33 517.48 Da。VP1的N端为天冬氨酸,在哺乳动物网织红细胞体外、酵母体内、大肠杆菌体内的半衰期分别约为1.4 h,3 min,>10 h。在水溶液中(A280 nm),VP1半胱氨酸残基若全部形成二硫键,则消光系数为44 475 M-1·cm-1,若二硫键完全打开,则消光系数为44 350 M-1·cm-1。VP1理论等电点为8.46,是碱性蛋白,其不稳定系数为40.04(>40),归类为不稳定蛋白,其脂肪族指数为63.64,总平均亲水系数为-0.428(<0),归类为亲水性蛋白。
CV-A6 VP1各二级结构预测结果显示α-螺旋约占22.62%,β-折叠约占22.95%,β-转角约占2.62%,无规则卷曲约占51.80%(图2)。
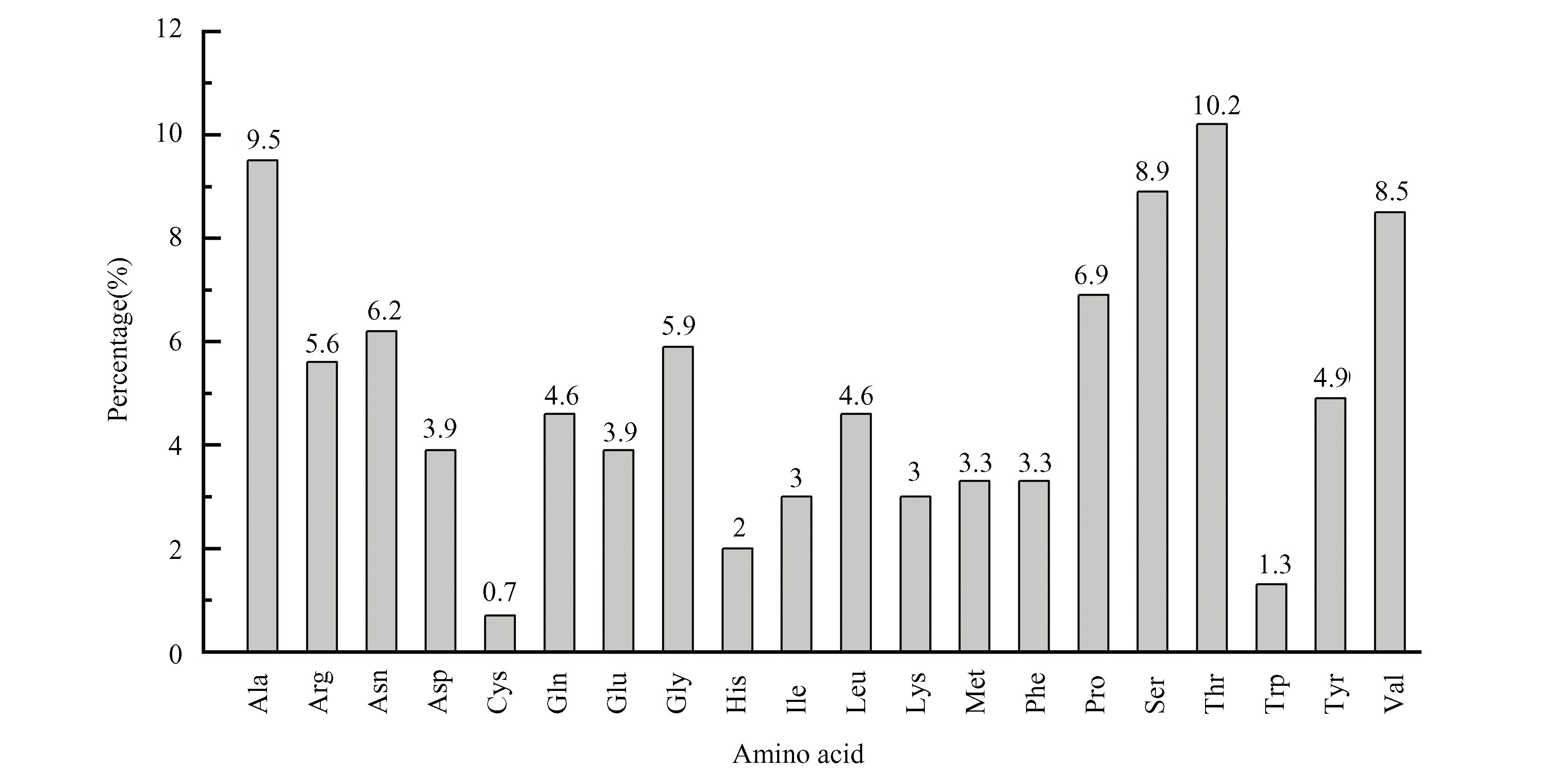
图1CV-A6 VP1氨基酸组成
Fig.1Amino acid composition of CV-A6 VP1
图2CV-A6 VP1二级结构预测
Fig.2Prediction of secondary structure of CV-A6 VP1
注:(a)不同位点所对应的各二级结构,h为α-螺旋,用蓝色表示,e为β-折叠,用红色表示, t为β-转角,用绿色表示,c为无规则卷曲,用橙色表示;(b)各二级结构得分.(扫本文首页二维码见彩图).
CV-A6 VP1经同源建模,与多个模板比对分析,从中选出评分排名第一的c5xs5A_(图3(a)),其氨基酸序列一致性为98%,可信度可达100%;对于该模型,ProSA-web在线评估结果(图3(b))显示,其Z-score值为-4.31,位于已知X-ray,核磁共振(Nuclear magnetic resonance,NMR)结构的蛋白质结构范围内,可以识别为天然蛋白质结构。PDBsum结果显示(图3(c)),模型中100%的氨基酸残基的二面角都在合理的范围内,符合立体化学能量规则。
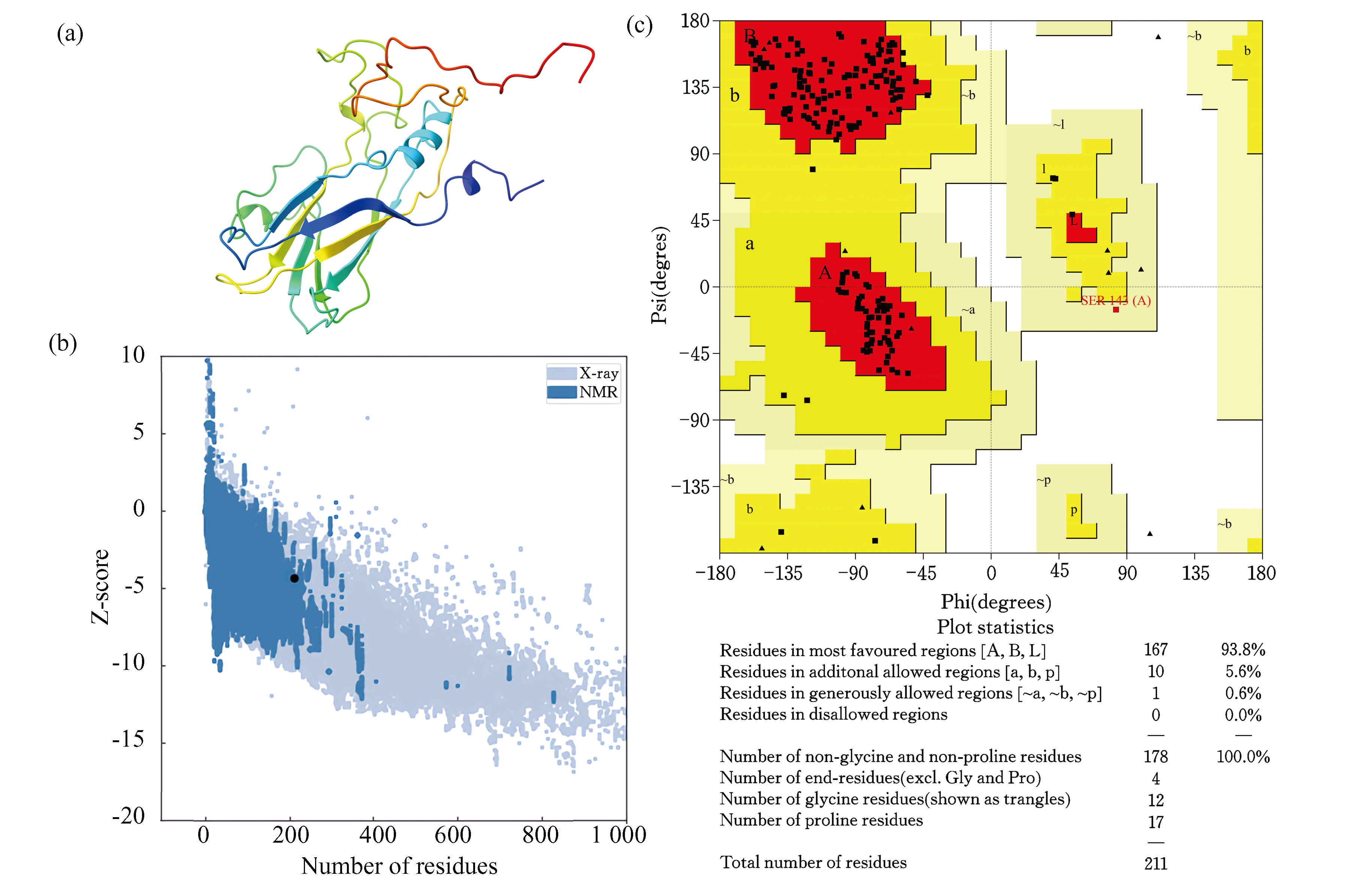
图3CV-A6 VP1三级结构的预测与验证
Fig.3Prediction and validation of the tertiary structure of CV-A6 VP1
注:(a)Phyre2同源建模所得的三级结构模型,蛋白质的N端到C端为模型中的蓝色到红色;(b)ProSA-web验证模型在能量上的合理性,来自不同来源(X-ray、NMR)的结构组以不同的颜色区分,实心黑点为模型;(c)PDBsum验证模型立体化学结构的合理性,横纵坐标表示肽链中α碳的二面角,phi(φ)为α碳左边C-N键的旋转角度,psi(ψ)为α碳右边C-C键的旋转角度,正方形黑点为各氨基酸残基(非Gly和非Pro)所处的空间位置.(扫本文首页二维码见彩图).
CV-A6襄阳分离株VP1区核苷酸和氨基酸同源性分析比对显示(图4),该毒株与CV-A6的A/B1/B2/C1/C2/D1/D2/D3a/D3b基因型及基因亚型的原型株和参考株VP1区核苷酸(氨基酸)一致性分别为83.3%(96.1%),83.3%(96.7%),84.7%(94.8%),85.4%(94.4%),84.0%(94.1%),90.4%~93.4%(97.7%~99.0%),89.3%~90.1%(97.0%~98.7%),96.9%~99.0%(98.4%~99.7%),93.6%~94.6%(97.4%~98.0%),与EV-A71/CV-A16/CV-A10的原型株和参考株的核苷酸(氨基酸)一致性分别为57.6%(57.0%),59.3%(57.7%~58.8%),58.4%(61.5%),29.4%~30.1%(61.2%~61.9%),61.8%(63.5%),27.2%~61.9%(61.8%~62.5%)。其中,相较其他CV-A6基因型及基因亚型,D3a亚型的核苷酸一致性相对较高,说明该分离株与D3a亚型毒株很可能同源,而CV-A6基因型与基因亚型之间的氨基酸一致性相差不大,则说明其在空间结构上具有很大程度的相似性。不同肠道病毒之间的核苷酸、氨基酸一致性均较低,说明肠道病毒VP1区序列在进化过程中具有较高的突变性,进而产生一定的差异。另外,在分析比对中,与原型株Gdula相比,CV-A6襄阳分离株VP1区共有12个位点的氨基酸发生了变异,分别是S5T,I8V,N10S,T14A,S32T,Q98L,D138N,G160S,S194T,F261L,S279T,F305S。
CV-A6襄阳分离株同GenBank上下载的肠道病毒原型株及国内外参考株共33株所构建的VP1区基因系统进化树显示(图5),该分离株属于D3亚型中的D3a分支,与CV-A6原型株Gdula及国外(如日本、法国等)分离株处于不同分支,亲缘关系较远,与2011年后的CV-A6中国毒株汇聚成簇,其中与来自广西、广东地区毒株亲缘关系较近。
2.2 CV-A6 VP1抗原表位
DNAstar v8.1.3 Protean预测显示,CV-A6 VP1有多处亲水性高、柔韧性好、抗原性强、表面可及性大、正负电荷分布区域(图6)。
基于二级结构结果及各氨基酸性质,通过ABCpred,Bepipred 2.0和BCPREDS在线分析工具综合得到潜在线性B细胞抗原表位(表1)。
图4CV-A6 VP1区核苷酸和氨基酸同源性分析
Fig.4Homology analysis of nucleotide and amino acid in the VP1 region of CV-A6
注:上三角为核苷酸同源性分析,下三角为氨基酸同源性分析,●为本研究CV-A6襄阳分离株.
表1CV-A6 VP1潜在线性B细胞抗原表位及分析
Table1Potential linear B-cell antigenic epitopes and analysis of CV-A6 VP1
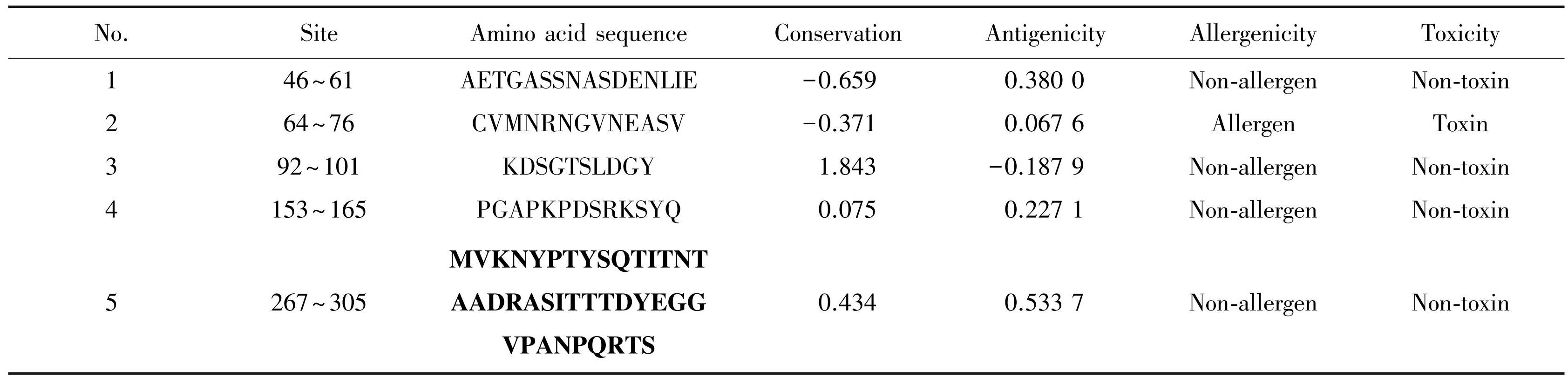
注:加粗的氨基酸序列为抗原表位分析后的优势抗原表位.
ElliPro和DiscoTope-2.0预测所得结果见图7,二者重叠区域即为潜在构象性B细胞抗原表位。
各等位基因预测所得结果中显示为强弱结合的肽段即为本研究所需的潜在T细胞抗原表位,这些肽段与各等位基因的对应关系见图8。通过热图可得高覆盖且多基因关联的潜在T细胞抗原表位(表2)。
图5CV-A6 VP1区基因系统进化树
Fig.5Phylogenetic tree of CV-A6 VP1 region gene
注:在CV-A6中,白色字体为A型,绿色字体为B1亚型,黄色字体为B2亚型,粉色字体为C1亚型,红色字体为C2亚型,蓝色字体为D1亚型,棕色字体为D2亚型,黑色字体为D3a亚型分支,灰色字体为D3b亚型分支。其中,加粗的黑色字体为本研究CV-A6襄阳分离株.(扫本文首页二维码见彩图).
表2CV-A6 VP1潜在T细胞抗原表位及分析
Table2Potential T cell antigenic epitopes and analysis of CV-A6 VP1

注:加粗的氨基酸序列为抗原表位分析后的优势抗原表位.
上述潜在B,T细胞抗原表位经不同在线工具分析后的保守性、抗原性、致敏性、毒性和免疫原性结果见表1、表2。其中,保守性得分小于1.000的为保守位点,介于1.000到2.000之间的为较易突变位点,大于2.000的为易突变位点。抗原性与免疫原性得分越高,抗原性与免疫原性就越强。由于构象性B细胞抗原表位不连续且来自于模板序列而非自身序列,故无法分析。
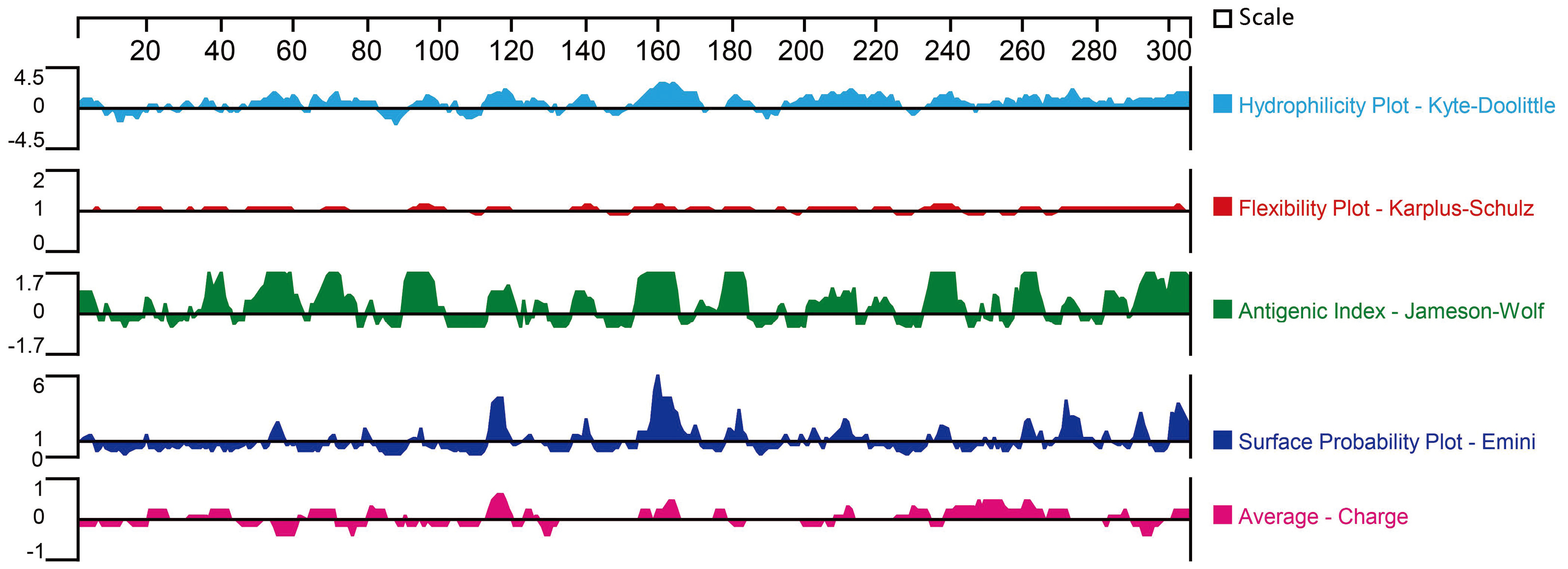
图6CV-A6 VP1的亲水性、柔韧性、抗原性、表面可及性、电荷分布预测
Fig.6Prediction of hydrophilicity, flexibility, antigenicity, surface accessibility, and charge distribution of CV-A6 VP1
注:亲水性高(>0);柔韧性好(>1);抗原性强(>0);表面可及性大(>1);正电荷分布(>0),负电荷分布(<0).
图7CV-A6 VP1潜在构象性B细胞抗原表位
Fig.7Potential conformational B-cell antigenic epitopes of CV-A6 VP1
注:在模型中,红色空心为ElliPro预测结果,黄色空心为DiscoTope-2.0预测结果,紫色实心为二者重叠部分;在氨基酸残基中,下划线为ElliPro预测结果,未做标记的为DiscoTope-2.0预测结果,灰色高亮为二者重叠部分.(扫本文首页二维码见彩图).
3 讨论
自我国自主研发的EV-A71疫苗于2015年面世以来,HFMD的防治取得了显著成效,但该疫苗只能预防EV-A71的感染,对其他能引起HFMD的病毒预防效果有限。研究表明[27],随着EV-A71灭活疫苗的广泛使用,HFMD的病例中由EV-A71感染导致的逐渐减少,而CV-A6,CV-A10和CV-A16比例却大幅提升,研发多价疫苗成为必然趋势。本文通过生物信息学技术,针对CV-A6 VP1的主要特性及表位进行预测分析,研究结果为后期HFMD监测以及多价疫苗的设计与研制提供理论基础。

图8不同肽段与HLA等位基因的对应关系热图
Fig.8Heat map of the correspondence between different peptides and HLA alleles
注:横坐标为不同肽段的起始位点,纵坐标为HLA等位基因.
本研究通过理化性质初步得出CV-A6 VP1为碱性、不稳定、亲水性蛋白,提示其暴露在病毒表面且在酸性环境中不稳定。此结果不仅说明该蛋白为表面可及蛋白,还解释了CV-A6病毒衣壳对酸的敏感,在低pH环境中很容易解离成12S五聚体[28]。理化性质预测结果为病毒蛋白的进一步研究提供诸多信息,提高了病毒蛋白相关研究的工作效率。在结构特性中,蛋白质的二级结构与抗原表位有着密切关系。α-螺旋和β-折叠为刚性结构,对蛋白质骨架起到稳定的作用,不易结合抗体,而结构相对较松散、易发生扭曲、盘旋并突出到蛋白表面的β-转角与无规则卷曲则有利于与抗体结合,故其常作为潜在抗原表位的判断依据。SOPMA二级结构分析显示,CV-A6 VP1以无规则卷曲为主,提示该蛋白上可能存在多个潜在抗原表位。以c5xs5A_构建的模型经ProSA-web,PDBsum验证,该模型质量较好、结构合理,说明其可用于后文的构象性B细胞抗原表位预测,并为后续分子对接、动态模拟等病毒蛋白亲和力研究助力。基于氨基酸序列的蛋白质二级和三级结构预测将有助于疫苗的研制,极大地降低了时间成本和金钱成本。
Song等[29]根据国内外CV-A6 VP1区序列将其分为A,B,C,D四个基因型,其中A基因型只有原型株Gdula,B,C,D基因型分为B1~B2,C1~C2,D1~D3亚型,而D3亚型又可进一步细分为D3a,D3b两个分支,中国大陆在2013年之后主要流行D3亚型,且主要集中在D3a分支。CV-A6襄阳分离株分析显示,其与D3a亚型毒株同源性较高,这与国内流行现状相符。该分离株还与CV-A6基因型及基因亚型在空间结构上有着很大的相似性,提示它们之间可能存在某些相同的功能。不同肠道病毒VP1区序列的比较结果显示其之间存在差异,而这种差异可以用于肠道病毒血清分型及分子流行病学研究。同时,VP1区序列较高突变性可能使病毒的变异不断累积,进而大幅提高其变异株的致病性和毒性,诱导新一轮HFMD的爆发[30]。本研究中的CV-A6襄阳分离株VP1区有12个氨基酸位点发生变异,均为氨基酸替换。史永林、李楠等[31-32]研究显示,安徽出现过S5T,I8V,N10S,T14A,N27S,A29T/V,S32T,Q98L,G160S,I174V,S194T,F261L,S279T,A283T,S305T的氨基酸变异,云南出现过I8V,N10S,T14A,S32T,Q98L,G160S,S194T,F261L,S279T,F305S的变异,而襄阳在S5T,I8V,N10S,T14A,S32T,Q98L,G160S,S194T,F261L,S279T,F305S也发生了变异,由此推测,这些位点容易发生变异,且不同地域、气候、人群可能是导致这11个氨基酸位点变异的主要原因。Zhou等[33]研究认为CV-A6毒株VP1区域(位点96~98,102,106,151,160,165,174和216)的氨基酸变异可能与主要的中和抗原表位相关,Wang等[34]研究发现的CV-A6引起小鼠致死模型中,致死毒株为5T,30A,137N和242V。CV-A6襄阳分离株VP1区S5T,Q98L,G160S的变异是否与传播力、致病力、毒力有关,还需进一步开展变异位点和流行病学监测分析。基于VP1区基因序列构建的系统进化树显示,CV-A6襄阳分离株属于D3亚型中的D3a分支,其流行很可能与D3a亚型分支毒株具有较强的毒力和感染性有关。该分离株与来自广西、广东地区毒株的遗传距离短,亲缘关系较近,这说明襄阳市CV-A6毒株可能来自2个不同传染源,可见CV-A6襄阳临床分离株在进化上不是独立的,而是与国内广西、广东地区的CV-A6分离株共进化共循环。其次,这些分离株(广西、广东)所在地区位于湖北省附近,而HFMD多由接触传播引起,可能是人口流动给予病毒跨省传播的机会。本研究不足之处在于CV-A6襄阳分离株仅1株,对分析CV-A6 VP1区序列特点缺乏一定的代表性,并且因为每年流行的CV-A6较多,不排除还存在其他基因亚型的可能。
病毒抗原表位对抗病毒免疫机制研究具有重要意义,可为疫苗的研发提供参考。徐莉、姚学君、Hadj等[35-37]对于B细胞抗原表位的预测比较单一,为避免单一分析方法的局限性和提高结果的准确性,本研究采用不同方法对CV-A6 VP1的抗原表位进行预测分析,以各结果的重叠表位作为潜在抗原表位。对于线性B细胞抗原表位的预测,本研究将更容易与抗体结合的二级结构及各氨基酸性质区域作为潜在抗原表位的优先选项,并结合ABCpred,Bepipred 2.0和BCPREDS的预测结果,最终筛选获得5个潜在线性B细胞抗原表位。此外,本研究还采用基于相邻残基的突出指数值进行聚类分析的ElliPro和基于蛋白质结构、表位倾向评分的DiscoTope2.0对构象性B细胞抗原表位进行综合预测,最终得到7个潜在构象性B细胞抗原表位。在预测线性与构象性B细胞抗原表位时,不同的分析方法所确定的重叠肽段区域将有更高的概率诱导相关的免疫应答,不过这需要体外和体内免疫激活实验的进一步确认。不同的抗原分子激活细胞免疫应答时需要不同的基因位点进行提呈,本研究选择了中国CWD 2.2版中频率大于1%的HLA I类、HLA II类等位基因。其中,由于HLA II类等位基因DQ型的α链和β链均是多态性的,组合形式复杂且高频搭配不明确,故本研究未作选取。不同于Deng,Ullah等[38-39]选择高人群覆盖率的HLA等位基因预测T细胞抗原表位,本研究在此基础上还从预测结果中选取重复出现的潜在T细胞抗原表位,其能与较多的HLA等位基因相关联,减少了HLA的遗传限制,实现少数抗原表位产生广泛的人群保护。最后,本研究还分析了这些潜在B,T细胞抗原表位的保守性、抗原性、致敏性、毒性和免疫原性,从中选取各性质相对较好的表位作为优势抗原表位,其具有高保守不易突变、易被T细胞捕获并快速诱导免疫应答、非过敏原、无毒等特点,但优势CTL抗原表位有所不同,在致敏性分析中均为过敏原,这需要在疫苗设计时进一步证实,并规避对其过敏的人群。另外,第92~101位的KDSGTSLDGY分析显示其较易突变,这可能是CV-A6毒力改变的原因之一[40]。
综上所述,本研究采用生物信息学方法对CV-A6 VP1进行了系统分析,预测得到VP1的理化性质、二级结构、三级结构、同源性、变异位点、亲缘关系,且获知该蛋白中存在多个潜在B,T细胞抗原表位以及基于潜在表位之上的优势B,T细胞抗原表位。这些主要特性与抗原表位将为CV-A6 VP1的深入了解以及多价疫苗的研究等提供理论依据与参考,对HFMD的预防和治疗具有重要意义。




