摘要
空间转录组(Spatial transcriptomics, ST)测序技术可以捕获多个细胞的空间位置信息,但无法达到单细胞分辨率,阻碍了对细胞类型异质性空间模式和基因表达特异性的解析。针对ST数据,本文提出了基于DenseNet网络结构和CORAL域自适应理论的细胞类型解卷积算法(STDN)。STDN通过学习引入的单细胞转录组(Single cell RNA sequencing, scRNA-seq)数据的细胞类型信息,利用迁移学习模型将其迁移到ST数据上,从而达到预测ST数据中每个捕获位点(Spot)的细胞类型组成及比例的目的。本文通过4组scRNA-seq真实数据及模拟的配套ST数据,表明STDN可以有效地恢复细胞类型转录谱及其在Spots内的比例,且优于其它解卷积算法。STDN对小鼠海马体和人类胰腺导管腺癌的ST数据进行解卷积,确定了组织中的多种细胞类型,解析了组织和癌症的高度异质性,为研究疾病的致病机理奠定了基础。
Abstract
Spatial transcriptomics sequencing technology captures spatial location information of multiple cells, but single-cell resolution cannot be achieved, which hampers the analysis of spatial patterns of cell type heterogeneity and gene expression specificity. The cell type deconvolution algorithm (STDN) based on DenseNet network structure and CORAL domain adaptive theory is proposed for spatial transcriptomics data. STDN learns cell type information about introduced single-cell RNA sequencing (scRNA-seq) data and migrates it to ST data using a transfer learning model. Thus, the purpose of predicting the cell type composition and proportion of each capture site (Spot) in the ST data is achieved. In this paper, four factual scRNA-seq datasets and simulated matching ST datasets show that STDN can effectively recover cell type transcription profiles and their proportions in Spots, and is superior to other deconvolution algorithms. By deconvolution of ST data from mouse hippocampus and human pancreatic ductal adenocarcinoma, STDN identifies multiple cell types in tissues, resolves the high heterogeneity of tissues and cancers, and laid a foundation for studying the pathogenesis of the disease.
Keywords
描绘组织或疾病的不同细胞类型的空间结构对于解析组织或疾病中细胞功能和分子结构至关重要[1]。空间转录组测序技术的出现,使得从空间环境中研究组织的基因表达谱成为可能[2]。空间转录组测序技术对每个捕获位点(Spot)进行测序,在获取基因表达信息的同时保留了空间位置信息,然而,每个Spot包含多个细胞,测量值则为潜在异质细胞类型细胞混合物的平均基因表达。单细胞测序技术在将实体组织解离成单个细胞进行测序的过程中,虽然丢失了空间位置信息,但可得到单个细胞的转录本信息[3]。因此,利用空间和基因组信息解析每个Spot中的细胞类型及其占比,即对ST数据进行细胞类型反卷积,成为了解析细胞类型的空间位置和表征复杂组织结构的关键步骤[4]。
最近大量针对ST数据的细胞类型解卷积算法被开发,大致分为基于统计学习和机器学习两类算法。SPOTlight[5]采用非负矩阵分解回归作为核心算法,考虑先验信息,借助细胞类型的Marker基因初始化基矩阵与系数矩阵,使用非负最小二乘法(Non-negative least squares)计算Spot的系数矩阵及其细胞类型组成,从推断复杂组织内细胞类型及状态的空间分布。SpatialDWLS[6]通过细胞类型富集分析确定可能存在于每个Spot的细胞类型,利用阻尼加权最小二乘法(Damped least squares)选择使总体相对错误率最小的权重,从而定量估计每个Spot的细胞类型的精确组成。RCTD[7]以带有细胞类型注释的scRNA-seq数据作为参考,在消除样本批次效应的同时识别ST数据每个Spot的细胞类型,再通过拟合统计模型精确解卷积。Cell2location[8]是基于贝叶斯模型构建的,考虑了数据样本的不同技术来源,借用了跨位置的统计强度,从而实现单细胞测序数据和空间转录组数据的整合。该算法可解析空间转录组测序数据中的细粒度细胞类型,并具有创建不同组织的综合细胞图谱的功能。CARD[9]是一种基于条件自回归的解卷积方法,它建立在非负矩阵分解模型的基础上,基于条件自回归建模假设,综合考虑scRNA-seq数据的细胞类型特异性基因表达信息和空间相关性对ST数据进行卷积。Stereoscope[10]基于单细胞转录组和空间转录组数据均遵循负二项分布(Negative binomial distribution)的假设,使用概率模型描述单细胞转录组和空间转录组数据之间的关系,对空间转录组的细胞类型混合物进行引导解卷积,从而将单细胞转录组细胞类型映射到空间转录组。
近些年,机器学习被广泛用于空间转录组数据解卷积算法的研究。DSTG[11]使用共享最近邻学习ST模拟数据和ST真实数据之间的链接图,以获得的链接图为基础,通过半监督图卷积网络学习局部图结构和基因表达模式的潜在表示,进而预测ST真实数据中的细胞类型组成。STRIDE[12]对带细胞类型标签注释的scRNA-seq数据进行主题建模,根据在scRNA-seq数据中获得的基因-主题分布,使用隐狄利克雷分布(Latent dirichlet allocation,LDA)估计Spot-主题分布,而后整合Spot-主题分布和细胞类型-主题分布计算各Spots包含每种细胞类型的概率,将这种概率视为每种细胞类型在该Spot中所占的比例,从而达到对ST数据进行细胞类型分解的目的。DestVI[13]是一种用于ST数据中细胞类型的多分辨率解卷积的贝叶斯模型。该算法引入了解码器神经网络的变分推理模型,它使用条件深度生成模型学习离散的细胞类型特异性分布和连续亚细胞类型潜在变化。在Spots中的转录本数量遵循负二项分布的前提假设下,DestVI分别为scRNA-seq数据(scLVM)和ST数据(stLVM)构建了不同的潜在变量模型(LVM). stLVM使用由scLVM训练的解码器神经网络,并使用最大后验(MAP)估算细胞类型比例。张柳[14]构建了一种基于统计学习方法和迁移学习的方法,该模型将原本用于图片识别的子空间对齐领域自适应迁移学习方法用于scRNA-seq和ST数据,通过对齐源域和目标域的子空间实现ST数据的降维,批次效应校正和细胞类型预测。CellDART[15]从单细胞转录组数据中随机选择细胞构成一个细胞类型比例已知的伪Spot,从伪Spot的基因表达中提取细胞部分信息训练神经网络模型,应用于空间转录组数据的不同Spot,有望帮助阐明细胞的空间异质性及其在各种组织中的紧密相互作用。
尽管存在多种ST数据解卷积算法,但这些算法仍存在数据信息提取不够充分、对数据敏感性高等问题。例如,基于统计学习方法的SPOTlight和RCTD没有将捕获的位置信息合并到模型空间分解中,SpatialDWLS估计稀有细胞类型的比例时偏差较大;基于机器学习方法的DSTG高度依赖建模图卷积神经网络的链接图的质量,CellDART特异性不够高等。因此,迫切需要研究新方法,深度整合单细胞和空间转录组数据中的互补信息,实现多源生物数据的整合,从而更准确推断每个组织Spot的细胞组成。
本文开发了一种基于深度迁移学习模型的细胞类型解卷积算法STDN,用于解析ST数据中每个Spot的细胞类型组成。STDN是基于DenseNet和相关对齐(CORrelation alignment,CORAL)[16]搭建的,通过共享基因集建立scRNA-seq数据集和ST数据集之间的联系,再引入迁移学习中的领域自适应技术来充分挖掘两类数据集潜在的信息特征,从而利用从scRNA-seq数据中学到的细胞类型知识来解决ST数据中每个Spot的细胞类型识别问题,更精确地实现ST数据的细胞类型解卷积。
1 方法
1.1 STDN算法
为了整合scRNA-seq和ST数据,以实现ST数据解卷积,本文提出了STDN算法,图1的三幅子图分别展示了STDN建模的整体流程(a)、STDN模型训练过程(b)和STDN模型预测过程(c)。
STDN的输入为scRNA-seq和ST数据,输出为ST数据中每一个Spot包含的细胞类型及其占比。首先,对scRNA-seq和ST数据进行共享特征选择。然后,STDN基于DenseNet模型,使用基因表达数据和细胞类型标签进行模型训练,在隐藏层使用CORAL减少单细胞转录组和空间转录组之间的差异,学习提取与细胞类型相关的特征用于模型预测。最后,使用训练好的模型对空间转录组数据进行预测,将单细胞转录组数据的细胞类型标签转移到Spot上,从而对每个Spot中的细胞类型及其占比进行估计。
图1(a)STDN流程图;(b)STDN模型训练;(c)STDN模型预测
Fig.1(a) Pipeline of STDN; (b) STDN model training; (c) STDN model prediction
1.2 共享特征选择
特征选择是机器学习中必要的数据预处理步骤,旨在从已有的特征集合中选择与任务相关的特征子集以提高模型运算效率,同时确保不丢失重要信息。生物基因测序数据具有高维度的特点,因此在分析之前尤其需要进行特征选择。生物大数据的分析过程中容易遇到特征过多造成的维数灾难问题,与此同时,去除不相关特征还可以排除无关元素的干扰,降低学习任务的难度,提高任务解决的效率。
除此之外,特征基因需在scRNA-seq数据和ST数据中均存在,则在模型训练之前对scRNA-seq和ST数据的基因取交集,从而得到共享特征空间,称为“共享基因集”。
1.3 STDN模型训练
STDN引入了一种深度迁移学习模型,迁移学习是从源域获取解决问题的知识并储存,然后应用该知识来解决目标域中类似问题的一种机器学习方法。
STDN模型以处理好的scRNA-seq数据和ST数据的基因表达矩阵以及scRNA-seq数据的细胞类型one-hot矩阵为输入,基因表达矩阵行为细胞或Spot,列为基因。经过3层5次聚合的DenseNet网络训练,选择Sigmoid激活函数,同时以细胞类型分类错误损失、度量scRNA-seq和ST基因表达之间潜在差异的CORAL损失和防止过拟合的L2正则化损失3种损失函数构成加权损失函数,损失函数在各层间反向传播信息,训练模型。具体模型训练流程如图1(b)所示。
STDN使用CORAL度量源域和目标域中数据分布差异,这是一种非对称转换,它计算的是源域和目标域特征的二阶统计量(协方差)之间的距离。设源域训练样本为,标签为LS={yi},i∈{1,···,L}. 目标域数据为,其中d为网络的输出个数,即神经元个数,目标域无标签。CORAL损失计算公式如下:
(1)
其中表示Frobenius范数,CS和CT分别表示源域和目标域的特征协方差矩阵,计算公式如下:
(2)
(3)
其中nS和nT分别表示源域样本和目标域样本个数,DijS和DijT分别表示源域和目标域第i个样本的第j个特征。
STDN模型使用了3层DenseNet网络,训练了5个模型进行聚合。下面以单层网络为例介绍STDN模型设置:首先,使用一个共同的隐藏层来合并scRNA-seq和ST的基因表达矩阵,隐藏层激活函数设置如下:
1)利用隐藏层使用Sigmoid激活函数将基因表达矩阵转化至较低维度,隐藏层激活函数设置为
(4)
(5)
其中,X表示基因表达矩阵,θHidden表示隐藏层权重,bHidden表示隐藏层偏差。
2)添加细胞类型输出层。空间转录组学数据可以有分类输出或无输出,无输出意味着没有匹配的细胞类型。将细胞类型层激活函数设置为
(6)
其中,X表示基因表达矩阵,θClass表示细胞类型层权重,bClass表示细胞类型层偏差。
接下来,添加输出层以预测空间转录组数据各Spot的细胞类型及比例。为了训练模型,需要分别计算分类输出和CORAL两种损失函数,公式如下:
(7)
(8)
其中,LossClass表示分类输出损失函数,LossCORAL表示CORAL损失函数。YType, i表示真实细胞类型,X表示输入的基因表达矩阵。XCell,XSpot 分别代表scRNA-seq数据基因表达矩阵和空间转录组数据基因表达矩阵,最小化CORAL损失是映射scRNA-seq数据和ST数据之间的数据代表性分布的关键。
另外,为了避免过拟合,模型添加一个L2正则化损失。因此,该模型总体损失函数是上述三种损失函数的加权和,见(9)式。
(9)
其中,λ1,λ2,λ3表示三种损失函数的权重,可以用来调整每个损失项和正则化项的重要性。损失函数反向传播达到模型训练的目的。
1.4 模型预测
训练好的迁移学习模型被应用于ST数据细胞类型及其占比预测,具体流程如图1(c)所示。模型输出是Sigmoid激活函数的输出,先对输出结果进行KNN平滑处理以消除随机噪声,后将输出值域为[0,1]的结果转换为类似相关系数的关联得分,公式如下:
(10)
其中AS表示关联得分(Associate score),Preds_Smooth表示平滑处理后的模型输出预测值,L表示预测标签数目。该关联得分代表包含某种类型细胞的可能性,最终得到以Spot为行,以细胞类型为列的得分矩阵,反映ST数据每个Spot包含的某种细胞类型的可能性。
1.5 模型效果评估指标
本文使用JS散度(Jensen-shannon divergence,JSD)[17]进行模型预测性能评估,这是一种衡量两个概率分布之间差异的信息熵方法,它对KL散度(Kullback-leibler divergence)进行了改进,解决了KL散度的不对称问题。KL散度及JS散度计算公式见(11)式、(12)式。
(11)
(12)
其中,P(x)表示真实细胞类型的分布,Q(x)表示训练后的STDN模型预测的细胞类型分布。JS散度的值域范围是[0,1],值越小表示两种分布越接近。可以通过这种方式计算所有Spots的JS散度,然后将所有Spots的JS散度的平均值作为评估指标,称为JSD分数。JSD分数越低说明预测细胞类型和真实细胞类型的分布越相似,则表明模型预测性能越好。
2 结果与讨论
2.1 基准测试
2.1.1 模拟数据集介绍
从文献[18]中收集了4组配套的scRNA-seq数据和ST模拟数据。4组scRNA-seq数据中2组数据来自人类健康胰腺组织,另外2组分别来自小鼠健康胰腺组织和小鼠健康气管组织,配套的ST数据由scRNA-seq数据模拟生成。
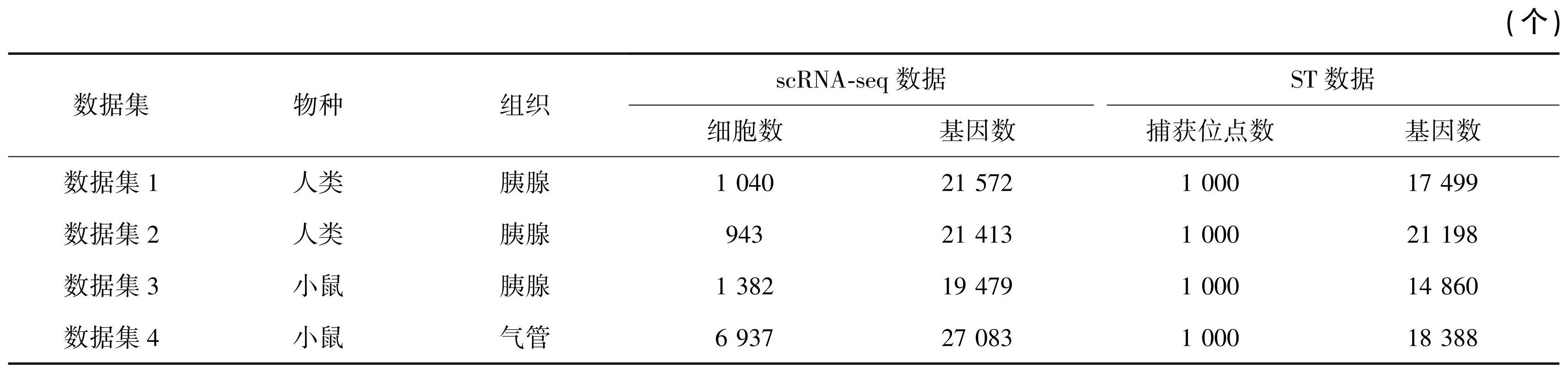
参考RCTD和Stereoscope的空间转录组数据模拟生成方法,设计了配套空间转录组数据的模拟生成过程。对于每个模拟Spot,首先采样细胞数目在5~15的均匀分布和采样细胞类型数目在2~6的均匀分布。然后假设这些细胞类型分布的可能性是相等的,并从scRNA-seq数据的每种细胞类型中随机分配细胞到该Spot。为了获得每个Spot的基因表达值,将一个Spot上所有细胞的基因表达值进行求和作为该模拟Spot的基因表达值。参考RCTD中空间转录组模拟数据集的构造方法,使用Scuttle包(http://bioconductor.org/packages/release/bioc/html/scuttle.html)将每个Spot的Count计数降采样到原始值的10%. 通过计算对应于细胞类型的细胞数来获得每个Spot上细胞类型的百分比,将每个模拟数据集的Spots数量设置为1 000。4组配套的scRNA-seq数据和ST模拟数据详细信息见表1。
表1scRNA-seq数据和ST模拟数据信息
Table1scRNA-seq data and the information of ST simulated data
2.1.2 ST模拟数据的解卷积结果
为了可比较性,本文为不同数据集设置统一的超参数:训练步骤为2 000,空间转录组数据一次训练所选取的样本数(Batch size)为200,单细胞数据一次训练所选取的样本数(Batch size)为50,隐藏层节点数为50,Dropout率为50%,3种损失函数的权重均设置为3。
将模型预测结果与ST模拟数据集的基本事实进行比较,从而评估模型解卷积性能。将空间转录组模拟数据的真实细胞类型分布和模型预测的细胞类型分布进行相关性分析,得到真实-预测细胞类型相关矩阵,可视化结果如图2所示,图中展示了4组数据真实细胞类型和预测细胞类型概率分布矩阵的相关性。观察4组数据相关性图发现,相关性图中对角线位置相关系数更高,说明真实细胞类型和预测细胞类型相同的关联性更高,不同细胞类型相关性低,表明模型预测结果和基本事实吻合度较高,说明了STDN具有较高的准确性。
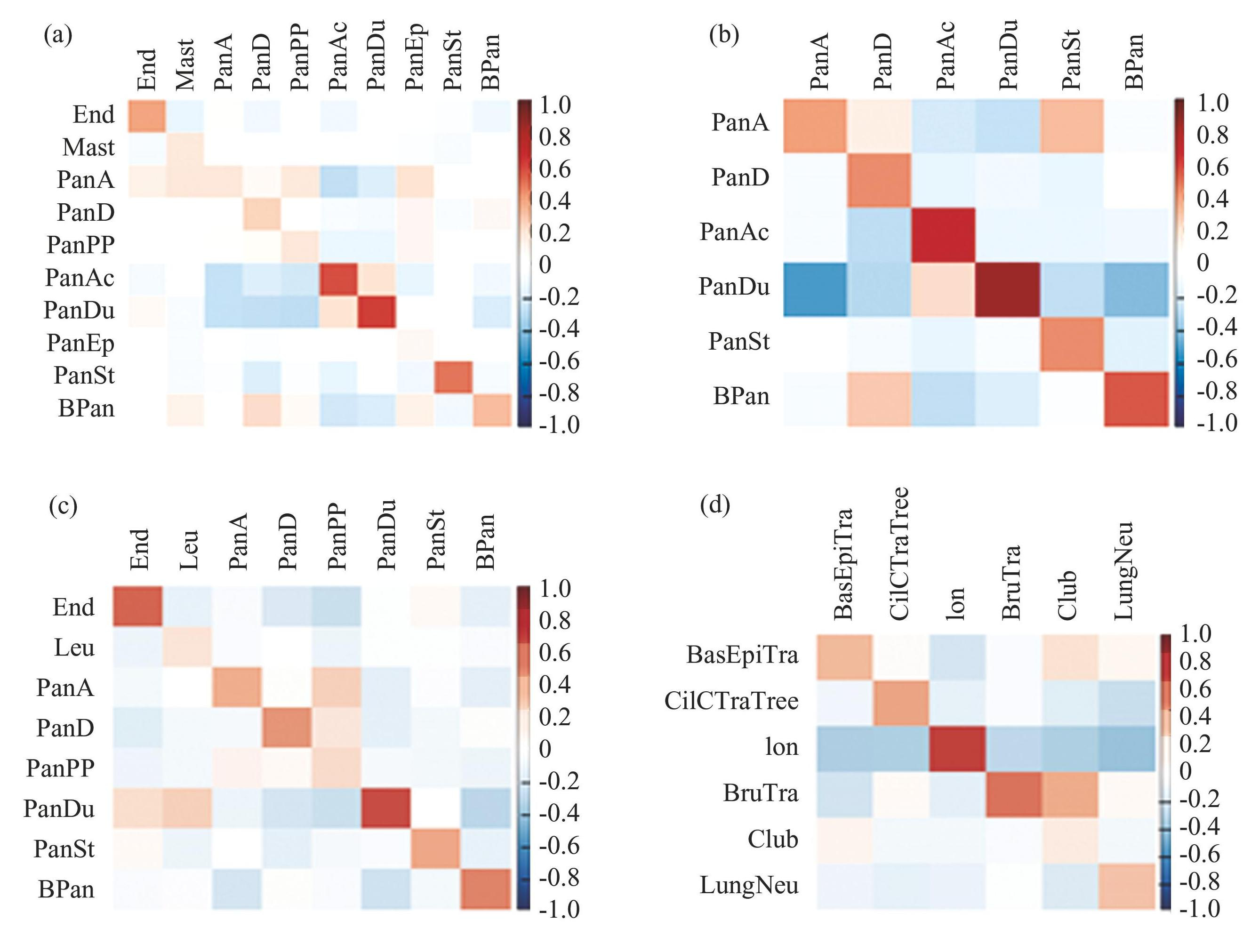
图2真实-预测细胞类型相关性图
Fig.2Heatmap of ground truth and predicted value
注:(a)ST模拟数据集1;(b)ST模拟数据集2;(c)ST模拟数据集3;(d)ST模拟数据集4.
为了更好地测试本算法的预测性能,本文将4组模拟的ST数据已知细胞类型和STDN算法预测的细胞类型进行对比,用JSD指标度量预测细胞类型分布和真实细胞类型分布之间的差异。通过计算得到的所有Spots的JS散度,以这些JS散度的平均值为指标评估模型解卷积性能。本文利用JSD指标,将STDN算法与五种经典解卷积算法(RCTD,Seurat,SpatialDWLS,SPOTlight和DSTG)进行比较。最终得到的指标值如图3,直观地展示了各算法解卷积性能的优劣。结果表明,相比于其他算法,STDN在4组ST模拟数据集中表现较好,JS散度指标值较低,分别为0.41,0.25,0.37,0.36,说明STDN性能基本优于其他算法。
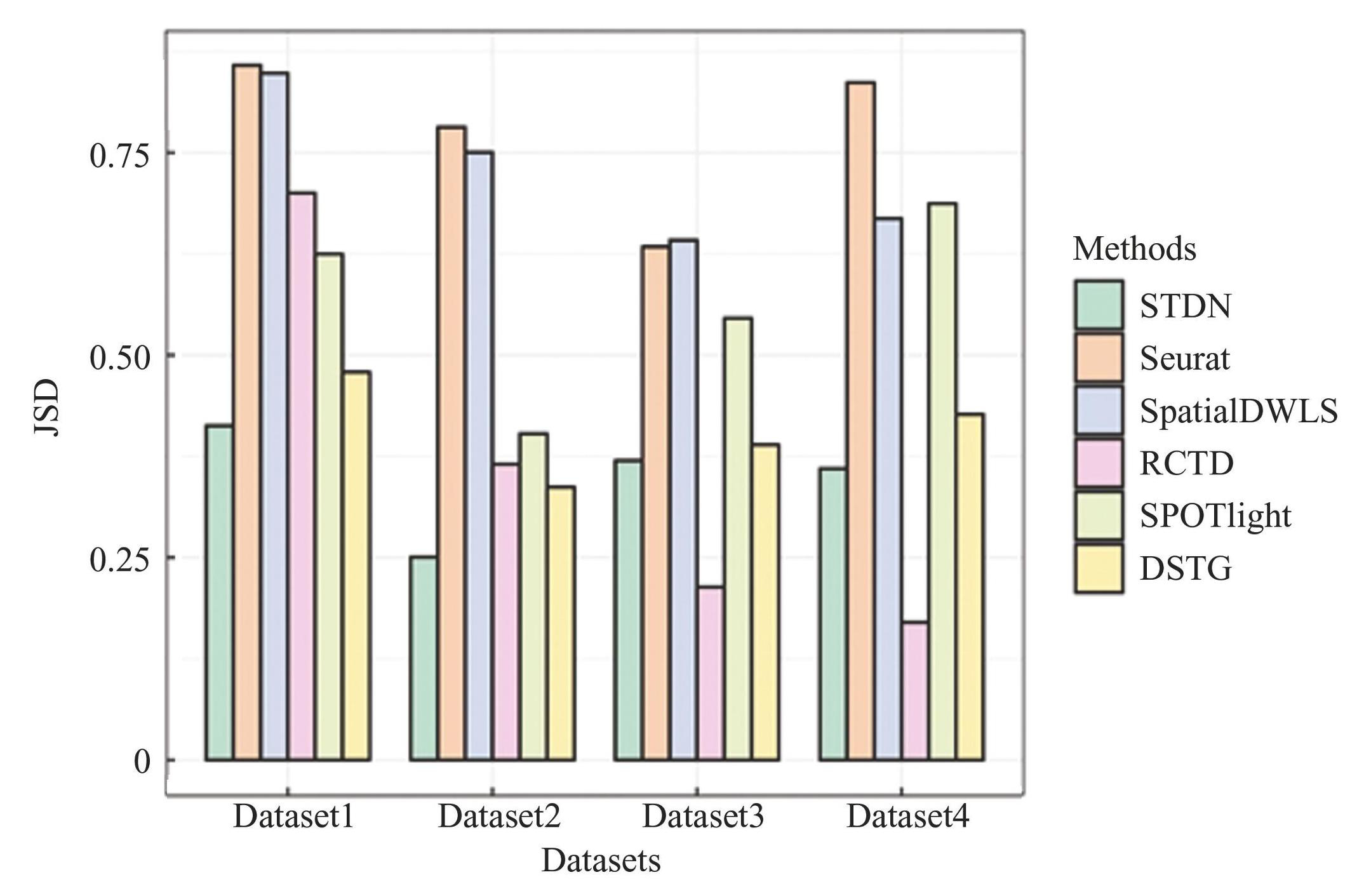
图3模型预测性能比较
Fig.3Prediction performance on different methods
2.2 小鼠大脑海马体数据分析
2.2.1 小鼠大脑海马体数据集
从文献[19]和GEO数据库中收集了配对的出生后7 d小鼠大脑海马体scRNA-seq数据和ST数据,GEO数据序列号为GSM4800800,GSM4800808。
2.2.2 实验结果分析
为了更直观地展示解卷积结果,本文将细胞类型预测关联得分投影到ST数据组织切片的空间位置,结果如图4所示,包括出生后7 d小鼠大脑海马体空间转录组测序的组织切片图像(图4(a))和7种细胞类型的预测结果(图4(b)~4(g))。图4(b)~4(g)分别反映了星形胶质细胞、内皮细胞、上皮细胞、成纤维细胞、小胶质细胞、神经元细胞、少突胶质细胞这7种类型细胞被每个Spot包含的可能性。每幅图横轴表示ST数据空间位置横坐标,纵轴表示ST数据空间位置纵坐标。图中一个点对应一个Spot,颜色表示关联得分的大小,越接近红色表示与对应细胞类型关联性越强,即该Spot含有该类型细胞的可能性越大,越接近紫色表示该Spot含有该类型细胞的可能性越小。
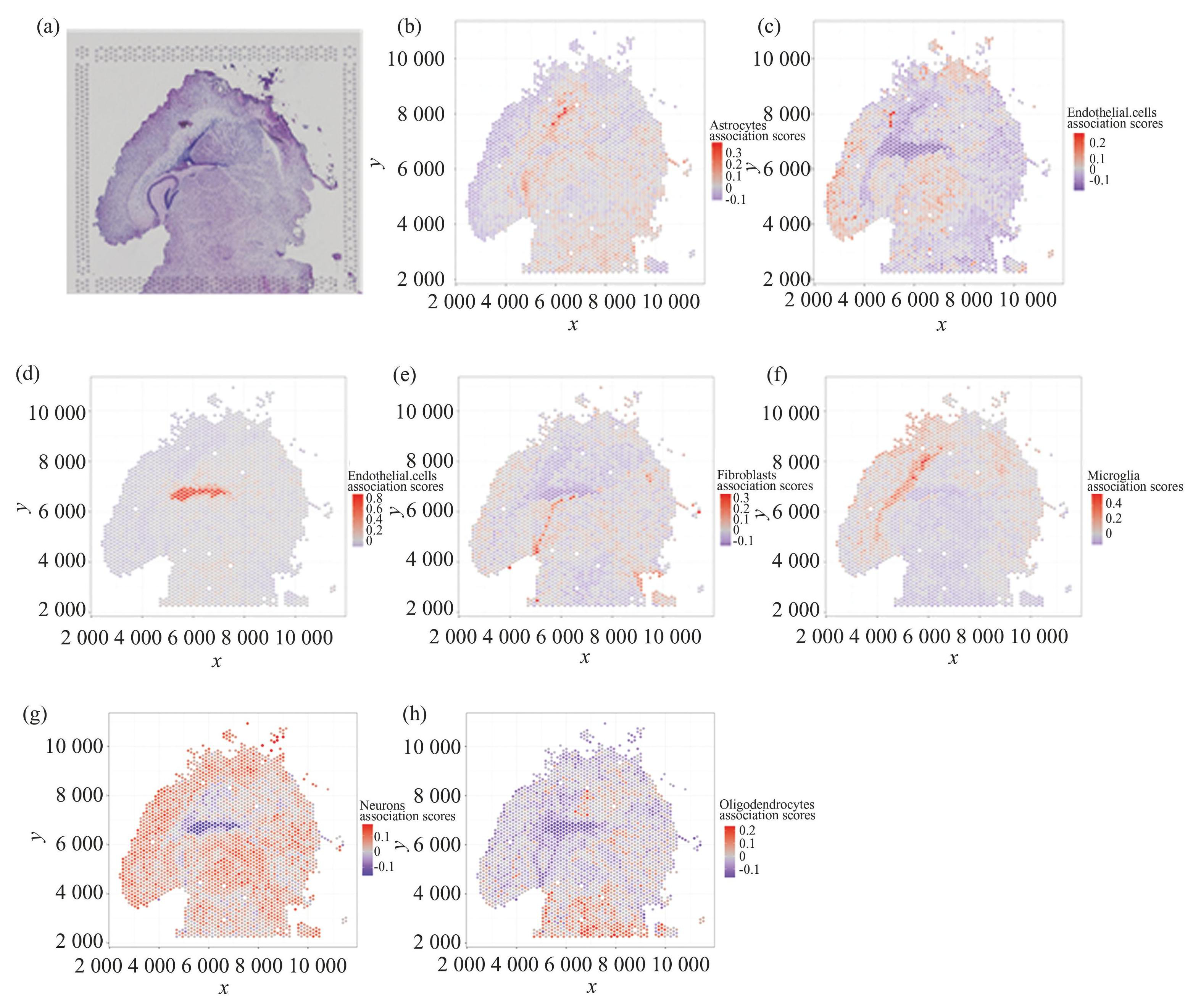
图4出生后7 d小鼠大脑海马体ST数据解卷积关联得分图
Fig.4Deconvolution correlation score map of mouse brain hippocampus ST data7 days after birth
注:图中x,y均代表Spot在组织切片中的空间位置坐标;(a)组织切片图;(b)星形胶质细胞关联得分;(c)内皮细胞关联得分;(d)上皮细胞关联得分;(e)成纤维细胞关联得分;(f)小胶质细胞关联得分;(g)神经元细胞关联得分;(h)少突胶质细胞关联得分.(扫本文首页二维码见彩图).
由图4可以明显地观察到细胞类型预测情况与组织切片的染色情况在一定程度上是一致的。例如,图4(b)表示的星形胶质细胞高关联得分部位对应组织切片图的深紫色部位,图4(d)表示的上皮细胞高关联得分部位对应组织切片图偏左上位置水平分布的浅紫色部位,图4(e)表示的成纤维细胞高关联得分部位对应组织切片图偏左下角凹陷处的浅色区域。已有研究表明组织切片着色情况与细胞的种类有关,所以本文的空间转录组数据Spots解卷积结果与真实染色情况吻合,验证了STDN算法的准确性。
另外,STDN模型Sigmoid激活函数的输出转化为行和为1的概率矩阵(Spots×细胞类型),矩阵值被解释为将Spots分配给对应细胞类型的概率,这是一个Spot中细胞属于该类型的比例近似值。将每个Spot的细胞类型组成及其比例绘制成饼图投影在对应的组织空间位置上得到图5所示饼状散点图。由图5可以看出某些区域的某种细胞类型占比较大,具有明显的区域特征,如在相应位置,绿色对应的上皮细胞占比较大,这与图4(a)的组织学染色图像高度一致。STDN预测的结果说明了小鼠海马体组织的异质性,更直观说明了细胞类型分布与空间位置有关的观点[4]。
图5出生后7 d小鼠大脑海马体的ST数据解卷积饼状散点图
Fig.5ST data deconvolution pie scatter plot of mouse brain hippocampus 7 days after birth
注:图中x,y均代表Spot在组织切片中的空间位置坐标.(扫本文首页二维码见彩图).
2.3 人类胰腺导管腺癌数据异质性分析
2.3.1 人类胰腺导管腺癌数据集
本文分析的人类胰腺导管腺癌数据来自未经治疗的胰腺导管腺癌患者胰腺导管切片,整合scRNA-seq数据和ST数据进行空间转录组解卷积旨在识别癌变和非癌变区域。其中scRNA-seq数据和配对的ST数据来自文献[20]和GEO数据库,GEO数据序列号为GSM3036909,GSM3036911。
2.3.2 实验结果分析
人类胰腺导管腺癌数据实证分析以人类胰腺导管腺癌空间转录组测序的组织切片图像和组织学家基于H&E染色(Hematoxylin and eosin(H&E)stains,苏木精和伊红染色)标记的多个组织区域(癌变区、胰腺区、导管区和间质区)[20]为基本事实进行对照,见图6(a)。将交集处理后的基因表达矩阵及scRNA-seq细胞类型注释作为输入训练STDN模型,用训练好的模型对人类胰腺导管腺癌ST数据进行解卷积,将Sigmoid激活函数的输出转化为行和为1的概率矩阵,表示ST数据每个Spot的细胞类型及其比例,绘制饼状散点图,如图6(b)所示。
由图6(b)可以观察到切片右上角区域的Cancer_clone_A及Cancer_clone_B细胞类型占比较大,具有明显的区域特征,这与图6(a)的癌变区标注位置保持一致;左侧边缘区域导管组织细胞占比较大,这与图6(a)的导管区标注位置也是一致的。数据分析结果表明人类胰腺导管腺癌ST数据解卷积结果与人工标注情况吻合,进一步验证了STDN模型解卷积结果的准确性。图6(b)显示了不同类型在癌变区和正常组织区域呈现出明显不同的空间分布模式,说明了胰腺导管腺癌具有高度的异质性。
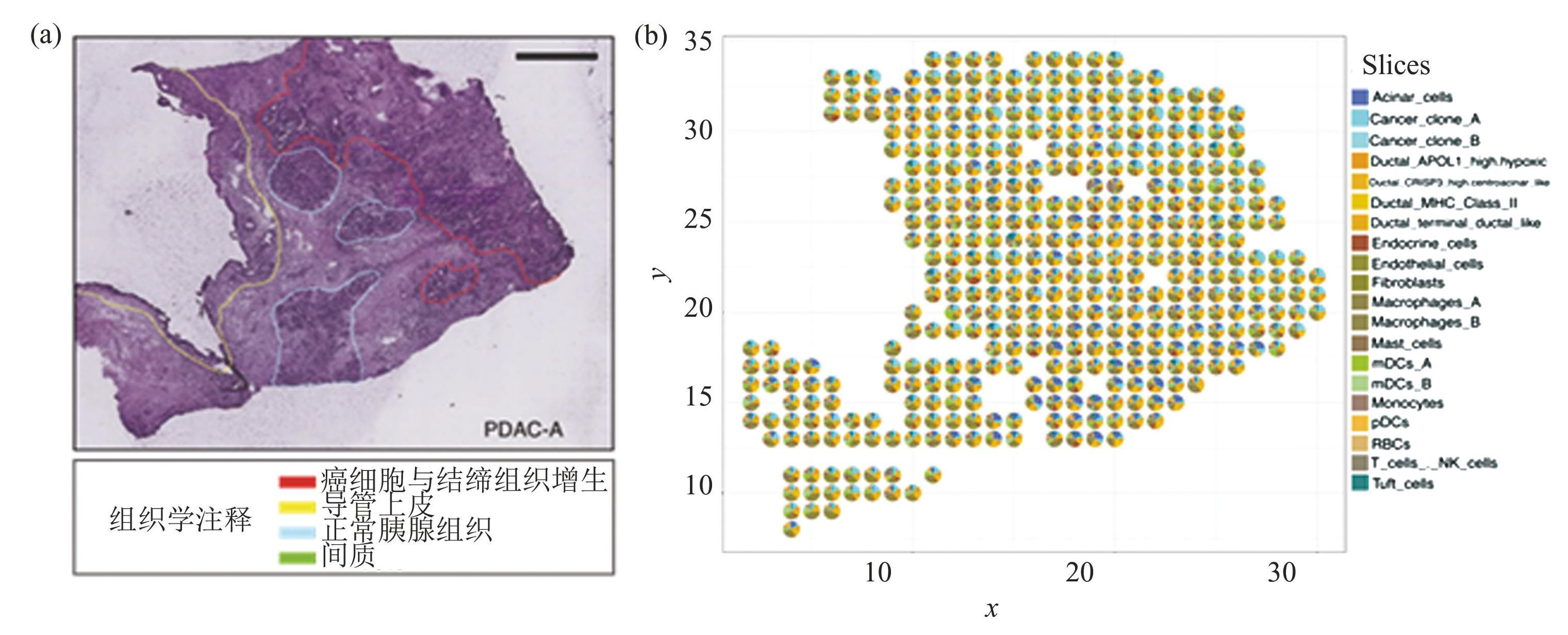
图6人类胰腺导管腺癌组织切片及ST数据解卷积结果
Fig.6Human pancreatic ductal adenocarcinoma tissue slice and ST data deconvolution result
注:图中x,y均代表Spot在组织切片中的空间位置坐标;(a)组织切片染色及区域标注[9];(b)解卷积饼状散点图.(扫本文首页二维码见彩图).
正确地识别癌变区至关重要,同时为了更直观地比较解卷积结果与实际切片染色情况,本文将其关联得分投影到ST数据组织切片的空间位置,得到如图7所示的20种细胞类型中与癌变及区域识别相关的2种细胞类型的预测结果。与人类胰腺导管腺癌ST数据解卷积饼状散点图6(b)类似,图7再次验证了算法解卷积结果的准确性。显然,STDN模型为组织切片右上角区域赋予了更高的癌变细胞(Cancer_clone_A,Cancer_clone_B)关联得分,意味着这片区域更可能是癌变区域,这与图6(a)人类胰腺导管腺癌切片H&E染色图像及癌变区域标注高度一致。
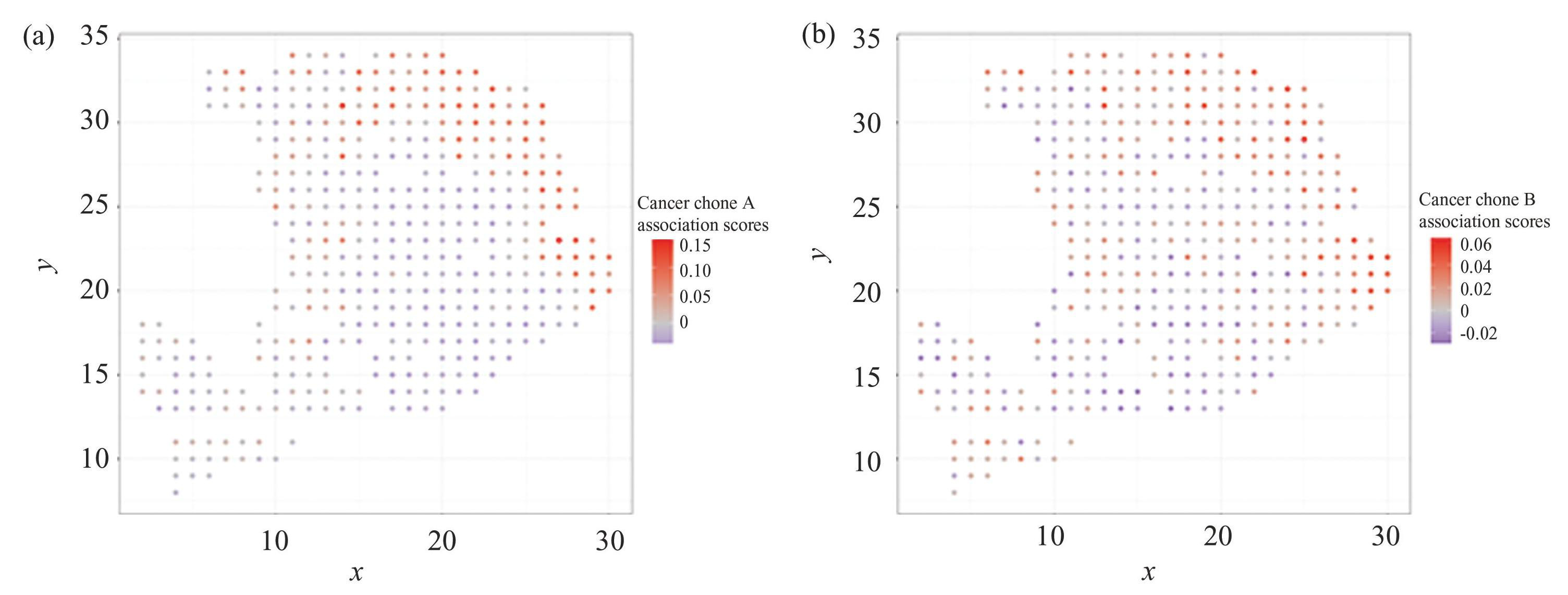
图7人类胰腺导管腺癌ST数据部分细胞类型关联得分图
Fig.7Correlation score map of partial cell types in human pancreatic ductal adenocarcinoma ST data
注:图中x,y均代表Spot在组织切片中的空间位置坐标;(a)癌症克隆细胞A关联得分;(b)癌症克隆细胞B关联得分.(扫本文首页二维码见彩图).
3 结论
本文充分利用了单细胞转录组数据和空间转录组数据特征的优势,将单细胞转录组数据作为参考信息,引入了卷积神经网络和领域自适应理论,提出了针对空间转录组数据的细胞类型解卷积算法。然后,利用scRNA-seq数据及模拟的配套ST数据将本算法与其他经典的解卷积算法比较,并利用小鼠大脑海马体数据和人类胰腺导管癌数据进行了实证分析,结果表明,本算法在模拟数据和真实数据上均具有较好的性能。
本文基于DenseNet网络和领域自适应理论构建了STDN解卷积算法。鉴于不对称转换更灵活,通常能在域自适应任务中产生更好的性能;同时考虑基因测序数据稀疏、高维的特点,STDN使用非对称转换CORAL度量scRNA-seq数据和ST数据的分布差异,将带有细胞类型标签的scRNA-seq数据作为参考,同时最小化细胞类型分类错误、scRNA-seq数据和ST数据的潜在差异,推测ST数据中每个捕获位点的细胞类型组成及比例,达到ST数据解卷积的目的。
然而,STDN具有局限性,也是目前基于scRNA-seq参考数据集的解卷积算法存在的共性问题:ST数据的解卷积过于依赖scRNA-seq数据。首先,ST数据解卷积无法得出scRNA-seq数据中没有的细胞类型。其次,scRNA-seq数据中不准确的细胞类型注释会极大程度影响ST解卷积的效果。这是我们未来研究需要解决的问题。另外,理论上,STDN也可将ST数据的空间位置信息迁移到scRNA-seq数据的单个细胞上,从而对单细胞数据进行空间重建。无论是ST数据解卷积,还是scRNA-seq数据空间重建,均是二者信息互相迁移的过程。因此,我们将改进算法,同时达到ST数据解卷积和scRNA-seq数据空间重建的目的。
致谢: 陈子睿、杨博然和何田韵对这项工作的贡献相等。


