摘要
癌症精准治疗作为当今抗癌研究的前沿,通过深入挖掘癌症的临床病理和分子特征,为患者提供个体化治疗策略。相对于传统治疗方法,癌症精准治疗极大地提高了治疗的成功率。然而,在临床实践中,精准治疗尚处于早期阶段,如何高效地为患者制定个性化治疗方案仍然是一个挑战。合成致死作为一种基因遗传互作,是指当两个基因同时失活时细胞会死亡,而这两个基因单独失活时对细胞存活无显著影响。基于合成致死互作原理,研究人员能够高效整合不同组学数据,进行药效预测、药物重定位以及联合用药预测分析,从而为患者提供有效的治疗策略,为癌症精准治疗注入了新的活力。鉴于合成致死在精准治疗中的巨大潜力,本文全面总结了合成致死相关的数据资源和识别方法,探讨了合成致死在癌症精准治疗中的实际应用以及所面临的问题和挑战,并展望了未来的研究方向。
Abstract
At the forefront of contemporary anti-cancer research, precision therapy provides personalized treatment strategies by thoroughly mining the clinical pathology and molecular characteristics of cancer. Compared to conventional therapeutics, precision therapy significantly improves the success rate of cancer treatment. However, in clinical practice, precision therapy is still in its infancy, and efficiently designing personalized treatment plans for patients remains a challenge. Synthetic lethality, as a kind of genetic interaction, refers to the phenomenon that cell death occurs only when two specific genes are simultaneously inactivated, while individual inactivation of these genes has no significant impact on cell survival. Based on the principle of synthetic lethality, researchers can effectively integrate various omics data to predict drug efficacy, and to study drug repositioning and combination, all of which can provide effective treatment strategies for patients. Given the immense potential of synthetic lethality in precision therapy, in this review we comprehensively summarized data resources and methodologies pertaining to synthetic lethality, and deliberated upon both the applicability and the challenges of synthetic lethality in cancer precision therapy. We also discussed the insightful perspectives on this topic.
Keywords
癌症的高度遗传异质性使得传统癌症治疗方法难以达到预期效果。精准治疗作为一种有望克服癌症异质性的方法,已经成为癌症治疗中不可忽视的策略。癌症精准治疗以个体化为导向,根据癌症的分子特征制定治疗方案[1]。在癌症精准治疗研究的推动下,癌症治疗范式已由传统的以癌症类型为核心的模式,转变为基于基因的个体化治疗模式。在新的治疗模式中,靶向治疗占据重要地位,它通过抑制癌症驱动基因消灭癌细胞[2-5]。然而,靶向治疗仍面临诸多局限,例如:部分癌症驱动基因并不适合作为药物靶点[6-8]。合成致死原理的引入为靶向药物研究提供了全新的思路。基于合成致死理论,与驱动基因具有合成致死互作的基因可以作为替代的治疗靶点[9]。基于合成致死的靶向治疗策略不仅补充了治疗靶点,还能够精准地杀死癌细胞[10]。此外,合成致死还在药物重定位、药效预测以及联合用药等方面展现出重要的应用价值[11]。合成致死原理为癌症精准治疗提供了新的思路和方法,有望提高药效预测能力,促进癌症的个体化治疗。综上,合成致死为癌症精准治疗带来了崭新的前景。
1 合成致死原理
合成致死基因互作最早由Calvin Bridge在酵母和果蝇中发现,指的是一种特殊的基因互作关系:当两个基因同时突变时,会导致细胞死亡,而基因单独突变时不影响细胞的生存能力[12-13]。类似地,癌细胞也存在合成致死现象。如图1所示,单个基因突变对细胞活性无显著影响,但当两个基因同时突变时,细胞则会死亡。这种现象通常由功能互补的基因同时失活引起[14]。
随着研究的深入,引起合成致死的基因组事件不再局限于基因突变,还包括基因低表达、基因拷贝数变异和基因甲基化异常等情况[15]。例如:CDK2在BRAF突变的黑色素瘤中过度激活,而抑制CDK12的表达能够与BRAF突变共同作用,导致黑色素瘤细胞死亡[16];在许多癌症类型中,CCNE1基因的扩增与PKMYT1基因的抑制会产生合成致死效应[17];在胶质母细胞瘤中,VRK2的甲基化与VRK1的抑制会导致癌症细胞活性降低[18]。在癌症治疗领域,合成致死是指一个基因失活和另一个基因被药物抑制导致的细胞死亡现象[19]。此外,合成致死具有环境依赖性,癌症异质性、肿瘤微环境以及外部刺激均会对合成致死互作产生影响[20]。例如:53BP1基因的缺失可以抑制PARP1和BRCA1之间的合成致死互作[21]。
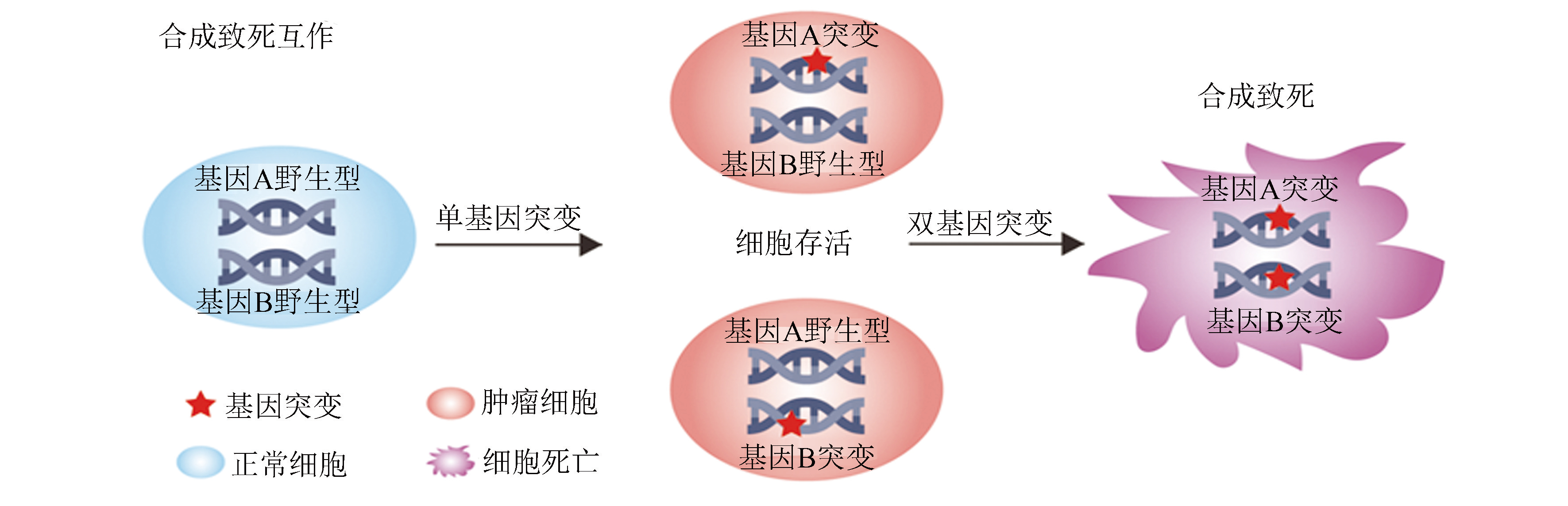
图1合成致死互作原理
Fig.1Principle of synthetic lethality
2 合成致死识别方法
合成致死应用于癌症精准治疗的关键,在于如何识别稳健的合成致死基因对。为此,科研工作者已经提出了多种合成致死识别方法。这些方法根据实验类型可分为湿实验筛选和生物信息学筛选两大类。湿实验方法旨在找到可靠的合成致死互作基因对,为进一步研究打下了坚实基础。早期,合成致死互作的识别主要依赖酵母杂交技术。随着基因编辑技术的进步,科研工作者得以在人类细胞系中进行全基因组的合成致死筛选。高通量测序技术产生了丰富的癌症多组学数据,这为合成致死的识别提供了坚实的数据基础。基于这些数据,科研工作者开发了多种生物信息学方法,用于鉴定合成致死,这些方法能够迅速地进行全基因组的初筛,进而减小湿实验的范围,节约时间和成本。
2.1 基于湿实验的合成致死识别方法
湿实验通常在模式生物或细胞系中,借助酵母筛选技术[22]、基因干扰技术[23]和药物干扰技术[24]进行合成致死筛选。目前,湿实验已经识别出大量的合成致死互作基因对,这些基因对一方面补充了现有的药物靶点;另一方面揭示了合成致死在不同组学数据中的规律,为合成致死识别的生物信息学方法提供了重要线索。虽然湿实验在合成致死识别中具有重要作用,但是仍然需进一步改进实验技术,以降低实验误差,提高可靠性和准确性。
2.1.1 酵母筛选技术
合成遗传阵列(Synthetic genetic array,SGA)推动了大规模合成致死筛选的研究,该方法通过将携带已知基因突变的酵母细胞株与携带其它基因突变的细胞株进行芯片杂交,获得携带双突变的细胞株,然后根据其存活状态来确定两个基因的互作关系[25-26]。在识别到酵母中的合成致死互作后,可以利用基因在不同物种之间的同源性,将这些合成致死互作映射到人类基因组中[27]。然而,该技术存在一些局限性:①由于酵母与人类在基因组和细胞结构方面存在差异,因此在酵母中发现的合成致死并不能直接适用于人类细胞;②酵母作为单细胞生物,缺乏人类器官和组织等复杂结构,因此无法反映人体组织对合成致死的调控作用。
2.1.2 基因干扰技术
RNAi技术和CRISPR-Cas9技术是开展人类全基因组合成致死筛选的关键工具。RNAi技术通过小干扰RNA(Small interfering RNA,siRNA)或微小RNA(microRNA,miRNA)沉默细胞中特定的基因,进行合成致死筛选[28]。与RNAi技术不同,CRISPR-Cas9系统以DNA为靶标,其通过单向导RNA(Single guide RNA,sgRNA)精确敲除基因组中的任意基因,以进行全基因组的合成致死筛选[29]。然而,基因功能筛选实验通常在离体细胞系中进行,因而存在以下几点不足:①细胞系脱离肿瘤微环境,而癌细胞与肿瘤微环境之间存在紧密互作。因此,细胞系实验难以准确地反映癌症组织内的基因互作;②癌症细胞系来源的多样性不足,例如:在10%的急性髓性白血病(Acute myeloid leukemia,AML)患者中存在IDH1突变,然而癌症细胞系百科全书(Cancer cell line encyclopedia,CCLE)所收录的1 000个细胞系中没有一个携带IDH1突变的AML细胞系[30];③RNAi和CRISPR-Cas9引起的基因组不稳定和脱靶效应,可能导致假阳性结果。此外,基因不完全抑制可能导致假阴性的结果[31-32]。
2.1.3 药物筛选技术
药物筛选技术通常利用一种外源性的化合物处理细胞,然后观察这些化合物对细胞、生物体或生物过程的影响,以识别潜在的治疗靶点或药物[33]。药物筛选技术可用于识别与药物具有合成致死互作的基因,其通过对携带特定突变的细胞系或特定基因被敲除的细胞系进行药物处理,以揭示基因和药物之间的合成致死互作[34]。该方法的缺陷在于药物对靶点的抑制程度以及药物的副作用,会干扰合成致死互作的筛选。
2.2 基于生物信息学的合成致死识别方法
考虑到人类基因组有近2亿个基因对,运用湿实验在全基因组进行合成致死筛选,显然是不切实际的。然而,随着高通量测序数据的不断积累,研究人员可以利用生物信息学方法进行合成致死筛选,进而缩小湿实验的筛选范围,从而减少实验所需时间和成本。如图2所示,合成致死识别的生物信息学方法主要包括两个关键步骤。首先,通过统计学模型从癌症多组学数据中挖掘与合成致死相关的特征;其次,借助特征组合或人工智能算法构建合成致死预测模型。根据模型构建方法的不同,合成致死识别的生物信息学方法可划分为四类:①基于特征组合的方法;②基于机器学习的方法;③基于深度学习的方法;④基于网络的方法。
2.2.1 基于特征组合的方法
基于特征组合的方法通常依据现有的生物学假设,采用统计学模型从癌症多组学数据中挖掘与合成致死相关的特征。随后,将获得的特征进行组合,认为同时满足多项特征的基因对可能存在合成致死互作。DAISY便是这类方法的代表,其基于以下三个假设:①基因组生存适应性(Genomic survival of the fittest,SoF):合成致死基因对在癌细胞中同时失活,导致细胞死亡;②基因重要性:当合成致死基因对中一个基因失活时,另一个基因的重要性增加;③基因共表达:合成致死基因对倾向于参与密切相关的生物学过程[35]。虽然这类方法加速了合成致死的识别,但是特征的预测性能受数据质量和数据分布的影响。当数据质量较差或者分布不均衡时,这类方法可能无法提供良好的预测性能。例如:当癌症基因组的突变负荷较低时,基于基因突变数据获得的特征可能无法准确反映基因合成致死互作[36-37]。
2.2.2 基于机器学习的方法
机器学习在合成致死预测中具有重要作用,其首先通过整合癌症多组学数据,提取合成致死相关的特征;接着,结合已知的合成致死标签,训练机器学习模型,从而预测未知的合成致死互作。例如:Das等[38]开发了一款名为DiscoverSL的R包,该R包结合癌症多组学数据和癌型特异的合成致死标签数据,构建了随机森林分类器,用以预测癌症特异的合成致死互作。然而,虽然机器学习能够有效整合癌症多组学数据,为合成致死预测提供更全面的信息,但由于历史和学术的原因,科研工作者往往更倾向于对热点基因的研究。这种倾向性导致实验验证的合成致死基因对存在严重的数据偏性,即某些热点基因在现有的SL标签中数量占绝对优势,而其他基因则缺乏研究。这种基因表示的不均衡导致模型性能估计过高,因为模型更多地依赖于这些热点基因,从而影响了模型的泛化能力[39]。为克服这一问题,构建更加全面且平衡的合成致死标签数据,能够有助于降低模型的过拟合。此外,运用适当的特征选择和模型调优方法,能够更准确地评估模型在未知数据上的性能,从而进一步提高模型的可靠性与泛化能力。
图2基于生物信息学的合成致死筛选基本流程
Fig.2Basic process of synthetic lethality screening method based on bioinformatics
2.2.3 基于深度学习的方法
因为癌症多组学数据在多个尺度上存在相关性,所以人工难以从中提取出优秀的特征[40]。深度学习作为机器学习领域的重要分支,在解决这一难题方面具有显著优势。其核心思想在于构建多层神经网络,以模拟人脑神经元之间的信息传递和处理方式。相对于传统的机器学习方法,深度学习的独特之处在于层次化的特征学习,使得模型能够自动从原始数据中学习到更高级、更抽象的特征表示。例如:Long等[41]运用基于上下文的图注意力网络,从若干个基因构成的特征图中学习合成致死预测所需的特征。研究结果显示,该方法在三个数据集上的预测性能均优于目前已有的14种方法。
2.2.4 基于网络的方法
基于网络的合成致死预测方法主要依赖于蛋白质互作网络、基因调控网络和代谢网络,然后通过分析生物网络中基因的拓扑特征以及基因对被敲除后对网络的影响来预测合成致死基因对。例如:Barrena等[42]将基因最小割集理论应用于代谢网络和基因调控网络,以揭示潜在的合成致死互作。与统计学方法相比,基于网络的方法能够更系统地了解基因的功能,并提高对合成致死机制的理解。然而,该类方法存在一些局限性:①基于网络的方法只考虑了基因之间的互作网络,而未充分考虑基因与患者等其他实体之间的关联;②目前,基于网络的方法无法充分利用其他有价值的特征信息,如基因序列和功能特征。
3 合成致死相关的数据资源
高通量测序技术现已在癌症研究中得到广泛应用,其产生的多组学数据不仅极大地丰富了癌症研究的数据资源,而且还为合成致死研究奠定了坚实的基础。这些多组学数据蕴含的信息多样,包括基因的共表达模式、基因突变的互斥现象以及患者的生存信息等。这些信息为科研工作者更准确地识别合成致死互作关系提供了有力的线索。此外,随着高通量基因编辑技术的进步,大规模的基因敲除实验数据源源不断地生成。这种数据为科研工作者供了更深层次的功能基因组学分析的可能,使他们能够进一步鉴定出对细胞生长至关重要的基因。目前,已经有大量的合成致死基因对被成功识别并收录在各个专业的数据库中。这些基因对为生物信息学方法的开发提供了宝贵的标签数据,不仅有助于验证生物信息学模型的准确性,更助力科研工作者更深入地理解合成致死互作的生物学功能和机制。同时,随着测序数据的不断积累和挖掘,合成致死在线分析平台也逐步开发完善,为科研工作者提供了方便快捷的在线分析工具,进一步推动了合成致死研究的发展和深入。接下来,本文将分别对合成致死相关的特征数据、合成致死标签数据以及合成致死在线分析平台进行总结。
3.1 合成致死相关的特征数据
合成致死相关的特征资源为合成致死的识别提供了丰富的信息,同时也推动了生物信息学在合成致死识别领域的应用。本文对与合成致死相关的特征资源进行了综合整理,并对其中有助于合成致死互作识别的信息进行了简要介绍(表1)。
虽然高通量测序技术为合成致死识别提供了丰富的特征信息,但在数据分析过程中仍然面临一些挑战。例如:高通量的基因组数据和基因敲除数据可能存在噪声,这将导致假阳性和假阴性的实验结果[43]。因此,研究人员需要采取适当的方法过滤和校正数据中的噪声,以提升结果的准确性和可靠性。此外,通过整合多个数据源和使用不同的实验进行验证,可以降低假阳性和假阴性的概率。随着实验技术的不断发展以及算法的改进,合成致死关系的识别将更加准确和可靠。
表1合成致死相关的特征信息数据库
Table1Synthetic lethal associated feature information databases

3.2 合成致死标签数据
随着高通量基因敲除技术的持续发展,越来越多的合成致死基因对被鉴定,并被收录在不同的数据库,为科研工作者提供了宝贵的资源。这些数据库收录的高可信的合成致死基因对,为机器学习模型提供了训练标准集。鉴于合成致死标签数据的重要性,表2对现有的合成致死标签数据库进行了汇总,并简要描述了其数据来源以及收录的合成致死类型。
表2合成致死标签数据库
Table2Synthetic lethal label databases

3.3 合成致死在线分析平台
尽管高通量测序技术和高通量基因编辑技术为合成致死互作识别提供了重要的数据资源,但这些数据的分析要求科研工作者具备一定的计算机编程和数据处理技能,同时也需要拥有充足的计算资源。因此,为协助科研工作者从这些数据中挖掘有价值的信息,构建简单易用的在线数据分析平台具有重要意义。表3对现有的合成致死在线分析平台进行了汇总。这些平台为合成致死研究提供了便捷工具,使得科研工作者能够更加高效地利用高通量组学数据进行合成致死互作的识别和分析,这进一步推动了合成致死互作在癌症精准治疗中的应用。
表3合成致死在线分析平台
Table3Synthetic lethality online analysis platform
4 合成致死在癌症精准治疗中的应用
合成致死在癌症精准治疗研究领域展现出重要的应用前景。首先,基于合成致死的靶向治疗药物研发为癌症精准治疗提供了更多有效的治疗药物。这些药物专注于特定的癌症标志物或致病机制,使得癌症治疗变得更加个体化和精准化;其次,借助合成致死原理,研究人员能够系统性地整合癌症多组学数据,实现药物重定位、联合用药预测以及药效评估等任务,为癌症精准治疗提供切实可行的治疗方案。综上所述,合成致死将有力地推动癌症精准治疗的发展与实践。
4.1 合成致死在靶向治疗中的应用
靶向治疗药物的设计旨在通过干扰癌细胞中的特定分子,从而精准地抑制癌细胞生长和增殖,并尽可能地减少对正常细胞的损害[60-61]。靶向治疗药物能为患者提供个性化的治疗方案,推动了癌症精准治疗的研究进展。目前,大部分靶向药物的靶点为癌症中异常的癌基因。然而,有些癌基因由于其分子结构的特殊性,难以被小分子药物干预。例如:两个常见的癌基因MYC和KRAS,由于缺乏合适的药物结合口袋,无法作为小分子药物的靶点[62-63]。此外,抑癌基因由于在癌症中处于失活状态,也难以作为药物的靶点。例如:在多种癌症中具有驱动作用的TP53基因,目前尚无靶向TP53的靶向治疗药物获得美国食品药品监督管理局(Food and drug administration,FDA)批准[64]。研究表明,与癌基因和抑癌基因存在合成致死互作的非癌基因,可能成为优秀的药物靶点,通过干预这些非癌基因,可以有效地消灭癌细胞[65]。目前,PARP抑制剂是首款基于合成致死策略开发的靶向治疗药物,且已获得FDA批准用于治疗存在基因同源重组修复缺陷的乳腺癌和卵巢癌患者。BRCA1/2和PARP1/2是一对经典的合成致死互作,其共失活导致细胞基因损伤修复功能缺失,从而破坏癌症基因组稳定性,引发癌细胞死亡[66-67]。大部分乳腺癌和卵巢癌都携带BRCA1/2突变。因此,PARP抑制剂通过抑制PARP1/2,可以有效杀死携带BRCA1/2突变的癌细胞。然而,目前仅有一款基于合成致死研发的抗癌症药物进入临床应用。未来通过进一步阐明合成致死互作机制,并深入挖掘癌症中的合成致死互作,将为临床提供更多有效的治疗药物。
4.2 合成致死在药物重定位中的应用
药物重定位是一种重要的药物研发策略,其核心思想在于将已经获得批准的药物或者正在研究中的化合物,应用于不同于其原始适应症的疾病或治疗领域,以探索其新的疗效和用途[68]。例如:赛来昔布最初被用于治疗骨关节炎和成人类风湿性关节炎,而后被发现适用于治疗结肠癌和乳腺癌[69]。与传统的新药研发相比,药物重定位旨在充分利用现有药物的安全性和药代动力学特性,从而加速新药的研发过程。
虽然药物重定位在促进癌症治疗研究方面发挥着重要作用,但如何高效整合不同分子组学数据以实现精准的药物重定位仍然是一个挑战。基于合成致死原理,科研工作者可以系统地整合多组学数据,开发药物重定位算法。合成致死驱动的药物重定位一般分为两个关键步骤。首先,通过整合分析不同组学数据,识别与癌症驱动基因存在合成致死互作的基因;然后,从已上市的药物中筛选出能够靶向这些基因的药物,实现药物重定位。例如:Feng等[70]的研究发现GNAQ与PTK2在葡萄膜黑色素瘤(Uveal melanoma,UM)中具有合成致死互作关系,并进一步证明了PTK2抑制剂能够显著抑制携带GNAQ突变的UM细胞生长。因此,该研究指出PTK2抑制剂可用于治疗携带GNAQ激活突变的UM。另外,Zhang等[58]系统地整合药物、合成致死和癌症三个层面的信息,构建了一种药物重定位打分系统。其研究结果表明,该系统能够准确地对155种药物进行重定位,并且大部分重定位的药物都得到了文献支持。综上所述,这些研究展示了合成致死在寻找潜在治疗靶点和药物重定位方面中的巨大潜力。
4.3 合成致死在联合用药中的应用
癌症作为一种多基因疾病,其发病机制涉及多个基因功能的紊乱。因此,仅仅针对单一靶点进行治疗,往往难以持久地控制癌症。例如:经过PARP抑制剂治疗后,有40%的卵巢癌患者出现耐药现象,基因同源重组修复功能的恢复被认为是其耐药的主要原因[71-72]。联合用药旨在通过同时靶向多个关键生物过程,以增强药物的疗效,从而有效应对单一药物治疗导致的耐药问题。合成致死可以帮助科研工作者迅速发现有效的联合用药方案。如图3所示,基于合成致死的联合用药预测算法主要依据两个原理:①若两种药物分别靶向癌症中的一对合成致死基因,那么它们可能产生联合用药效应。例如:Lee等[73]首先在癌症中识别一对最可信的合成致死基因对,随后选择两种分别靶向这对基因的药物,作为联合用药方案。此外,Verma等[74]通过合成致死筛选,发现靶向BET和CXCR2的药物组合,可用于治疗基质性的三阴性乳腺癌;②药物能抑制更多的合成致死互作基因对,其联合用药潜能更高。例如:Zhang等[58]通过整合多层次的信息构建合成致死知识图谱,然后运用整数规划算法在癌症中识别覆盖合成致死基因对数目最多的药物组合作为潜在的联合用药方案。综上所述,这些研究充分证实了合成致死可以助力科研工作者发现更有效的联合用药方案。
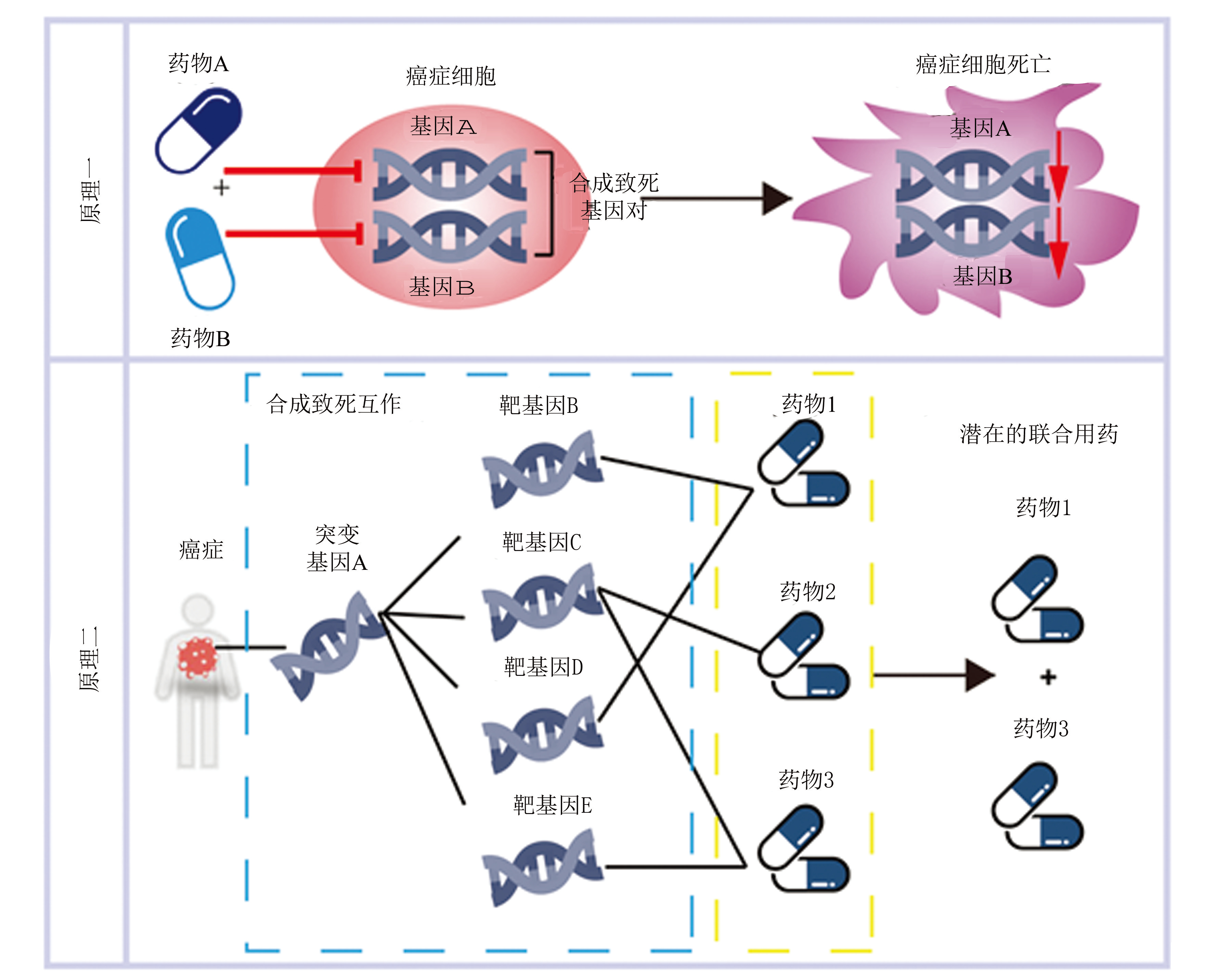
图3联合用药原理示意图
Fig.3Schematic diagram of combination therapy principle
4.4 合成致死在药物药效评估中的应用
目前,癌症精准治疗策略主要侧重于根据癌症基因特征预测药物反应[75]。例如:Shi等[76]的研究发现,奥西替尼在携带EGFR突变的局部晚期或转移性非小细胞肺癌中具有很好的疗效。然而,对于没有可靠药效标志物的患者,这种模式无法准确地预测药物反应。美国国家癌症研究所的研究表明,仅有20%的患者可以根据基因突变选用相应治疗药物[77-78]。近年来,研究人员发现通过整合癌症多组学数据,能够更好地预测药物反应。例如:WINTHER作为一项前瞻性的临床试验,揭示了通过整合基因组或转录组数据可以挖掘更为有效的药效标志物[79]。然而,这些研究局限于特定的临床场景或单一的治疗药物。基于合成致死原理的药效评估模型有望弥补这些不足。
基于合成致死原理的药效评估模型能够系统地整合癌症多组学数据,为患者从多种治疗药物中选择有效的药物。合成致死药效预测原理认为,若药物的靶点在癌症中涵盖越多的合成致死基因对,其治疗效果越显著(图4)。目前,已有大量研究证明合成致死在不同数据集均能准确评估药物反应,并为患者推荐有效的治疗药物。以Lee等[73]的研究为例,他们的工作在细胞系数据、人源异种移植模型(Patient-derived xenograft model,PDX)以及癌症组织数据中发现,药物靶点的合成致死伙伴基因在癌症中的失活比例能够有效地预测药物的疗效。
图4合成致死药效评估原理示意图
Fig.4Illustration of the principle of synthetic lethality drug efficacy prediction
鉴于合成致死在药效评估研究领域展现出的卓越潜力,Lee等[80]提出了SELECT框架。该框架的主要目标是从癌症转录组数据中挖掘合成致死基因对,并以此评估药物的药效,为患者推荐最有效的治疗药物。SELECT框架对35个靶向治疗数据集进行研究,结果表明其在80%的数据集中均能准确地预测药物反应。然而,SELECT框架依然存在一些不足:①在105种FDA批准的靶向药物中,SELECT框架无法为其中35种药物构建合成致死互作网络,导致其在这些药物中失去预测能力;②SELECT框架采用接收者操作特征曲线下面积(Area under the curve,AUC)评估模型性能,然而,AUC只是一种常见的模型评价指标,其未能全面考虑模型的临床实用性。为了弥补SELECT框架的不足,Dinstag等[81]在该框架的基础上,提出了ENLIGHT框架,以扩展模型的药物涵盖度并增强模型在临床转化中的能力。ENLIGHT算法主要从以下两个方面进行了性能的提升。首先,通过引入合成致活基因互作,该算法得以拓展其药物覆盖范围,从而更全面地考虑多种治疗选项;其次,ENLIGHT更重视预测模型的阳性预测率(Positive predictive value,PPV)。在临床癌症治疗场景中,一个卓越的预测模型需要具备出色的PPV表现。高PPV代表着模型能够在众多治疗选择中,更精确地识别出对患者真正有效的药物。
5 结论和展望
目前,合成致死机制的研究以及合成致死互作的识别已经取得了显著的进展,为合成致死在癌症治疗中的应用奠定了坚实的基础。一方面,合成致死在癌症靶向治疗领域的应用,为癌症治疗开辟了全新的途径;另一方面,合成致死在联合用药、药物重定位以及药效评估等方面的应用,推动了癌症精准治疗的发展。然而,尽管基于合成致死的抗癌研究已经取得了长足进步,但仍然面临诸多挑战:
1)合成致死的环境依赖性导致离体实验识别的合成致死互作,在大部分患者中无法复现。通过结合CRISPR技术和PDX模型,进行体内合成致死筛选有望解决这一难题。此外,解析环境对合成致死互作的影响机制,有利于解决合成致死互作无法复现的问题。
2)数据驱动的合成致死方法需要解决两个关键问题:首先,如何从高噪声和高维度的生物大数据中,准确提取合成致死相关的特征信息;其次,如何获得高可信的合成致死标签,从而提升模型的预测性能。
3)癌症具有高度异质性,因此识别亚型特异的合成致死互作,可能更有助于发现有效的治疗方案。单细胞测序技术为解析肿瘤异质性提供了有力工具,因此,整合单细胞测序数据用于合成致死互作的识别,可以有效地克服癌症异质性问题。
4)为了促进基于合成致死的抗癌研究成果的临床转化,科研工作者应当以临床需求为出发点,加强临床研究的深度和广度,并建立适用的药物筛选平台和评估体系,以确保其在临床环境中的可行性和安全性。


