结肠癌是消化系统常见的恶性肿瘤之一,发病率在全部恶性肿瘤中排名第四位,其形成发展与环境和遗传因素有关[1]。对于结肠癌患者,手术和辅助治疗后仍有5%~30%的复发率,因此结肠癌是一个重要的全球健康问题[2]。
坏死性凋亡作为肿瘤免疫治疗靶点,已受到越来越多的关注,目前相关文章还较少[3]。坏死性凋亡通过在肿瘤微环境中释放DAMP, 细胞因子和趋化因子,激活树突状细胞, CD4和CD8 T细胞,继而浸润肿瘤组织,形成炎性免疫微环境,产生抗肿瘤效应[4-5]。坏死性凋亡的肿瘤细胞也会吸引髓系抑制细胞和肿瘤相关巨噬细胞,产生肿瘤相关的免疫抑制[6]。在实验小鼠模型中,接种坏死性癌细胞可诱导有效的抗肿瘤免疫[7]。因此,以坏死性凋亡为靶点诱导抗肿瘤免疫是一种可行的策略,尤其是当细胞凋亡被阻断时[8]。
非编码RNA (ncRNA) 是指由基因组转录而成的不编码蛋白质的RNA分子,长非编码RNA (lncRNA) 是长度超过200个核苷酸的非编码RNA[9]。哺乳动物中lncRNA占总RNA的4%~9%,从染色质重塑、转录调控及转录后加工等多方面实现对基因表达的调节,能够控制细胞的发育和分化,并对疾病的诊断、进展监测及靶向治疗产生一定的影响[10-11]。目前虽然在特定细胞类型中lncRNA的研究已经取得了较大进展,但免疫细胞中lncRNA的功能研究还远远落后[12]。获得更多与坏死性凋亡相关的lncRNA可以帮助我们清楚地理解坏死性凋亡和lncRNA在免疫治疗中的作用[13]。本研究拟通过构建结肠癌坏死性凋亡相关lncRNA的预后模型,进行肿瘤细胞免疫浸润分析,并筛选结肠癌潜在的治疗药物,为结肠癌患者的免疫治疗提供理论基础。
1 材料和方法 1.1 TCGA转录组数据下载与整理从TCGA数据库(https://portal.gdc.cancer.gov) 下载结肠癌病人转录组和临床组数据,通过提取转录组数据,得到基因在样本中的表达量矩阵,并获得每个样本中编码蛋白的mRNA和lncRNA的表达量,然后根据坏死性凋亡基因的列表文件和mRNA的矩阵文件来提取坏死性凋亡基因的表达量。
1.2 共表达预测差异表达的坏死性凋亡lncRNA根据坏死性凋亡相关基因和lncRNA的表达文件进行共表达分析(相关系数> 0.4,P < 0.001),获得网络关系文件和节点属性文件,可绘制共表达网络图。通过lncRNA在正常样本和肿瘤样品中的表达量获得显著差异表达的lncRNA(|LogFC|≥1, FDR < 0.05 and P < 0.05)。
1.3 筛选预后相关lncRNA及模型构建将差异lncRNA的表达量与结肠癌病人的生存时间,生存状态等数据合并,查找预后相关lncRNA,并构建预后模型。将样本分为训练(Train) 组和测试(Test) 组,使用训练组样本构建预后模型,使用测试组验证模型的准确性。首先对训练组数据进行单因素Cox分析,对lncRNA进行循环,将lncRNA的表达量和生存时间、生存状态进行比较,保留满足条件(P < 0.05) 的lncRNA,接着对显著表达的lncRNA进行Lasso回归分析,进一步筛选Lasso回归中显著的lncRNA,并根据其表达量进行Cox模型构建,得到模型公式(lncRNA的表达量乘以其系数之和)。根据公式计算训练组中各样本的风险得分,并根据风险得分的中位值将病人划分为高、低风险组,同样计算出测试组的风险得分,并根据训练组中的风险得分将测试组病人划分为高、低风险组,最后进行训练组、测试组及所有病人的生存曲线、ROC曲线、风险曲线和独立预后分析。
1.4 诺谟图(Nomogram)模型构建通过单因素和多因素独立预后分析,观察构建的模型是否可以独立于其它的预后性状作为独立的预后因子。单因素分析是将年龄、性别、病理阶段等分别与生存时间、生存状态进行比较,如果P < 0.05,说明该因素与病人预后相关,多因素分析是将年龄、性别、病理阶段等因素一次性输入,与生存时间和生存状态比较,考虑各因素之间的相互作用。
1.5 GSEA富集分析使用GSEA在线软件(https://www.gsea-msigdb.org/gsea/login.jsp) 筛选高低风险组的富集通路(P < 0.05, FDR < 0.25)。
1.6 免疫浸润分析免疫细胞相关性分析根据R软件(R 4.0.2) limma, scales, ggplots2, ggtext, reshape2, tidyverse和ggpubr包选取TCGA数据库中XCELL, TIMER, QUANTISEQ, MCPcounter, EPIC, CIBERSORT-ABS和CIBERSORT平台结肠癌病人数据进行分析。单样本基因富集分析(ssGSE) 应用R软件中的GSVA, limma和GSEABase包进行,高低风险组及不同分型结肠癌病人的肿瘤微环境评分采用R软件中的ggpubr包进行。免疫检查点应用ggpubr和ggplot2包进行分析。
1.7 筛选治疗结肠癌的潜在药物结肠癌病人药物敏感性用IC50来表示,其在高低风险组及不同分型间的差异使用R软件中的pRRophetic包进行分析。
2 结果 2.1 提取坏死性凋亡基因的表达量通过TCGA数据库下载并整理结肠癌病人的表达和临床数据,共得到39个正常样本和398个结肠癌肿瘤样本,进一步获取坏死性凋亡基因在正常和肿瘤样本中的表达量文件。
2.2 共表达预测坏死性凋亡基因相关的lncRNA根据坏死性凋亡相关基因和lncRNA的表达文件进行共表达分析,并绘制共表达网络图,共获得1 859个坏死性凋亡基因相关的lncRNA (图 1(a))。通过lncRNA在正常样本和结肠癌样本中的表达量绘制火山图,筛选出961个显著差异表达的lncRNA (|LogFC|≥1, FDR < 0.05且P < 0.05) (图 1(b))。
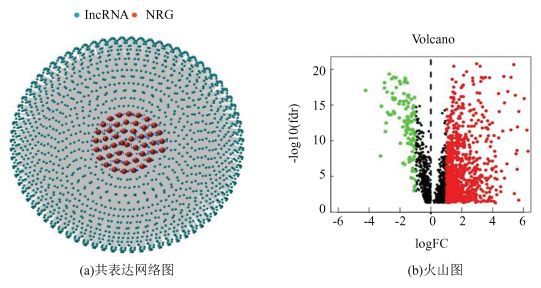
|
图 1 坏死相关lncRNA筛选 Figure 1 Screening of necrosis-related lncRNA in colon cancer patients 注:图(a)中NRG: 坏死性凋亡基因,红色代表坏死性相关基因,蓝色代表与坏死性基因具有共表达关系的lncRNA;图(b)中红色代表上调lncRNA,绿色代表下调lncRNA.(扫本文首页二维码见彩图). |
通过单因素Cox分析提取预后lncRNA (P < 0.05),绘制森林图(图 2(a))和热图(图 2(b)),根据单因素显著的lncRNA的表达量、生存时间和生存状态,绘制Lasso回归图(图 2(c)),并进行交叉验证(图 2(d))。然后通过Lasso回归显著lncRNA的表达量进行Cox模型构建,得到模型公式,从而获取每个样本的风险得分为:AC010973.2×1.6905+AC104819.3×(-2.2257)+ LINC00513×(-0.5379)+ABALON×(-2.1099)+LINC02257×1.4632+MIR3150BHG×(-2.987)+MACORIS×1.6446+AL357054.4×2.5179+LINC01857×0.7415+AP001469.3×1.569+AC093382.1×1.4422。根据模型公式中的11个lncRNA绘制桑基图,发现TRAF2,BRAF,CFLAR和BACH2等坏死性凋亡基因与之密切相关,且所有lncRNA均上调(图 2(e))。
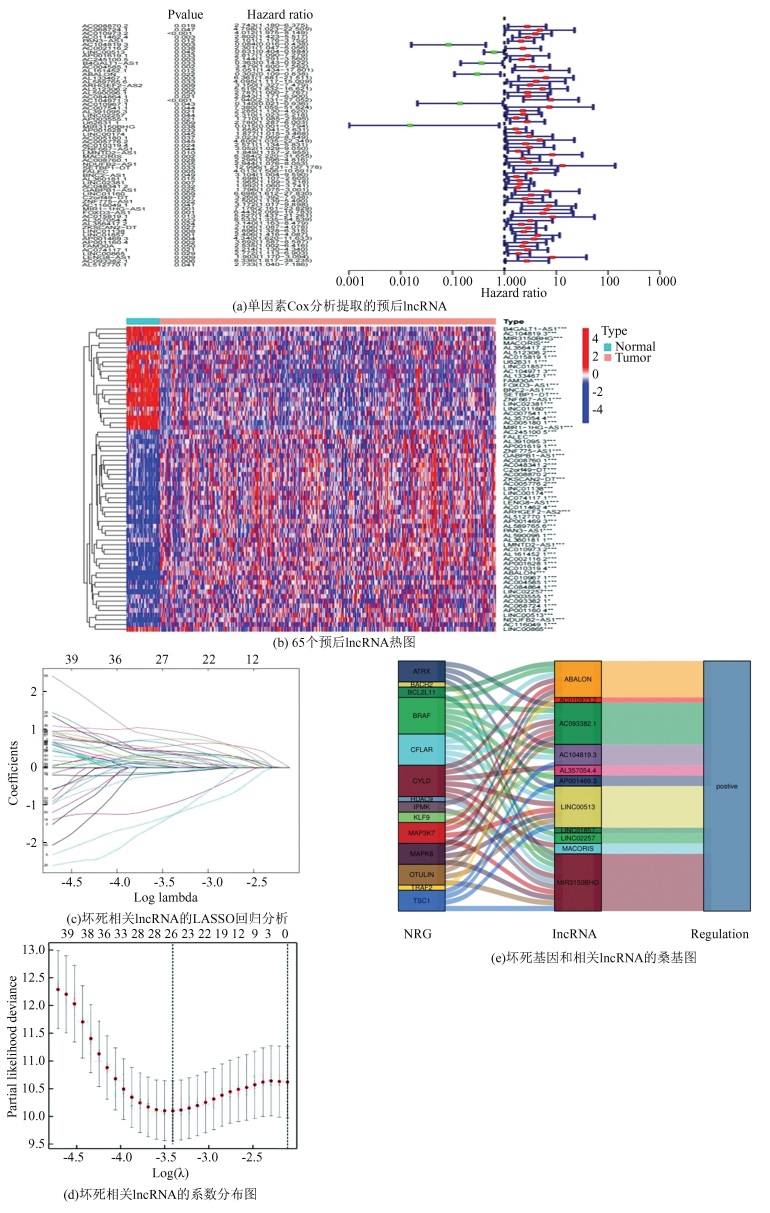
|
图 2 结肠癌中坏死性凋亡预后相关lncRNAs的提取 Figure 2 Extraction of lncRNAs associated with necroptosis prognosis in colon cancer 注:图(b)中横坐标代表样品,对样品进行分组,蓝色为正常组样品,红色为肿瘤组样品,纵坐标代表预后相关lncRNA,三个星号代表说明这个lncRNA在正常和肿瘤组之间差异的P < 0.001,两个星号代表P < 0.01,蓝色代表低表达,红色代表高表达.(扫本文首页二维码见彩图). |
将训练组、测试组和所有病人的lncRNA表达量根据高低风险(P < 0.05) 进行生存分析,发现生存曲线在高低风险组间差异显著,且高风险组病人较低风险组病人更早死亡,且预后差,因此可以证明构建的模型能够区分高低风险组病人(图 3(a))。根据风险评分公式,分别绘制高低风险组训练组、测试组和所有结肠癌病人相关lncRNA的风险曲线图,从中可以看出高风险组病人风险得分更高(图 3(b));且随着风险值的增大,AC010973.2, LINC02257, MACORIS, AL357054.4, LINC01857, AP001469.3, AC093382.1表达上升,为高风险的lncRNA,而AC104819.3, LINC00513, ABALON, MIR3150BHG表达下降,为低风险的lncRNA (图 3(c))。
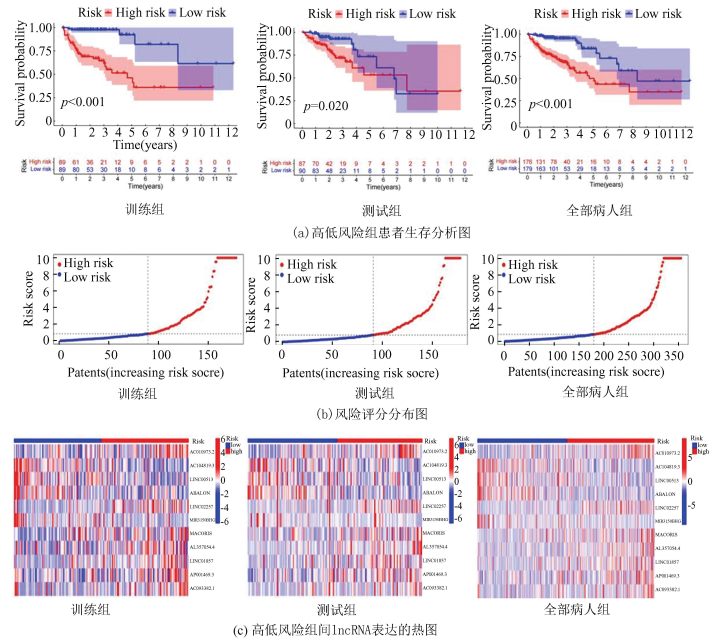
|
图 3 11个坏死相关lncRNA在训练组、测试组和所有病人组的预后图 Figure 3 Prognosis value of the 11 necroptosis-related lncRNAs model in the train, test, and entire sets |
单因素预后分析中,风险评分的风险比(Hazard radio, HR) 和95% 置信区间(Confidence interval, CI) 分别为1.013和1.009-1.017。多因素预后分析中,风险评分的风险比和95% 置信区间分别为1.01和1.006-1.014,此外,我们还发现了年龄和分期两个独立的预后因素(Age: 1.032和1.01-1.054, P=0.005; Stage: 2.611和1.947-3.5, P < 0.001) (图 4(a))。根据风险得分、年龄和分期三个独立的预后因素,绘制了预测1年、3年和5年生存期的列线图,并通过校准图证明了使用诺谟图预测病人生存期具有良好的效果(图 4(b))。
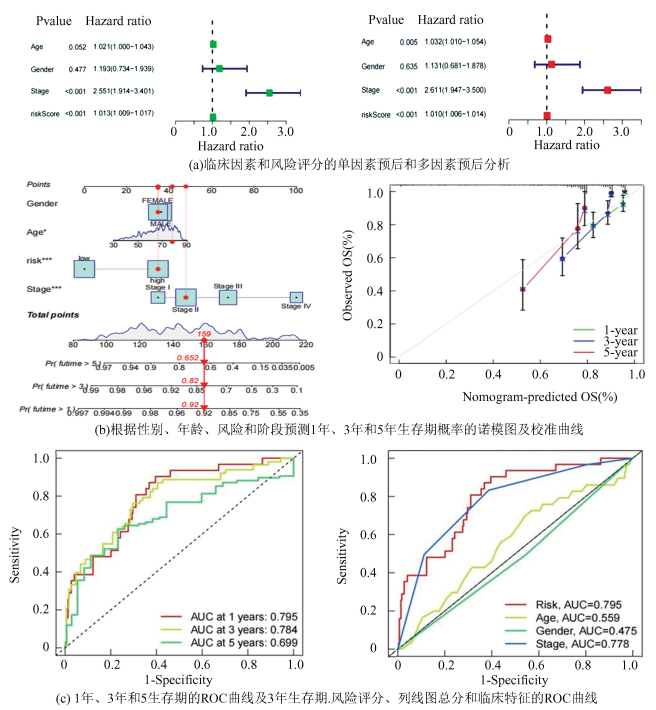
|
图 4 风险模型的诺模图和评估 Figure 4 Nomogram and assessment of the risk model |
ROC曲线被用来评估模型对预后的敏感性和特异性,曲线下面积越大,使用模型去预测病人生存期的准确性越高。从图 4(c)中可以看出,1年、3年、5年ROC曲线下面积分别为0.795, 0.784和0.699。以1年的风险模型为例,将风险评分与其他临床性状进行联合ROC曲线分析,发现风险评分(0.795), 年龄(0.559)和分期(0.778)的曲线下面积均较高,表明使用构建的诺谟图模型预测病人的生存期具有一定的依据。
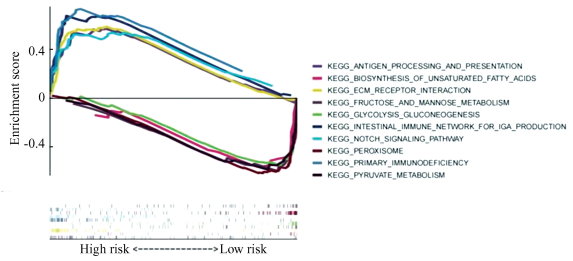
2.5 GSEA为了研究生物学功能在风险组间的差异,使用GSEA软件分别确定了5条高风险组和5条低风险组中活跃的KEGG通路(图 5)。其中高风险组中的“抗原加工与提呈”和低风险组的“用于免疫球蛋白A产生的肠道免疫网络”与“原发性免疫缺陷”三条富集通路与免疫有关(P < 0.05, FDR < 0.25, |NES|>1.5),因此,接下来在模型中进行免疫分析。
|
图 5 高低风险组中活跃的KEGG通路 Figure 5 Active KEGG pathways in high and low risk groups |
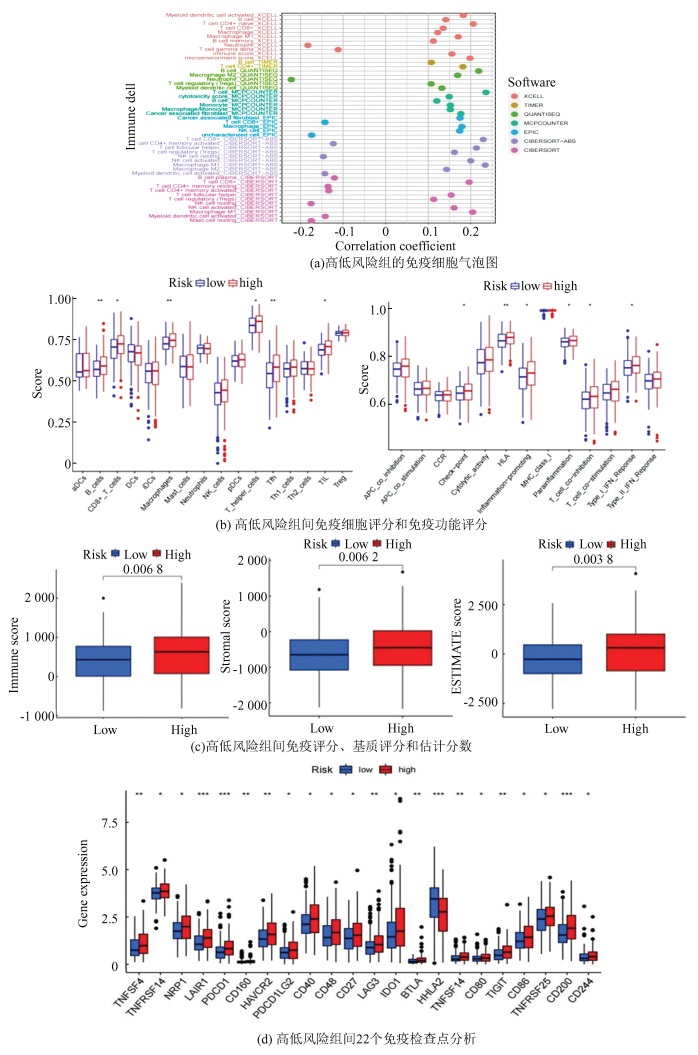
根据不同平台各种免疫细胞在结肠癌中的表现做气泡图(图 6(a)),相关系数为正表明该免疫细胞与病人风险得分之间呈正相关,为负表明呈负相关,结果发现,多种免疫细胞均与不同平台上的高危人群相关联。单样本基因集富集分析(ssGSE)可用于量化免疫浸润,计算免疫细胞在肿瘤组织中的比例,通过免疫细胞和免疫功能在高低风险组中表现的箱线图,发现B细胞、CD8+ T细胞、巨噬细胞、辅助性T细胞, 滤泡辅助性T细胞和肿瘤浸润淋巴细胞在高风险组中表达量更高,并且与检查点、人类白细胞抗原、促进炎症、副炎症, T细胞共抑制和Ⅰ型干扰素反应等功能相关(图 6(b))。基质细胞得分和免疫细胞得分在高低风险组中存在差异(P < 0.05),并且高风险组得分更高,表明高风险组与免疫细胞关联更大(图 6(c))。大部分免疫检查点基因在高低风险组间具有差异,并且在高风险组中高表达,如:HAVCR2(TIM3), LAG3, PDCD1LG2(PD-L2) 和IDO1等,说明这些免疫检查点在结肠癌细胞中被激活(图 6(d))。
|
图 6 结肠癌肿瘤免疫细胞及免疫检查点分析 Figure 6 Analysis of immune cells and immune checkpoints in colon cancer |
采用Wilcoxon符号秩检验方法分析药物在高低风险组间敏感性的差异,筛选结肠癌免疫治疗相关药物,共发现瑞卡帕布、伊利斯莫、长春花碱、帕纳替尼、奥拉帕尼、来那度胺, 尼洛替尼等20种药物在高低风险组间具有差异,并且高风险组病人的IC50值更低,说明高风险组病人对该种药物更敏感(表 1)。
| 表 1 免疫疗法相关药物在高危和低危人群中的预测 Table 1 Prediction of immunotherapy related drugs in high-risk and low-risk populations |
本研究中,构建了11个坏死凋亡相关lncRNA的预后模型,并通过高低风险组划分进行了生存分析、独立预后分析、诺谟图模型构建及GSEA、免疫细胞浸润和药物筛选,其结果可为结肠癌的免疫治疗和潜在药物的确定提供较好的理论基础。
差异表达的lncRNA AC010973.2是结肠癌的诊断和预后生物标志物[14]。TRAF2与lncRNA AC010973.2相关,可增加肿瘤对免疫疗法的易感性[15]。LINC02257是结肠癌的独立预后生物标志物,在不同癌症中作为免疫治疗增强子RNA,调节PI3K-Akt信号通路[16]。BRAF与LINC02257相关,是多种实体瘤的重要治疗靶点之一,可增加细胞增殖,抑制细胞凋亡,目前针对BRAF突变患者,已获批了治疗黑色素瘤、甲状腺癌、结直肠癌、非小细胞肺癌等多种靶向治疗药物[17-19]。另一个与LINC02257相关的基因CFLAR,是天然免疫调控网络重要分子,在调控细胞内的凋亡信号通路中发挥至关重要的作用[20]。LINC01857是一种新型lncRNA,在乳腺癌[21]、胃癌[22]和胶质瘤[23]的进展中起到很大作用,但在结肠癌中的作用还未见报道。与LINC01857相关的BACH2在调节性T细胞中能够直接抑制CD25,削弱白介素2受体信号通路,调控调节性T细胞的稳态和滤泡调节性T细胞的分化,BACH2过表达的细胞显著降低了PD-1,TIM-3,TIGIT,LILRB4和KLRG1等免疫抑制性分子的表达量[24-25]。其它的lncRNA目前均未见报道,而这些新获得的坏死性凋亡相关lncRNA可为结肠癌治疗的深入研究提供理论依据。
免疫检查点抑制剂是最成功的免疫疗法之一,在多种癌症治疗上表现出了卓越的疗效[26-29]。本研究中,多个免疫检查点在高低风险组间具有差异,如:HAVCR2(TIM3),LAG3,PDCD1LG2(PD-L2)和IDO1等。虽然已有较多的CTLA-4,PD-1和PD-L1靶向药物在临床上得到应用,但一部分癌症患者对治疗没有反应或产生耐药性,而新一代免疫检查点抑制剂(LAG-3,TIM-3)已成为当前免疫治疗的焦点,其中TIM-3在功能失调的T细胞上表达,与多种癌症的预后不良有关[30]。未来,我们需要进行强有力的生物标志物研究,开发免疫检查点抑制剂,以确保癌症治疗最大的临床疗效。
在本研究获得的20种预测的潜在药物中,AS601245作为一种抗炎JNK抑制剂,在诱导细胞反应和影响CaCo-2结肠癌细胞的基因表达谱方面发挥一定的作用[31]。2022年美国临床肿瘤学会年会发布的最新研究成果表明坏死性凋亡与结直肠癌发生发展及抗肿瘤活性相关,且AS601245对高风险组人群具有更好的疗效。全反式维甲酸(ATRA)目前已用于白血病的治疗且疗效较好,它通过下调白血病细胞DNA损伤修复关键因子Chk1,从而增强FLT3-ITD阳性白血病对化疗药物的敏感性[32]。ATRA也可诱导结肠癌细胞株LoVo和RKOD的分化和凋亡[33-34]。环巴胺(Cyclopamine)可以诱导上调半乳糖凝集素-9表达的结直肠癌HT29细胞凋亡,从而抑制结直肠癌的发展[35]。尼洛替尼(Nilotinib)可以通过靶向盘状蛋白结构域受体1抑制结肠癌细胞的侵袭[36]。其它药物还未发现在结肠癌中的治疗作用,未来可作为潜在药物进行机制和用途的深入研究。
| [1] |
RAWLA P, SUNKARA T, BARSOUK A. Epidemiology of colorectal cancer: Incidence, mortality, survival, and risk factors[J]. Przeglad Gastroenterologiczny, 2019, 14(2): 89-103. DOI:10.5114/pg.2018.81072 (  0) 0) |
| [2] |
BHANDARI A, WOODHOUSE M, GUPTA S. Colorectal cancer is a leading cause of cancer incidence and mortality among adults younger than 50 years in the USA: A SEER-based analysis with comparison to other young-onset cancers[J]. Journal of Investigative Medicine, 2017, 65(2): 311-315. DOI:10.1136/jim-2016-000229 (  0) 0) |
| [3] |
GALLUZZI L, KEPP O, CHAN F K, et al. Necroptosis: Mechanisms and relevance to disease[J]. Annual Review of Pathology Mechanisms of Disease, 2017, 12(1): 103-130. DOI:10.1146/annurev-pathol-052016-100247 (  0) 0) |
| [4] |
WANG Ruicong, LI Hongda, WU Jianfeng, et al. Gut stem cell necroptosis by genome instability triggers bowel inflammation[J]. Nature, 2020, 580(4): 386-390. DOI:10.1038/s41586-020-2127-x (  0) 0) |
| [5] |
SPROOTEN J, WIJNGAERT P D, VANMEERBEEK I, et al. Necroptosis in immuno-oncology and cancer immunotherapy[J]. Cells, 2020, 9(8): 1823. DOI:10.3390/cells9081823 (  0) 0) |
| [6] |
XIE Yadong, ZHAO Yifan, SHI Lei, et al. Gut epithelial TSC1/mTOR controls RIPK3-dependent necroptosis in intestinal inflammation and cancer[J]. The Journal of Clinical Investigation, 2020, 130(4): 2111-2128. DOI:10.1172/jci133264 (  0) 0) |
| [7] |
DALEY D, MANIi V R, MOHAN N, et al. Dectin 1 activation on macrophages by galectin 9 promotes pancreatic carcinoma and peritumoral immune tolerance[J]. Nature Medicine, 2017, 23(4): 556-567. DOI:10.1038/nm.4314 (  0) 0) |
| [8] |
YAN Jiong, WAN Peixiang, CHOKSI S, et al. Necroptosis and tumor progression[J]. Trends in Cancer, 2022, 8(1): 21-27. DOI:10.1016/j.trecan.2021.09.003 (  0) 0) |
| [9] |
SHAKIL A B, SYED M A, PEERZADA T M, et al. Long non-coding RNAs: Mechanism of action and functional utility[J]. Non-coding RNA Research, 2016, 1(1): 43-50. DOI:10.1016/j.ncrna.2016.11.002 (  0) 0) |
| [10] |
ATIANAND M K, CAFFREY D R, FITZGERALD K A. Immunobiology of Long Noncoding RNAs[J]. Annual Review of Immunology, 2017, 35(1): 177-198. DOI:10.1146/annurev-immunol-041015-055459 (  0) 0) |
| [11] |
O'BRIEN S, SCHEURLEN K, ROCHET A, et al. Increased expression of long non-coding RNA H19 is associated with colon cancer recurrence[J]. Journal of Surgical Research, 2022, 269: 59-68. DOI:10.1016/j.jss.2021.08.004 (  0) 0) |
| [12] |
KOPP F, MENDELL J T. Functional classification and experimental dissection of long noncoding RNAs[J]. Cell, 2018, 172(3): 393-407. DOI:10.1016/j.cell.2018.01.011 (  0) 0) |
| [13] |
STATELLO L, GUO C J, CHEN L L, et al. Gene regulation by long non-coding RNAs and its biological functions[J]. Nature Reviews Molecular Cell Biology, 2021, 22(2): 96-118. DOI:10.1038/s41580-020-00315-9 (  0) 0) |
| [14] |
LIU Xinhong, XIAO Caizhi, TAN Fang, et al. Discovered differentially expressed lncRNA AC010973.2 can act as a diagnostic and prognostic biomarker for colon adenocarcinoma[J]. Translational Cancer Research, 2020, 9(10): 6275-6286. DOI:10.21037/TCR-20-2011 (  0) 0) |
| [15] |
VREDEVOOGD D W, KUILMAN T, LIGTENBERG M A, et al. Augmenting immunotherapy impact by lowering tumor TNF cytotoxicity threshold[J]. Cell, 2020, 180(2): 404-405. DOI:10.1016/j.cell.2019.06.014 (  0) 0) |
| [16] |
XIAO Junbo, LIU Yajun, YI Jun, et al. LINC02257, an enhancer RNA of prognostic value in colon adenocarcinoma, correlates with multi-Omics immunotherapy-related analysis in 33 cancers[J]. Frontiers in Molecular Biosciences, 2021, 8: 646786. DOI:10.3389/fmolb.2021.646786 (  0) 0) |
| [17] |
SAKAI T, MATSUMOTO S, NAKAGAWA T, et al. Large scale clinico-genomic analyses amongpatients with BRAF-mutated non-small cell lung cancers (NSCLC) identified by nationwide genomic screening project (LC-SCRUM-Japan)[J]. Journal of Clinical Oncology, 2020, 38(suppl 15): 9590. DOI:10.1200/JCO.2020.38.15_suppl.9590 (  0) 0) |
| [18] |
GEOERGER B, BOUFFET E, WHITLOCK J A, et al. Dabrafenib + trametinib combination therapy in pediatric patients with BRAF V600mutant lowgrade glioma: Safety and efficacy results[J]. Journal of Clinical Oncology, 2020, 38(suppl 15): 10506. DOI:10.1200/JCO.2020.38.15_suppl.10506 (  0) 0) |
| [19] |
SUBBIAH V, LASSEN U, E'LEZ E, et al. Dabrafenib plus trametinib in patients with BRAFV600E-mutated biliary tract cancer (ROAR): A phase 2, open-label, single-arm, multicentre basket trial[J]. Lancet Oncology, 2020, 21: 1234-1243. DOI:10.1016/S1470-2045(20)30321-1 (  0) 0) |
| [20] |
WANG Pixiao, JI Yanxiao, ZHANG Xiaojing, et al. Targeting CASP8 and FADD-like apoptosis regulatorameliorates nonalcoholic steatohepatitis in mice and nonhuman primates[J]. Nature Medicine, 2017, 23: 439-449. DOI:10.1038/nm.4290 (  0) 0) |
| [21] |
LI Qian, LI Bao, LU Changliang, et al. LncRNA LINC01857 promotes cell growth and diminishes apoptosis via PI3K/mTOR pathway and EMT process by regulating miR-141-3p/MAP4K4 axis in diffuse large B-cell lymphoma[J]. Cancer Gene Therapy, 2021, 28(9): 1046-1057. DOI:10.1038/s41417-020-00267-4 (  0) 0) |
| [22] |
CHEN G R, SUN W, ZHENG K, et al. LINC01857 promotes the development of gastric cancer by regulating microRNA-200b[J]. European Review for Medical and Pharmacological Sciences, 2020, 24(7): 3648-3656. DOI:10.26355/eurrev_202004_20827 (  0) 0) |
| [23] |
HU Guozhang, LIU Naijie, WANG Han, et al. LncRNA LINC01857 promotes growth, migration, andinvasion of glioma by modulating miR-1281/TRIM65 axis[J]. Journal of Cellular Physiology, 2019, 234(12): 22009-22016. DOI:10.1002/jcp.28763 (  0) 0) |
| [24] |
YAO Chen, LOU Guohua, SUN Hongwei, et al. BACH2 enforces the transcriptional and epigeneticprograms of stem-like CD8(+) T cells[J]. Nature Immunology, 2021, 22(3): 370-380. DOI:10.1038/s41590-021-00868-7 (  0) 0) |
| [25] |
ZHANG H, DAI D, HU Q, et al. Bach2 attenuates IL-2R signalling to control treg homeostasis and Tfr development[J]. Cell Reports, 2021, 35(6): 109096. DOI:10.2139/ssrn.3696755 (  0) 0) |
| [26] |
MORAD G, HELMINK B A, SHARMA P, et al. Hallmarks of response, resistance, and toxicity to immune checkpoint blockade[J]. Cell, 2021, 184(Suppl 4): 5309-5337. DOI:10.1016/j.cell.2021.09.020 (  0) 0) |
| [27] |
AJONA D, ORTIZ-ESPINOSA S, LOZANO T, et al. Short-term starvation reduces IGF-1 levels to sensitize lung tumors to PD-1 immune checkpoint blockade[J]. Nature Cancer, 2020, 1(1): 75-85. DOI:10.1038/s43018-019-0007-9 (  0) 0) |
| [28] |
YUAN Hong, LIU Jing, ZHANG Jun. The current landscape of immune checkpoint blockade in metastatic lung squamous cell carcinoma[J]. Molecules, 2021, 26(5): 1392. DOI:10.3390/MOLECULES26051392 (  0) 0) |
| [29] |
PINTER M, JAIN R K, DAN G D. The current landscape of immune checkpoint blockade in hepatocellular carcinoma: A Review[J]. JAMA Oncology, 2020, 7(1): 113-132. DOI:10.1001/jamaoncol.2020.3381 (  0) 0) |
| [30] |
ANDREWS L P, YANO H, VIGNALI D A A. Inhibitory receptors and ligandsbeyond PD-1, PD-L1 and CTLA-4: Breakthroughs or backups[J]. Nature Immunology, 2019, 20(11): 1425-1434. DOI:10.1038/s41590-019-0512-0 (  0) 0) |
| [31] |
CERBONE A, TOALDO C, PIZZIMENTI S, et al. AS601245, an Anti-inflammatory JNK inhibitor, and clofibrate have a synergistic effect in inducing cell responses and in affecting the gene expression profile in CaCo-2 colon cancer cells[J]. PPAR Research, 2012, 2012(1): 269751-269751. DOI:10.1155/2012/269751 (  0) 0) |
| [32] |
WANG Wenliang, JIANG Zongru, WANG Li, et al. All-trans retinoic acid exerts selective anti-FLT3-ITD acute myeloid leukemia efficacy through downregulating Chk1 kinase[J]. Cancer Letters, 2020, 473: 130-138. DOI:10.1016/j.canlet.2019.12.045 (  0) 0) |
| [33] |
陆爱国, 郁宝铭, 郑民华. ATRA诱导结肠癌细胞株LoVo分化与凋亡[J]. 肿瘤, 2000, 20(1): 4-7. LU Aiguo, YU Baoming, ZHENG Minhua. All-trans-retinoic acid induced differentiation and apoptosis of colorectal cancer cell line LoVo[J]. Tumor, 2000, 20(1): 4-7. DOI:10.3781/j.issn.1000-7431.2000.01.002 (  0) 0) |
| [34] |
左莉. ATRA及其衍生物对结肠癌RKO细胞生物学行为的影响及其机制的研究[D]. 合肥: 安徽医科大学, 2014. DOI: 10.7666/d.D522618. ZUO Li. The effect of ATRA and ATPR on colorectal cancer cell and its possible molecular mechanisms[D]. Hefei: Anhui Medical University, 2014. DOI: 10.7666/d.D522618. (  0) 0) |
| [35] |
赵鑫, 唐亚萍, 陈琳琳, 等. Galectin-9调控SHH信号通路影响结直肠癌HT29细胞凋亡[J]. 中国病理生理杂志, 2019, 35(3): 459-465. ZHAO Xin, TANG Yaping, CHEN Linlin, et al. Galectin-9 regulates SHH signaling pathway to influence apoptosis of colorectal cancer HT29 cells[J]. Chinese Journal of Pathophysiology, 2019, 35(3): 459-465. DOI:10.3969/j.issn.1000-4718.2019.03.013 (  0) 0) |
| [36] |
LEROY C, ROBERT B, SIMON V, et al. 442 The Abl tyrosine kinase inhibitor Nilotinib inhibits invasive properties of colon cancer cells by targeting the discoidin domain receptor 1[J]. European Journal of Cancer Supplements, 2010, 8(5): 113-113. DOI:10.1016/S1359-6349(10)71243-X (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


