尿毒症(Uremia,UA)的字面意思是“血液中的尿素”,是慢性肾脏疾病(Chronic kidney disease, CKD)患者发展到终末期肾脏疾病,即肾小球滤过率 < 15 ml/min/1.73 m2时具有的特征[1]。尿毒症综合征是指CKD患者出现全身器官功能障碍的各种症状和体征,可导致许多蛋白结合物和水溶性代谢物在血浆中蓄积,即尿毒症溶质,这些毒素的堆积严重危害着患者们的健康[2]。尿毒症综合征是常见的全球性公共卫生问题,患者需要通过定期透析或肾移植进行肾脏替代治疗,且常常会面临皮肤瘙痒,严重贫血,心脑血管疾病等风险,给患者和社会都带来了巨大的负担。一项横断面研究表明中国CKD患者人数约为1.195亿,总体患病率为10.8%[3],据中国血液净化学会调查估计,2008年中国大陆终末期肾病维持性血液透析或腹膜透析患者的患病率为71.9/百万人口,患病率每年增加52.9%[4]。
因此,寻找新的诊断和治疗手段是临床工作的关键,以防止患者发展至终末期肾病,出现尿毒症的表现。自噬是在某些生理或病理条件下细胞内蛋白质和衰老细胞器的动态自消化循环,是细胞生存和健康所必需的生物过程,对细胞生长、凋亡和维持内环境稳态起着重要作用[5]。越来越多的研究表明,自噬在多种CKD中发挥肾脏保护作用,包括IgA肾病、糖尿病肾病和膜性肾病等[6-7]。
基因表达综合(Gene expression omnibus,GEO)数据库是一个存储多种生物信息的基因表达数据库,可以用于检索任何物种来源的遗传信息,通过提供基因表达测量数据信息平台,使更多的研究者能够进行更深入的数据挖掘[8]。本研究对GSE37171和GSE38750基因芯片进行分析,通过R语言对关键差异表达基因(Differentially expressed gene,DEG)进行筛选和深度挖掘,旨在建立基于UA差异表达自噬相关基因(AT-DEGs)的诊断模型。通过这种新模型,提高UA诊断的准确性和便利性,并为临床工作和UA机制研究提供更多的候选生物标志物。
1 材料和方法 1.1 数据来源根据以下标准选择了两个数据集GSE37171和GSE38750:①包含UA患者和健康人的样本;②样本数不少于30个。其中训练集采用基于GSE37171数据集,包括75个UA患者血液样本和40个健康人血液样本。以GSE38750数据集为验证集,包括15个来自UA患者的血液样本和19个健康个体的血液样本。
1.2 数据处理分别下载训练数据集GSE37171和验证数据集GSE38750的微阵列数据集的原始和串行矩阵文件。使用R包中的"RMA",从原始数据中提取探针表达矩阵并进行标准化,然后通过平台注释文件转换为基因表达矩阵。对同一基因对应的多个探针进行取平均值。使用Perl脚本获得转化后的基因表达矩阵后,使用R的"Limma"包对标准化后的基因表达矩阵进行分析。
1.3 自噬相关差异基因的筛选和分析为了鉴定UA和健康个体之间的差异表达基因(DEGs),使用"Limma"包根据Adj.P.Val < 0.05和|logFC| < 1筛选DEGs。同时从人类自噬数据库(Human autophagy database,HADb,http://www.autophagy.lu)中获取与人类疾病相关的自噬基因(Autophagy-related genes,ATGs),然后使用R中的"VennDiagram"包对ATGs和DEGs取交集,得到与自噬相关的DEGs(AT-DEGs)。分别通过"heatmap"和"ggplot2"包绘制热图和火山图,从而对AT-DEGs进行可视化分析。
1.4 功能富集分析使用R软件对UA和健康个体之间的DEGs进行基因集富集分析(Gene set enrichment analysis,GSEA),从而可视化UA和健康个体之间存在区别的生物通路。对二者之间的AT-DEGs进行基因本体(Gene ontology,GO), 京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)分析并可视化,筛选条件为Adj.P.Val < 0.05,进一步研究二者间与自噬相关的DEGs活跃的生物途径和通路。通过Metascape数据库对AT-DEGs进行DisGeNet富集分析,以分析哪些疾病与其关系最为密切。
1.5 基于AT-DEGs的风险评分诊断模型的建立与评估使用“glmnet”包对AT-DEGs进行最小绝对收缩和选择算子(Least absolute shrinkage and selection operator,Lasso)回归分析,以约束参数,防止过拟合,减少偏倚。然后对lasso回归产生的UA特征基因进行多因素logistic分析。以P < 0.05作为差异具有统计学意义,将各AT-DEG表达水平(α)乘以相应系数(β)计算风险评分。建立列线图模型对结果进行评估和分析,预测相关AT-DEGs在UA中的发生率。最后使用“pROC”包评估预测模型的准确性,并计算ROC曲线下面积(AUC)。
1.6 列线图的构建与验证使用"RMS"构建列线图,汇总logistic回归分析得到的有意义的参数,并整合多个预测因子评估UA的发生概率。采用Harrell一致性指数(C-index)计算预测结果与实际观测结果一致的概率,并通过"Hmisc"和"RMS"绘制校准曲线,将分析结果可视化,评价基于AT-DEGs的风险评分模型的预测能力。
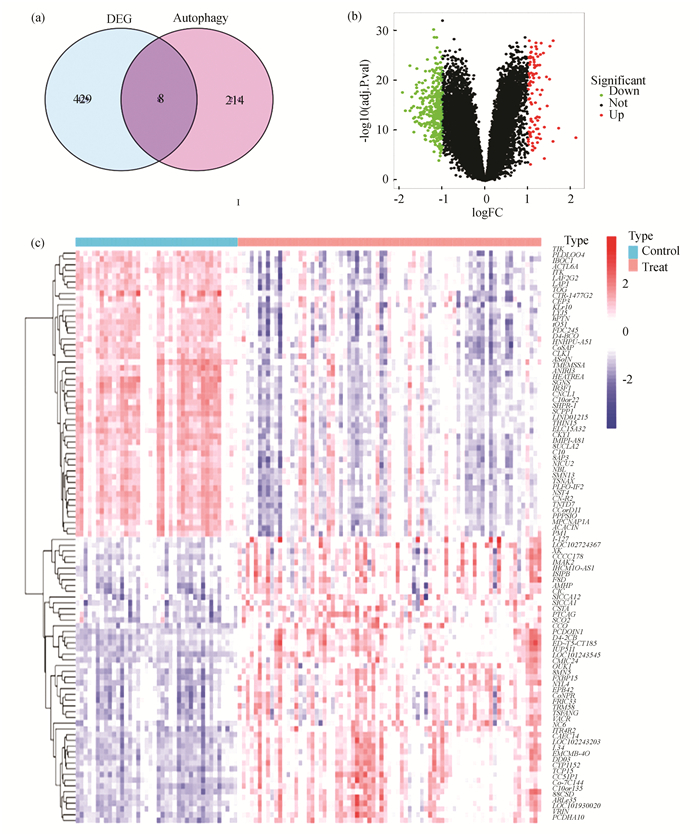
2 结果 2.1 DEGs的鉴定对训练数据集GSE37171中所含数据进行分析,共筛选出437个DEGs,其中上调基因98个,下调基因339个。将这些DEGs与从HADb中收集到的222个ATGs进行比较,发现有8个AT-DEGs存在交集(图 1(a)),包括上调的FKBP1B及下调的NAF1,FOS,BIRC6,CXCR4,SIRT1,CDKN1B,HIF1A。通过火山图(图 1(b))和热图(图 1(c))可以看到8种AT-DEGs在UA患者和正常个体之间的表达水平存在显著差异。
|
图 1 AT-DEGs鉴定结果的可视化:(a)DEGs与ATGs的交集维恩图;(b)AT-DEGs的火山图;(c)AT-DEGs的热图 Figure 1 Visualization of the results of AT-DEGs identification: (a) Intersection Venn diagram of DEGs and ATGs; (b) Volcano plot of AT-DEGs; (c) Heat map of AT-DEGs |
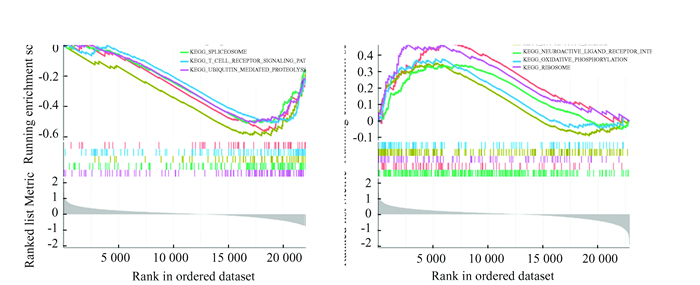
对UA与正常个体间的437个DEGs进行GSEA富集分析,结果提供了42条潜在的生物学通路,如图 2所示。结直肠癌、RNA降解、剪接体、T细胞受体信号通路, 泛素介导的蛋白水解通路是正常组最活跃的生物学通路,而心肌收缩, 凯格-亨廷顿病, 神经活性配体受体相互作用, 氧化磷酸化, 核糖体通路是UA组中最活跃的生物学通路。
|
图 2 DEGs的GSEA富集分析:(a)正常组;(b)UA组 Figure 2 GSEA enrichment analysis of DEGs: (a) Normal group; (b) UA group |
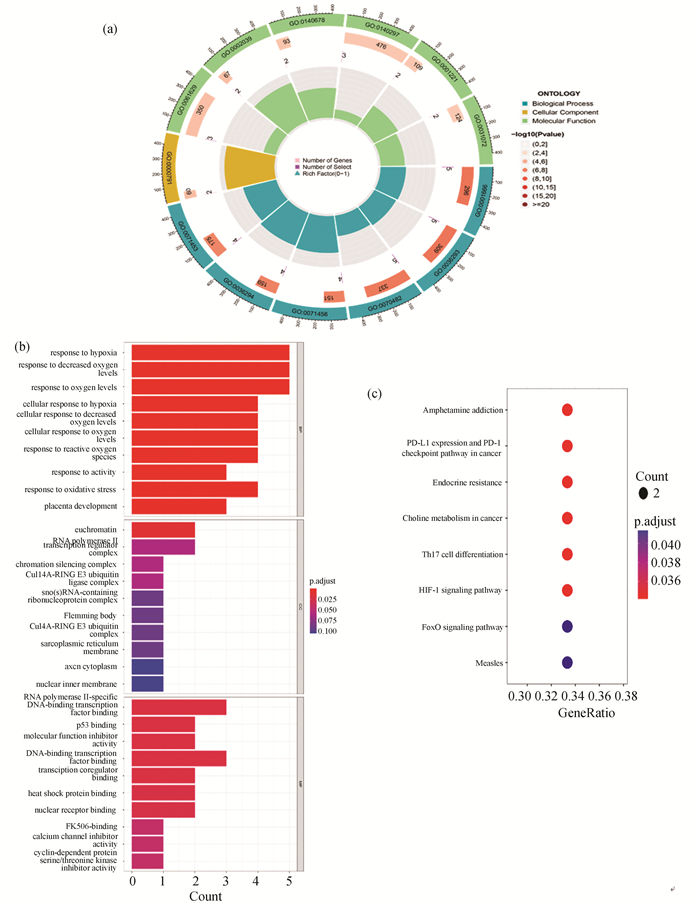
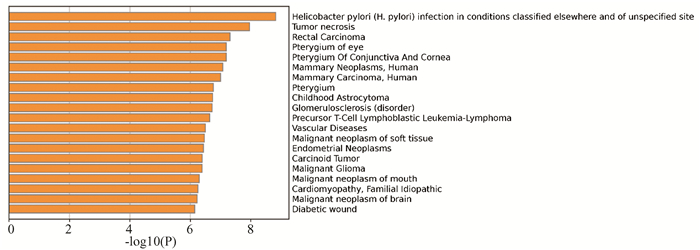
使用R软件对8个AT-DEGs进行富集分析。如图 3(a), 3(b)所示,AT-DEGs的GO功能富集分析包括细胞成分(Cellular component,CC), 生物过程(Biological process,BP)和分子功能(Molecular function, MF)三个部分,主要包括细胞对缺氧的反应, 常染色质, p53结合, DNA结合转录因子结合, 热休克蛋白结合等生物功能。图 3(a)中,第一个圈代表BP,CC,MF三个类别;第二个圆圈代表每个类别上富集DEGs的数目,颜色越红代表交集基因在上面富集的越显著;第三个圆圈代表在每个类别上富集的AT-DEGs的数目;第四个圆圈代表富集的DEGs/AT-DEGs的比例。如图 3(c)所示,这8个AT-DEGs富集的KEGG通路主要包括与自噬相关的生物代谢通路,涵盖细胞凋亡、细胞衰老和细胞周期以及免疫炎症相关疾病,如苯丙胺成瘾, PD-L1表达与PD-1检查点通路在肿瘤中的表达, 内分泌抵抗, 癌症中的胆碱代谢, Th17细胞分化, HIF-1信号通路, FoxO信号通路等。Metascape对AT-DEGs的DisGeNET分析结果如图 4所示,提示AT-DEGs除肾小球硬化症外,还与幽门螺杆菌, 直肠癌、白血病、乳腺癌等免疫功能相关疾病联系紧密。
|
图 3 AT-DEGs富集分析图:(a) GO功能富集圆圈图;(b) GO功能富集分析柱状图;(c) KEGG通路富集分析气泡图 Figure 3 Enrichment analysis of AT-DEGs: (a) GO functional enrichment circle diagram; (b) GO functional enrichment analysis bar chart; (c) Bubble map of KEGG pathway enrichment analysis |
|
图 4 AT-DEGs的DisGenNET功能富集柱状图 Figure 4 DisGenNET functional enrichment bar graph of AT-DEGs |
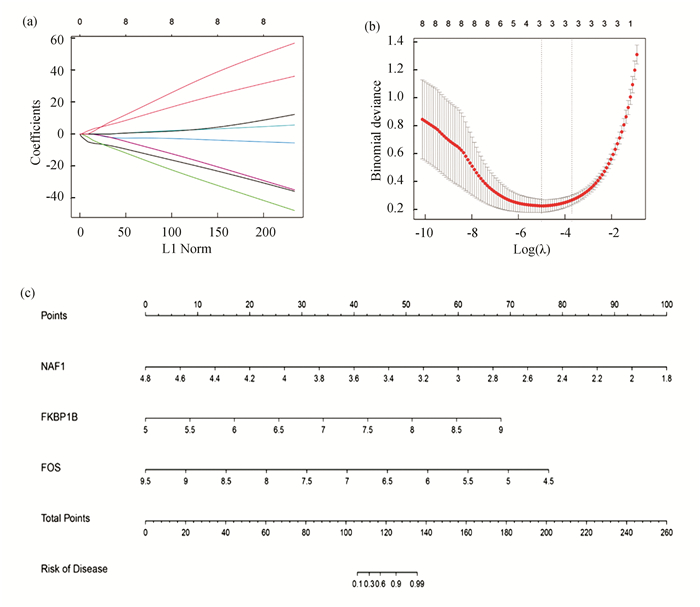
采用Lasso回归分析以最小化诊断模型的偏倚,在保证最佳拟合误差的同时使参数尽可能简单,降低过拟合概率,选择最合适的调谐参数λ(图 5(a))。在训练集的8个候选基因中,共得到3个与诊断相关的关键AT-DEG基因:NAF1, FKBP1B和FOS。使用这3个关键基因构建预测模型(图 5(b))。模型构建采用如下公式:风险评分= NAF1*-7.586+FKBP1B *3.881+ FOS*-3.521。随后建立诊断模型列线图,进一步将结果可视化并应用于临床。如图 5(c)所示,根据3个关键AT-DEGs在人体中的表达水平,医生或患者可以将其定位到列线图对应的尺度上,并计算相应的评分。每个基因的累积表达分数为总分。从总分向下投影的风险评分可用于评估个体罹患UA的风险概率。
|
图 5 采用Lasso回归分析筛选合适的AT-DEGs构建诊断预测模型:(a)Lasso回归基因系数图;(b)Lasso回归交叉验证图;(c)基于核心AT-DEGs,建立预测UA诊断的列线图 Figure 5 Using Lasso regression analysis to screen suitable AT-DEGs to construct a diagnostic prediction model: (a) Lasso regression gene coefficient map; (b) Lasso regression cross-validation plot; (c) Based on the core AT-DEGs, a nomogram was established to predict UA diagnosis |
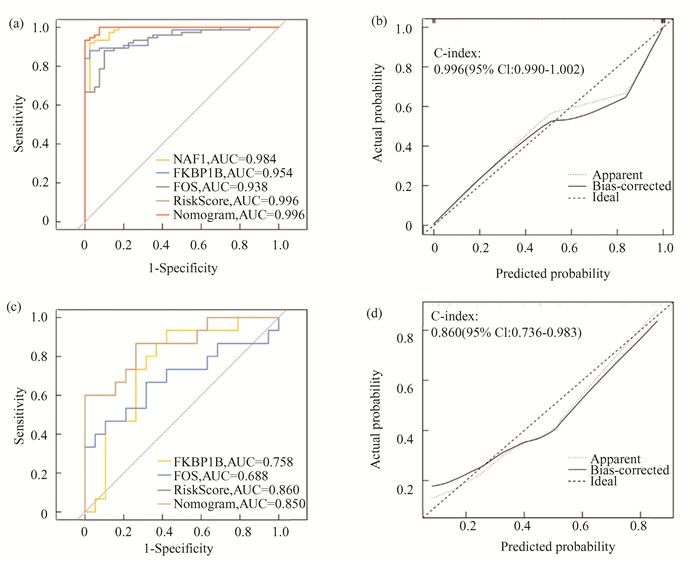
采用AUC曲线验证AT-DEGs相关诊断模型的准确性。训练集风险评分的AUC曲线下面积为0.996,表明模型具有较高的准确率(图 6(a))。验证集AUC面积为0.860,证实AT-DEGs诊断模型具有较高的预测准确性(图 6(c))。采用C-index对列线图进行检验。结果如图 6(b), 6(d)所示,训练集和验证集的C-index分别为0.996和0.860,表明模型的预测能力值得肯定。基于训练集和验证集的AUC曲线下面积图,提示FKBP1B和FOS基因具有最高的诊断准确性。
|
图 6 AT-DEGs诊断预测模型的准确性评价:(a)基于训练数据集的UA诊断模型ROC曲线;(b)训练数据集C-index校准曲线;(c)基于验证数据集的UA诊断模型ROC曲线;(d)验证数据集C-index校准曲线 Figure 6 Accuracy evaluation of the AT-DEGs diagnostic prediction model: (a) ROC curve of UA diagnostic model based on training dataset; (b) C-index calibration curve of training dataset; (c) ROC curve of UA diagnostic model based on the validation dataset; (D) C-index calibration curve of the validation dataset |
“尿毒症”一词在1840年由皮奥利和L’he ritier首次提出,目前尿毒症在临床上被用来描述一种具有许多相关特征的复杂综合征[9]。尿毒症综合征是由于大量化合物进行性滞留所致,这些化合物在正常情况下由健康的肾脏排泄。由于这种积累,这些化合物被称为尿毒症滞留溶质,这些化合物的累积会对许多身体功能产生负面影响,并导致逐渐的内源性中毒。出现尿毒症的患者往往会伴随贫血、疲劳、睡眠障碍、皮肤瘙痒、食欲减退等症状,严重影响患者的生活质量[10]。目前没有特定的时间点可以界定肾功能进行性丧失患者的尿毒症发作,且尿毒症的早期症状(如疲劳)是非特异性的,因此很难以识别[11]。此外,目前现代医学可以减缓肾衰竭的进展,但只能通过替代肾功能的透析或者肾脏移植的方式来治疗尿毒症。然而,在某些情况下,患者接受透析治疗数十年后的总体结果往往不尽如人意,他们的生活质量通常很低[12]。因此,临床上亟需寻求新的诊断和治疗手段,以防止患者发展至终末期肾病,出现尿毒症的表现。
高通量芯片技术的应用使生物信息学方法可以更加快速高效地筛选与疾病发生和发展机制相关的核心基因,为疾病的诊断、治疗和新药设计提供依据。自噬是细胞的一种受控机制,可以对有缺陷和不必要的细胞成分系统性降解和回收[13],它具有抗炎和促炎作用,在肾脏生理学和体内平衡中起着重要作用[14],在各种急性肾损伤、肾小球疾病和衰老模型中的上皮肾细胞和足细胞中被证实具有肾脏保护作用[15]。
在本次研究中,将GEO数据库中获得的437个DEGs与HADb数据库中的222个ATGs取交集,筛选出8个AT-DEGs。同时,制作健康组和UA组之间的DEGs热图和火山图,以更好地呈现可视化结果。对这8个AT-DEGs进行GO, KEGG和DisGeNET富集分析。发现AT-DEGs的GO功能富集主要集中在细胞对缺氧的反应, 常染色质, p53结合, DNA结合转录因子,热休克蛋白结合等生物功能上。富集的KEGG通路主要包括与自噬相关的生物代谢通路,如细胞凋亡、细胞衰老和细胞周期以及免疫炎症相关的疾病,如苯丙胺成瘾, 内分泌抵抗, 癌症中的胆碱代谢, Th17细胞分化, HIF-1信号通路, FoxO信号通路等。DisGeNET分析显示,AT-DEGs除肾小球硬化症外,还与幽门螺杆菌, 直肠癌、白血病、乳腺癌等免疫功能相关疾病相关。采用lasso回归分析生成包含3个关键基因的最优回归模型,之后使用验证数据集进行进一步验证。其中FKBP1B和FOS在验证集ROC曲线下面积均大于0.5,提示其诊断能力值得信赖。
低氧诱导因子(HIF)-1是调控低氧反应基因的转录因子,是治疗CKD患者肾性贫血的潜在靶点,目前,小分子HIF抑制剂被用于激活HIF-α信号通路,并通过恢复促红细胞生成素的产生来提高血浆血红蛋白水平[16]。CKD及其相关的炎症反应会刺激蛋白质能量消耗,这是一种导致肌肉质量损失的并发症,肌肉萎缩主要是通过自噬/溶酶体和蛋白酶体途径加速蛋白质降解引起的,FOXO1控制肌肉生长抑制素的表达,肌肉生长抑制素是一种限制肌肉生长并导致CKD诱导的肌肉萎缩的生物因子[17]。有研究表明,敲低SCP4可显著抑制FOXO1/3a介导的Atrogin-1和MuRF-1的表达,防止CKD小鼠肌肉萎缩[18]。IL-17A是免疫细胞Th17分泌的标志性细胞因子,在新月体性和增殖性肾炎的实验模型中,Th17/IL-17轴在肾组织损伤中发挥重要作用,而这些侵袭性自身免疫性肾脏疾病可显著促进终末期肾病[19]。
FK506结合蛋白1B(FK506 binding proteins 1B,FKBP1B)是FKBPs家族中的一员[20]。FKBP属于亲免素家族的肽基-脯氨酰顺式/反式异构酶,是一种广泛分布且系统发育保守的非淋巴特异性蛋白,存在于多种真核生物中[21],可以与FK506、雷帕霉素、环孢菌素等免疫抑制药物结合,其中就包括CKD患者经常使用的免疫抑制剂他克莫司[22]。FKBP家族成员分布广泛,被报道能够调节糖皮质激素信号、经典和非经典NF-κB信号, mTOR/Akt和TGF-β信号通路[23-25]。一项研究表明,miR-23a-3p可通过靶向调控FKBP5抑制NF-κB信号通路而改善脓毒症诱导的急性肾损伤,而FKBP51被认为可通过增加TIMP3的自噬降解来增加透明细胞肾细胞癌的侵袭和迁移[26]。此外,有研究发现FKBP1B在肾移植急性排斥患者中表达上调,FKBP1B mRNA水平升高与同种异体肾移植功能恶化和急性排斥反应风险增加有关[27]。
Fos蛋白家族主要包括c-Fos,FosB,Fra-1和Fra-2,这些Fos蛋白可以使Jun家族蛋白形成结构高度稳定的同源或异源二聚体,调控细胞的生命活动[28]。c-Fos在多种如增殖、分化、自噬, 凋亡的细胞生物过程中起重要作用,从而可以参与多种肾脏病的发生及发展[29-31]。有研究表明,c-Jun和c-Fos的活性在常染色体显性多囊肾病(PKD)患者的肾囊组织和多囊肾病畸形小鼠的肾囊组织中发生改变,c-Fos的表达显著增加,而PKD是引起终末期肾脏病的常见原因之一[32]。在5/6肾被切除的尿毒症大鼠模型组中,c-Fos和c-Jun基因表达也有所增强[33]。有学者发现扶正化瘀方可通过改善血清肌酐、尿素氮水平,减轻肾组织胶原过度沉积,降低TNF-α,IL-1β,IL-6水平,抑制MAPK/FOS信号分子的表达,从而发挥抗肾纤维化作用[34]。
综上所述,本研究中确定的AT-DEGs或关键基因可能通过上述途径参与UA的进展。FKBP1B和FOS可能是与自噬相关的诊断UA的关键基因。本研究首次利用生物信息学技术筛选UA自噬相关的高灵敏度, 高特异性诊断标志物,构建诊断模型,以减少侵入性检测,为UA的早期诊断和靶向药物研究提供参考。本研究的不足之处在于,虽然GEO数据库中包含了世界各大研究机构的研究成果,但基因库的全面性仍有欠缺,纳入的基因有限,因此后续还需要挖掘更多数据库以及使用动物或细胞实验来进行更全面、更深入的研究,以提高该研究领域的准确性和全面性。
| [1] |
COHEN G. Immune dysfunction in uremia 2020[J]. Toxins (Basel), 2020, 12(7): 439. DOI:10.3390/toxins12070439 (  1) 1) |
| [2] |
NIGAM S K, BUSH K T. Uraemic syndrome of chronic kidney disease: Altered remote sensing and signalling[J]. Nature Reviews Nephrology, 2019, 15(5): 301-316. DOI:10.1038/s41581-019-0111-1 (  1) 1) |
| [3] |
ZHANG Luxia, WANG Fang, WANG Li, et al. Prevalence of chronic kidney disease in China: A cross-sectional survey[J]. Lancet, 2012, 379(9818): 815-822. DOI:10.1016/S0140-6736(12)60033-6 (  1) 1) |
| [4] |
ZUO L, WANG M. Current burden and probable increasing incidence of ESRD in China[J]. Clinical Nephrology, 2010, 74(Suppl 1): S20-S22. (  1) 1) |
| [5] |
LEVINE B, KROEMER G. Biological functions of autophagy genes: A disease perspective[J]. Cell, 2019, 176(1-2): 11-42. DOI:10.1016/j.cell.2018.09.048 (  1) 1) |
| [6] |
CHOI M E. Autophagy in kidney disease[J]. Annual Review of Physiology, 2020, 82: 297-322. DOI:10.1146/annurev-physiol-021119-034658 (  1) 1) |
| [7] |
TANG Chengyuan, LIVINGSTON M J, LIU Zhiwen, et al. Autophagy in kidney homeostasis and disease[J]. Nature Reviews Nephrology, 2020, 16(9): 489-508. DOI:10.1038/s41581-020-0309-2 (  1) 1) |
| [8] |
王潇, 杨智勇. 基于GEO数据库的肥厚型心肌病差异表达基因分析[J]. 中国医科大学学报, 2023, 52(4): 313-317. WANG Xiao, YANG Zhiyong. Identification of differentially expressed genes of hypertrophic cardiomyopathy based on the gene expression omnibus database[J]. Journal of China Medical University, 2023, 52(4): 313-317. DOI:10.12007/j.issn.0258‐4646.2023.04.005 (  1) 1) |
| [9] |
ALMERAS C, ARGILÉS A. The general picture of uremia[J]. Seminars in Dialysis, 2009, 22(4): 329-333. DOI:10.1111/j.1525-139X.2009.00575.x (  1) 1) |
| [10] |
CLEMENTI A, VIRZÌ G M. Novel clinical updates in uremia[J]. Journal of Clinical Medicine, 2022, 11(13): 3791. DOI:10.3390/jcm11133791 (  1) 1) |
| [11] |
MEYER T W, HOSTETTER T H. Uremia[J]. The New England Journal of Medicine, 2007, 357(13): 1316-1325. DOI:10.1056/NEJMra071313 (  1) 1) |
| [12] |
LEE M B, BARGMAN J M. Survival by dialysis modality-who cares?[J]. Clinical Journal of The American Society of Nephrology, 2016, 11(6): 1083-1087. DOI:10.2215/CJN.13261215 (  1) 1) |
| [13] |
MIZUSHIMA N, KOMATSU M. Autophagy: Renovation of cells and tissues[J]. Cell, 2011, 147(4): 728-741. DOI:10.1016/j.cell.2011.10.026 (  1) 1) |
| [14] |
LIN T A, WU V C, WANG C Y. Autophagy in chronic kidney diseases[J]. Cells, 2019, 8(1): 61. DOI:10.3390/cells8010061 (  1) 1) |
| [15] |
FOUGERAY S, PALLET N. Mechanisms and biological functions of autophagy in diseased and ageing kidneys[J]. Nature Reviews Nephrology, 2015, 11(1): 34-45. DOI:10.1038/nrneph.2014.201 (  1) 1) |
| [16] |
NATALE P, PALMER S C, JAURE A, et al. Hypoxia-inducible factor stabilisers for the anaemia of chronic kidney disease[J]. Cochrane Database of Systematic Reviews, 2022, 8(8): Cd013751. DOI:10.1002/14651858.CD013751.pub2 (  1) 1) |
| [17] |
ALLEN D L, UNTERMAN T G. Regulation of myostatin expression and myoblast differentiation by FoxO and SMAD transcription factors[J]. American Journal of Physiology-cell Physiology, 2007, 292(1): C188-C199. DOI:10.1152/ajpcell.00542.2005 (  1) 1) |
| [18] |
LIU Xinyan, YU Rizhen, SUN Lijing, et al. The nuclear phosphatase SCP4 regulates FoxO transcription factors during muscle wasting in chronic kidney disease[J]. Kidney International, 2017, 92(2): 336-348. DOI:10.1016/j.kint.2017.02.031 (  1) 1) |
| [19] |
KREBS C F, PANZER U. Plasticity and heterogeneity of Th17 in immune-mediated kidney diseases[J]. Journal of Autoimmunity, 2018, 87: 61-68. DOI:10.1016/j.jaut.2017.12.005 (  1) 1) |
| [20] |
GANT J C, BLALOCK E M, CHEN K C, et al. FK506-Binding protein 12.6/1b, a negative regulator of [Ca2+], rescues memory and restores genomic regulation in the hippocampus of aging rats[J]. Journal of Neuroscience, 2018, 38(4): 1030-1041. DOI:10.1523/JNEUROSCI.2234-17.2017 (  1) 1) |
| [21] |
张少森, 鞠久君, 朱雪洁, 等. FKBP1B在乳腺癌中的表达及其临床意义[J]. 医学研究杂志, 2022, 51(5): 59-63+29. ZHANG Shaosen, JU Jiujun, ZHU Xuejie, et al. Expression and Clinical Significance of FKBP1B in Breast Cancer[J]. Journal of Medical Research, 2022, 51(5): 59-63+29. DOI:10.11969/i.issn.1673-548X.2022.05.014 (  1) 1) |
| [22] |
GUO Zufeng, HONG S Y, WANG Jingxin, et al. Rapamycin-inspired macrocycles with new target specificity[J]. Nature Chemistry, 2019, 11(3): 254-263. DOI:10.1038/s41557-018-0187-4 (  1) 1) |
| [23] |
LI Yingxiang, LIU Chengdong, BAI Xuanxuan, et al. FK506-binding protein 5 regulates cell quiescence-proliferation decision in zebrafish epithelium[J]. Febs Letters, 2023, 597(14): 1868-1879. DOI:10.1002/1873-3468.14670 (  1) 1) |
| [24] |
ALQUDAH A, ABUDALO R, QNAIS E, et al. The emerging importance of immunophilins in fibrosis development[J]. Molecular and Cellular Biochemistry, 2023, 478(6): 1281-1291. DOI:10.1007/s11010-022-04591-1 |
| [25] |
QUIST-L∅KKEN I, ANDERSSON-RUSCH C, KASTNES M, H, et al. FKBP12 is a major regulator of ALK2 activity in multiple myeloma cells[J]. Cell Communication And Signaling, 2023, 21(1): 25. DOI:10.1186/s12964-022-01033-9 (  1) 1) |
| [26] |
MAO Shaowei, ZHANG Di, CHEN Luan, et al. FKBP51 promotes invasion and migration by increasing the autophagic degradation of TIMP3 in clear cell renal cell carcinoma[J]. Cell Death & Disease, 2021, 12(10): 899. DOI:10.1038/s41419-021-04192-8 (  1) 1) |
| [27] |
BONEZI V, GENVIGIR F D V, SALGADO P C, et al. Differential expression of genes related to calcineurin and mTOR signaling and regulatory miRNAs in peripheral blood from kidney recipients under tacrolimus-based therapy[J]. Annals of Translational Medicine, 2020, 8(17): 1051. DOI:10.21037/atm-20-1757 (  1) 1) |
| [28] |
王晶, 姜辉, 高雅晨, 等. 原癌基因c-Fos与肾脏疾病关系研究进展[J]. 中华实用诊断与治疗杂志, 2019, 33(12): 1236-1238. WANG Jing, Jiang Hui, GAO Yachen, et al. Relationship between proto-oncogene c-Fos and renal diseases[J]. Journal of Chinese Practical Diagnosis and Therapy, 2019, 33(12): 1236-1238. DOI:10.13507/j.issn.1674-3474.2019.12.025 (  1) 1) |
| [29] |
ZHOU Xue, WANG Ning, ZHANG Yuefeng, et al. Expression of CCL2, FOS, and JUN may help to distinguish patients with IgA nephropathy from healthy controls[J]. Frontiers in Physiology, 2022, 13: 840890. DOI:10.3389/fphys.2022.840890 (  1) 1) |
| [30] |
张媛媛. 外周血T细胞c-Jun和c-Fos在复发型儿童原发性肾病综合征中的表达及临床意义[D]. 河南新乡: 新乡医学院, 2022. ZHANG Yuanyuan. Expression and clinical significance of c-Jun and c-Fos inperipheral blood T cells of children with relapsing idiopathic nephrotic syndrome[D]. Xinxiang, Henan: Xinxiang Medical University, 2022. |
| [31] |
MING Jie, SANA SRGL, DENG Xijin. Identification of copper-related biomarkers and potential molecule mechanism in diabetic nephropathy[J]. Frontiers in Endocrinology, 2022, 13: 978601. DOI:10.3389/fendo.2022.978601 (  1) 1) |
| [32] |
LE N H, VAN DER WAL A, VAN DER BENT P, et al. Increased activity of activator protein-1 transcription factor components ATF2, c-Jun, and c-Fos in human and mouse autosomal dominant polycystic kidney disease[J]. Journal of The American Society of Nephrology, 2005, 16(9): 2724-2731. DOI:10.1681/ASN.2004110913 (  1) 1) |
| [33] |
IMANISHI Y, KOYAMA H, INABA M, et al. Phosphorus intake regulates intestinal function and polyamine metabolism in uremia[J]. Kidney International, 1996, 49(2): 499-505. DOI:10.1038/ki.1996.71 (  1) 1) |
| [34] |
WANG Fan, WANG Siyuan, WANG Jing, et al. Pharmacological mechanisms of Fuzheng Huayu formula for Aristolochic acid I-induced kidney fibrosis through network pharmacology[J]. Frontiers in Pharmacology, 2022, 13: 1056865. DOI:10.3389/fphar.2022.1056865 (  1) 1) |
 2024, Vol. 22
2024, Vol. 22



