2. 上海交通大学 数学科学学院,上海 200240
2. School of Mathematical Science, Shanghai Jiaotong University, Shanghai 200240, China
化合物-蛋白质相互作用(Compound-protein interactions, CPIs)的鉴定对于新药研发过程中的靶点发现,网络药理学探索,蛋白质功能识别,药物重定位等至关重要[1-2]。现有的化合物-蛋白质相互作用预测方法主要分为三类:基于生物化学的湿实验(Wet tests)方法, 基于结构的计算方法, 基于机器学习(Machine learning,ML)和深度学习(Deep learning,DL)的计算方法。①湿实验如动力学实验, 光谱分析, 细胞热转变分析, 电化学方法等[3-7]需要大量的人力, 物力和经费的投入,是非常昂贵和耗时的过程;②基于结构的计算方法主要是通过分子对接[8-11]的方法,筛选能与靶蛋白有效结合的药物小分子化合物,但由于已知的配体数量不足、蛋白质的三维结构未知等原因,该方法的应用受到限制;③基于机器学习和深度学习的计算方法因其大规模测试能力和可靠的预测结果而最受关注[12-14]。
目前,研究人员已经开发了多种机器学习方法如朴素贝叶斯分类器(NBC)[15]、k-最邻近算法(KNN)[16]、随机森林(RF)[17]、支持向量机(SVM)[18]、梯度提升决策树(GBDT)[19]等,并将这些算法模型应用于CPI预测。Nanni等[20]采用分子指纹和蛋白质描述符作为特征向量,输入到支持向量机模型中预测CPIs。He等[21]用28个常见的官能团表示化合物,用特别设计的伪氨基酸组成结合氨基酸组成表示蛋白质序列,再将通过使用最大相关最小冗余(mRMR)方法得到的最佳特征输入K最近邻模型(KNN)中预测药物-靶点相互作用关系。Farshid等[22]利用靶点的结构信息和进化信息来生成包括二级结构图案, 扭转角和结构概率, 可访问表面积等在内的蛋白质特征,将这些特征和化合物分子指纹(811位)组合后输入到其提出的iDTI-ESBoost模型中识别药物-靶点相互作用关系。
近几年来,作为机器学习中的一个新的研究范式,以深度神经网络[23]、卷积神经网络[24]、循环神经网络[25]等为代表的深度学习方法逐渐兴起,其因在语音识别、图像识别及自然语言处理等许多领域的出色表现而变得越来越受欢迎,将深度学习方法应用于药物发现的研究一直在不断增加,开发出了多种用于CPI预测的深度学习算法。Tian等[26]提出利用深度神经网络(DNN)预测化合物-蛋白质互作对, 蛋白质靶点特征使用氨基酸组成, 伪位置特异性评分矩阵, 联合三联组成, 过渡和分布, 自相关系数和结构特征等,药物化合物使用结构指纹进行编码。Xie等[27]利用来自LINCS项目L1000数据库的转录组数据开发了一个基于深度学习算法的框架来预测潜在的药物靶标相互作用。上述方法[21-22, 26-27]应用于CPIs的预测具有一定效果,但缺点在于特征提取需要大量人工的参与,因此当数据量很大时,这些方法就存在明显的局限性,不仅需要许多专家知识,而且涉及复杂的数据处理和算法优化过程,限制了ML和DL算法在CPI领域的进一步开发与应用。
近年来图神经网络(Graph neural network,GNN)尤其是图卷积网络(Graph convolutional network, GCN)的快速发展将深度学习的应用扩展到图领域,相关方法[28-30]也被应用到药物发现中。图(Graph)是一种数据格式,图中的节点表示图网络中的实体,边表示实体之间的连接关系。同质图(Homogeneous graphs)只有一种类型的节点和边,如蛋白互作[28]、药物间互作[29]、基因互作[30]等。相对应的异质图(Heterogeneous graphs,HetG)包括多种类型的图结构,如CPI[31], RNA-疾病关联[32], 生物知识图谱[33]等。Nguyen等[34]提出使用图神经网络预测药物-靶标亲和力,预测性能较优。Tsubaki等[35]将卷积神经网络(CNN)和图卷积网络(Graph convolutional network, GCN)结合起来,即使用GCN处理化合物分子图,CNN处理蛋白质序列,从而进行端对端(End-to-End)的化合物-蛋白质相互作用预测,取得了较优结果,但预测性能和稳定性仍有进一步提升的空间。
GCN应用节点之间的连接作为卷积滤波器来执行邻域聚合,在处理以CPI为代表的生物图(Graph)结构的数据时展现出优势。但GCN本身也存在一些缺陷,最为典型的是其在处理复杂大图时遇到的“邻居爆炸”现象(节点表示和随机梯度的计算复杂度会随着消息传递层数的增加而呈指数级上升),以及多层GCN堆叠所致的过平滑或过拟合问题(当神经网络更深入时,节点在聚合操作之后往往具有相似表示)。国内外学者提出了多种图采样技术以减少消息传递所牵涉的节点个数来降低训练成本,最常见的包括节点采样(如GraphSAGE[36],PinSage[37],VRGCN[38])和层采样(如FastGCN[39],ASGCN[40])等。
为了提升CPI预测的特征提取效率、泛化能力及稳定性,本文提出基于Word2vec[41]表示学习和基于GCN的端对端化合物-蛋白质相互作用预测方法,利用节点特征和拓扑网络来构造图,并基于子图采样策略来实现,具体而言:Word2vec-GCN基于图上的边(Edge)自适应地采样子图,并在每个子图上运行模型,这不仅可以避免邻居爆炸现象,还可以降低模型的复杂性和内存消耗。首先利用Word2vec表示学习方法学习到化合物与蛋白质的特征向量表示,再输入到图神经网络(GCN)中以预测化合物-蛋白质的相互作用,最后将预测结果分别进行交叉验证、独立测试及对比验证。在Binding-DB大规模数据集上运行的结果表明,本文模型相对于传统ML、DL方法和他人提出的先进方法,分类效果更优。
1 数据与处理 1.1 数据库本论文主要选用结合亲和力数据库(The binding database,Binding-DB[42])来进行数据集的构建。Binding-DB数据库是加州大学实验室开发的一个免费的结合亲和力公共数据库,主要关注小分子类药化合物和蛋白质靶点的相互作用亲和力,同时,提供通路信息,化合物ZINC编号以及其他信息,研究者通过网络查询数据库可以获取化合物分子和蛋白质靶点之间的结合数据,可用于药物研发和结合预测模型的构建。Binding-DB数据库包含由120万种化合物和9 000种蛋白质靶标组成的270万个结合数据(Binding data),其中58万个化合物和4 500个蛋白质组成的126万个结合数据被人工标注。Binding-DB的数据来源包括PDB,PubMed相关文献报道数据,专利信息,PubChem BioAssay数据库和Chembl记录数据,数据库定期进行更新并发布版本。
1.2 数据预处理为了保证数据质量,本研究做如下处理:
1) IC50值可以表明抑制剂与底物结合能力,考虑到成药性,去除IC50值大于300 nM的CPI对,同时去除IC50值缺失的CPI对。
2) 考虑到小分子靶向药物主要是指分子量 < 1 000的有机化合物,因此去除数据库中的无机物和分子量>1 000的类药分子。
3) 为了降低蛋白质冗余度,解决“数据泄露”问题,将数据库中蛋白序列一致性>75%的蛋白质靶点进行过滤。
经过上述处理后的数据集包括了36 014个小分子化合物和2 099个蛋白质靶点,并可能产生超过7 500万个CPI对。其中83 676条被确认的CPI对被称为正样本,其余尚未确认的作为未标记数据处理。按照1∶1的数量配比原则,从未标记数据集中随机挑选相同数量的83 676条样本,定义为负样本。随机挑选正负样本数据集中的CPI数据的80%用作训练集,剩余20%用作测试集。
2 方法定义化合物分子集合=[M1, M2, …, Mn]、氨基酸序列集合=[P1, P2, …, Pm],二者互作的标签集合={0, 1},其中,1表示互作的类别标签,0表示不互作的类别标签。化合物-蛋白质对的互作预测任务是一个二分类学习模型S: (M, P)→L,模型输入化合物分子M和氨基酸序列P,输出类别标签L。
本文方法的整体流程如图 1所示,主要过程如下:
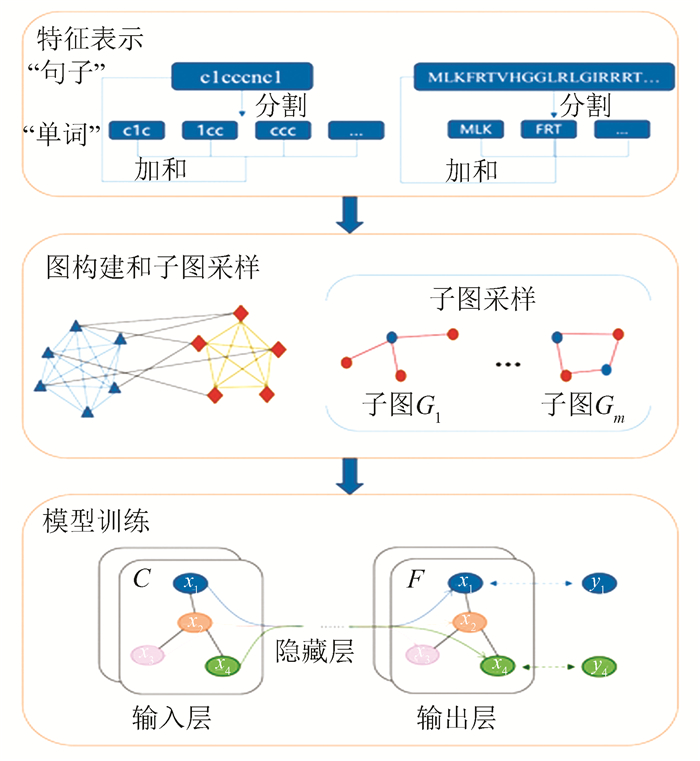
|
图 1 本文方法流程图 Figure 1 Workflow chart of the proposed method |
1) 基于Word2vec表示学习方法实现对化合物分子和蛋白质序列的特征向量化的表示。
2) 将特征输入到图神经网络中,反复调优测试,得到分类预测结果。
2.1 Word2vec表示学习Word2vec是一款基于统计语言模型构建的利用深度学习来训练生成词向量的工具。Word2vec的主要原理是通过预测上下文或目标单词的方法学习单词的向量表示,具体为通过一个简单的神经网络来学习单词的向量表示,该神经网络包括一个输入层(One-hot热编码向量), 一个隐藏层(单词嵌入向量)和一个输出层(Softmax激活函数)。Word2vec有两种实现形式——连续词袋模型(Continuous bag-of-Words, CBOW)和跳字模型(Skip-gram)。本论文采用Skip-gram模型进行化合物药物和蛋白质靶标的向量计算。
神经网络的训练过程为:输入层(1×V维)→输入矩阵*权重矩阵W(V×N)=隐藏层H(1×N)→隐藏层*W'(N×V)→输出层(C*1×V)→softmax输出概率→反向传播训练→最优化权重参数矩阵W→得到词的词向量,完成词嵌入。
为了表示药物分子,将SMILES视为“句子”,将每三个不重叠的字符(代表原子、化合键、分支等)视为“单词”。对于蛋白质序列,我们将它们视为“句子”,每三个不重叠的氨基酸视为一个“单词”。
本研究Word2vec的执行主要是利用Python工具包Gensim完成。选用的方法为Skip-Gram,设置词向量的维度=100,窗口大小=3,负采样(Negative sampling)的数量=12,其余使用默认参数。
2.2 图卷积神经网络(GCN)定义图结构G=(V, E):V表示节点的集合,V={化合物分子集合,氨基酸序列集合}=[M1, M2, …, Mn, P1, P2, …, Pn],E表示边的集合,eij=(ei, ej)∈E表明化合物与蛋白质存在相互作用。A代表图G的邻接矩阵,Aij=1代表化合物节点Mi和蛋白质节点Pj之间存在边。节点特征由Word2vec方法得到。
图卷积的核心思想是利用边的信息对节点信息进行聚合,从而生成新的节点表示。计算公式如下:
| $ H^{(l+1)}=\sigma\left(\widetilde{D}^{-\frac{1}{2}} \widetilde{A} \widetilde{D}^{-\frac{1}{2}} H^{(l)} W^{(l)}\right) $ |
上述公式中:
GCN训练流程为:
1) 输入图结构信息,经过GCN前向传播最终输出每个节点的特征,输出结果与期望值之间产生误差。
2) 进行反向传播,计算有标签的节点的训练损失,对误差运行梯度下降算法来动态调整权重参数直至最优,实现监督分类。
3) 将反向传播后得到的全局权重矩阵代入GCN前向传播公式,计算得到与期望值最接近(即最精确)的预测值输出结果,整个模型达到最优。
上述GCN训练中使用了子图采样方法来代替传统GCN算法中通常使用的微批量(Mini-batch)构造。该方法主要思想是从训练图中抽取几个子图,并在每个子图上建立一个完整的GCN。这种方法使样本中的节点能够提供信息并相互帮助,而不会在GCN层中传播期间涉及批外的节点,使用该算法可以避免传统GCN算法中经常出现的“邻居爆炸”问题。为了确保训练过程的准确性,该采样方法使用了一种基于拓扑结构的边缘采样器,采样器以很大的概率对边缘进行采样,通过考虑同一子图中的节点相互影响的程度来计算边缘概率。为了避免偏差,我们在聚合节点信息和计算微批量损失的过程中对边缘采样概率进行了归一化,来减少图神经网络中的偏差;通过使全图卷积网络中节点聚合的方差最小化定义了最佳边缘采样概率。本研究GCN模型基于Pytorch框架构建完成,相关的算法和数据已在github上公开(网址https://github.com/zgn002/Word2vec-GCN)。
对于均衡数据集,选取曲线下面积(Area under curve,AUC)、准确率、精密度,召回率和F1分数作为模型的评价指标。本研究进行了10次5折交叉验证,其模型表现结果均值作为最终算法准确性的估计。
3 结果分析 3.1 Word2vec-GCN在BindingDB数据集上的分类性能图 2展示Word2vec-GCN模型在Binding-DB数据集上的分类性能总结。结果表明,本模型在CPI预测任务中训练集AUC、准确性,精确度和召回率以及F1分数分别达到0.998 7,0.995,0.996 2,0.997和0.995 2。较高的预测准确度表明,本模型不仅实现了CPI异质性特征的有效提取与融合,还可以更好地聚合邻居信息、节点特征和自适应地捕获化合物-蛋白质空间的拓扑结构。
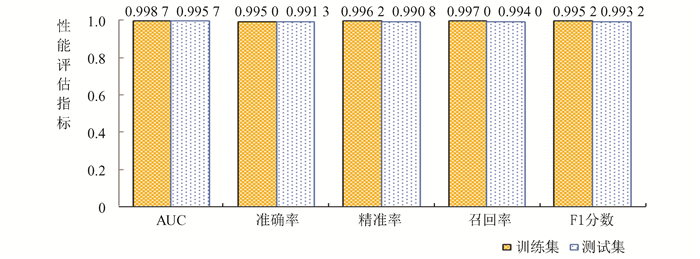
|
图 2 Word2vec-GCN在Binding-DB数据库训练集和测试集上的分类性能 Figure 2 Results of classification performance of our methods on Binding-DB training sets and test sets |
同时,模型在测试集上AUC、准确性、精确度和召回率以及F1分数分别达到0.995 7,0.991 3,0.990 8,0.994和0.993 2。表明了模型具有较高的鲁棒性和可扩展性。模型在测试集上准确性降低可能有以下两点原因:①由于负样本是随机选择未标记的数据而构建,出现假阴性的化合物-蛋白质互作对的概率在较小的数据集中会更高;②测试集中化合物-蛋白质互作对的分布是非常不均衡的,与训练集的分布有所偏离,这也会影响模型的准确性。
3.2 Word2vec-GCN和传统ML和DL方法模型表现对比为了进一步说明Word2vec-GCN在化合物-蛋白质互作预测中的优越性,还将其与一些经典的机器学习和深度学习算法进行了比较,包括随机森林(RF)、支持向量机(SVM)、深度神经网络(DNN)、梯度提升决策树(GBDT)。上述模型的预测结果如表 1所示。根据结果,本文模型优于其他经典的ML和DL算法,本文方法的AUC分别比SVM,GBDT,RF和DNN高46.75%,2.59%,2.30%和5.28%。
| 表 1 Word2vec-GCN在与四个经典ML/DL分类器的模型表现结果比较 Table 1 Results of classification performance of our method compared with four classic ML and DL methods on BindingDB testsets |
基于BindingDB测试集,树模型方法(即GBDT和RF模型)对CPI的分类任务没有产生重要差异,并且所有方法都获得了同样高的AUC分数、准确度、精确性,召回率和F1分数。作为深度学习方法,GCN和DNN模型在网络结构和参数数量上较为复杂。与DNN模型相比,GCN更善于处理以CPI为例的图结构数据。虽然在上述评估方法中,基于树的方法如GBDT和RF都优于DNN架构,但由于DNN模型参数复杂,进一步微调(Fine-tuning)可能会提高DNN模型的预测性能。而SVM方法在CPIs预测任务中取得了五个模型中最差的模型性能,原因可能在于SVM通常在线性数据集上表现良好。尽管许多核函数,如高斯径向基(Gaussian RBF)核函数,也可以处理非线性情况,但它们往往需要大量的相似性特征和大量的计算资源,而在最坏的情况下,一个完全不可分割的数据,如环状数据,使用SVM往往表现不佳。
3.3 Word2vec-GCN与他人先进方法的模型表现对比为了进一步证明本文所提出方法的优越性,Word2vec-GCN方法与以下五种他人先进方法进行了比较。
1) Tsubaki等[35]开发了一种新的CPI预测方法GNN-CNN,该方法通过结合用于化合物结构提取的GCN和用于蛋白质序列表示的CNN,进行端到端表示学习。
2) Tian等[26]提出了一种DL-CPI方法,该方法使用增强深度神经网络来有效地学习化合物-蛋白质对的表示。DL-CPI可以通过分层提取来学习化合物-蛋白对的有用特征,从而实现良好的预测性能。
3) Ye等[43]提出了一种基于多元分类策略的药物-靶标相互作用预测方法—MCSDTI,首先根据相互作用的数量将靶点分为两组,然后设计并采用不同的分类策略来准确预测每组药物-靶点相互作用。
4) Rayhan等[44]提出的CFSBoost是一种简单且计算成本低廉的集成增压分类模型,使用进化和结构特征识别和预测药物-靶标相互作用。
5) Zhang等[45]同样基于Word2vec方法提出了SPVec模型学习药物-靶点相互作用对的特征表示,并将特征向量输入GBDT模型中在DrugBank[46]独立测试集中预测二者相互作用。
本方法与上述5种先进方法的模型表现结果比较如表 2所示。本方法在所有模型性能评估指标上都优于上述五种方法,AUC评分、准确性、精确度,召回率和F1评分分别比次优方法高2.63%,2.06%,2.44%,1.93%和2.51%。在使用相同Word2vec特征提取方法前提下,GCN模型结果优于SPVec-GBDT模型,说明了GCN在表示化合物、蛋白质等生物分子结构以及分子之间的功能关系和集成异质数据方面的优秀性能。Word2vec-GCN方法不仅可以自适应地聚合邻居信息与节点特征,还可以利用化合物-蛋白质空间的拓扑结构。与同为端对端表示学习的GNN-CNN方法相比,Word2vec比CNN更加适用于蛋白质序列的特征提取。这可能是由于CNN固定大小的卷积核只能捕获蛋白质序列滑动窗口内的子序列,而所有子序列的依赖关系难以获取。与CFSBoosting和MCSDTI方法相比,本方法不仅取得了更优的模型表现,而且省去了手工构建特征的繁琐过程。总之,表 2体现了本方法的有效性和高效性。
| 表 2 Word2vec-GCN方法与5种先进方法的模型表现结果比较 Table 2 Results of classification performance of our method compared with five state-of-art models |
从前几节讨论可以得出,本文提出的基于表示学习的图神经网络Word2vec-GCN方法在化合物-蛋白质互作预测任务中展现出了优秀的模型性能和良好的泛化能力。在真实世界中,准确的预测出潜在化合物-蛋白质互作对于促进药物重定位,提高靶点识别效率,加速药物研发过程意义重大。本研究使用Word2vec-GCN方法对Binding-DB数据集中的所有未标记CPIs存在相互作用的概率进行了预测,预测得分越高,说明互作可能性越大。其得分前五名的化合物-蛋白质对如表 3所示。通过检索多个生物信息学数据库(如PubChem,ChEMBL,KEGG等)和相关文献,最终有4个化合物-蛋白质对得到外部证据的证实。为了直观表现出化合物-蛋白质对相互作用,基于结构的分子对接图如图 3所示。由于P33402蛋白数据库中未收录三级结构,故未在图 3中进行展示。排名第三的化合物-蛋白质作用对虽然未在外部数据库中找到实验数据,分子对接结果表明了二者的相互作用。进一步证明Word2vec-GCN方法预测出的五个药物-靶标对的有效性。
| 表 3 Word2vec-GCN预测排名前五的新化合物-蛋白质对 Table 3 Top five novel CPIs predicted by Word2vec-GCN |

|
图 3 Word2Vec-GCN预测排名1,3,4,5的新化合物-蛋白质对分子对接结果 Figure 3 Molecular docking results of rank 1, 3, 4, 5 novel DTIs predicted by Word2Vec-GCN |
Word2vec-GCN方法优势可分为三个方面:①无需专家知识,表示学习自动表征化合物和蛋白质连续、稠密、低维特征,有效避免维数灾难、信息有限等传统特征提取方法的局限性;②GCN模型可以充分利用聚合邻居信息、节点特征和自适应地捕获化合物-蛋白质空间的拓扑结构,从而实现较高的模型精度,同时兼具鲁棒性和可扩展性;③该方法是端对端(End-to-End)学习,即直接从原始数据得到预测结果,极大节省人力、时间和成本。
本文研究内容不足之处在于:①Word2vec表示学习方法本质上属于深度学习方法,深度学习方法的“黑箱模型”使得计算得出的特征向量每一维度代表的含义难以解释,目前暂时缺少适用于像CPI预测等生物问题的可解释性模型方法;②相对于ML方法,GCN方法的参数众多,调参工作量大,后续研究可以尝试引入自动化调参工具,进一步提高Word2vec-GCN方法的预测效率;③仅采用计算方法预测,对于化合物-蛋白质互作发生机制的研究不够深入全面,缺少体内或体外实验支撑。在后续研究中,需要与生物实验结合,提高模型的实用性与准确性。
5 结论1) 模型在训练集和测试集上均取得较高的模型表现,表明了模型具有优秀的预测性能、鲁棒性和可扩展性。
2) 与经典的ML和DL方法(即SVM,GBDT,RF和DNN)和最先进的MDA预测方法(即DL-CPI,GNN-CNN,CFSBoosting和MCSDTI)相比,本研究所提出的Word2vec-GCN模型可以实现更好的预测性能。
3) 本模型重新预测了Binding DB中所有未标记的CPI,并且来自其他数据库的外部证据证实了前五个预测的新CPI中的四个。这表明,Word2vec-GCN可以在新发现的化合物和蛋白质靶点中发现可靠的CPI,可以促进药物重定位、提高靶点识别效率、加速药物研发过程。
| [1] |
薛斌, 李益洲, 李梦龙. 基于化学信息学方法预测药物副作用的研究进展[J]. 计算机与应用化学, 2019, 36(5): 487-490. XUE Bin, LI Yizhou, LI Menglong. Research progress in predicting drug side effects based on chemical informatics methods[J]. Computers and Applied Chemistry, 2019, 36(5): 487-490. DOI:10.16866/j.com.app.chem201905009 (  0) 0) |
| [2] |
YABUUCHI H, NⅡJIMA S, TAKEMATSU H. Analysis of multiple compound-protein interactions reveals novel bioactive molecules[J]. Molecular Systems Biology, 2011, 7(1): 472. DOI:10.1038/msb.2011.5 (  0) 0) |
| [3] |
宗可昕, 张晶, 陈云雨, 等. PLK1 PBD靶向抗肿瘤抑制剂的筛选及活性研究[J]. 中国医药生物技术, 2016, 11(1): 13-20. ZONG Kexin, ZHANG Jing, CHEN Yunyu, et al. Screening and anti-tumor activity of PLK1 PBD targeted antitumor inhibitors[J]. China Medical Biotechnology, 2016, 11(1): 13-20. DOI:10.3969/j.issn.1673-713X.2016.01.004 (  0) 0) |
| [4] |
孙国章, 何莹, 尹照瑞. 灯盏花素与牛血清白蛋白相互作用的光谱学研究[J]. 山西中医药大学学报, 2020, 21(3): 192-195. SUN Guozhang, HE Ying, YIN Zhaorui. Spectroscopic study of the interaction between breviscapine and bovine serum albumin[J]. Journal of Shanxi University of Chinese Medicine, 2020, 21(3): 192-195. DOI:10.19763/j.cnki.2096-7403.2020.03.10 (  0) 0) |
| [5] |
JAFARI R, ALMQVIST H, AXELSSON H, et al. The cellular thermal shift assay for evaluating drug target interactions in cells[J]. Nature Protocols, 2014, 9: 2100-2122. DOI:10.1038/NPROT.2014.138 (  0) 0) |
| [6] |
VILAR S, HRIPCSAK G. Leveraging 3D chemical similarity, target and phenotypic data in the identification of drug-protein and drug-adverse effect associations[J]. Journal of Cheminformatics, 2016, 8: 35. DOI:10.1186/s13321-016-0147-1 (  0) 0) |
| [7] |
MOLINA D M, JAFARI R, IGNATUSHENKO M. Monitoring drug target engagement in cells and tissues using the cellular thermal shift assay[J]. Science, 2013, 341(6141): 84-87. DOI:10.1126/science.1233606 (  0) 0) |
| [8] |
李玲, 李佳蔚, 张月梅. 基于分子对接预测靶点ACE2和IL-6R研究归芪白术方治疗新型冠状病毒肺炎的物质基础及其作用机制[J]. 甘肃中医药大学学报, 2020, 37(2): 1-9. LI Ling, LI Jiawei, ZHANG Yuemei, et al. Material basis and action mechanism of Guiqi Baizhu Fang in the treatment of COVID-19 based on molecular docking prediction for target ACE2 and I-6R[J]. Journal of Gansu University of Chinese Medicine, 2020, 37(2): 1-9. DOI:10.16841/j.issn1003-8450.2020.02.01 (  0) 0) |
| [9] |
MA D L, CHAN D S H, LEUNG C H. Drug repositioning by structure based virtual screening[J]. Chemical Society Reviews, 2013, 42(5): 2130-2141. DOI:10.1039/c2cs35357a (  0) 0) |
| [10] |
贾聪敏. 基于分子振动特征的药物靶点识别及活性预测模型研究[D]. 北京: 北京中医药大学, 2019. JIA Congmin. Research on drug target recognition and activity prediction model based on molecular vibration characteristics[D]. Beijing: Beijing University of Chinese Medicine, 2019. (  0) 0) |
| [11] |
CHOW E, KLEPEIS L. New technologies for molecular dynamics simulations[J]. Comprehensive Biophysics, 2012, 9: 86-104. DOI:10.1016/B978-0-12-374920-8.00908-5 (  0) 0) |
| [12] |
JACOB L, VERT J P. Protein-Ligand Interaction Prediction: An improved chemogenomics approach[J]. Bioinformatics, 2008, 24(19): 2149-2156. DOI:10.1093/bioinformatics/btn409 (  0) 0) |
| [13] |
BLEAKLEY K, YAMANISHI Y. Supervised prediction of drug-target interactions using bipartite local models[J]. Bioinformatics, 2009, 25(18): 2397-2403. DOI:10.1093/bioinformatics/btp433 (  0) 0) |
| [14] |
COELHO E D, ARRAIS J P, OLIVEIRA J L. Ensemble-based methodology for the prediction of drug-target interactions[C]. Process of the 29th IEEE International Symposium on Computer-Based Medical Systems. Washington, USA: IEEE, 2016: 36-41. DOI: 10.1109/CBMS.2016.67.
(  0) 0) |
| [15] |
MADHUKAR N S, KHADE P K, HUANG L, et al. A bayesian machine learning approach for drug target identification using diverse data types[J]. Nature Communication, 2019, 10(1): 5221. DOI:10.1038/s41467-019-12928-6 (  0) 0) |
| [16] |
郭真俊. 基于异构网络的药物靶标交互作用预测方法研究[D]. 长春: 长春工业大学, 2022. GUO Zhenjun. Research on prediction method of drug target interaction based on heterogeneous networks[D]. Changchun: Changchun University of Technology, 2022. (  0) 0) |
| [17] |
HO T K. The random subspace method for constructing decision forests[C]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 1998, 20(8): 832-844. DOI: 10.1109/34.709601.
(  0) 0) |
| [18] |
CORTES C, VAPNIK V. Support-Vector Networks[J]. Machine Learning, 1995, 20(3): 273-297. DOI:10.1007/BF00994018 (  0) 0) |
| [19] |
FRIEDMAN J H. Greedy function approximation: A gradient boosting machine[J]. Annals of Statistics, 2001, 29(5): 1189-1232. DOI:10.1214/aos/101320345 (  0) 0) |
| [20] |
NANNI L, LUMIN A, BRAHNAM S. A set of descriptors for identifying the protein-drug interaction in cellular networking[J]. Journal of Theoretical Biology, 2014, 359(24): 120-128. DOI:10.1016/j.jtbi.2014.06.008 (  0) 0) |
| [21] |
HE Zhisong, ZHANG Jian, SHI Xiaohe, et al. Predicting drug-target interaction networks based on functional groups and biological features[J]. PloS one, 2010, 5(3): e9603. DOI:10.1371/journal.pone.0009603 (  0) 0) |
| [22] |
RAYHAN F, AHMED S, SHATABDA S, et al. iDTI-ESBoost: identification of drug target interaction using evolutionary and structural features with boosting[J]. Scientific Reports, 2017, 7(1): 17731. DOI:10.1038/s41598-017-18025-2 (  0) 0) |
| [23] |
LIU Weibo, WANG Zidong, LIU Xiaohui, et al. A survey of deep neural network architectures and their applications[J]. Neuro Computing, 2017, 234(234): 11-26. DOI:10.1016/j.neucom.2016.12.038 (  0) 0) |
| [24] |
K IM, P. Convolutional neural network[M]. Berkeley, CA: MATLAB Deep Learning, 2017.
(  0) 0) |
| [25] |
MARHON S, CAMEROM C, KREMER S. Recurrent neural networks[M]. Berlin, Heidelberg: Handbook on Neural Information Processing, 2013: 49.
(  0) 0) |
| [26] |
TIAN Kai, SHAO Mingyu, WANG Yang. Boosting compound-protein interaction prediction by deep learning[J]. Methods, 2016, 110: 64-72. DOI:10.1016/j.ymeth.2016.06.024 (  0) 0) |
| [27] |
XIE Lingwei, HE Song, SONG Xinyu, et al. Deep learning-based transcriptome data classification for drug-target interaction prediction[J]. BMC Genomics, 2018, 19(7): 667. DOI:10.1186/s12864-018-5031-0 (  0) 0) |
| [28] |
YUAN Qianmu, CHEN Jianwen, ZHAO Huiying. Structure-aware protein-protein interaction site prediction using deep graph convolutional network[J]. Bioinformatics, 2022, 38(1): 125-132. DOI:10.1093/bioinformatics/btab643 (  0) 0) |
| [29] |
CHANHEE P, JINUK P, SANGHYUN P. AGCN: Attention-based graph convolutional networks for drug-drug interaction extraction[J]. Expert Systems with Applications, 2020, 159: 113538. DOI:10.1016/j.eswa.2020.113538 (  0) 0) |
| [30] |
YUAN Ye, BAR-JOSEPH Z. GCNG: Graph convolutional networks for inferring gene interaction from spatial transcriptomics data[J]. Genome Biology, 2020, 21: 300. DOI:10.1186/s13059-020-02214-w (  0) 0) |
| [31] |
WAN Fangping, HONG Lixiang, XIAO An, et al. NeoDTI: Neural integration of neighbor information from a heterogeneous network for discovering new drug-target interactions[J]. Bioinformatics, 2019, 35(1): 104-111. DOI:10.1093/bioinformatics/bty543 (  0) 0) |
| [32] |
RAO A, VG S, JOSEPH T, et al. Phenotype-driven gene prioritization for rare diseases using graph convolution on heterogeneous networks[J]. BMC Med Genomics, 2108, 11(1): 57. DOI:10.1186/s12920-018-0372-8 (  0) 0) |
| [33] |
MONA A. Neuro-symbolic representation learning on biological knowledge graphs[J]. Bioinformatics, 2017, 33(17): 2723-2730. DOI:10.1093/bioinformatics/btx275 (  0) 0) |
| [34] |
NGUYEN T, LE H, QUINN T P. GraphDTA: Predicting drug-target binding affinity with graph neural networks[J]. Bioinformatics, 2020, 37(8): 1140-1147. DOI:10.1093/bioinformatics/btaa921 (  0) 0) |
| [35] |
TSUBAKI M, TOMⅡ K, SESE J. Compound-protein interaction prediction with end-to-end learning of neural networks for graphs and sequences[J]. Bioinformatics, 2019, 35(2): 309-318. DOI:10.1093/bioinformatics/bty535 (  0) 0) |
| [36] |
HAMILTON W, YING R, LESKOVEC J. Inductive representation learning on large graphs[C]. Neural Information Processing Systems. Long Beach, California, USA, 2017: 1025-1035. DOI: 10.48550/arXiv.1706.02216.
(  0) 0) |
| [37] |
REX Y, RUINING H, KAIFENG C, et al. Graph convolutional neural networks for web-scale recommender systems[C]. Proceedings of the 24th ACM SIGKDD International Conference on Knowledge Discovery & Data Mining London, United Kingdom: ACM, 2017: 974-983. DOI: 10.1145/3219819.3219890.
(  0) 0) |
| [38] |
CHEN Jianfei, ZHU Jun, SONG Le. Stochastic training of graph convolutional networks with variance reduction[J]. arXiv: 1710.10568v3, 2018. DOI: 10.48550/arXiv.1710.10568.
(  0) 0) |
| [39] |
CHEN Jie, MA Tengfei, XIAO Cao. FastGCN: Fast learning with graph convolutional networks via importance sampling[J]. arXiv: 1801.10247, 2018. DOI: 10.48550/arXiv.1801.10247.
(  0) 0) |
| [40] |
YU Z. AS-GCN: Adaptive semantic architecture of graph convolutional networks for Text-Rich networks[C]. 2021 IEEE International Conference on Data Mining (ICDM). Auckland, New Zealand: IEEE, 2021: 837-846. DOI: 10.1109/ICDM51629.2021.00095.
(  0) 0) |
| [41] |
MIKOLOV T, CHEN Kai, CORRADO G, et al. Efficient estimation of word representations in vector space[J]. arXiv preprint arXiv: 1301.3781, 2013. DOI: 10.48550/arXiv.1301.3781.
(  0) 0) |
| [42] |
GILSON M K, LIU Tiqing, BAITALUK M, et al. BindingDB in 2015: A public database for medicinal chemistry, computational chemistry and systems pharmacology[J]. Nucleic Acids Research, 2016, 44(D1): D1045-D1053. DOI:10.1093/nar/gkv1072 (  0) 0) |
| [43] |
YE Qing, ZHANG Xiaolong, LIN Xiaoli. Drug-target interaction prediction via multiple classification strategies[J]. BMC Bioinformatics, 2022, 22(12): 461. DOI:10.1186/s12859-021-04366-3 (  0) 0) |
| [44] |
RAYHAN F, AHMED S, FARID D, et al. CFSBoost: Cumulative feature subspace boosting for drug-target interaction prediction[J]. Journal of Theoretical Biology, 2019, 464: 1-8. (  0) 0) |
| [45] |
ZHANG Yufang, WANG Xiangeng, KAUSHIK A C, et al. SPVec: A word2vec-inspired feature representation method for drug-target interaction prediction[J]. Frontiers in Chemistry, 2020, 7: 895. DOI:10.3389/fchem.2019.00895 (  0) 0) |
| [46] |
DAVID S. DrugBank: A knowledgebase for drugs, drug actions and drug targets[J]. Nucleic Acids Research, 2008, 36: 901-906. DOI:10.1093/nar/gkm958 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


