2. 南昌大学 食品科学与技术国家重点实验室,南昌 330047;
3. 南昌大学 食品学院,南昌 330047
2. State Key Laboratory of Food Science and Technology, Nanchang University, Nanchang 330047, China;
3. College of Food Science and Technology, Nanchang University, Nanchang 330047, China
抗体一般是由于外来物质进入机体后,机体的免疫系统在抗原刺激下识别并由免疫细胞分泌的免疫球蛋白分子,能特异性识别和结合抗原,消灭外来的有害物质。正是由于抗体的结合延展性,使其成为生物治疗药物的主要类别(10种畅销药物中的6种,市场价值1 000亿美元)[1]。近三十多年来,科学家将抗体应用于疾病的诊断和治疗,尤其在癌症治疗领域,取得了显著的成就。然而,抗体蛋白质本身很大,这使得输送抗体到靶点细胞很困难,特别是在一些特殊情况下,如肿瘤穿透。
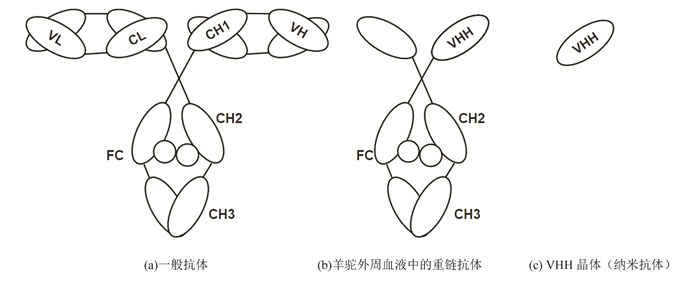
1993年,Hamers等[2]首次报道在骆驼科动物的血清中存在一种奇特的抗体。与传统抗体(图 1(a))相比,这种抗体天然缺失轻链,只由两条重链组成,且重链缺乏CH1区,称之为重链抗体(Heavy-chain antibodies,HCABs),如图 1(b)所示。由于缺乏CH1区,HCABs可以很快地从效应B细胞中分泌出去,及时发挥效应。由于不存在轻链,HCABs的抗原结合部位只有重链的可变区(Variable domain of the heavy chain,VHH)。而单独克隆并表达出来的VHH结构具有与原重链抗体相当的结构稳定性以及与抗原的结合活性。VHH晶体直径2.5 nm,长4 nm,分子量只有15 KDa,为传统抗体大小的十分之一,是已知的可结合目标抗原的最小单位,因此也被称作纳米抗体(Nanobody, Nb)[3],如图 1(c)所示。纳米抗体兼具传统抗体与小分子药物的优势,且具有比传统抗体更好的治疗特性,逐渐成为生物医药与临床诊断试剂中的新兴力量。
|
图 1 纳米抗体与其他抗体的对比 Figure 1 Comparison between nanobody and other antibodies |
作为一种特殊的蛋白质分子,纳米抗体的三维结构往往决定了纳米抗体与抗原的结合强度,即抗体亲和力,乃至对靶点的治疗效果。在进行纳米抗体的药物筛选和研发过程中,都需要参考大量现有的纳米抗体序列和其结构数据。尽管这部分数据目前常常以公开的形式存放在网络等公共领域,且数量也正在迅速增长,但其来源的异质性和缺乏标准化阻碍了纳米抗体信息的可靠收集,不利于进一步的产品研发。
为了有效整合当前已公开的纳米抗体的庞大数据,有必要建立用于免疫信息学的纳米抗体数据库。近期已经有美国和波兰的开发者创建了INDI纳米抗体集成数据库[4],该数据库收集了PDB[5], NCBI[6]等5个来源的纳米抗体序列数据库,并提供序列搜索, CDR3区段搜索和文本搜索功能。但其搜索结果基本是纳米抗体的序列信息,不直接包含抗体的结构信息,不利于针对抗原筛选或设计高亲和力的纳米抗体。
1.3 本文的工作和贡献本文提出一个包含结构信息的纳米抗体数据库系统NANO。该系统收集和存储了2 160条纳米抗体的序列,提取了所有纳米抗体序列的骨架区(Framework region,FR), 互补决定区(Complementarity determining region,CDR), 3D坐标数据这些结构信息,提供文本、序列、氨基酸分区等基本搜索,以及物种、亲和力和序列长度高级搜索等功能。可促进新型纳米抗体特异性计算方案的开发,帮助实现纳米抗体在医药, 食品等领域的进一步突破。
本文的主要工作和贡献如下:
1) 收集了INDI, PDB, NCBI和SAbDab-nano等各公共数据库的纳米抗体公开资料,经过严格地数据筛查与清洗,形成了比较全面的纳米抗体结构数据集。
2) 构建了基于MySQL的纳米抗体结构数据库,将数据集批处理导入数据库。
3) 开发了一个访问纳米抗体结构数据的Web系统,前后端交互采用MVC设计模式,利用Django框架,实现了系统的浏览、抗体检索等核心功能。
4) 实验比较了本文提出的NANO系统与INDI, PDB, SAbDab-nano和NCBI数据库在检索字段、检索功能覆盖和检索时间性能方面的表现。
2 相关工作 2.1 INDIINDI数据库是由名为Natural Antibody的公司创建的[4],该公司专注于收集, 生成和分析抗体数据,以实现新型生物治疗的端到端计算发现。INDI数据库整理了来自多种公共渠道的纳米抗体:专利、基因库、新一代测序存储库、结构库、科学出版物,并配备了强大的纳米抗体特异性序列, CDR3和文本搜索。但INDI数据库的搜索结果只包含纳米抗体的序列信息,不包含其结构信息。
2.2 PDB蛋白质数据库(Protein data bank,PDB)是通过X射线单晶衍射、核磁共振、电子衍射等实验手段确定蛋白质、多糖、核酸、病毒等生物大分子的三维结构数据库[5]。其内容包括生物大分子的原子坐标, 参考文献, 1级和2级结构信息, 晶体结构因数以及NMR实验数据等。PDB数据库以统一的格式化文件存储数据,每个文件对应一个特定的蛋白质结构,允许用户用各种方式以及布尔逻辑组合(AND, OR和NOT)进行检索,可检索的字段包括功能类别、PDB代码、名称、作者、空间群、分辨率、来源、入库时间、分子式、参考文献、生物来源等。
PDB数据库中的纳米抗体信息主要涉及以下几个方面:
1) 纳米抗体的结构:PDB数据库中收录了许多纳米抗体的晶体结构,包括单链变异型(Single-domain antibody,sdAb)、人类重链抗体(Human heavy chain antibody,HCAb)等。这些结构数据可以帮助研究者了解纳米抗体的三维结构, 构象变化等信息。
2) 纳米抗体与靶标的相互作用:PDB数据库中还收录了许多纳米抗体与其靶标(如病毒、细菌、癌细胞等)的复合物结构。这些数据可以帮助研究者了解纳米抗体与靶标之间的相互作用机制,为开发新型纳米抗体药物提供参考。
3) 纳米抗体在药物研发中的应用:PDB数据库中还收录了一些关于纳米抗体在药物研发中的应用案例。例如,一些针对癌细胞表面分子的纳米抗体已经被开发成为临床上使用的药物。这些数据可以帮助研究者了解纳米抗体在药物研发中的应用前景和挑战。
PDB数据库中的纳米抗体相关数据可以为研究者提供丰富的结构, 相互作用和应用信息。但PDB库不是专门针对纳米抗体的结构数据库,纳米抗体信息在PDB库中也是作为复合物分子的一部分,若要单独检索纳米抗体并不方便。
2.3 NCBINCBI是指美国国家生物技术信息中心(National center for biotechnology information,NCBI),是美国国立卫生研究院分馆美国国家医学图书馆(NLM)的一部分[6]。NCBI拥有GenBank和PubMed等一系列与生物技术和生物医学相关的数据库,以及用于分析包括蛋白质在内的生物分子的结构和功能的工具软件。
NCBI数据库中纳米抗体相关的信息非常丰富,包括了文献、序列和结构等方面的数据。
1) 通过NCBI数据库中的文献信息,可以了解到纳米抗体在各种领域中的应用情况,如生物医学、生物工程、食品安全等。同时,还可以了解到纳米抗体在不同物种中的表达情况和功能特点。
2) NCBI数据库中的序列信息可以帮助研究者进行基因克隆和表达等实验操作。此外,还可以通过比对序列来分析不同纳米抗体之间的差异和相似性,并进一步探究其结构与功能之间的关系。
3) NCBI数据库中的结构信息可以帮助研究者深入了解纳米抗体分子的三维结构及其与靶标分子之间的相互作用机制。这对于设计新型纳米抗体以及优化已有纳米抗体具有重要意义。
但NCBI不是单独针对纳米抗体的数据库,虽然从NCBI数据库可检索到纳米抗体的氨基酸序列、基因序列和测序序列和部分结构信息,但这些信息是分散的,没有以纳米抗体为中心关联起来。
2.4 SAbDab-nanoSAbDab由Oxford protein informatics group (OPIG)根据一项开放创新协议建立的公开的实验确定的抗体结构数据库,SAbDab-nano是SAbDab的一个纳米抗体(仅重链抗体)子数据库[7]。SAbDab-nano数据库提供以下功能:
1) 数据库收集:SAbDab-nano纳米抗体数据库收集了全球各地的纳米抗体结构信息,包括其PDB编号、序列、抗原等方面的信息。
2) 数据库搜索:用户可以通过多种方式检索纳米抗体结构信息, 使用PDB编号搜索特定条目;也可按属性搜索纳米抗体的子集,例如物种、实验方法, 特定位置的残基等。
3) 数据库浏览:SAbDab-nano纳米抗体数据库提供了一个直观的浏览界面,用户可以通过浏览器查看各种类型的纳米抗体结构信息。
4) 数据库分析:SAbDab-nano纳米抗体数据库还提供了一些分析工具,可以帮助用户分析和比较不同纳米抗体结构之间的差异和相似性。
但是,SAbDab-nano不支持查看纳米抗体的氨基酸序列,按序列搜索出来的子集也包含非纳米抗体,且不包含抗体的核酸序列和亲和力数据。
3 纳米抗体结构数据库系统 3.1 数据收集收集的纳米抗体结构信息主要来源于PDB数据库和INDI数据库,覆盖了绝大部分从科学文献和专利中收集的纳米抗体。构成纳米抗体结构数据库的数据集内容和具体数据收集策略如下:
1) 在INDI数据库下载了结构纳米抗体元数据,该数据包含了纳米抗体的ID, 分辨率和生物来源等一些基本信息。通过元数据中的ID字段得到纳米抗体的PDB编号,再到PDB数据库中使用搜索及下载功能获取纳米抗体PDB文件,得到纳米抗体的结构信息。
2) 除了通过INDI数据库的结构纳米抗体元数据获取纳米抗体的结构信息,还可以根据与纳米抗体相关的关键词收集相应的纳米抗体信息。目前收集了使用“nanobody”和“vhh”等关键词检索出的纳米抗体信息。通过使用PDB数据库提供的批量下载,多图像批量下载插件,收集了这些纳米抗体的PDB文件和3D视图。
采用上述收集策略收集的数据共有1 137条,跨越了PDB和INDI等数据来源。后续会进一步完善数据,主要的做法有配置PDB数据库同步、利用代码搜索出其中的纳米抗体记录、在数据库中导入实验预测生成的抗体序列和结构数据等。
3.2 数据处理PDB文件是蛋白质三维数据结构文件,完整的PDB文件会包含非常多的信息,比如分子类别、蛋白质的一级结构、二级结构信息、3D坐标信息等。将收集的PDB文件和3D视图全部上传至项目的static文件夹中,通过分析PDB文件结构从对应部分提取出需要的纳米抗体数据及结构信息。数据提取完毕后,通过一定的方法对数据进行清洗,将无效、错误的数据剔除,留下干净的数据。
3.2.1 数据提取需要搜集的纳米抗体数据字段有:检索号、分子名、物种来源、亲和力、氨基酸序列、序列长度、核酸序列、氨基酸分区、检索名、源库检索号、3D视图、3D坐标。为系统中的每一个纳米抗体结构数据设计了唯一的检索号(ID)。具体命名规则为:来源标识(1位字母)_PDB编号_结构链号_版本号(1位数字)。以来源于PDB数据库的纳米抗体分子9 043为例,PDB数据库存储了它与乳糖渗透酶及硫代半乳糖的复合物结构(编号6vbg)。该抗体有两条不同结构的链,在NANO系统中逐一被存储,检索号分别为“P_6vbg_C_1”和“P_6vbg_D_1”。“P”为来源标识,表示来源于PDB数据库。“6vbg”为该纳米抗体分子在PDB中的编号。“C”和“D”为该分子两条结构链的链号。“1”表示版本号。分子名是PDB文件中COMPND MOLECULE字段后的信息,每条链有对应的分子名,因此我们在收集分子名数据时,要在PDB文件中找纳米抗体氨基酸链对应的分子名。PDB文件的SOURCE字段后是化合物来源信息,SOURCE 2后面记录的就是该蛋白质的所属物种。
亲和力是指单个生物分子与其配体/结合配偶体间相互结合的强度,一般通过平衡解离常数(KD)进行测量和报告,KD值越小,配体对于其目标的结合亲和力就越大[8]。本文中纳米抗体的亲和力是指抗原-抗体之间结合的强度。纳米抗体有较长的CDR3区,能形成暴露的凸环状结构,可以识别隐藏的表位位点,因此具有高亲和力的特点。NANO数据库收集的纳米抗体亲和力数据均来自于MOAD数据库[9](http://www.bindingmoad.org)。该数据库是RCSB数据库的子集,专门收录蛋白质-配体晶体结构数据信息,同时提供由实验而得的亲和力数据。我们从MOAD数据库中获取纳米抗体-抗原的亲和力数据,若没有找到相关记录,就在NANO系统中将该抗体的亲和力表示为0。
氨基酸是含有碱性氨基和酸性羧基的有机化合物,氨基酸序列是氨基酸相互连接形成的肽链顺序,每一条链的氨基酸序列以氨基酸名称全称的形式记录在PDB文件的SEQRES标题后,将提取出的氨基酸序列转换成对应的缩写形式,如GLY甘氨酸转换成G,未知的氨基酸用X代替。序列长度是氨基酸序列的长度。
核酸序列是DNA或RNA中碱基的排列顺序。根据中心法则我们可以知道,核酸序列作为遗传信息的载体,核酸序列中的遗传信息被转录成为信使RNA(mRNA),转运RNA(tRNA)通过自身的反密码子识别mRNA上的密码子,将该密码子对应的氨基酸转运至核糖体(含rRNA)翻译成氨基酸序列[10],氨基酸序列再通过脱水缩合得到蛋白质。由于PDB文件中没有核酸序列的信息,因此我们需要通过氨基酸序列反推得到核酸序列。使用欧洲生物信息研究EMBL-EBI所提供的反向序列翻译工具EMBOSS Backtranseq进行序列反推(https://www.ebi.ac.uk/Tools/st/emboss_backtranseq)。该工具将蛋白质序列按照氨基酸表进行反向翻译,将每个氨基酸翻译成对应的密码子。由于存在密码子的多义性,可能会出现多种可能的DNA或RNA序列。Backtranseq会生成所有可能的序列,并按照用户指定的密码子使用偏好进行排序,选择最优的核酸序列返回[11]。将所有的氨基酸序列整理到一个文件中,选择物种Escherichia coli K12(大肠杆菌K12)的密码子使用偏好,利用Backtranseq的开发接口将氨基酸序列批量转成核酸序列。
氨基酸分区记录的是蛋白质的结构信息。纳米抗体有CDR1, CDR2和CDR3三个高变区和FRl、FR2, FR3和FR4四个骨架区。可变区指抗体分子的轻链或重链中靠近N端氨基酸序列变化较大的区域,可分为高变区(Hypervariable region,HVR)和骨架区FR。高变区氨基酸残基的组成和排列次序比可变区内其他区域更易变化,由于序列与抗原决定簇互补,故高变区又称为互补决定区CDR,其中CDR3最为高变。骨架区FR的可变性要低于CDR。CDR区和FR区的起止位置在不同的编号方案中有不同的定义。采用了IMGT编号方案[12],如表 1所示,截取氨基酸序列相应的部分分别存到对应的字段中。
| 表 1 IMGT方案中CDR区和FR区的定义 Table 1 Definition of FR and CDR regions in IMGT scheme |
检索名是纳米抗体用于数据库搜索的关键词,在PDB文件的KEYWDS标签后面,运用BioPython建立一个PDBParser对象,运用PDBParser对象解析PDB文件,产生Structure对象,Structure对象有个属性叫header,它可以将头记录映射到其相应值的Python字典,运用这个字典中的关键字keywords提取出纳米抗体的检索名。源库检索号是我们获取纳米抗体信息的来源,通过点击源库检索号可链接到其他数据库。我们在收集数据时就收集好了每个纳米抗体的3D图像,于是将3D图像在项目中的存放路径存在数据库中,当需要查看纳米抗体的3D图时,直接访问相应路径下的图像。
3D坐标数据在PDB文件的ATOM标签之后,有X轴、Y轴、Z轴的数据,将每一条链的XYZ坐标分别存在三个文本文件中,有多种结构的链坐标不相同,在文本文件中标明出坐标信息属于第几个结构(MODEL n)。将每条链的坐标文件上传至项目的static文件夹中,在数据库中记录每个文件的路径,若需要访问纳米抗体的3D坐标信息,直接读取相应路径下的文件进行展示。提取出所有数据字段之后,将其保存在一个csv文件中,总计5 131条。
3.2.2 数据清洗由于在收集数据时,通过关键词Nanobody和VHH在PDB数据库搜索获得的蛋白质不一定属于纳米抗体,因此在数据清洗部分将不需要的数据删除。INDI数据库提供了关于纳米抗体信息文件下载的功能,Structural nanobody metadata的文件中包含所有纳米抗体在INDI数据库中的编号,将该文件中的编号提取出来,再从中提取出PDB编号。根据这些PDB编号在INDI数据库中搜索这个PDB编号对应的基因哪几条链属于纳米抗体,将不属于纳米抗体的链删除,如果没有搜索结果,就查看相应的PDB文件,如果COMPND MOLECULE标签后的分子名包含NANOBODY, NB等标识纳米抗体链的信息,就保留数据,否则将其删除。
由于通过PDB文件直接提取出的氨基酸序列含有非纳米抗体序列的片段,因此还需要对序列进行清洗,只保留类似“QVQL......TVSS”这种纳米抗体序列,同时要对3D坐标数据进行清理,只保留对应序列部分的坐标。经过数据清洗,删除了不属于纳米抗体的数据,得到了干净的数据,最终有2 160条纳米抗体序列结构信息。
3.3 数据库结构设计将处理后的纳米抗体数据存储在MySQL数据库中,这些数据均为静态数据,基本不会变动,且均为关系型数据。MySQL关系型数据库管理系统的存储速度快、灵活性高,为了便于后续的增删改查,将纳米抗体数据存入MySQL数据库。根据需求分析,数据库中的实体主要如表 2。
| 表 2 静态数据库实体 Table 2 Static database entities |
每个纳米抗体序列包含一至多条链,但是其检索号可以区分每一个纳米抗体下面的具体链,因此将检索号作为主键,将每一条链的信息存储在App01_chain表中。部分纳米抗体的亲和力数据可能存在缺失,FR1-CDR3的结构信息部分不存在。因此这些列设置允许为空。
数据库字段名和具体含义的对应关系如表 3, 数据库模型设计图如图 2所示。
| 表 3 字段含义说明 Table 3 Description of data fields |

|
图 2 数据库模型设计图 Figure 2 Database model diagram |
纳米抗体结构数据库系统NANO提供Web访问界面。系统部署在腾讯云服务器,系统开发采用Python+Django +MySQL的技术方案。用户可通过http://124.221.201.63:8000/index访问数据库系统,执行纳米抗体结构的浏览与检索功能。系统前端采用Layui+Bootstrap框架,进行合理布局与设计,提供给用户最直观、舒适的体验。后端开发负责核心功能的实现,如数据浏览、文本搜索、序列搜索, 氨基酸分区搜索以及高级搜索功能,在功能实现的基础上保证了时间效率。
NANO系统的优势在于可直接搜索和呈现纳米抗体结构信息,结构信息体现在CDR, FDR, 亲和力等方面。CDR区是抗体识别与抗原结合的区域,直接决定抗体的特异性,重链CDR3区域通常被认为对抗原抗体结合起关键作用,因此在没有其他信息的情况下,人们习惯优先选择对抗体CDR3区域进行随机突变。骨架区FR的主要作用是稳定CDR区的空间构型,以利于抗体CDR与抗原决定簇间的精细结合。抗体亲和力体现了一个抗体分子和抗原分子或半抗原的一个决定簇起反应的能力,是评价抗体质量的重要指标,反应了抗体与相应抗原之间的结合力度,亲和力越高,抗体与相应的抗原之间的结合程度越高,对抗体的应用具有重要的指导作用。纳米抗体的结构信息对于设计具有高亲和力的纳米抗体具有重大意义,因此本系统在数据浏览部分直观地展示了纳米抗体结构信息,并且提供了结构信息的搜索功能。
3.4.1 数据浏览本系统提供了数据浏览功能,以便于将整个数据库中的所有纳米抗体结构信息直观地呈现给用户。用户点击“详情”可查看该纳米抗体序列的详细信息,点击“查看”可查看该纳米抗体序列的3D视图,点击“跳转”可跳转至数据来源PDB。每个纳米抗体序列包含一至多条链,且序列数据较为庞大,对搜索效率具有挑战性。这里选择在浏览整个数据库时只展现序列的部分重要信息字段,如名称, CDR, FR, 亲和力等,在详情页面,利用MySQL数据库的全文索引,提高检索速度。数据浏览功能的界面截图如图 3所示。

|
图 3 NANO数据库系统的数据浏览功能 Figure 3 Data browsing function of NANO database system |
由于纳米抗体序列与其丰富的属性信息相关,为方便用户检索,本系统提供了全站搜索功能,将用户输入的字符串与纳米抗体的所有信息匹配,展示匹配到的纳米抗体,来方便与数据库中的数据进行交互。然而,数据来自于INDI, PDB, NCBI等不同的数据库,为解决不同来源的信息多样性,对数据进行了统一的处理,提取同一组字段集合,与用户输入的关键字进行匹配,例如输入某个序列的检索号字段1BZQ,搜索结果将展示所有字段包含1BZQ的纳米抗体信息。
为提升搜索效率,同样采用全文索引进行搜索。首先,创建app01_chain的全文索引,由于全文索引需要索引字段是char, varchar, text等文本类型,而app01_chain表中的字段的类型都是varchar,所以所有字段均符合创建全文索引的要求。考虑到文本搜索功能需求是搜索所有字段中包含相似文本的纳米抗体数据,因此本文将所有字段都建立了全文索引。其次,将数据库配置文件my.cnf中的最小查询字符串的长度设为3。然后,在使用全文索引进行查找时,有两种检索模式:自然语言全文检索和布尔全文检索。考虑到自然语言全文检索中存在停用词,且检索方式更适用于自然语言处理,而本文的检索主要是序列、抗体名称、抗体编号等,这些都不属于自然语言,因此在使用全文搜索时,主要采用布尔全文搜索模式。最后,结果显示为一个交互式表格,列出了3D视图查看、详情信息查看、源链接跳转按钮和文本字段。用户可以点击查看详细信息,并在详情页面选择是否下载文本信息。
3.4.3 序列搜索该功能用于搜索氨基酸序列中包含局部相似序列的纳米抗体。首先对用户输入的序列进行文本预处理,去除空格字符,并做大小写转化。接着创建好氨基酸序列字段proch的全文索引,然后采用布尔全文搜索模式,例如搜索包含“QVQLVESGGGSVQAGGSLRLSCAA”这个氨基酸序列的纳米抗体信息,可以使用以下SQL语句:
SELECT * FROM app01_chain WHERE MATCH (proch) AGAINST (‘QVQLVESGGGSVQAGGSLRLSCAA’ IN BOOLEAN MODE);
最后,将查询到的信息返回给前端页面,以交互表格的形式展现。若用户已确认输入的关键字内容是氨基酸序列,可以使用该功能,相较于全站文本搜索,它的搜索范围更小,速度更快且结果更加准确。
3.4.4 氨基酸分区搜索该功能支持根据氨基酸分区进行特异性搜索,搜索氨基酸分区(FR,CDR)中包含相似序列的纳米抗体结构。利用MySQL全文检索搜索出匹配结果,结果显示在一个交互式的表格中,该表格允许用户浏览结果,并可跟踪到序列的详情页面。
3.4.5 高级搜索该功能用于搜索指定物种、亲和力、序列长度的纳米抗体。用户输入上述三种信息后,系统将采用模糊匹配的方式,将符合条件的纳米抗体结果以表格的形式呈现,用户可选择查看任一序列的详情页面。
4 实验分析为了检验本文提出的纳米抗体结构数据库NANO的功能和性能,使用NANO进行了数据检索实验,并将实验结果与INDI, PDB, NCBII, SAbDab-nano等数据库系统进行了比较。
4.1 检索字段INDI数据库有超过1 140 894条的纳米抗体信息,通过PDB号搜索纳米抗体,会显示出所有属于该纳米抗体的链的信息,点击单条链,可以查看纳米抗体的结构文档元数据,包含PDB编号、标题、作者、日期、分辨率、物种来源、实验技术、链的基本信息,还可以查看到纳米抗体的序列信息,包含氨基酸序列、CDR区、分配生殖系。本项目的数据暂时只有两千多条,今后还需继续完善。相比数据内容而言,比INDI数据库缺少了部分结构文档元数据的信息,多了结构信息等,如分子名、亲和力、核酸序列、FR区、检索名、源库检索号,3D视图及3D坐标数据,有利于进行纳米抗体的结构分析。
PDB是最主要的收集生物大分子(蛋白质、核酸和糖)2.5维(以二维的形式表示三维的数据)结构的数据库,它不止包含纳米抗体数据。通过PDB号来搜索纳米抗体,会显示3D图、很详细的PDB文档数据信息、高分子含量、大分子数据如蛋白质特征视图。PDB数据库不只是纳米抗体数据库,在数据展示方面不只是展示纳米抗体的信息,本研究的数据相比PDB数据库而言,多了分子名、亲和力、核酸序列、氨基酸分区、3D坐标信息。
NCBI通过PDB号搜索纳米抗体,展示了纳米抗体的结构文档元数据、结构信息、可以查看该PDB号对应的蛋白质所有氨基酸序列的信息并提供了数据下载。本研究的数据库在序列数据展示部分只展示了纳米抗体链的序列,并且对序列进行清洗,只保留属于纳米抗体氨基酸序列部分。数据相比NCBI数据库而言,多了分子号、核酸序列、氨基酸分区、检索名、3D坐标数据。
SAbDab-nano数据库是SabDab数据库的一个子数据库,可以通过多种方式进行检索,其搜索结果字段包括PDB编号(源库检索号)、抗体结构的链标识符、物种来源、结构分辨率、实验方法、配体、互补决定区CDR1,CDR2,CDR3。本研究的数据库相比SAbDab-nano数据库而言,没有结构链标识符、结构分辨率、配体信息,但有分子名、核酸序列、抗体骨架区(FR)、检索名、3D坐标数据。
NANO系统返回的检索结果与其他数据库系统的字段对比如表 4所示。
| 表 4 NANO与其他数据库系统检索结果的字段对比 Table 4 Comparison among NANO and the other database systems in data fields of search results |
INDI数据库提供了三种搜索功能,即可变区序列搜索, CDRH3搜索和文本搜索。相比于INDI,本文数据库的所有搜索功能不只是展示最优匹配结果,而是展示所有匹配到的相似结果,因此更有利于命中用户的搜索目标。其次,NANO提供氨基酸分区搜索功能,而本文数据库不仅可搜索CDRH3区的序列,还提供了FR区域的序列搜索。
PDB数据库和本文数据库都提供了文本搜索、序列搜索、高级搜索,而由于两个数据库设计的侧重点不同,在功能上,PDB提供了化学相似性搜索功能,而本文未提供;本文数据库配备了氨基酸分区搜索功能,而PDB未提供。
NCBI的检索功能包括文本搜索、序列搜索, 指定搜索字段的高级搜索和Entrez浏览检索(搜索与NCBI链接的基因序列数据库的分子生物数据和书目文献资料)等检索方法,但不提供氨基酸分区搜索功能。
SAbDab-nano数据库提供了文本搜索、CDR区搜索、氨基酸序列搜索, PDB代码搜索和属性搜索,由于NANO数据库和SAbDab-nano数据库设计的侧重点不同,SAbDab-nano存储的许多信息与实验相关,因此会提供相应的实验信息搜索,而本文系统未提供。另外,SAbDab-nano提供的PDB编号搜索相当于根据检索号搜索,而在本文数据库中,使用文本搜索可以根据检索号进行检索。其次,SAbDab-nano与本文数据库的文本搜索功能不完全相同,SAbDab-nano的文本搜索是对输入的关键词或关键短语进行检索,例如"novel coronavirus"或"antigen binding",而本文数据库的文本搜索不限于对关键词搜索,而是可以对每个字段进行搜索;SAbDab-nano数据库的属性搜索功能与本文的高级搜索功能较为相似,但搜索的字段有差别,SAbDab-nano的属性搜索需要输入物种、实验方法、分辨率等,本文的高级搜索需要输入物种、亲和力、序列长度。最后,本文数据库配备了FR分区搜索功能,而SAbDab-nano未提供。
本文数据库与其他四种数据库的功能对比如表 5所示,可以看出,本文数据库在检索功能上覆盖较广,既有广泛所需的搜索方式,也有自己独特的检索功能,后续也将参考其他数据库特有的检索方式,丰富系统功能。
| 表 5 NANO与其他数据库系统的功能对比 |
本文的实验均由实验室PC机完成,具体的软硬件配置如表 6所示。
| 表 6 硬件配置和软件环境 Table 6 Hardware configuration and software environment |
用于测试数据搜索功能的测试用例如表 7所示。
| 表 7 数据检索测试用例 Table 7 Test cases of data retrieval |
实验使用Chrome浏览器,首先对上述8个测试用例进行测试,主要的对比内容包括搜索时间和搜索结果。具体的比较结果如表 8和图 4所示,时间单位为秒。
| 表 8 五个数据库系统在8个测试用例上的时间效率结果 Table 8 Time efficiency results of 5 database systems in 8 test cases |
|
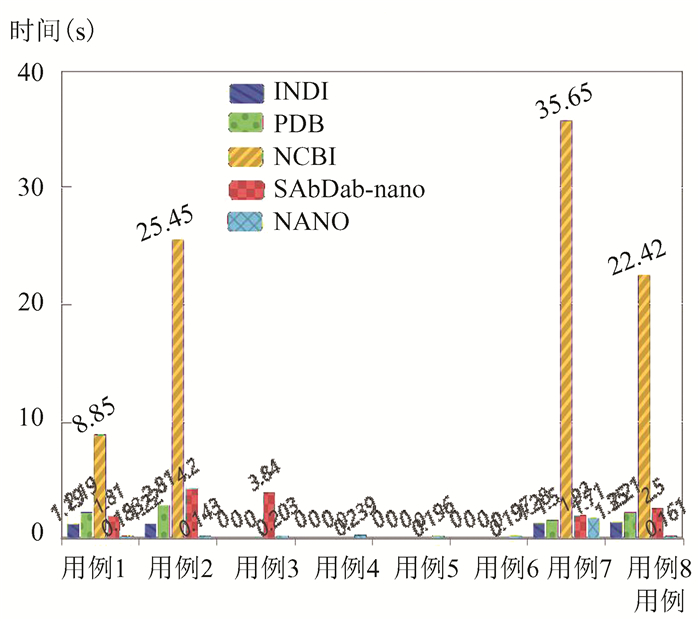
图 4 NANO与其他数据库系统的全文搜索时间比较 Figure 4 Comparison among NANO and the other database systems in full-text search time |
从实验结果可以看出,NCBI数据库搜索所用的平均时间是五个数据库中最长的,PDB和SAbDab-nano次之,NANO平均所用时间最短。本文数据库在搜索效率上占非常大的优势,除了第7条用例以外,本文数据库搜索耗时均为四个数据库中最短的,且对于多种不同类型的字段信息,均能够准确匹配到结果。INDI,PDB,NCBI数据库在用例3, 4, 5, 6无搜索结果,其原因可能是全站文本搜索功能中不包括氨基酸序列的搜索和对CDRH3区的搜索,或者数据库的存储字段未覆盖CDRH3。SAbDab-nano数据库对于第4, 5, 6条用例无搜索结果,可能是数据库中未存储该纳米抗体的信息。
综上所述,相较于其他四种数据库,本文数据库在时间效率、功能多样性方面均占优势,但数据的存储量没有其他三大数据库庞大,在后续会逐渐增加数据,并扩大功能覆盖范围。
5 结论与未来工作随着对纳米抗体的研究越来越深入,纳米抗体不仅能在医疗方面以及生物信息方面发挥极大的作用,在越来越多的新兴领域也展示出它极高的应用价值[13]。本文提出了一个纳米抗体结构数据库系统NANO。该系统收集和存储了2 160条纳米抗体的序列和结构信息,提供文本、序列、氨基酸分区等基本搜索以及物种、亲和力和序列长度等高级搜索功能。与INDI, PDB, NCBI, SAbDab-nano等现有数据库系统的比对实验表明,NANO系统在检索字段, 检索功能覆盖和检索时间性能方面均表现出色。
本课题组已在生物纳米抗体多样性及抗黄曲霉毒素纳米抗体进化方面进行了相关研究,利用高通量测序技术总结了该抗体重链可变区VHH的关键特征。接下来我们将把NANO系统应用于该抗体的编辑和制备,以提高其亲和力。并且,将导入更多数据源的纳米抗体序列数据和可靠的结构预测数据,以更通用的分子三维结构格式呈现纳米抗体的结构,并提供数据源更新和实验录入功能,以增强NANO纳米抗体结构数据库系统的实用性。
| [1] |
秦秀峰. 全合成纳米抗体文库的设计、构建及鉴定[D]. 天津: 天津大学, 2017. QIN Xiufeng. The design, construction and identification of a fully synthetic nanobody library[D]. Tianjin : Tianjin University, 2017. (  0) 0) |
| [2] |
HAMERS-CASTERMAN C, ATARHOUCH T, MUYLDERMAN S, et al. Naturally occurring antibodies devoid of light chains[J]. Nature, 1993, 363(6428): 446-448. DOI:10.1038/363446a0 (  0) 0) |
| [3] |
李若微. 核糖体展示天然纳米抗体文库筛选HIF-1α单域重链抗体的应用基础研究[D]. 天津: 天津大学, 2018. DOI: 10.27356/d.cnki.gtjdu.2018.000063. LI Ruowei. An applied basic research of the ribsome-displayed non-immunised nanobody library used for selection of single domain heavy chain antibodies targeting HIF-1α[D]. Tianjin : Tianjin University, 2018. DOI: 10.27356/d.cnki.gtjdu.2018.000063. (  0) 0) |
| [4] |
DESZYNSKI P, MLOKOSIEWICZ J, VOLANAKIS A, et al. INDI-integrated nanobody database for immunoinformatics[J]. Nucleic Acids Research, 2022, 50(D1): D1273-D1281. DOI:10.1093/nar/gkab1021 (  0) 0) |
| [5] |
BURLEY S K, BERMAN H M, BHIKADIYA C, et al. RCSB Protein Data Bank: biological macromolecular structures enabling research and education in fundamental biology, biomedicine, biotechnology and energy[J]. Nucleic Acids Research, 2019, 47(D1): D464-D474. DOI:10.1093/nar/gky1004 (  0) 0) |
| [6] |
SAYERS E W, BOLTON E E, BRISTER J R, et al. Database resources of the national center for biotechnology information in 2023[J]. Nucleic Acids Research, 2023, 51(D1): D29-D38. DOI:10.1093/nar/gkac1032 (  0) 0) |
| [7] |
SCHNEIDER C, RAYBOULD M I J, DEANE C M. SAbDab in the age of biotherapeutics: updates including SAbDab-nano, the nanobody structure tracker[J]. Nucleic Acids Research, 2022, 50(D1): D1368-D1372. DOI:10.1093/nar/gkab1050 (  0) 0) |
| [8] |
AZIMZADEH A, VAN REGENMORTEL M H. Antibody affinity measurements[J]. Journal of Molecular Recognition, 1990, 3(3): 108-116. DOI:10.1002/jmr.300030304 (  0) 0) |
| [9] |
SMITH R D, CLARK J J, AHMED A, et al. Updates to Binding MOAD (Mother of All Databases): Polypharmacology Tools and Their Utility in Drug Repurposing[J]. Journal of Molecular Biology, 2019, 431(13): 2423-2433. DOI:10.1016/j.jmb.2019.05.024 (  0) 0) |
| [10] |
陈霄. DNA遗传算法及应用研究[D]. 杭州: 浙江大学, 2010. CHEN Xiao. Research on DNA genetic algorithms and applications[D]. Hangzhou: Zhejiang University, 2010. (  0) 0) |
| [11] |
MADEIRA F, PEARCE M, TIVEY A R N, et al. Search and sequence analysis tools services from EMBL-EBI in 2022[J]. Nucleic Acids Research, 2022, 50(W1): W276-W279. DOI:10.1093/nar/gkac240 (  0) 0) |
| [12] |
BARKER D J, MACCARI G, GEORGIOU X, et al. The IPD-IMGT/HLA Database[J]. Nucleic Acids Research, 2023, 51(D1): D1053-D1060. DOI:10.1093/nar/gkac1011 (  0) 0) |
| [13] |
孙山, 谭星, 庞晓燕, 等. 纳米抗体技术应用的最新进展[J]. 生物工程学报, 2022, 38(3): 855-867. SUN Shan, TAN Xing, PANG Xiaoyan, et al. Recent advances in the application of nanobody technology: a review[J]. Chinese Journal of Biotechnology, 2022, 38(3): 855-867. DOI:10.13345/j.cjb.210464 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


