据WHO最新统计,2020年全球肝癌新增病例约90万例,被认为是第七种常见的癌症和第三种致命的癌症,其中,肝细胞癌(Hepatocellular carcinoma,HCC)占所有原发性肝脏恶性肿瘤的75%,且在亚洲和非洲死亡率最高[1]。HCC主要风险因素包括乙肝病毒(Hepatitis B virus,HBV)或丙型肝炎病毒(Hepatitis C virus,HCV)感染以及长期饮酒、糖尿病或肥胖相关的非酒精性脂肪性肝炎,其中HBV感染导致了约50%的HCC,是最为突出的风险因素[2]。尽管如此,HCC的发病机制仍不完全明晰。
既往HCC相关生物信息学研究发现,在HCC中许多基因均可作为枢纽基因在HCC发生发展过程中发挥作用,如李琳等[3]发现BUB1, CDC20, KIF20A, RACGAP1和CEP55可能参与肝细胞癌的致癌, 侵袭或复发。孙维华等[4]发现在伴HBV感染的HCC中,SHARPIN可能作为枢纽基因促进细胞迁移。丁庆林等[5]发现CFHR4与HCC预后不良和免疫浸润水平显著相关。但这些生物信息学研究均未排除HBV对差异基因表达的影响,在HCC中,HBV相关的差异基因可能与其他因素(如酒精暴露、糖尿病及非酒精性脂肪肝炎等)相关的差异基因有所不同。为探讨HBV与其他因素导致HCC的共同机制并挖掘共有差异基因,将HBV相关的HCC与其他因素导致的HCC单独进行差异基因分析是有必要的。
综上,为了排除HBV可能对差异基因表达分析产生的干扰,本研究首先利用生物信息学手段将慢性HBV感染(Chronic hepatitis B,CHB)相关HCC患者与非HBV相关HCC两组患者中,癌组织和癌旁组织的转录组测序数据分别进行差异基因表达分析,并对共有差异基因进行了功能富集分析、蛋白质互作网络分析和生存分析等,以挖掘HCC发生发展和预后相关的共性标志分子。随后对筛选到的候选基因进行生物学实验验证,进一步解释候选基因在HCC发生发展过程中发挥的作用。这些工作,有望体外筛选鉴定出与HCC发生发展相关的生物标志物,为HCC的诊断, 预后和靶向治疗提供新线索。
1 材料与方法 1.1 数据来源及预处理数据来源于癌症基因组图谱(The cancer genome atlas,TCGA,https://www.cancer.gov/tcga)数据库中的肝细胞癌数据(Liver hepatocellular Carcinoma,LIHC),包含377例HCC患者肝癌组织样本和50例癌旁组织样本全基因转录组测序数据及患者临床信息。该数据采用R语言的RTCGA软件包下载,过滤缺失值后对数据进行标准化及log2转换。把377例HCC组织样本按照是否存在HBV感染分为CHB_HCC组(慢性HBV感染,270例)和Not_CHB_HCC组(非HBV感染,107例),将50个癌旁组织样本中的7例感染HBV的样本剔除后,得到样本数为43,作为对照组。
1.2 差异基因表达分析采用R语言中的Limma软件包进行差异基因表达分析。比较两组HCC组织样本与癌旁组织样本基因表达,|logFC|大于mean(abs(logFC)) + 2*sd(abs(logFC))且校正后的p值小于0.05的基因被定义为差异表达基因。
1.3 基因富集分析采用R语言中的ReactomePA软件包进行京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)信号通路的基因集富集分析(Gene set enrichment analysis, GSEA)。
1.4 PPI网络分析采用STRING数据库进行PPI网络分析。将富集信号通路中的差异基因导入STRING数据库,物种选择为“Homo sapiens”,得到基因相互作用数据,随后将数据导入CytoScape软件,并利用CytoNCA软件包对各基因的Betweenness centrality(BC)值进行计算,按BC值对代表基因的圆形的大小和颜色进行排序,将BC值前9的基因定义为hub基因。
1.5 生存分析采用网页工具GSCA(http://bioinfo.life.hust.edu.cn/GSCA/#)[6]对TCGA肝癌数据中的hub基因进行生存分析,包括无疾病间隔生存期(Disease free interval,DFI)、疾病特异性生存期(Disease specific survival,DSS)、总生存期(Overall survival,OS)和无进展生存期(Progression free survival,PFS),将P < 0.05的基因定义为对生存期有显著影响的基因。
1.6 临床组织样本及细胞系经湖北工业大学中德生物医学中心伦理委员会批准,获得每位医疗参与者的书面同意,并遵循《赫尔辛基宣言》的伦理标准进行研究[7],采用的所有HCC组织样本(10个HCC癌组织与对应的10个癌旁组织)来自武汉大学中南医院肝胆疾病研究院肝脏移植手术标本,HepG2细胞系来源于本实验室。
1.7 细胞和组织RNA提取、反转录及qRT-PCR细胞RNA提取:取长满10 cm培养皿的细胞置于生物安全柜中,PBS清洗3次,吸弃PBS,加入TRIZOL Reagent 700 μL,室温孵育5 min,加入1/5体积的氯仿剧烈颠倒15 sec,室温孵育2 min,4 ℃,12 000 rpm,离心15 min。吸取上层含有RNA的水相入新管,加入等体积异丙醇沉淀RNA,室温孵育10 min,4 ℃,12 000 rpm,离心10 min。弃上清,加入1 mL 75%的乙醇洗涤RNA沉淀,轻弹沉淀,使其浮在酒精中,静置1~2 min,让75%的乙醇充分接触沉淀,充分溶解有机试剂,然后4 ℃离心5 min。轻轻吸弃上清。4 ℃,7 500 rpm,离心5 min。重复步骤清洗5次,前4次用75%乙醇洗,最后一次用无水乙醇洗。敞口放置5~10 min至酒精全部挥发,用20 μL DEPC水溶解RNA。用紫外分光光度计测量RNA含量,-20 ℃保存。
组织样本RNA提取:取临床样本各30 mg,液氮研磨,加入TRIZOL Reagent 700 μL,随后步骤与细胞RNA提取一致。
1) 反转录:取上述提取的RNA 1 μg,按照诺唯赞公司提供的HiScript Ⅲ 1st Strand cDNA Synthesis Kit说明书进行RNA反转录,得到cDNA。
2) qRT-PCR:将上述cDNA 1 μL,SYBR Green(G441-02,诺唯赞,南京) 10 μL,H2O 6 μL,正反引物各1μL加入八联管中,随后将八连管置于qRT-PCR仪(Roche lightCycler 96)中,采用三步扩增法扩增cDNA检测荧光信号。变性温度95 ℃,10 s,退火温度60 ℃,30 s,延伸温度72 ℃,20 s,共扩增40个循环。本文中所用引物序列如下:CDKN2A Forward Primer: GTGCTCACTCCAGAAAACTC; CDKN2A Reverse Primer: AATGTCCTGCCTTTTAACGTAG; ACTB Forward Primer: CCTGGCACCCAGCACAAT; ACTB Reverse Primer: GGGCCGGACTCGTCATAC。
1.8 Western Blot1) 蛋白提取:取液氮研磨后的30 mg临床组织样本或PBS清洗三次后的细胞,向其中加入700 μLNP40(已加入蛋白酶体抑制剂),于冰上裂解30 min,吹打均匀后,4 ℃,12 000 rpm离心15 min,取上清,得到蛋白溶液,取10 μL用于浓度测定,余下溶液中加入1/4体积的SDS loading,100 ℃煮样10 min,-80 ℃保存。将10 μL蛋白溶液稀释适当比例后,使用BSA蛋白浓度测定试剂盒(G3522-1,捷倍斯,广州)进行浓度测定。
2) 电泳:制备12%的下层分离胶与3%的上层浓缩胶,分别加入到垂直电泳槽中,待凝固后加入样品(40 μg蛋白/孔),80 V电压下运行35 min,待蛋白条带被压平后,150 V电压运行45 min。将分离胶从目的蛋白条带处切开,转移到湿转的转膜滤纸上,裁剪合适大小的PVDF膜(甲醇活化30 s)覆盖于分离胶上,合上转膜板,将转膜板放入转膜槽中,向转膜槽中倒入湿转转膜液,200 mA电流转膜80 min。转膜后的PVDF膜置于抗体孵育盒中,加入5%脱脂牛奶封闭2 h,随后加入相应的p16(sc-1661,Santa cruz biotechnology,USA)或p14Arf(74560,Cell signaling technology,USA)一抗,4 ℃孵育过夜。回收一抗,TBST清洗PVDF膜3×10 min,加入二抗,孵育2 h。TBST清洗3×10 min,将PVDF膜置于C-DiGit数码化学发光扫描仪(Li-cor,USA),每张膜上加入现配的ECL reagent(佰欧乐基,武汉)100 μL,显影。管家基因GAPDH作为内部对照用于数据校正。
1.9 p16Ink4a在HepG2细胞系中的瞬时转染选择对数生长期的HepG2细胞,吸弃培养基,更换新鲜完全培养基。将过表达CDKN2A的p16Ink4a质粒(PLVX载体,北京擎科生物科技有限公司)10 μL加入无血清DMEM中,混匀后加入10 μL Neofect转染试剂(北京玛因科技有限公司),静置20 min,将混匀后的溶液均匀加入HepG2细胞培养皿中,转染48 h得到HepG2_p16细胞后进行后续实验。
1.10 CCK8增殖试验使用上海Biosharp公司的CCK8试剂进行细胞增殖检测。选取对数生长期的HepG2_p16或HepG2细胞,接种于96孔板(2 000个细胞/孔)中,培养体系为100 μL完全培养基。分别在37 ℃培养24,48,72 h时,加入CCK8 10 μL,37 ℃孵育1 h后,使用酶标仪在450 nm处检测吸光度值。
1.11 划痕实验选取对数生长期的HepG2_p16或HepG2细胞,接种7×105个细胞于六孔板中,每组三个复孔,37 ℃过夜培养。观察细胞密度于90%时,使用200 μL枪头在直径和距离直径1cm处平行划痕三道,37 ℃培养0,6,12,24,48 h在显微镜观察并拍照,计算细胞迁移率。细胞迁移率= (0 h划痕宽度—N h培养后划痕宽度)/ 0 h划痕宽度× 100%,N在本实验中分别代表 6,12,24,48,72 h。
1.12 绘图火山图,维恩图及GSEA富集分析图采用R语言中的ggplot2,biomaRt,enrichplot软件包进行绘图;PPI图由Cytoscape绘制,统计分析图由GraphPad Prism 8绘制。
1.13 统计分析采用SPSS 23.0处理数据,计量数据以x±s表示。多组间比较采用Paired-t检验得到p值(pvalue, P),同时,生物信息学部分的多重检验使用Bonferroni-Holm法校正后得到校正后的p值(p.adjust)。P小于0.05或P和p.adjust都小于0.05则认为P < 0.05,表示差异有统计学意义。
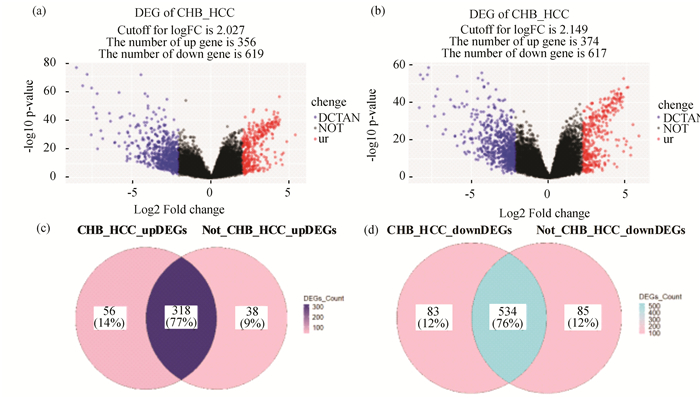
2 结果 2.1 HCC与癌旁组织差异基因比较分析本研究将TCGA数据库中HCC转录组数据进行挖掘,提取出377例患者的转录组表达矩阵与对应的临床信息(包含临床分期、患者年龄、乙肝抗原抗体检测结果等),并将患者分为CHB_HCC组(107例有HBV感染的癌组织样本和部分对应癌旁组织样本)和Not_CHB_HCC组(270例无HBV感染的癌组织样本和部分对应癌旁组织样本),分别对CHB_HCC组和Not_CHB_HCC组中癌组织和癌旁组织基因表达矩阵进行分析。本研究发现,相对于癌旁组织,CHB_HCC组中有356个基因表达上调,619个基因表达下调(图 1 (a));而在Not_CHB_HCC组上调和下调的基因数分别为374和617(图 1 (b))。
|
图 1 107例慢性HBV感染相关的HCC组(CHB_HCC)及含270例非慢性HBV感染相关的HCC组(Not_CHB_HCC)的差异基因分析 Figure 1 Differential gene analysis of the HCC group containing 107 cases associated with chronic HBV infection (CHB_HCC) and the HCC group containing 270 cases associated with non-chronic HBV infection (Not_CHB_HCC) 注:(a) CHB_HCC组差异基因火山图,|logFC|值大于2.027且校正后P < 0.05的基因为差异基因,红色点为上调基因,蓝色点为下调基因; (b) CHB_HCC组差异基因火山图,|logFC|值大于2.149且校正后P < 0.05的基因为差异基因,红色点为上调基因,蓝色点为下调基因; (c) 两组差异基因中上调差异基因的维恩图,左圈为CHB_HCC组的部分上调差异基因个数,右圈为Not_CHB_HCC组的部分上调差异基因个数,中间为二者共有上调差异基因的个数; (d) 两组差异基因中下调差异基因的维恩图,左圈为CHB_HCC组的部分下调差异基因个数,右圈为Not_CHB_HCC组的部分下调差异基因个数,中间为二者共有下调差异基因的个数.(扫本文首页二维码见彩图). |
值得注意的是,两组差异基因的共有差异基因高达852个,且并无共有差异基因在两组中上下调不一致的情况,共有差异基因数占所有差异基因总数的77%左右(图 1 (c)和1 (d))。由此可见,HBV感染相关的HCC虽然与无HBV感染的HCC在基因表达上有所差异,但差异基因绝大多数相同,这提示其他因素导致的HCC可能与HBV相关HCC可能具有类似或相关的一些机制。
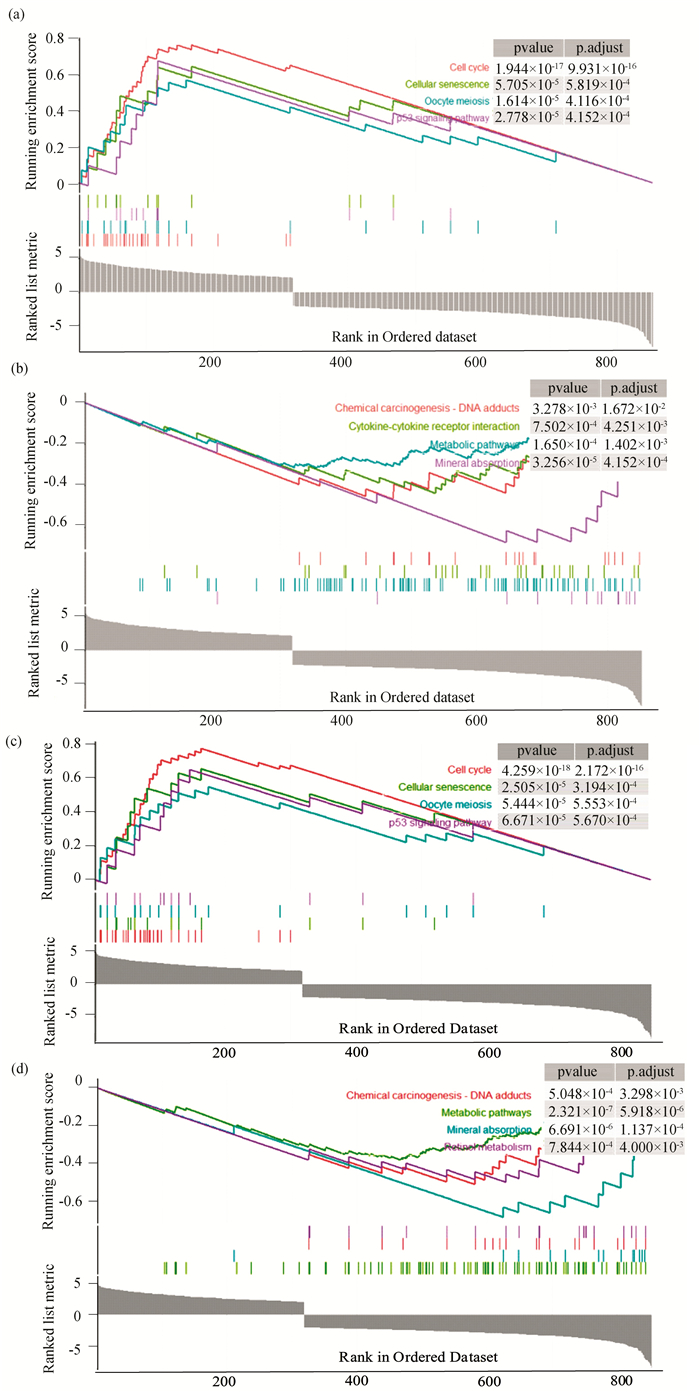
2.2 KEGG信号通路的GSEA富集分析为探究两组差异基因的生物学功能,本研究对共有差异基因进行GSEA_KEGG分析,将共有基因中的上调基因分别在CHB_HCC组(图 2 (a))和Not_CHB_HCC组(图 2(b))中进行富集,下调基因为共有基因中的下调基因分别在CHB_HCC组(图 2(c))和Not_CHB_HCC组(图 2 (d))中进行富集。富集分析结果显示,上调基因主要富集在细胞周期(Cell cycle),细胞衰老(Cellular Senescence)等通路(P < 0.05),下调基因主要在矿质吸收(Mineral absorption),化学致癌-DNA加合(Chemical carcinogenesis-DNA adducts)等通路富集(P < 0.05)。提示这些差异基因确实参与了细胞周期及癌症发生发展的相关过程。
|
图 2 852个共有差异基因在KEGG通路中的GSEA富集分析图 Figure 2 Plots of GSEA enrichment analysis of 852 shared differential genes in KEGG pathway 注:在CHB_HCC组中上调基因的富集结果(a)及下调基因的富集结果(b);在Not_CHB_HCC组中上调基因的富集结果(c)及下调基因的富集结果(d)。不同颜色的曲线代表不同的通路,P < 0.05的通路为显著富集的通路.(扫本文首页二维码见彩图). |
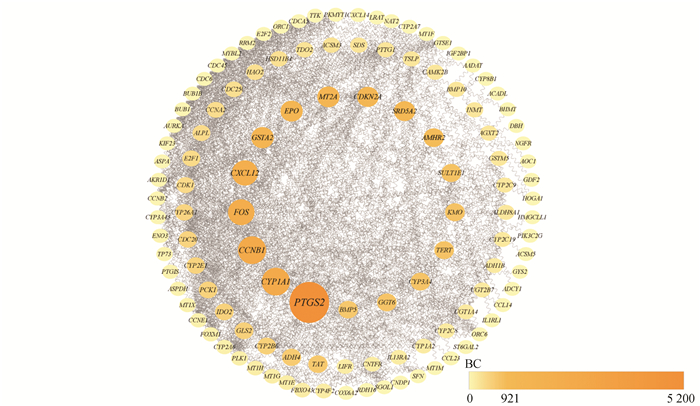
为探究差异基因间相互作用,构建了GSEA_KEGG富集通路中的130个共有差异基因的PPI网络,得到92个基因与其他基因或其对应的蛋白有相互作用,将BC值排在前9的基因作为hub基因,它们分别是前列腺素氧化环化酶2(Prostaglandin-endoperoxide synthase 2,PTGS2),细胞色素P450家族1亚家族A成员1(Cytochrome P450 family 1 subfamily A member 1,CYP1A1),周期蛋白B1(Cyclin B1,CCNB1),FOS原癌基因,AP-1转录因子亚基(Fos proto-oncogene, AP-1 transcription factor subunit,FOS),C-X-C基序趋化因子配体(C-X-C motif chemokine ligand 12,CXCL12),谷胱甘肽S转移酶2(Glutathione S-transferase alpha 2,GSTA2),促红细胞生成素(Erythropoietin,EPO),金属硫蛋白2A(Metallothionein 2A,MT2A),细胞周期蛋白依赖性激酶抑制剂2A(Cyclin dependent kinase inhibitor 2A,CDKN2A)(图 3)。
|
图 3 92个共有差异基因的PPI网络图 Figure 3 PPI network diagram of 92 shared differential genes 注:每一个圆圈代表一个蛋白或基因,圆圈大小或颜色深浅代表其在网络中的Betweenness centrality(BC)值的大小.(扫本文首页二维码见彩图). |
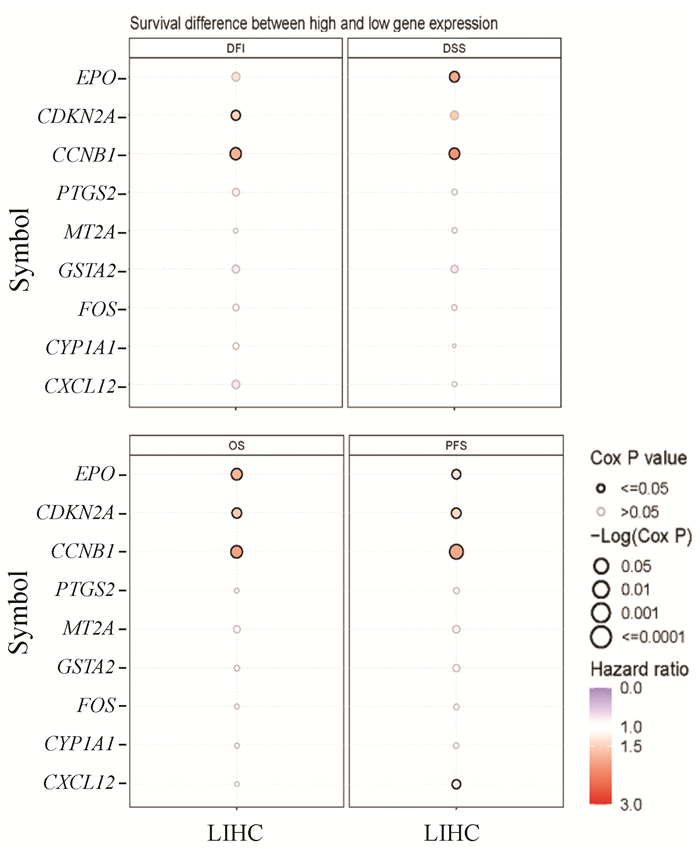
对PPI网络中BC值排名前9的hub基因分别进行DFI生存期、DSS生存期、OS生存期及PFS生存期分析,得到对生存结果具有显著性影响的基因结果如图 4。在OS与PFS生存分析中,CCNB1, EPO及CDKN2A均与生存期显著相关(图 4,P < 0.05),而CXCL12与PFS不相关(图 4,P=0.05)。在DFI生存分析中,仅CDKN2A与CCNB1的高表达与DFI生存期的减少显著相关,而在DSS生存分析中,仅EPO及CCNB1的高表达与DSS生存期减少显著相关(P < 0.05)。综上,仅CCNB1, EPO及CDKN2A与HCC患者的生存期显著相关。
|
图 4 GSCA网页工具分析9个关键基因与HCC生存期的相关性 Figure 4 The GSCA web tool analyzed the association of 9 key genes with HCC survival 注:对PPI网络中的9个hub基因进行生存分析,红色代表风险比(Hazard ratio)大于1,即基因表达量与生存期成反比;相反,蓝色代表风险比小于1,即基因的表达量高低与生存期成正比。圆圈越大则p值越小,圆圈未高亮表示P>0.05,反之,圆圈高亮则表示P≤0.05,P < 0.05定义为与生存期显著相关的基因.(扫本文首页二维码见彩图). |
值得注意的是,在HCC中高表达的EPO,CCNB1,CDKN2A中,EPO在肝脏中具有促进红细胞或血管生成的作用[8-10],CCNB1具有调节细胞周期促进肿瘤细胞增殖迁移侵袭的作用[11-12],而CDKN2A却是广为人知的抑癌基因,编码两种常见的抑癌蛋白,即p16Ink4a和p14Arf,p16Ink4a主要被认为在pRb及CDK4/6,p38 MAPK和PI3K/AKT/mTOR[13]等通路介导的衰老和细胞周期异常中发挥作用,从而抑制肿瘤发生发展[14];p14Arf可与线粒体蛋白p32相互作用促进凋亡[15],同时,与多种转录因子作用,如Myc,NF-κB和HIF-1α,但其研究最广泛的功能是与MDM2直接相互作用使之沉默,从而稳定p53不被蛋白酶体降解,以此发挥抑癌的功能。尽管对于上述CDKN2A相关的抑癌功能有诸多研究,但其在HCC中的过表达作用尚不清楚。因此,为探究这一反常现象,首先开展了qRT-PCR及Western Blot实验检测CDKN2A在临床组织中的表达水平。
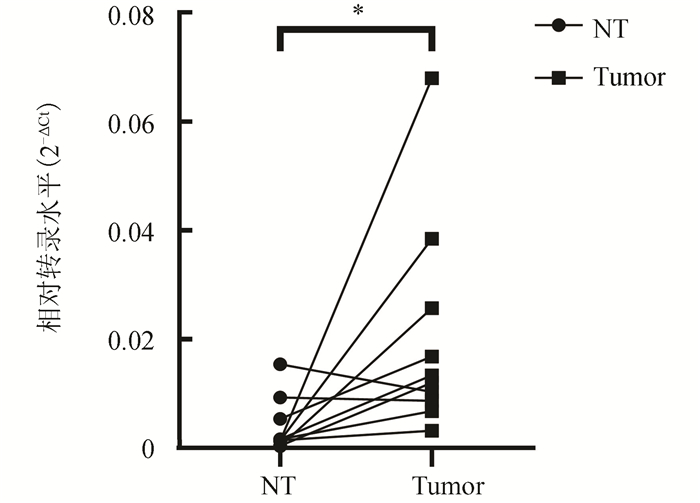
2.5.1 qRT-PCR为了验证CDKN2A在转录水平的改变,本文运用了qRT-PCR对HCC组织及癌旁组织进行了验证,发现CDKN2A在HCC组织中的表达显著高于癌旁组织(图 5,P < 0.05),与差异分析结果一致。另外,相对于内参基因的转录水平,CDKN2A的转录水平在癌组织及癌旁组织中均表达较低,仅为管家基因GAPDH转录水平的0.02倍左右(图 5)。
|
图 5 qRT-PCR检测HCC和癌旁组织中CDKN2A的表达 Figure 5 Expression of CDKN2A in HCC and adjacent non-tumorous tissues was detected by qRT-PCR 注:10对HCC样本及其对应癌旁中的CDKN2A的转录水平,NT为癌旁组织,Tumor为HCC组织,*为P < 0.05. |
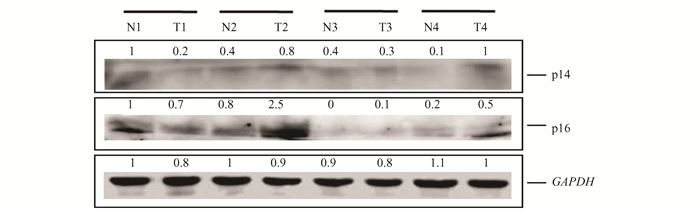
CDKN2A编码2种抑癌蛋白,即p14Arf和p16Ink4a[16],为了验证两种蛋白在HCC样本中的表达量,本文进行了WB实验,证实p14Arf蛋白在标本中未发现差异,而部分样本中的p16 Ink4a蛋白在癌组织和癌旁组织中存在差异,且p16 Ink4a在癌组织中高表达(图 6 N2T2),而在另一部分癌组织样本及其对应癌旁组织中,p16Ink4a表达无差异或无表达。
|
图 6 Western Blot检测CDKN2A在HCC组织中的表达 Figure 6 Expression of CDKN2A in HCC tissues was detected by Western Blot 注:取四例HCC患者的癌组织和对应的癌旁组织,分别检测p14Arf和p16Ink4a的蛋白表达水平。N为癌旁组织,T为肿瘤组织。N和T后面的数字相同则表示同一位病人,N与T之间的短线表示总是以相同病人的N和T二者的数据进行比较. |
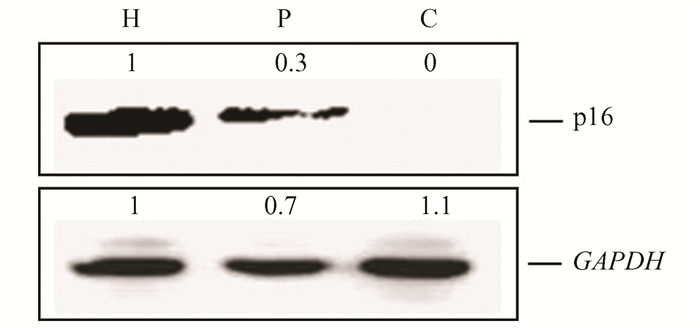
为探究p16Ink4a对HepG2细胞增殖、迁移能力的影响,将p16Ink4a质粒导入到HepG2细胞系中,使p16Ink4a在HepG2细胞中过表达。Western blot结果显示导入了p16Ink4a质粒的HepG2细胞系(HepG2_p16)中的p16Ink4a蛋白的表达显著高于空白对照HepG2细胞系组(图 7),这说明CDKN2A基因在HepG2_p16细胞中过表达。
|
图 7 Western Blot检测CDKN2A在HepG2_p16细胞中的过表达 Figure 7 Stable expression of CDKN2A in HepG2_p16 cells was detected by Western Blot 注:P为过表达p16Ink4a的293T细胞,H为过表达p16Ink4a的HepG2细胞(HepG2_p16),C为空白对照HepG2 Control细胞. |
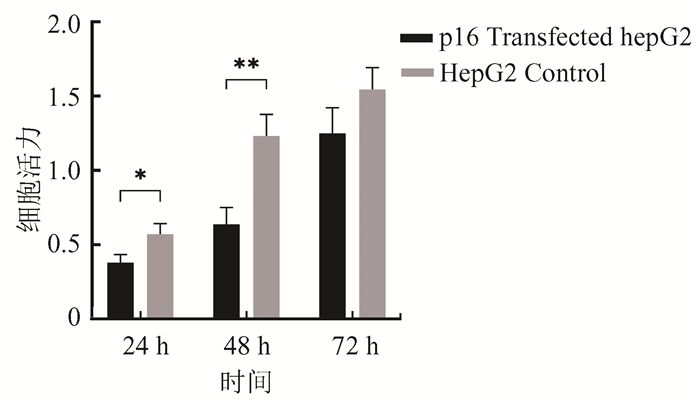
在CCK8增殖实验中,过表达p16Ink4a的HepG2_p16细胞的增殖能力在24 h和48 h时显著弱于对照组(图 8),证实了p16Ink4a蛋白作为抑癌蛋白具有抑制细胞增殖的能力。
|
图 8 CCK8检测p16Ink4a过表达对HepG2细胞增殖的影响 Figure 8 Effect of over-expression of p16Ink4a on proliferation of HepG2 was detected by the CCK8 assay 注:*为P < 0.05,**为P < 0.01,未标注表示无统计差异. |
为探究p16Ink4a是否对细胞迁移能力有所影响,进行了划痕实验,结果显示在划痕72 h后,HepG2_p16细胞的迁移能力明显低于对照HepG2细胞,这提示p16Ink4a具有抑制细胞迁移的能力(图 9)。
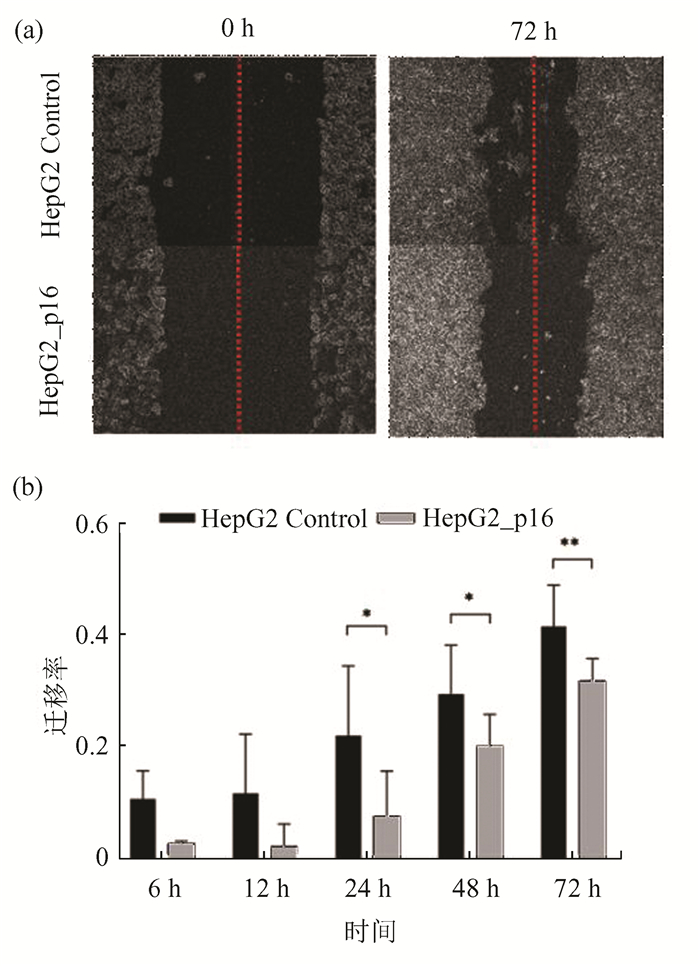
|
图 9 划痕实验结果 Figure 9 Results of scratching experiments 注:(a)分别对HepG2_p16细胞和HepG2 Control细胞进行划痕,并在6,12,24,48,72 h于显微镜下对划痕状态拍照并计算迁移率; (b)计算两组细胞在6,12,24,48,72 h时迁移率并作柱状图, *为P < 0.05,**为P < 0.01,未标注表示无统计差异. |
利用生物信息学方法,对TCGA数据库中LIHC数据集进行挖掘并进行差异分析,筛选共得到852个差异基因。对差异基因进行富集分析及PPI网络分析后得到9个关键基因(PTGS2,CYP1A1,CCNB1,FOS,CXCL12,GSTA2,EPO,MT2A,CDKN2A),随后对9个基因进行生存分析得到CCNB1、EPO和CDKN2A与生存期显著相关。然而,qRT-PCR结果显示仅CDKN2A在HCC组织中转录并与癌旁组织具显著差异。CDKN2A基因编码p14Arf与p16Ink4a两种蛋白,在所有标本中,p14Arf无显著差异,而部分标本中p16Ink4a的Western Blot结果与qRT-PCR结果一致,但另一部分标本的Western Blot结果显示p16Ink4a表达无差异或低表达。为进一步探究p16Ink4a在HCC中的功能,构建了过表达p16Ink4a的HepG2细胞系,使用该细胞系开展了p16Ink4a对癌细胞的增殖及迁移功能验证,发现p16Ink4a能够显著抑制HepG2细胞增殖及迁移。
综上所述,本研究发现并证实CDKN2A在HCC组织中转录水平较癌旁组织显著升高,其编码的p16Ink4a蛋白表达水平在部分HCC组织中较癌旁组织显著升高。同时,本研究验证了p16Ink4a对肝癌细胞系(HepG2)具有抑制增殖和迁移的作用,但这并不能解释其在HCC组织中转录水平及蛋白水平的过表达。p16Ink4a是一种抑癌蛋白,主要参与结合CDK4并抑制其活性,从而抑制细胞周期,其主要受E47, Myc, ITSE, JunB, PPARα, 转录因子Est和Sp等调节[17]。研究表明,p16Ink4a与衰老相关,但仅在年老的动物中,而不是在发育过程中[18]。在一些癌症中,如人乳头瘤病毒(HPV)相关的头颈部癌症中,p16Ink4a可受HPV调控,并作为检测HPV状态的标志物[19];在脂肪瘤性肿瘤中,p16Ink4a单独或合并MDM2展现出了区分非典型脂肪瘤/分化良好的脂肪肉瘤和深层脂肪瘤的重要价值[20];在2/3的乳腺癌中,显示出高水平的p16Ink4a与帕博西尼耐药相关[21],但在HCC中,其过表达机制及相关功能尚不明晰。
TCGA项目团队发现在HCC中CDKN2A启动子高度甲基化,且与TERT改变高度相关[22]。尽管CDKN2A的沉默通常与其DNA高甲基化有关,但Jim Smith等提出,基因的甲基化可能通过三种途径激活基因的转录,即阻碍转录抑制因子结合,解除沉默子和绝缘子调控的并增加增强子的调控,以及促进可变启动子的激活,同时,这些过表达机制可能与人类早期胚胎发育相关细胞、诱导多能干细胞和肿瘤发生发展及转移相关[23]。因此,CDKN2A的甲基化过表达调控可能对于癌症的早期发生或发展具有促进作用。此外,康宁等[24]研究发现,在结肠癌中,CDKN2A可能参与上皮细胞的间质转化(Epithelial-mesenchymal transition,EMT);刘早渠等[25]通过生物信息学手段研究发现,在HCC中,CDKN2A可能与免疫浸润相关;同时,罗志文等[26]研究发现CDKN2A可能导致免疫应答下调从而增加转移性结直肠癌的复发风险。研究表明,CDKN2A编码的p16Ink4a蛋白在癌症中的过表达分为两种情况: ①p16Ink4a在良性或癌前病变中过表达,这种过表达继发于癌基因诱导的衰老;②恶性病变中的过表达,这种过表达是由Rb途径失活引起的,Rb失活将p16Ink4a从其负反馈控制中释放出来,导致这种蛋白质水平的反常增加,从而试图抑制不受控制的细胞复制,但缺乏功能性pRb的细胞对p16Ink4a异位表达导致的细胞积累G0/G1 DNA具有抗性,因此,p16Ink4a或许可用于识别pRb失活,或者识别良性与恶性病变[27]。综上,CDKN2A在HCC组织中的上调或许可以从其甲基化调控、衰老、EMT、病毒感染或免疫相关途径中得到诠释。
| [1] |
SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 (  0) 0) |
| [2] |
LLOVET J M, KELLEY R K, VILLANUEVA A, et al. Hepatocellular carcinoma[J]. Nature Reviews Disease Primers, 2021, 7(1): 6. DOI:10.1038/s41572-020-00240-3 (  0) 0) |
| [3] |
LI Lin, LEI Qingsong, ZHANG Shujun, et al. Screening and identification of key biomarkers in hepatocellular carcinoma: Evidence from bioinformatic analysis[J]. Oncology Reports, 2017, 38(5): 2607-2618. DOI:10.3892/or.2017.5946 (  0) 0) |
| [4] |
孙维华, 朱祥, 尧晨光, 等. 伴HBV感染性肝癌调控枢纽基因筛选与初步鉴定[J]. 生物信息学, 2016, 14(4): 203-212. SUN Weihua, ZHU Xiang, YAO Chenguang, et al. Screening and preliminary identification of regulatory hub genes for the HBV-related hepatocellular carcinoma[J]. China Journal of Bioinformatics, 2016, 14(4): 203-212. DOI:10.3969/jissn.1672-5565.2016.04.02 (  0) 0) |
| [5] |
DING Qinglin, LI Hanluo, XU Zhigao, et al. Identification of CFHR4 associated with poor prognosis of hepatocellular carcinoma[J]. Frontiers in Oncology, 2022, 12: 812663. DOI:10.3389/fonc.2022.812663 (  0) 0) |
| [6] |
LIU Chunjie, HU Feifei, XIE Guiyan, et al. GSCA: An integrated platform for gene set cancer analysis at genomic, pharmacogenomic and immunogenomic levels[J]. Briefings in Bioinformatics, 2023, 24(1): bbac558. DOI:10.1093/bib/bbac558 (  0) 0) |
| [7] |
FU Xiaowei, ZHU Yaqiong, ZHENG Bingbing, et al. KIFC1, a novel potential prognostic factor and therapeutic target in hepatocellular carcinoma[J]. International Journal of Oncology, 2018, 52(6): 1912-1922. DOI:10.3892/ijo.2018.4348 (  0) 0) |
| [8] |
WEIDEMANN A, JOHNSON R S. Nonrenal regulation of EPO synthesis[J]. Kidney International, 2009, 75(7): 682-688. DOI:10.1038/ki.2008.687 (  0) 0) |
| [9] |
JELKMANN W. Regulation of erythropoietin production[J]. Journal of Physiology-London, 2011, 589(Pt 6): 1251-1258. DOI:10.1113/jphysiol.2010.195057 (  0) 0) |
| [10] |
RIBATTI D, TAMMA R. Hematopoietic growth factors and tumor angiogenesis[J]. Cancer Letters, 2019, 440-441: 47-53. DOI:10.1016/j.canlet.2018.10.008 (  0) 0) |
| [11] |
KRENNING L, FERINGA F M, SHALTIEL I A, et al. Transient activation of p53 in G2 phase is sufficient to induce senescence[J]. Molecular Cell, 2014, 55(1): 59-72. DOI:10.1016/j.molcel.2014.05.007 (  0) 0) |
| [12] |
RONG Minhua, LI Jiandi, ZHONG Luyang, et al. CCNB1 promotes the development of hepatocellular carcinoma by mediating DNA replication in the cell cycle[J]. Experimental Biology and Medicine, 2022, 247(5): 395-408. DOI:10.1177/15353702211049149 (  0) 0) |
| [13] |
XU Yingxi, LI Na, XIANG Rong, et al. Emerging roles of the p38 MAPK and PI3K/AKT/mTOR pathways in oncogene-induced senescence[J]. Trends in Biochemical Sciences, 2014, 39(6): 268-276. DOI:10.1016/j.tibs.2014.04.004 (  0) 0) |
| [14] |
LI Junan, POI M J, TSAI M D. Regulatory mechanisms of tumor suppressor P16(INK4A) and their relevance to cancer[J]. Biochemistry, 2011, 50(25): 5566-5582. DOI:10.1021/bi200642e (  0) 0) |
| [15] |
SEO J, SEONG D, LEE S R, et al. Post-Translational Regulation of ARF: Perspective in Cancer[J]. Biomolecules, 2020, 10(8): 1143. DOI:10.3390/biom10081143 (  0) 0) |
| [16] |
CLURMAN B E, GROUDINE M. The CDKN2A tumor-suppressor locus--a tale of two proteins[J]. New England Journal of Medicine, 1998, 338(13): 910-912. DOI:10.1056/NEJM199803263381312 (  0) 0) |
| [17] |
LI J, POI M J, TSAI M D. Regulatory mechanisms of tumor suppressor P16(INK4A) and their relevance to cancer[J]. Biochemistry, 2011, 50(25): 5566-5582. DOI:10.1021/bi200642e (  0) 0) |
| [18] |
WAGNER K D, WAGNER N. The senescence markers p16INK4A, p14ARF/p19ARF, and p21 in organ development and homeostasis[J]. Cells, 2022, 11(12): 1966. DOI:10.3390/cells11121966 (  0) 0) |
| [19] |
RIBEIRO E A, MALEKI Z. p16 immunostaining in cytology specimens: Its application, expression, interpretation, and challenges[J]. Journal of the American Society of cytopathology, 2021, 10(4): 414-422. DOI:10.1016/j.jasc.2020.12.003 (  0) 0) |
| [20] |
HE M, AISNER S, BENEVENIA J, et al. p16 immunohistochemistry as an alternative marker to distinguish atypical lipomatous tumor from deep-seated lipoma[J]. Applied Immunohistochemistry & Molecular Morphology, 2009, 17(1): 51-56. DOI:10.1097/PAI.0b013e3181719223 (  0) 0) |
| [21] |
MOLDOGAZIEVA N T, ZAVADSKIY S P, SOLOGOVA S S, et al. Predictive biomarkers for systemic therapy of hepatocellular carcinoma[J]. Expert Review of Molecular Diagnostics, 2021, 21(11): 1147-1164. DOI:10.1080/14737159.2021.1987217 (  0) 0) |
| [22] |
CANCER GENOME ATLAS RESEARCH NETWORK. Comprehensive and integrative genomic characterization of hepatocellular carcinoma[J]. Cell, 2017, 169(7): 1327-1341. DOI:10.1016/j.cell.2017.05.046 (  0) 0) |
| [23] |
SMITH J, SEN S, WEEKS R J, et al. Promoter DNA hypermethylation and paradoxical gene activation[J]. Trends in Cancer, 2020, 6(5): 392-406. DOI:10.1016/j.trecan.2020.02.007 (  0) 0) |
| [24] |
KANG Ning, XIE Xiaoli, ZHOU Xue, et al. Identification and validation of EMT-immune-related prognostic biomarkers CDKN2A, CMTM8 and ILK in colon cancer[J]. Bmc Gastroenterology, 2022, 22(1): 190. DOI:10.1186/s12876-022-02257-2 (  0) 0) |
| [25] |
LIU Zaoqu, ZHANG Yuyuan, SHI Chengcheng, et al. A novel immune classification reveals distinct immune escape mechanism and genomic alterations: Implications for immunotherapy in hepatocellular carcinoma[J]. Journal of Translational Medicine, 2021, 19(1): 5. DOI:10.1186/s12967-020-02697-y (  0) 0) |
| [26] |
LUO Zhiwen, CHEN Xiao, ZHANG Yefan, et al. Development of a metastasis-related immune prognostic model of metastatic colorectal cancer and its usefulness to immunotherapy[J]. Frontiers in Cell and Developmental Biology, 2020, 8: 577125. DOI:10.3389/fcell.2020.577125 (  0) 0) |
| [27] |
ROMAGOSA C, SIMONETTI S, LOPEZ-VICENTE L, et al. p16(Ink4a) overexpression in cancer: A tumor suppressor gene associated with senescence and high-grade tumors[J]. Oncogene, 2011, 30(18): 2087-2097. DOI:10.1038/onc.2010.614 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


