生物小分子如microRNA在生物过程中发挥着重要的作用,可以对靶基因表达进行正向或负向调控。人类细胞中大多数蛋白的表达水平一定程度上都受到一种或多种microRNA的调控[1]。microRNA是一类约22个核苷酸组成的内源非编码RNA,广泛存在于动物、植物和病毒等物种中。成熟的microRNA分子与Argonaute等蛋白形成RNA诱导的沉默复合体(RNA-induced silencing complex,RISC)抑制基因表达。message RNA(mRNA)是在基因被RNA聚合酶(RNA polymerase)转录后形成,可以将基因的遗传信息转录保存下来。microRNA通常可以通过加速靶mRNAs30的脱腺苷化和降解来负向调控基因表达[2]。microRNA通过调控靶向基因,进而调控下游的生物过程。预测潜在的microRNA与基因的关系可以帮助更好的理解疾病的发生机理,为精准医疗提供更多潜在的靶点。
目前有关microRNA靶基因的确定主要靠生物学实验方法和计算机预测方法。传统的鉴定microRNA靶基因的生物学实验方法,实验成本高,实验耗时长。目前采用的microRNA靶基因预测方法大多是基于规则定义的软件模拟方法和机器学习(Machine learning)方法。Miranda[3],miRBase[4], PITA[5]和TargetScan[6]等工具都是采用了基于规则定义的软件模拟方法。但microRNA与基因的靶向调控关系属于生物过程,会受到不同细胞内多种因素的干扰。因此基于规则定义的计算机模拟方法并不能准确识别microRNA的靶基因。随着机器学习的不断发展,更多基于机器学习的microRNA靶基因预测方法被提出。如TargetSpy[7],miREE[8], MultiMiTar[9]和miRTPred[10]等。这些方法首先生成大量的生物学特征,随后利用机器学习方法从大量特征中选取出对microRNA靶基因预测影响较大的多个特征。机器学习方法相较于软件模拟方法在预测准确率方面有所提高,能够缓解生物实验的高成本问题。但机器学习方法需要使用人工定义的描述符作为模型的输入特征。例如,靶点结合稳定性、位点的类型以及跨物种的靶位点的保守性等特性。人工定义的特征描述符无法全面的概述microRNA与基因序列所蕴含的潜在信息,这对采用这些特征的预测方法有不利的影响。
深度学习(Deep learning)是机器学习的一个子领域,深度学习技术在生物信息领域中的应用,降低了microRNA靶基因预测的难度。深度学习本质上是一类大型的人工神经网络。深度学习方法能够通过构建非线性模块来处理原始的输入数据,将原始数据转换为维度更高,层次更为抽象的特征表示。深度学习模型的输入通常是不包含人工描述符的microRNA和基因的序列数据。特征的定义由深度学习模型本身来实现,这缓解了人工定义特征存在的局限性。在包含复杂特征表示的数据中,深度学习已经被证明是进行分类任务的有效方法[11]。通过内部的多层神经网络,深度学习方法可以从输入的原始数据中提取出有利于模型训练的抽象特征。相对于人工定义的特征,深度神经网络所提取的特征更加全面,这对模型的训练是有利的。
2015年Cheng等[12]提出了miRTDL算法模型,该模型是一种基于卷积神经网络(CNN) 的microRNA靶基因预测算法。2018年Wen等[13]提出了一种基于堆叠去噪自编码器的DeepMiRTar模型预测microRNA的靶基因。miRTDL和DeepMiRTar虽然使用深度学习实现了microRNA靶向基因的预测,但输入特征不是原始数据,没有利用深度神经网络进行特征的自动提取,模型的输入仍然存在人工设计的特征,无法准确表示原始数据中蕴含的潜在信息。因此以上两种方法存在跟软件模拟方法和机器学习方法相同的局限性。同年,Pla等[14]提出了miRAW算法模型。该模型通过输入microRNA和基因3’UTR的原始数据,自动提取原始数据中蕴含的高级抽象特征。2020年Zheng等[15]提出了CNNMirTarget算法模型。该模型通过扫描mRNA全长来预测microRNA与基因的靶向关系,使用CNN自动提取和整合原始序列中的潜在特征,缓解了人工设计特征存在的局限性。
本文设计并实现了一种新的microRNA靶向基因预测模型TransformerMGI。首先,使用基于图卷积网络的GP-GCN方法和预训练模型DNA2Vec分别对microRNA和基因的碱基序列进行向量化。其次,通过引入幂归一化改进了经典的Transformer模型,并将microRNA和基因向量化后的矩阵输入该模型。然后,模型内部进行Attention的关联运算,最后经过非线性激活函数Softmax输出microRNA靶向该基因的概率。在不同数据集上的实验结果表明,本文提出的TransformerMGI模型可以完全依赖序列信息特征对microRNA靶向基因进行准确的预测。
1 材料和方法 1.1 用于模型训练的microRNA和基因数据集本文采用的用于预测microRNA靶基因的数据集分为两部分,一部分是microRNA与基因的序列数据,另一部分是二者的靶向关系。microRNA与基因的表达形式是由碱基组成的字符序列,microRNA与靶基因靶向关系数据有两种取值,可以为Functional和None-Functional。前者代表microRNA和基因存在靶向调控关系,后者代表不存在靶向调控关系。本文使用的microRNA序列信息以及基因序列信息数据集来源于miRTarSeq数据库[16]。本文采用的microRNA与基因的靶向关系数据来源于miRTarBase公开数据库。经过人工初步筛选,最终有10 781条microRNA和基因互作数据参与本文模型训练。
此外,本文从Hafner等[17]的PAR-CLIP实验研究中获取48个正样本数据作为独立数据集,用于在测试阶段检验模型鉴定和识别正样本的能力。该独立数据集在Ding等[18]和Wang等[19]的研究中被使用过。PAR CLIP应用于microRNA靶基因分析能够明显地降低microRNA结合位点的假阳性预测频率并且减小microRNA结合位点搜寻空间的范围,可以在全基因组范围内鉴定AGO2蛋白在不同RNA上的结合位点。本文数据集的部分数据如表 1所示。
| 表 1 数据集展示 Table 1 Dataset display |
由于组成microRNA序列和基因序列的碱基类别和碱基序列长度是不同的,因此本文采用两种不同的特征提取方法分别提取microRNA与基因的特征信息。一方面,本文采用基于GP-GCN的特征提取方法对microRNA序列进行特征提取。另一方面,引入了DNA2Vec预训练模型对基因序列的特征信息进行提取。
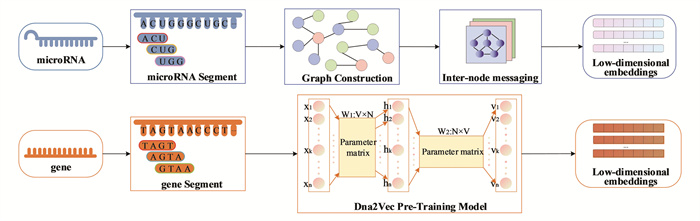
1.2.1 基于GP GCN的microRNA矩阵化流程相对于传统方式来说,图数据具有比序列更强的数据和知识表示能力,不仅能表示样本(节点)的独立特性,还能表达相同类型甚至不同类型样本之间的关系。因此,为了实现对microRNA字符串序列的数值表示,本文将基于图卷积网络的GP-GCN方法[20]引入到对microRNA序列的矩阵化中。microRNA矩阵化流程如图 1所示。
|
图 1 microRNA和基因的矩阵化表示流程 Figure 1 Matrix representation of microRNAs and genes |
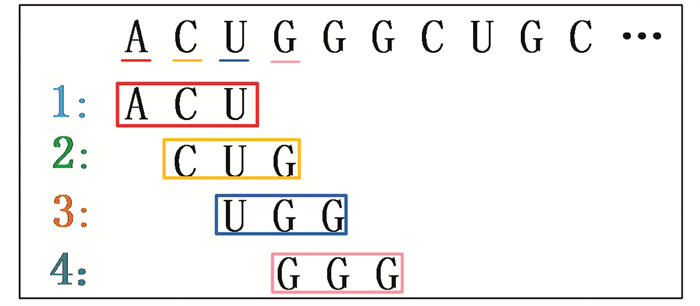
在图 1中,对于给定的一条microRNA序列s,首先利用如图 2所示的“滑动窗口”方式进行碱基序列切分,得到若干个长度为k的k聚体(k-mer)[21]。其次通过多个k-mer之间的位置关系建立Graph。然后通过图卷积网络节点间的消息传递运算,最终得到每一个k-mer的低维向量表达。
|
图 2 “滑动窗口”分词操作示意图 Figure 2 Schematic diagram of the "sliding window" word segmentation operation |
在图 2中,对于给定的一个microRNA碱基序列m和一个正整数k,m可以切分为多个长度为k的microRNA碱基子序列。k值越大,k-mer就能跨过更多长度较短的重复序列,但较大的k值会导致k-mer的数量变少。k-mer之间彼此互联,k-mer数量的减少不利于不同位置碱基特征的提取。通过参考文献[22-23]中对k值的设定,并考虑microRNA序列的长度问题,本文将k值设置为3,可以得到若干个重叠的k-mer。microRNA矩阵化的具体流程如图 3所示。
|
图 3 GP-GCN的处理流程图 Figure 3 GP-GCN processing flowchart |
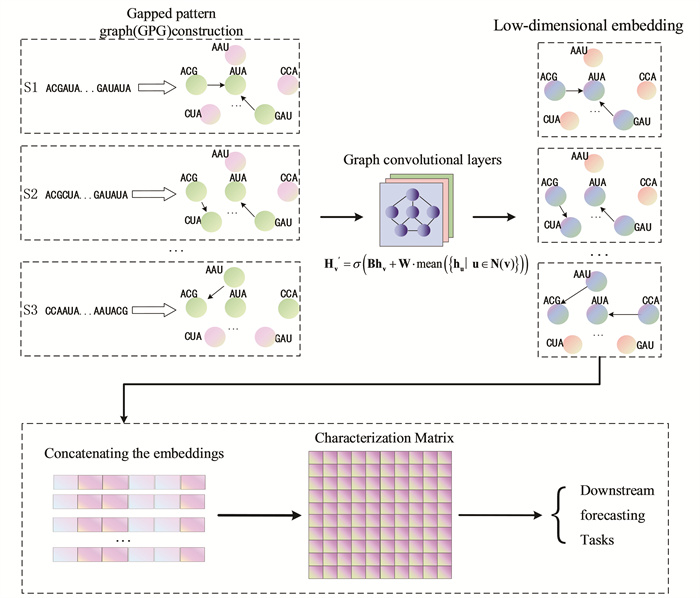
在图 3中,首先,对于给定的一条microRNA序列s构建一个Graph用于学习图数据中节点和边之间的关系,Graph的点对应k-mer,Graph的边对应k mer之间的interaction。点的特征是k-mer出现的频率。边的特征是这两个k-mer连在一起出现的频率。
由于遗传变异和外部环境影响,基因组上会出现单核苷酸多态性(Single nucleotide polymorphisms,SNP)。microRNA的SNP会影响microRNA的功能和生物过程,从而影响表型[24]。因此,为了避免SNP变异情况发生对预测结果产生不利的影响,本文引入了间隙形态图(Gapped pattern graphs,GPG)[20]来减弱SNP现象对核苷酸序列建模的影响。
GPG通过考虑k-mer之间设置间隔距离来忽略SNP对于序列整体建模的影响。间隔距离越大,建模可以容忍的发生SNP现象的碱基的个数越多。通过考虑GPG中不同的间隔距离可以得到多个GPG子图。
例如: 对于一条序列S {ACGUAGC},通过k值为3的“滑动窗口”可以得到若干个长度为3的k-mer{ACG,CGU,GUA,UAG,AGC},这些k-mer对应Graph中的每一个顶点。当不考虑间隔距离时,对于Graph的两个顶点v1和v2分别对应ACG和UAG。当且仅当ACG和UAG在S中相邻时,v1和v2之间才有边。在S中由于ACG和UAG相邻,因此顶点v1和v2之间有边。当考虑间隔距离时,设间隔距离d=1。对于Graph的两个顶点v1和v2分别对应ACG和AGC,当且仅当ACG和AGC在S中相隔1个碱基时,v1和v2之间才有边。因此考虑不同的间隔距离,序列S可以得到不同的子图。
其次,在完成Graph建立的基础上,本文使用图卷积神经网络进行节点间的消息传递,将单个k-mer的信息聚合到其他k-mer上。最后,将所有k-mer的embeddings拼接在一起,得到microRNA的表征矩阵用于下游预测任务。GP GCN中单个顶点的特征计算如公式1所示[20] :
| $ \left.h^{\prime}{ }_v=\sigma B h_v+W \cdot mean\left(\left\{h_u \mid u \in N(v)\right\}\right)\right) \# $ | (1) |
在公式1,hv是图 3中顶点v的特征,N(v)是图 3中所有顶点的集合,与顶点v相关的边从顶点v发出,σ 是非线性激活函数,上式中的mean计算输入特征的平均值,W,B是使用图卷积网络进行消息传递时要学习的参数矩阵。
本文引用的GP-GCN是一款开源框架,该框架使用Pytorch和Pytorch_geometric编写,可以将碱基序列编码成深度学习的向量化输入。
1.2.2 基于DNA2Vec的基因特征提取方法本文使用DNA2Vec[25]预训练模型编码基因序列数据。DNA2Vec基于Word2Vec[26]模型,使用人类基因组序列作为训练资源,将1.2.1小节中提到的k mers嵌入到了100维连续向量空间中。因此学习的向量包含更多的基因序列潜在信息。
使用DNA2Vec对基因序列进行编码能够捕获更多的潜在信息。在DNA2Vec中,DNA载体的总和类似于碱基串联的事实在实验中得到了验证[25]。这表明在DNA序列上使用词嵌入确实捕获了有用的信息。对预训练的词向量进行适当的微调是有利的,可以使向量更适合特定任务。因此本文使用了图 2所示的“滑动窗口”方式对基因碱基序列进行切分。在基因的碱基序列切分时将滑动窗口大小设置为4,切分得到若干个长度为4的基因碱基子串。基因序列矩阵化流程如图 1所示。在图 1中,本文对碱基序列进行了切分,并从预训练好的DNA2Vec模型中提取了相应的向量表示。所获得的嵌入向量具有100个维度。由于基因序列可以被“滑动窗口”划分为多个碱基子串,因此每个基因序列都可以表示为数值矩阵的结构化形式。
1.3 microRNA与基因的靶向关系预测模型TransformerMGI将microRNA序列和基因序列类比为自然文本序列,通过基于图卷积神经网络的GP-GCN方法和DNA2Vec预训练模型分别对二者的特征信息进行提取,并利用改进的TransformerMGI模型来学习二者在序列结构方面的特性和联系,进而预测microRNA和基因之间相互作用的概率。
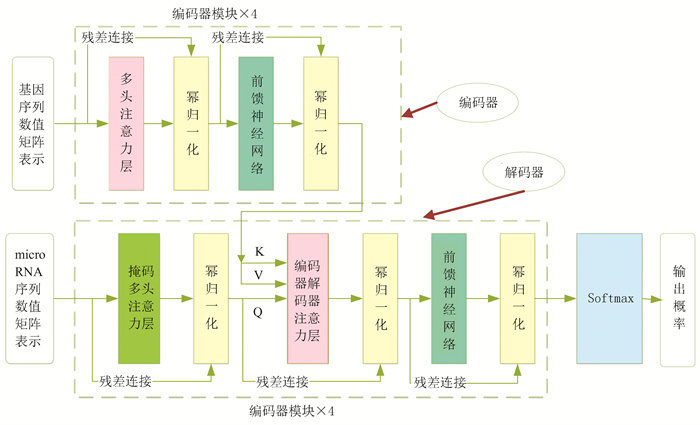
本文提出的基于TransformerMGI的microRNA与基因靶向关系预测模型架构图如图 4所示。在图 4中,本文提出的TransformerMGI模型使用编码器解码器架构。模型深度为8层,编码器和解码器各占4层。编码器端由4个相同的编码器模块堆叠而成,每一个编码器模块又包含多头自注意力层和前馈神经网络层两个子层和幂归一化操作。解码器端由4个结构相同的解码器模块堆叠而成,解码器模块的结构类似于编码器,但除了解码器模块包含的多头注意力层和前馈神经网络层外还包含一个掩码多头注意力层。
|
图 4 基于TransformerMGI的microRNA与基因靶向关系预测模型架构图 Figure 4 Architecture diagram of TransformerMGI-based microRNA and gene target relationship prediction model |
此外,在编码器和解码器模块的各个子层之间加入了残差连接和归一化操作。残差连接的加入可以一定程度上对由于梯度爆炸引起的网络退化问题有所缓解,而归一化的加入可以使得数据的分布更加趋于稳定规范和优化空间,从而达到加速模型收敛速度的目的。
将经过DNA2Vec模型提取后得到的基因表征嵌入矩阵传入编码器,经过多头注意力层对基因特征数据进行聚合,生成新的数据表示。随后经过前馈神经网络层进行非线性映射得到编码器的输出。编码器输出传入解码器的第一个解码器模块。同时,经过GP GCN方法提取得到的microRNA表征嵌入矩阵传入解码器,经过掩码注意力层进行运算得到新的特征信息。掩码注意力层输出的新的microRNA特征信息和编码器传来的基因的特征信息同时传递到第一个解码器模块的编码器解码器交互注意力层。目的是学习microRNA数据与基因数据的特征,对二者的数据进行关联度计算。随后将编码器解码器交互注意力层的输出传入解码器的前馈神经网络层,得到解码器的输出。最后经过全连接输出层,预测出microRNA与基因存在靶向调控关系的概率。
1.3.1 编码器如图 4所示,编码器由4个编码器模块堆叠而成。每一个编码器模块又由2个子模块组成,每一个子层之间都使用了残差连接和幂归一化操作。
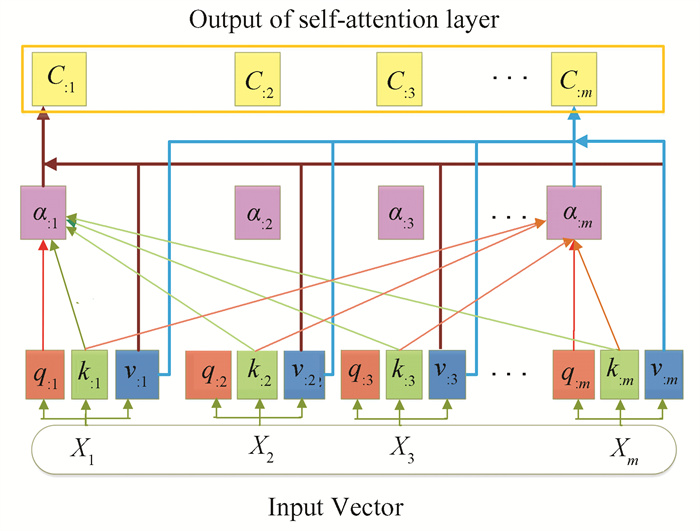
编码器模块的第一个子层是多头自注意力层,多头注意力模块可以增强模型对不同位置的关注能力,形成多个子空间,让模型去关注不同方面的信息,进而增强注意力的表现能力。多头注意力机制的本质是利用多组参数矩阵进行多个独立的Attention计算,并将最后得到的结果做集成。多头注意力机制一定程度上防止了过拟合现象的发生。具体自注意力机制计算过程如图 5所示。
|
图 5 注意力机制计算过程图 Figure 5 Attention mechanism calculation process diagram |
在图 5中,在输入的基因碱基序列中,单个xi是一个固定长度的向量表示经过切分后单个基因碱基子串的向量表达,多个xi组成一个矩阵形式输入到注意力层,通过与自注意力层的参数矩阵进行多次运算最后得到对应m个ci向量,ci依赖于所有的xi向量,每一个ci向量中都包含了所有xi的表征信息,改变任意一个xi的值,ci就会发生变化。
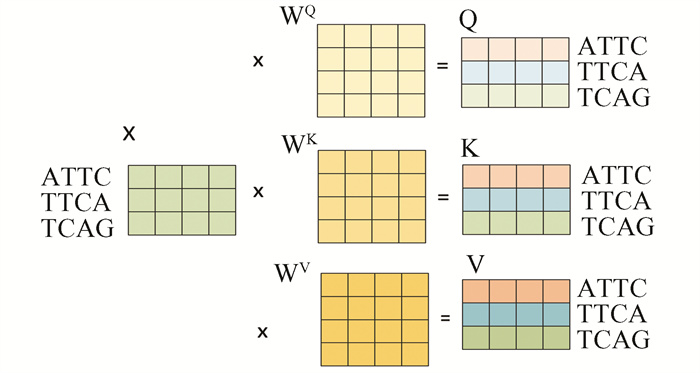
输入的m个xi向量用来表示m个碱基子串的特征,通过与权重参数矩阵WQ,WK,WV相乘分别生成查询向量q、键向量k、值向量v,其矩阵相乘过程如图 6所示。
|
图 6 Q, K和V的计算过程 Figure 6 Calculation process of Q, K, and V |
在图 6中,假设某基因序列长度经过碱基序列切分后,得到“ATTC”, “TTCA”, “TCAG”三个碱基子串。通过使用DNA2Vec预训练模型的权重参数,可以获取到这三个碱基子串对应的向量表达xi,随后三个向量拼接为一个数值矩阵的形式记为X。X分别与WQ,WK,WV三个权重参数矩阵相乘可以分别得到这三个xi的查询向量、键向量以及值向量,分别记作Q, K和V。Q, K和V由三个xi的查询向量qi, 键向量ki和值向量vi堆叠而成。每一个xi都可以依靠与参数矩阵相乘生成三个向量: qi,ki,vi。计算过程如公式2所示:
| $ \left\{\begin{array}{l} Query: q_{: i}=W_Q x_i \\ Key : k_{: i}=W_K x_i \\ Value : v_{: i}=W_V x_i \end{array}\right. $ | (2) |
在公式2中,WQ,WK和WV是根据输入向量初始化而成,可以通过后期训练不断优化改变。通过上述运算,可以计算出每一个输出向量的查询向量、键向量和值向量。
在得到每个xi对应的qi,ki,vi向量后,通过比较每一个的查询向量qi与所有输入向量的键向量ki的关系得到权重值αi。αi计算过程如公式3所示:
| $ \alpha: j=Softmax(K T q: j) \in R m $ | (3) |
在公式3中,矩阵K是由所有的键向量ki组成。结果α: j表示当前查询向量q与所有的键向量k的匹配程度。匹配程度越高权重值越大。经过了Softmax函数处理,α: j的所有值相加等于1。
通过得到的权重值αi乘以权重值所在输入向量xi的值向量vi,得到一个全局向量cj,计算过程如公式4所示:
| $ c: j=\alpha 1 j v: 1+\cdots+\alpha m j v: m=V \alpha: j $ | (4) |
在公式4中,cj表示当前xi向量对应的上下文向量(Context Vector) cj的值。cj融合了所有碱基串的表征信息,依赖于所有的碱基串向量。若所有碱基串向量中的其中一个碱基串xi的向量值发生改变,会直接性的改变qi,ki,vi向量的值,从而间接改变权重值αj和全局向量cj的值。
重复上述过程,可以计算出每一个xi对应的全局向量cj的值。注意力机制更一般的表达形式如公式5所示:
| $ Attention(Q, K, V)=Softmax\left(\frac{Q K^T}{\sqrt{d_K}}\right) \cdot V $ | (5) |
在公式5中,Q值、K值和V值分别代表注意力机制中的查询向量、键向量和值向量,dK表示特征向量的维度。Q, K和V的值通过与参数矩阵WQ, WK和WV运算得到。在注意力机制运算过程中,将dk作为缩放因子,可以在一定程度上避免因为方差过大造成的训练不稳定问题。
归一化是深度神经网络架构中的关键部分。归一化可以保证数据伸缩不变形和数据的权重伸缩不变形,可以有效避免网络层执行反向传播时权重过大或过小导致的梯度爆炸和梯度消失问题,有效地减少梯度弥散现象的发生,从而加速神经网络模型的训练。
然而,归一化却存在多种实现形式。例如批归一化(Batch normalization,BN)[27]常用于计算机视觉任务中。在自然语言处理中使用BN会使模型性能下降,因此层归一化(Layer normalization,LN)[28]成为大多数自然语言处理任务模型中的标准方案,如Vaswani等[29]提出的Transformer模型就使用了LN。尽管如此,相较于BN,LN在NLP中仍存在一些不足。一方面,LN会破坏神经网络所学习到的抽象特征,影响模型的收敛。另一方面,当BN与LN都适用的场景下,BN的效果一般比LN更优。这是因为对于不同数据,同一特征归一化得到的数据更不容易损失信息。
生物序列通常除了存在一定的空间结构和高维特性外,还存在序列元素之间的依赖关系。例如,碱基序列中不同位置的碱基存在相互依赖关系。LN在处理生物序列时存在缺陷,会破坏神经网络学习到的生物序列的抽象特征。因此,模型的泛化和预测能力会下降。
在基准Transformer模型中,使用层归一化对数据进行归一化。文本数据在训练过程中具有很大的方差值,在数据本身均值方差变化剧烈的情况下强制将输入的数据转换为均值为0方差为1的正态分布,会导致网络层中数据震荡剧烈,甚至极端的异常值。这会造成训练和测试时数据不一致。
文献[30]在探讨BN和LN的优缺点的同时,提出了幂归一化(Power normalization,PN)。PN基于BN进行了改进,是一种新的归一化方案。由于BN在处理文本数据时会导致性能下降,因此PN放宽了BN中的零均值均一化来缓解这个问题。此外,PN还结合了连续两次的平均值来稳定数据分布,以缓解数据分布震荡问题。
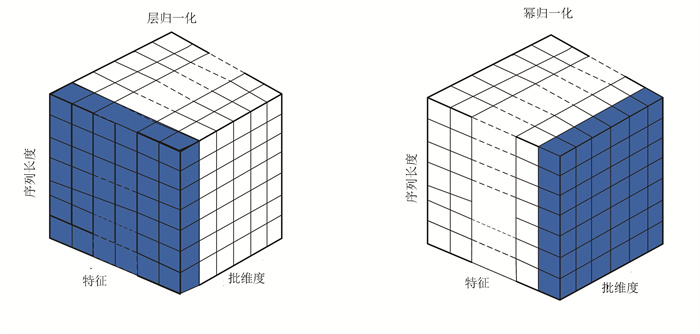
为了解决在处理生物数据时基准Transformer模型中层归一化存在的问题,并训练出泛化性更强的模型,本文引入幂归一化[30]模块替代基准Transformer模型中的层归一化模块。本文引入的幂归一化是针对整个批次计算的,而经典的层归一化是对一个样本中的所有特征进行计算的。层归一化和幂归一化的示意图如图 7所示。在实验部分的实验结果也表明,相对于层归一化,幂归一化的引入使得模型具有更好的性能表现。
|
图 7 层归一化和幂归一化 Figure 7 Layer normalization and power normalization |
本文的TransformerMGI模型解码器的结构如图 4下侧虚线框内所示,由4个解码器模块堆叠而成。每一个解码器模块由3个子层组成,除了包含编码器-解码器注意力层以及前馈神经网络层外,本文还使用了掩码多头注意力层,目的是屏蔽碱基序列中的无效区域。掩码多头注意力层利用掩码操作来覆盖解码器中所预测内容的下文,以此保证训练阶段和推理阶段的一致性。被添加的掩码多头注意力操作可以避免模型看到未来时刻的信息,使参数在更新时不产生效果。本文在解码器模块每一个子层之后还使用了残差连接和幂归一化。
在图 4中,解码器模块的第一个子层是掩码多头注意力层,第一个解码器模块将经过GP-GCN处理后产生的microRNA表征矩阵作为输入。除第一个解码器模块外,剩余的解码器模块的输入为前一个解码器模块的输出。由于各个microRNA的长度会有不一致的情况,因此在数据预处理阶段为了统一microRNA的长度,本文对不满足规定长度的microRNA数据的长度进行了padding操作,以此满足深度神经网络对数据的基本要求。但是使用padding操作所填充的位置上的信息是无意义的信息,不能让其参与到后续注意力机制的运算中,否则就会导致模型无法精准的关注模型本身需要重点关注的信息,增大相关性较低位置的信息对模型性能的影响。而在训练阶段,解码器部分的输入数据是microRNA整个序列信息,序列中不仅包含了已知部分序列信息,还包含了等待被预测的后续的未来信息。在这种情况下,掩码多头注意力操作掩盖了那些不应出现的未来时刻信息,使未来时刻信息在参数更新时不产生效果。
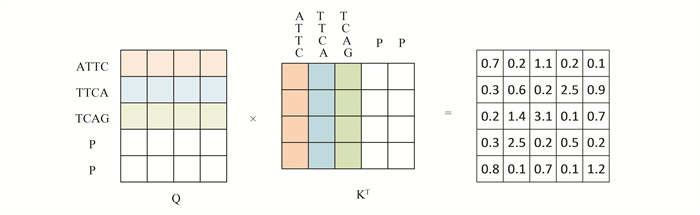
如图 8所示,P表示Padding的位置,右边的矩阵表示在不使用Mask的情况下通过注意力计算得到的注意力权重矩阵。相对于非Padding位置而言,注意力权重也给予了Padding位置的信息同样的关注。因此本文提出的TransformerMGI模型通过在数据集处理过程中提前记录每个样本Padding的实际位置,然后将注意力权重矩阵中对应位置的权重替换为负无穷便达到了忽略Padding位置信息的目的。Padding的掩码计算如图 9所示。
|
图 8 Padding时注意力计算过程图 Figure 8 Diagram of the attention calculation process when using Padding |

|
图 9 Padding的掩码计算过程图 Figure 9 Diagram of the mask calculation process when using Padding |
在图 9中,对于“ATTCAGPP”这个碱基序列来说,前3个子序列是正常的。因此后2个子序列包含了P,所以是Padding后的结果。因此,其Mask向量为[True,True,True,False,False]。通过此Mask向量可知,权重矩阵的最后两列被替换成了图 9中B矩阵中的-inf来表示负无穷,目的是在注意力计算过程中对Padding位置的关注度降到最低。本文在提出的TransformerMGI模型中掩码多头注意力机制提高了模型对重点信息的关注能力,减轻了相关性较低位置的信息对模型性能的影响。
1.4 模型评估指标在本文的实验过程中,选择二元交叉熵损失函数Loss(Binary cross entropy loss),ROC (Receiver operating characteristic)曲线下面积AUC(Area under curve)和精确率-召回率曲线PRC(Precision recall curve)下面积AUPRC作为模型的评价指标。
交叉熵描述的是两个概率分布之间的距离,交叉熵越小两个概率分布越接近。二元交叉熵是二分类任务中常用的一个的损失函数。当模型的预测值与真实标签值越接近损失越小,反之,损失会越大。二元交叉熵损失函数Loss计算方法如公式6所示:
| $ Loss =-[y \log \hat{y}+(1-y) \log (1-\hat{y})] $ | (6) |
在公式6中,y表示真实的标签值,
AUC表示ROC曲线下的面积。ROC是以假正例率(False positive rate,FPR) 为X轴、真正例率(True positive rate,TPR)为Y轴绘制的反应模型敏感性和精确性趋势走向的曲线。AUC的值越接近1,代表模型的分类性能越好;反之,模型分类性能越差。假正例率FPR和真正例率TPR计算方法如公式7和公式8所示:
| $ T P R=\frac{T P}{T P+F N \#} $ | (7) |
| $ F P R=\frac{F P}{F P+T N \#} $ | (8) |
在公式7和公式8中,TP表示数据集中被判定为正类,实际也为正类的样本数;FN表示数据集中被判定为负类,实际却为正类的样本数。FP表示数据集中被判定为正类,实际却为负类的样本数;TN表示数据集中被判定为负类,实际也为负类的样本数。
PRC可以对分类器的整体结果进行综合性评价。以查全率为X轴、查准率为Y轴绘制曲线,AUPRC的取值是曲线与X轴和Y轴所围成区域的面积。AUPRC表示“查准率=查全率”时的取值,值越大表明分类器性能越好。而且PRC与AUC相比对样本不均衡敏感,很适合在microRNA与基因靶向关系正负样本不均衡时评价分类器的好坏。查全率(Recall)如公式7所示,查准率(Precision)计算方法如公式9所示:
| $ Precision =\frac{T P}{T P+F P} \# $ | (9) |
为了在实验阶段通过优化实验得到性能更优的模型,本文将数据集进行了划分,80%用作训练和验证,20%用作测试。并且利用控制变量法,以表 2中的参数为基准,对模型参数进行优化并开展了实验。实验从以下三个参数优化角度进行了对比分析,三个优化角度分别是批大小,模型网络结构以及归一化。
| 表 2 TransformerMGI模型参数设置 Table 2 TransformerMGI model parameter setting |
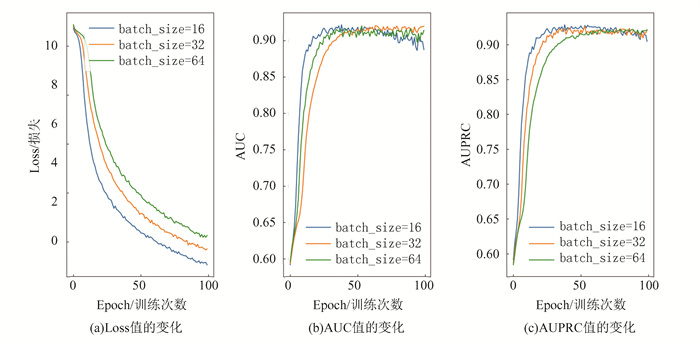
批大小(Batch size)即每一次训练中参与训练的microRNA与基因关系对样本的数量。批大小不仅影响模型的训练速度和模型优化,还影响每一个epoch训练模型的次数。批大小若设置过大,不仅对GPU显存要求较高而且会减低模型的泛化能力;批大小设置过小,所花费的训练时间会变长,同时会出现梯度震荡现象不利于模型的收敛。因此选择一个合适的批大小对模型训练很重要[31]。为了衡量模型性能和批大小之间的关系,本小节将批大小设置为16, 32和64,并分别进行了实验,实验结果如图 10所示。
|
图 10 使用不同批大小时评价指标的结果 Figure 10 Evaluate the results of the metric when using different batch sizes |
图 10分别表示在不同批大小的情况下,随着训练迭代次数的不断增加,模型Loss值、AUC值以及AUPRC值的变化。横坐标表示训练的迭代次数epoch。从图 10(a)可以看出当批大小等于16和32时,模型的Loss值下降速度较快下降幅度更大。从图 10(b)中可以看出在当批大小等于32时,模型的AUC值相对更高。但在图 10(c)中批大小选择32和64时的AUPRC值相近,但由于批大小等于32时,单个批次的数据进行训练耗费的系统资源更低。因此基于以上实验数据,本文选择的最优批大小为32。
2.1.2 网络结构的优化选择对于深度神经网络模型而言,增加模型的复杂程度有助于模型学习能力的提升[32]。模型可以通过增加模型的宽度或深度进而达到提高模型学习能力的目的。增加模型宽度可以使模型中的每一层学习到更加丰富的特征。增加模型的深度可以使模型拥有更强大的非线性表达能力,使模型学习更加复杂的变化,从而拟合更加复杂的数据输入。本文将AUC和AUPRC的值作为选择合适网络架构的重点关注因素。
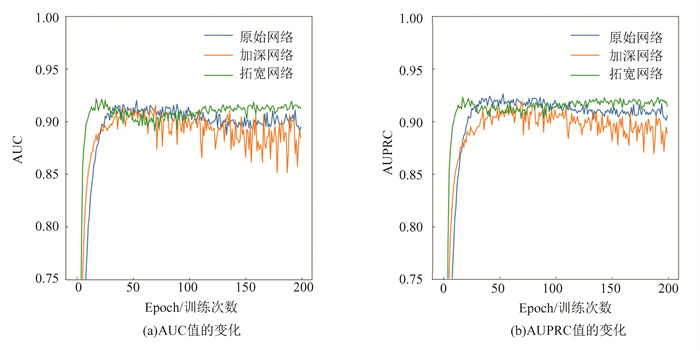
在本文中,增加模型深度意味着增加编码器模块和解码器端模块的堆叠数量。为衡量拓宽和加深网络对本文模型的影响,设定原始网络深度为4层,网络宽度为64;加深网络实验中设置网络深度为8层,宽度为64;拓宽网络中设置网络深度为4层,宽度为128。参数变量如表 3所示,实验结果如图 11所示。
| 表 3 加深和拓宽网络与原始网络实验参数表 Table 3 Deepen and broaden the experimental parameter table of the network and the original network |
|
图 11 网络结构改变后评价指标的结果 Figure 11 The results of the evaluation index after the network structure change |
如图 11所示,横坐标表示训练迭代次数epoch,模型使用训练集每迭代训练一轮,都会使用验证集进行一次验证。可以看出在相同的迭代次数下,加深网络的各项指标均低于原始网络。而拓宽网络的各项指标均略高于原始网络。这表明在使用控制变量法,控制其他参数不变的情况下,适当拓宽网络对本文模型起到了积极的影响。最终本文选择了性能较优的拓宽网络作为本文的模型。
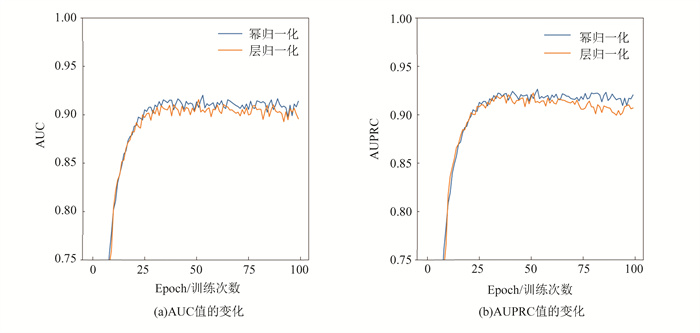
2.1.3 幂归一化和层归一化的优化选择本文将幂归一化引入到了模型中,取代了原始的层归一化。为了验证本文采用的幂归一化性能方面优于经典的层归一化,所以本文对二者进行了实验验证。验证结果如图 12所示。
|
图 12 幂归一化和层归一化的效果对比 Figure 12 Comparison of the effects of power normalization and layer normalization |
本文使用训练集每迭代训练一轮,都会使用验证集进行一次验证。在图 12中,横坐标表示训练迭代次数epoch,纵坐标表示模型对验证集预测所得的AUC和AUPRC的值。随着训练迭代次数的不断增加,幂归一化在各项评价指标方面的均略优于层归一化。从整体来讲,在100轮的迭代次数下,相较于层归一化,幂归一化在AUC和AUPRC指标方面均优于前者。这也表明在模型中引入幂归一化对模型的性能表现有着积极的影响,能够改善模型的预测效果。
2.2 TransformerMGI模型的性能测试AUC指标表示模型预估正样本得分大于负样本的概率,可以用来衡量模型的排序能力[33]。AUC指标所衡量的一个模型的预测能力与样本比例无关。在样本比例不均衡的情况下,AUPRC更能反应一个分类器的性能[34]。本文同时采用AUC和AUPRC两个评价指标,通过实验测试了本文数据集在不同模型上的性能表现。
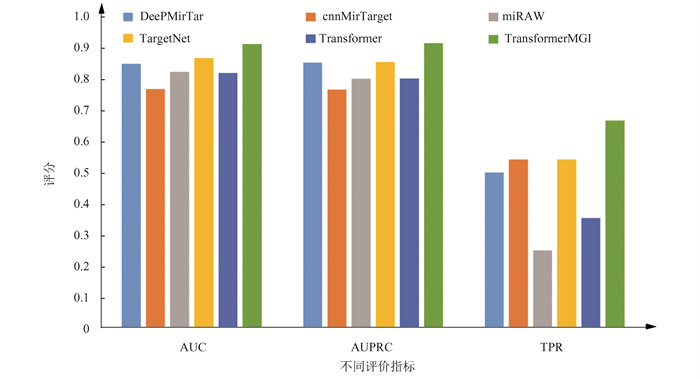
由于microRNA生物实验的最终目的是发现能与microRNA存在调控关系的靶基因,因此模型鉴定和识别正样本的能力很重要。为验证本文模型的有效性,本文使用了含48个正样本的PAR-CLIP数据集对本文模型进行测试,用于检验本文模型识别正样本的能力。将48个microRNA和基因互作对输入已经被训练集训练完毕的TransformerMGI模型中。模型会对48个互作对的靶向调控关系进行预测,并以TPR作为评价指标输出实验预测结果。实验结果如图 13所示。
|
图 13 不同算法模型在microRNA与基因靶向关系预测任务上的效果对比 Figure 13 Comparison of the effects of different algorithm models on the task of predicting the relationshipbetween microRNA and gene targeting |
此外,基于本文的数据集,本文选择了近些年发表的用于预测microRNA与基因靶向关系的深度学习模型与本文提出的模型进行了对比实验,比较了cnnMirTarget[15],TargetNet[35],miRAW[14],deepMiRTar[13]与本文提出的TransformerMGI模型在测试集上的预测表现,实验结果如图 13所示。
cnnMirTarget模型学习了microRNA和靶位点嵌合体中规范和非规范相互作用。TargetNet是一种基于残差神经网络预测模型。miRAW通过自定义规则选取潜在microRNA靶位点。而deepMiRTar使用了专家设计的特征。以上方法均未考虑子串在碱基序列中的位置依赖关系。
在图 13中,实验结果表明,相较于参与对比实验的其他模型,在特征提取阶段,基于图卷积网络的特征提取方法使得本文模型充分考虑了碱基子串之间的位置依赖关系,这使得本文在特征提取阶段能提取出比其他模型更准确的潜在信息。因此,本文模型在性能指标方面优于其他参与对比的模型。
对于PAR-CLIP独立数据集,本文模型能正确预测其中67%的数据,准确率高于参与对比实验的其他模型。由于PAR-CLIP独立数据集中的正样本数据都已经过生物实验证实。因此,这也证明了本文模型在鉴定和识别microRNA靶向基因方面的能力。
随着microRNA与基因靶向关系研究的不断深入,越来越多的研究表明microRNA对靶基因的失调会诱发各种恶性疾病[36]。例如,Xu等[37]的研究中提到了GNG7是食道癌、头颈部鳞状细胞癌,胰腺癌等疾病的肿瘤抑制基因。Tan等[38]的研究中提到GNG7的表达受microRNA的调控,microRNA可以通过调控靶基因GNG7从而影响食道癌的癌细胞表达水平。Teng等[39]的研究指出基因DNMT1的表达同样对食道癌治疗有潜在意义。此外,基因DNMT1表达还与乳腺癌细胞生存率低有关[39]。因此本文选取了与癌症病变有关联的GNG7和DNMT1基因对模型预测效果进行检验,通过模型预测得到了分别与GNG7和DNMT1存在靶向关系的microRNA,如表 4所示。
| 表 4 分别与GNG7和DNMT1存在靶向关系的microRNA Table 4 microRNAs with targeting relationships with GNG7 and DNMT1, respectively |
在表 4中,列举了模型预测得到的分别对基因GNG7和DNMT1存在靶向调控关系的microRNA的名称、microRNA序列以及生物实验支持证据。其中生物实验证据一列的数据通过miRTarBase数据库获得,Reporter assay, Western blot以及qpCR都是用于判断microRNA与基因是否存在靶向调控关系的生物学实验方法。这表明了本文模型预测的结果存在生物学实验的支持,印证了本文提出的TransformerMGI模型在microRNA与基因靶向关系预测方面的有效性。
3 结论1) 利用GP-GCN方法和DNA2Vec模型提取数据特征,提出了一个新的深度学习模型TransformerMGI来预测microRNA和基因的靶向关系。
2) 通过性能测试,TransformerMGI的预测结果优于其他深度学习方法,且部分预测结果已被生物实验证实。
3) TransformerMGI可以为后续microRNA靶向基因方面的研究提供更有价值的研究思路。
| [1] |
PU Mengfan, CHEN Jing, TAO Zhouteng, et al. Regulatory network of miR-NA on its target: Coordination between transcriptional andpost-transcriptional regulation of gene expression[J]. Cellu-lar and Molecular Life Sciences, 2019, 76: 441-451. DOI:10.1007/s00018-018-2940-7 (  0) 0) |
| [2] |
STAVAST C J, ERKELAND S J. The non-canonical aspectsof microRNAs: Many roads to gene regulation[J]. Cells, 2019, 8(11): 1465. DOI:10.3390/cells8111465 (  0) 0) |
| [3] |
ENRIGHT A, JOHN B, GAUL U, et al. MicroRNA targetsin Drosophila[J]. Genome Biology, 2003, 4: 1-27. DOI:10.1186/gb-2003-4-11-p8 (  0) 0) |
| [4] |
KOZOMARA A, BIRGAOANU M, GRIFFITHS-JONES S. miRBase: From microRNA sequences to function[J]. Nucleicacids research, 2019, 47(D1): D155-D162. DOI:10.1093/nar/gky1141 (  0) 0) |
| [5] |
KERTESZ M, IOVINO N, UNNERSTALL U, et al. The roleof site accessibility in microRNA target recognition[J]. Nature genetics, 2007, 39(10): 1278-1284. DOI:10.1038/ng2135 (  0) 0) |
| [6] |
AGARWAL V, BELL G W, NAM J W, et al. Predicting effective microRNA target sites in mammalian mRNAs[J]. elife, 2015, 4: e05005. DOI:10.7554/elife.05005 (  0) 0) |
| [7] |
STURM M, HACKENBERG M, LANGENBERGER D, et al. TargetSpy: A supervised machine learning approach formicroRNA target prediction[J]. BMC Bioinformatics, 2010, 11: 1-17. DOI:10.1186/1471-2105-11-292 (  0) 0) |
| [8] |
REYES-HERRERA P H, FICARRA E, ACQUAVIVA A, et al. miREE: miRNA recognition elements ensemble[J]. Bmc Bioinformatics, 2011, 12(1): 1-20. DOI:10.1186/1471-2105-12-454 (  0) 0) |
| [9] |
MITRA R, BANDYOPADHYAY S. MultiMiTar: A novelmulti objective optimization based miRNA-target predictionmethod[J]. PloS One, 2011, 6(9): e24583. DOI:10.1371/journal.pone.0024583 (  0) 0) |
| [10] |
MAJI R K, KHATUA S, GHOSH Z. A supervised ensem-ble approach for sensitive microRNA target prediction[J]. IEEE/ACM Transactions on Computational Biology andBioinformatics, 2018, 17(1): 37-46. DOI:10.1109/tcbb.2018.2858252 (  0) 0) |
| [11] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539 (  0) 0) |
| [12] |
CHENG Shuang, GUO Maozu, WANG Chunyu, et al. MiRTDL: A deeplearning approach for miRNA target prediction[J]. IEEE/ACM Transactions on Computational Biology and Bioinfor-matics, 2015, 13(6): 1161-1169. DOI:10.1109/tcbb.2015.2510002 (  0) 0) |
| [13] |
WEN Ming, CONG Peisheng, ZHANG Zhimin, et al. DeepMirTar: A deep-learning approach for predicting human miRNA targets[J]. Bioinformatics, 2018, 34(22): 3781-3787. DOI:10.1093/bioinformatics/bty424 (  0) 0) |
| [14] |
PLA A, ZHONG Xiangfu, RAYNER S. miRAW: A deep learn-ing-based approach to predict microRNA targets by analy-zing whole microRNA transcripts[J]. PLoS ComputationalBiology, 2018, 14(7): e1006185. DOI:10.1371/journal.pcbi.1006185 (  0) 0) |
| [15] |
ZHENG Xueming, CHEN Long, LI Xiuming, et al. Prediction of miRNAtargets by learning from interaction sequences[J]. PlosOne, 2020, 15(5): e0232578. DOI:10.1371/journal.pone.0232578 (  0) 0) |
| [16] |
中国计算机学会. 基于序列信息的microRNA和gene关系对预测[EB/OL]. (2021-09-24)[2023-05-12]. https://datafountain.cn/competitions/534/datasets. China Computer Federation, Prediction of microRNA and gene relationship based on sequence information[EB/OL](2021-09-24)[2023-05-12], https://datafountain.cn/competitions/534/datasets. (  0) 0) |
| [17] |
HAFNER M, LANDTHALER M, BURGER L, et al. Tran-scriptome-wide identification of RNA-binding protein andmicroRNA target sites by PAR-CLIP[J]. Cell, 2010, 141(1): 129-141. DOI:10.1016/j.cell.2010.03.009 (  0) 0) |
| [18] |
DING Jun, LI Xiaoman, HU Haiyan. TarPmiR: A new approach for mi-croRNA target site prediction[J]. Bioinformatics, 2016, 32(18): 2768-2775. DOI:10.1093/bioinformatics/btw318 (  0) 0) |
| [19] |
WANG Xiaowei. Improving microRNA target prediction by model-ing with unambiguously identified microRNA-target pairsfrom CLIP-ligation studies[J]. Bioinformatics, 2016, 32(9): 1316-1322. DOI:10.1093/bioinformatics/btw002 (  0) 0) |
| [20] |
GONG Jing, TONG Yin, ZHANG Hongmei, et al. Genome-wide i-dentification of SNPs in microRNA genes and the SNPeffects on microRNA target binding and biogenesis[J]. Hu-man Mutation, 2012, 33(1): 254-263. DOI:10.1002/hu-mu.21641 (  0) 0) |
| [21] |
MARCAIS G, KINGSFORD C. A fast, lock-free approachfor efficient parallel counting of occurrences of k-mers[J]. Bioinformatics, 2011, 27(6): 764-770. DOI:10.1093/bioinformatics/btr011 (  0) 0) |
| [22] |
EDGAR R. Syncmers are more sensitive than minimizers forselecting conserved k mers in biological sequences[J]. PeerJ, 2021, 9: e10805. DOI:10.7717/peerj.10805 (  0) 0) |
| [23] |
MARUYAMA O, LI Y, NARITA H, et al. CMIC: predic-ting DNA methylation inheritance of CpG islands with em-bedding vectors of variable-length k-mers[J]. BMC Bioin-formatics, 2022, 23(1): 1-20. DOI:10.1186/s12859-022-04916-3 (  0) 0) |
| [24] |
MIKOLOV T, CHEN K, CORRADO G, et al. Efficient es-timation of word representations in vector space[J/OL]. arXiv preprint arXiv: 1301.3781, 2013. https://arxiv.org/abs/1301.3781. DOI: 10.48550/arXiv.1301.3781.
(  0) 0) |
| [25] |
WANG Ruohan, NG Y K, ZHANG X, et al. A graph representa-tion of gapped patterns in phage sequences for graph convo-lutional network[J/OL]. bioRxiv, 2022: 2022. 08. 22.504727. https://www.biorxiv.org/content/10.1101/2022.08.22.504727v2. DOI: 10.1101/2022.08.22.504727.
(  0) 0) |
| [26] |
BA J L, KIROS J R, HINTON G E. Layer normalization[J/OL]. arXiv preprint arXiv: 1607.06450, 2016. https://arxiv.org/abs/1607.06450. DOI: 10.48550/arXiv.1607.06450.
(  0) 0) |
| [27] |
IOFFE S, SZEGEDY C. Batch normalization: Acceleratingdeep network training by reducing internal covariate shift[C]//International conference on machine learning. pmlr, 2015: 448-456. DOI: 10.48550/arXiv.1502.03167.
(  0) 0) |
| [28] |
NG P. dna2vec: Consistent vector representations of varia-ble-length k-mers[J/OL]. arXiv preprint arXiv: 1701.06279, 2017. https://arxiv.org/abs/1701.06279. DOI: 10.48550/arXiv.1701.06279.
(  0) 0) |
| [29] |
VASWANI A, SHAZEER N, PARMAR N, et al. Attentionis all you need[J/OL]. Advances in neural information pro-cessing systems, 2017, 30. https://arxiv.org/abs/1706.03762. DOI: 10.48550/arXiv.1706.03762.
(  0) 0) |
| [30] |
SHEN Sheng, YAO Zhewei, GHOLAMI A, et al. Powernorm: Re-thinking batch normalization in transformer[C]//Interna-tional Conference on Machine Learning. PMLR, 2020: 8741-8751. DOI: 10.48550/arXiv.2003.07845.
(  0) 0) |
| [31] |
RADIUK P M. Impact of training set batch size on the per-formance of convolutional neural networks for diverse data-sets[J]. Information Technology and Management Science, 2017, 20(1): 20-24. DOI:10.1515/itms-2017-0003 (  0) 0) |
| [32] |
WONG K K. DNMT1: A key drug target in triple-negativebreast cancer[C]//Seminars in cancer biology. AcademicPress, 2021, 72: 198 - 213. DOI: 10.1016/j.semcancer.2020.05.010.
(  0) 0) |
| [33] |
CARRINGTON A M, FIEGUTH P W, QAZI H, et al. A new concordant partial AUC and partial c statistic for imbal-anced data in the evaluation of machine learning algorithms[J]. BMC Medical Informatics and Decision Making, 2020, 20: 1-12. DOI:10.1186/s12911-019-1014-6 (  0) 0) |
| [34] |
HU Xia, CHU Lingyang, PEI Jian, et al. Model complexity of deeplearning: A survey[J]. Knowledge and Information Sys-tems, 2021, 63: 2585-2619. DOI:10.1007/s10115-021-01605-0 (  0) 0) |
| [35] |
MIN S, LEE B, YOON S. TargetNet: Functional microR-NA target prediction with deep neural networks[J]. Bioin-formatics, 2022, 38(3): 671-677. DOI:10.1093/bioin-formatics/btab733 (  0) 0) |
| [36] |
LAI X, EBERHARDT M, SCHMITA U, et al. Systems bi-ology-based investigation of cooperating microRNAs asmonotherapy or adjuvant therapy in cancer[J]. NucleicAcids Research, 2019, 47(15): 7753-7766. DOI:10.1093/nar/gkz638 (  0) 0) |
| [37] |
XU Shan, ZHANG Haibao, LIU Tianjie, et al. G Protein γ subunit 7 losscontributes to progression of clear cell renal cell carcinoma[J]. Journal of Cellular Physiology, 2019, 234(11): 20002-20012. DOI:10.1002/jcp.28597 (  0) 0) |
| [38] |
TAN Cheng, QIAN Xia, GUAN Zhifeng, et al. Potential biomarkers foresophageal cancer[J]. Springerplus, 2016, 5: 1-7. DOI:10.1186/s40064-016-2119-3 (  0) 0) |
| [39] |
TENG Ying, YU Xiying, YUAN Hui, et al. DNMT1 ablation sup-presses tumorigenesis by inhibiting the self-renewal of e-sophageal cancer stem cells[J]. Oncotarget, 2018, 9(27): 18896. DOI:10.18632/oncotarget.24116 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


