转座子为基因组中可以改变自身位置的独特DNA片段。研究表明,转座子几乎存在于所有生物的基因组中,可以发生转座并不断扩张,是基因组扩张的决定性因素,同时也对生物基因组结构和进化有着重要的影响[1]。原核转座子主要分为插入序列(Insertion sequence, IS),复合转座子及TnA家族。IS成员众多、结构简单,仅携带与转座和调节有关的基因[2]。其中IS200/IS605家族广泛分布于细菌和古菌,迄今已鉴定出153余种成员[3]。IS200作为家族创建者,最早发现于鼠伤寒沙门氏菌(Salmonella typhimurium)[4]。研究表明,IS200有着稳定的分布和高拷贝数量[2]。IS605最早发现于幽门螺旋杆菌(Helicobacter pylori),其编码的基因与IS200转座酶同源,因此统称为IS200/IS605家族[5]。该家族分子结构包括转座子左侧末端(Left end, LE)、右侧末端(Right end, RE),转座酶TnpA以及TnpB蛋白。TnpA不具有经典的IS转座酶催化结构域DDE特征,而是HuH核酸内切酶家族的一员,包含一个保守的氨基酸三联体,由组氨酸(H)-巨型疏水残基(u)-组氨酸(H)构成。TnpA转座酶能够切割和连接单链DNA,其催化中心为单个酪氨酸(Y),因此又称为Y1转座酶[3]。与CRISPR-Cas系统中的Cas9和Cas12不同,Y1转座酶不需要RNA引导转座过程。相反,Y1通过自身的结构域识别并选择其靶标DNA,并通过嵌入DNA和剪接目标DNA来实现转座。LE和RE含有回文序列,可形成亚末端发卡结构[6]。Y1特异地识别并结合这些短的末端二级结构,在特异位点剪切,形成环形单链DNA中间体,然后将其3’端插入目标单链DNA上富含AT的四核苷酸或五核苷酸位点,而5’端插入位点无特异性,靶位点不发生复制[3]。
IS200/IS605家族编码多种RNA导向的核酸酶,目前已发现的包括IscB家族、IsrB家族和TnpB家族[7]。研究表明,CRISPR-Cas9起源于IscB[8];TnpB与IscB进化关系较远,被认为是Cas12的祖先[9-10]。另外,TnpB可能也是一种较大蛋白质Fanzors的祖先,这种蛋白质被发现于多种真核转座子[11]。目前这三种核酸酶相关转座子系统的生物学功能仍未知,但推测这些核酸酶有利于Y1等转座酶催化反应、RNA导向的转座,或者和转座子一起发挥抗毒素等作用,从而确保IS200/IS605插入基因组[7]。此外,也有研究发现,在耐辐射球菌以及大肠杆菌中,TnpB对ISDra2的切除具有抑制作用[12]。
目前研究表明,IS200/IS605超家族分布广泛,但其在大肠杆菌(Escherichia coli)和沙门氏菌(Salmonella enterica)的分布种类、数量、结构和进化特性仍不明确,尤其是Y1关联转座子的挖掘,对于开发遗传编辑和流行病学研究工具潜力巨大。本研究分析了Y1关联转座子系统在E. coli和S. ente的遗传结构进化,可为挖掘RNA导向的转座子提供重要参考。
1 材料与方法 1.1 Y1关联转座子挖掘在美国国家生物技术信息中心(NCBI)网站(https://www.ncbi.nlm.nih.gov/)所提供的whole-genome shotgun conting数据库(WGS)中下载细菌蛋白NR(非冗余)序列,收集ISfinder(https://www-is.biotoul.fr/blast.php)中TnpA转座酶序列作为参考序列,使用Hmmsearch软件(v3.3.2)搜寻所有符合条件的Y1转座酶蛋白序列,设置E期望值为1×10-4。对于Y1转座酶含量最高的E. coli和S. ente,使用Usearch程序对所获序列进行聚类,要求相似性>80%,并获得代表序列的CDS序列。在Hmmsearch软件中根据代表CDS序列收集获得E. coli及S. ente的所有基因组拷贝(E期望值为1×10-4),前后延伸1.8 kb侧翼序列以保证序列的完整性。
1.2 插入序列识别使用MAFFT程序进行多重序列比对[13],将Y1相关转座子进行分类。在ISfinder网站提供的数据库中进行序列Blast比对,通过与注释序列的比对获得转座子信息。使用BioEdit软件(v7.2.0)确定转座子的边界并进行序列截取[14],前后保留50 bp以研究其插入位点及侧翼序列特征。对于部分相似性高却未能在ISfinder上找到注释序列的转座子,使用Hmmscan网站(https://www.ebi.ac.uk/Tools/hmmer/search/hmmscan)对其蛋白结构域进行分析,以确定其分类。若序列开放阅读框(orf)无Y1同源性,则舍弃该序列。将具有完整LE、RE且能够编码100 aa以上转座酶的序列视为完整转座子(IS605中TnpA>100 aa,TnpB>300 aa)。
1.3 转座子结构预测与进化分析使用Bioedit软件提取多拷贝转座子的LE,RE以及CDS。其中LE与RE使用EMBOSS explorer网站(https://www.bioinformatics.nl/emboss-explorer)构建一致序列,并在Oligo Analyzer网站(https://sg.idtdna.com/pages/tools/oligoanalyzer)中预测转座子DNA序列末端二级结构。通过BioEdit软件将CDS翻译为Y1转座酶和TnpB蛋白的序列,然后使用EMBOSS explorer网站构建它们的一致序列。同时根据得到的所有Y1转座酶蛋白序列,在Weblogo(https://weblogo.threeplusone.com/create.cgi)网站中绘制序列Logo图,并对其进行结构与变异位点分析。使用IBS软件(v1.0.3)绘制转座子结构[15]。通过Alpha-Fold网站(https://colab.research.google.com/github/sokrypton/ColabFold/blob/main/AlphaFold2.ipynb?authuser=0#scrollTo=kOblAo-xetgx)预测Y1转座酶结构,使用PyMol软件(v2.5.5)对其关键氨基酸残基及结构进行标注。
2 结果与分析 2.1 Y1ATs广泛分布于E. coli和S. ente采用上述Hmmsearch序列收集方法,在2 467个E. coli基因组中挖掘到8 645条Y1关联序列(Y1ATs),分布于2 389个基因组(占比96.84%),其中序列完整的Y1ATs占55.62%;在1 495个S. ente基因组中收集到8 316条Y1ATs序列,分布于1 202个基因组(占比80.4%),完整序列占比高达93.13%(表 1)。Y1ATs在E. coli中的含量略高于S. ente,但在S. ente中的完整序列比例远高于E. coli。此外,相对于E. coli,Y1ATs在S. ente基因组中有着更高的拷贝数(表 1)。
| 表 1 E. coli和S. ente中Y1ATs的分布概况 Table 1 Distribution overview of Y1ATs in E. coli and S. ente |
通过ISfinder已注释序列比对, 以及Hmmscan网站蛋白结构域预测,剔除非Y1相关序列后,在E. coli和S. ente共鉴定到了10类Y1ATs,均属于IS200/IS605超家族。2类为IS200(IS200C和IS200F),6类为IS605(IS609, ISEc46, ISEc41, ISSen6, ISEc44和ISKpn69)。其中7种含有完整拷贝(E. coli中的IS200C, IS609, ISEc46, ISEc44, ISEc41以及S. ente中的IS200F, ISSen6),1种仅有残缺拷贝(ISKpn69)。还有两类序列在ISFinder中尚未注释,但在基因组中有着较高的序列一致性,长度在2 500 bp左右。虽然在ISfinder网站的Blast比对中难以找到其同源序列,但通过Hmmscan网站的蛋白分析,发现这两个类群的序列都包含Y1转座酶,ISEc94包含一个165 aa的Y1蛋白和一个458 aa的FIhA家族蛋白,而ISEc95包含一个200 aa的Y1, Y2融合蛋白和一个343 aa的dipZ家族蛋白。因此将这两个类群的序列判定为新的Y1关联插入序列,根据ISfinder的命名规则将其命名为ISEc94和ISEc95,拷贝数分别为466和21,由于两者左右末端序列尚不清楚,难以界定是否有完整拷贝。
分析汇总含Y1完整拷贝的IS200和IS605转座子,发现在E. coli的2 467个基因组中89.38%的基因组含有IS605,33.04%的基因组含有IS200,642个基因组共同存在着IS200和IS605转座子(图 1(a))。E. coli基因组中存在4种IS605转座子的重叠插入(图 1(b))。在S. ente的1 495个基因组中,含IS200的基因组占比达到72.24%,含IS605的基因组占20.27%,195个基因组同时存在IS200和IS605转座子(图 1(c))。
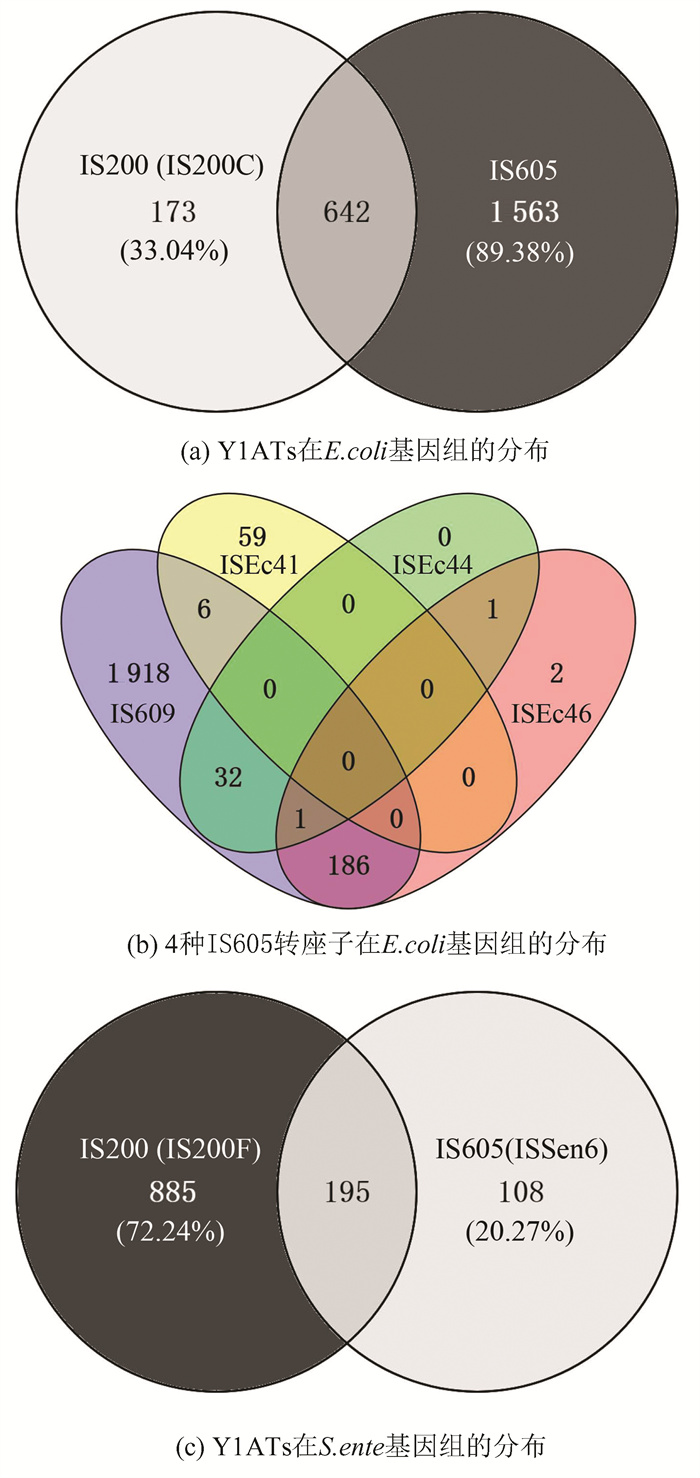
|
图 1 Y1ATs完整拷贝在E. coli和S. ente基因组的分布概况 Figure 1 The distribution overview of complete Y1ATs copies in the genomes of E. coli and S. ente 注:重叠部分表示含有两种或多种Y1变体的基因组. |
统计分析IS200和IS605在E. coli和S. ente基因组中的平均拷贝数,结果如图 2所示,IS200的基因组拷贝显著高于IS605。IS200在S. ente和E. coli基因组平均拷贝分别为7.2和4.75。IS605的平均拷贝均在1左右。根据拷贝数分布图可知,除IS200在少数基因组存在较高拷贝(约35),Y1ATs在大多数基因组中的拷贝数均较低(图 3)。
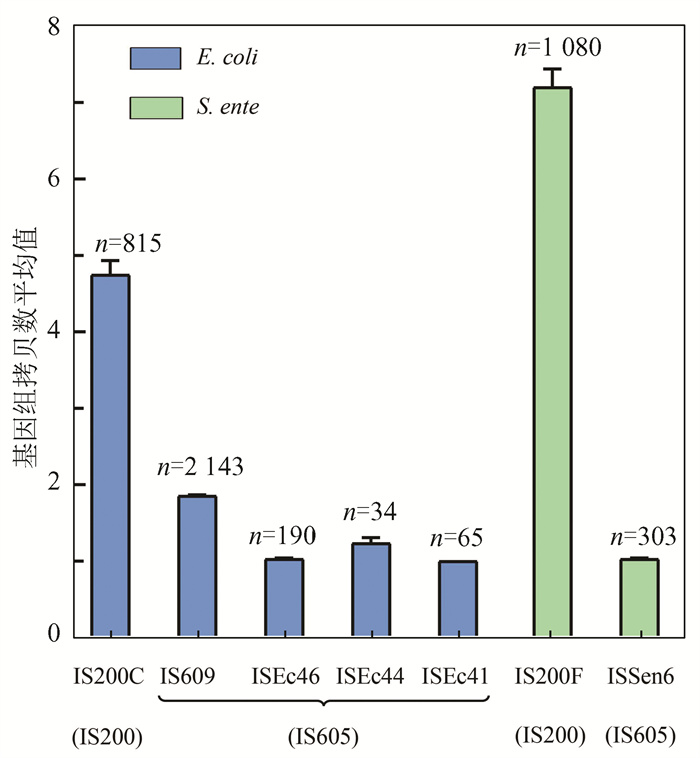
|
图 2 IS200和IS605转座子在两个菌属的平均拷贝数比较 Figure 2 Comparison of the average copy number of IS200 and IS605 transposons in two bacterial genera 注:X轴代表Y1ATs,Y轴代表Y1ATs在基因组中拷贝数的平均值;n表示基因组数量. |
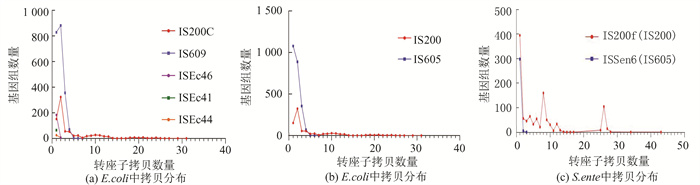
|
图 3 E. coli和S. ente中Y1ATs各家族拷贝分布 Figure 3 Copy distribution of Y1ATs families in E. coli and S. ente 图(a)中IS200C属于IS200家族,IS609、ISEc46、ISEc41、ISEc44属于IS605家族. |
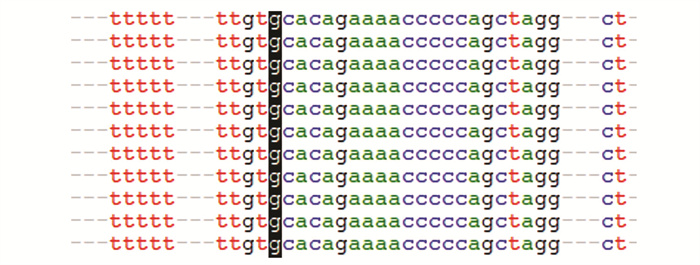
IS200C和IS200F全长大多在710 bp左右,中间为Y1转座酶编码序列(152 aa)(图 4(a))。个别序列会因为插入外源序列从而导致长度达到2 287 bp,编码的蛋白也增加到378 aa(表 2)。两端结构分为左末端(The left IS end, LE)和右末端(The right IS end, RE),均含有回文序列,形成发卡和茎环结构,而不是经典IS的末端反向重复序列(TIR)(图 4(b),图 5)。IS200C与IS200F左侧切割位点分别为TTGT和TTTT,未包含在转座子中,位于LE的左侧;右侧切割位点均为TTAT,包含在转座子中,位于RE末端。根据序列比对结果,推断IS200 3’端偏好插入T富集区(图 6)。
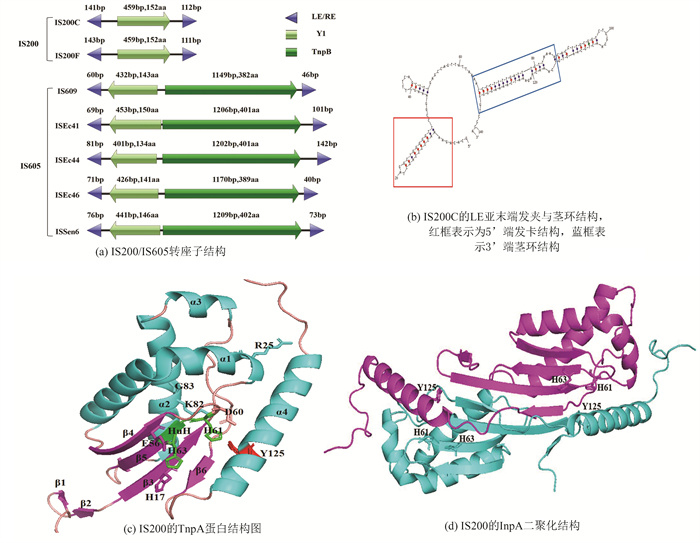
|
图 4 IS200和IS605家族转座子结构示意图 Figure 4 Schematic diagram illustrating the structure of the IS200 and IS605 transposon families 注:图(c)中α螺旋和β折叠分别用蓝色、粉色标出.绿色代表HuH基序,红色代表保守的Y125酪氨酸残基. |
| 表 2 E. coli和S. ente中Y1ATs的分类和结构组成 Table 2 Classification and structural composition of Y1ATs in E. coli and S. ente |

|
图 5 IS200C、IS200F的LE亚末端发夹与茎环结构DNA序列图 Figure 5 DNA sequence diagram of LE subterminal hairpin and stem-loop structure of IS200C and IS200F 注:蓝色部分配对形成发卡结构,绿色部分配对形成茎环结构. |
|
图 6 IS200转座子左侧末端LE的偏好插入位点 Figure 6 The preferred insertion site of LE at the left end of IS200 transposon 注:黑色区域为IS200转座子序列起始位置,ttgt为左侧切割位点,LE偏好插入T富集区. |
IS609, ISEc41, ISEc44, ISEc46和ISSen6的完整拷贝总长为1 748-1 879 bp,中间为Y1和TnpB两个基因的编码序列,两者方向反向且无重叠序列(图 4(a))。两端分别为LE和RE。Y1编码约143 aa,TnpB编码约400个aa。由于插入突变与重组的存在,个别转座子长达3 220 bp,Y1编码蛋白可增加到332个aa。ISSen6、ISEc44与ISEc41三者左、右侧切割位点相同,分别为CCAT和TCAA。IS609左、右侧切割位点分别为TTAT, TCAA;ISEc46左右切割位点分别为TTAG, TCAC(表 2)。
根据已报道的Y1转座酶晶体结构,们通过Alpha-Fold网站以及PyMol软件对拷贝数及序列完整性较高的IS200C和IS200F进行了Y1转座酶蛋白结构图绘制和关键氨基酸残基的标注(图 4(c)、4(d))。Y1转座酶的HuH基序由H61和H63组成,位于链β5上(图 4(c))。螺旋α4上的Y125是Y1转座酶中唯一严格保守的酪氨酸残基。HuH基序和Y125是TnpA与TIR结合的核心结构域,它们形成催化位点,对于催化DNA单链中磷酸二酯键的断裂至关重要[16]。E56、H63和四个水分子与Mn2+离子配位,形成催化结构中必须的金属配位球[17]。H17, D60和H61在维持金属结合位点方面也发挥着重要作用。K82和G83对于茎环DNA的结合至关重要。此外,保守的R25可能在Y1转座酶发生构象变化时与DNA相互作用,因此可能是必不可少的[16]。在转座过程中,Y1转座酶通过β折叠的合并形成二聚体(图 4(d))。
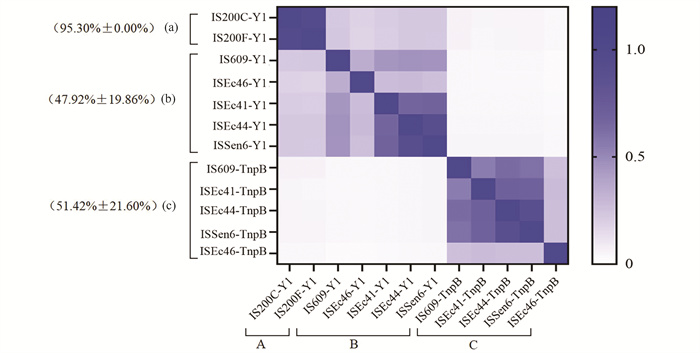
2.4 Y1和TnpB蛋白的系统进化分析使用EMBOSS explorer网站构建E. coli和S. ente中IS200/IS605超家族的Y1转座酶及TnpB蛋白一致序列(所用序列均为完整拷贝),并通过Bioedit软件计算各一致序列间相似性,结果如图 7所示。IS200家族Y1序列相似性高达95.3%,说明其在进化过程中转座酶区域高度保守,可能来自同一个古老祖先。IS605家族Y1平均相似性为47.92%±19.86%(26.8%~90.2%),TnpB蛋白序列平均相似性为51.42%±21.6%(25.9%~88.7%),提示两者遗传多样性均较高。此外,由表 2可以看出,IS200在基因组中的衍生以完整拷贝为主,而IS605在基因组中的衍生以残缺拷贝居多。使用WebLogo网站绘制完整拷贝IS200的Y1转座酶一致序列变异Logo图,其中IS200C约3 000条,遗传组成相对较为多样;IS200F约7 000条,序列一致性较高。序列间变异情况见图 8。
|
图 7 E. coli和S. ente中的Y1转座酶和TnpB蛋白进化特性 Figure 7 Evolutionary characteristics of Y1 transposases and TnpB proteins in E. coli and S. ente 注:(a)代表IS200的Y1转座酶相似性; (b)代表IS605的Y1转座酶相似性; (c)代表TnpB蛋白相似性.蓝色从深到浅表示序列相似性从高到低,序列平均相似性在左侧标出. |

|
图 8 IS200的Y1转座酶变异Logo图 Figure 8 Logo diagram of IS200 Y1 transposases mutation 注:字母大小表示其在序列中的占比. |
本研究结果表明,Y1ATs广泛分布于E. coli和S. ente两个菌属(占比>80%),提示Y1ATs存在大规模传播,并可能存在水平传播现象。IS200和IS605的拷贝数在两个菌属存在着显著差异,IS200在S. ente基因组中传播更广泛,而IS605在E. coli基因组中传播更为广泛,这说明在E. coli和S. ente中存在着Y1ATs进化动力学的差异[18]。IS通过基因组重排和有益基因的转移增加了遗传多样性和基因组可塑性[19]。IS200在基因组上的拷贝数显著高于IS605,其完整拷贝数也显著高于IS605。但研究表明IS200对其原始宿主沙门氏菌造成的自发变异很小,转座较为罕见[20]。原因之一可能是由于IS200LE端发夹结构与启动子重叠,导致了启动子的低表达[21]。IS200最初被视为惰性转座子的典例,但它在细菌属中的分布表明其有很长的进化历史[22],推测是IS200低频次的转座给自身提供了一定的进化优势。IS200的稳定分布与高拷贝与前人的研究结果一致,其高丰度提示可能存在功能性转座元件,为进一步挖掘活性Y1ATs奠定基础。
转座子的拷贝数与转座子的进化模式及插入年龄有重要关系[23]。在物种进化过程中,伴随着时间的推移,转座子累积了突变、重排等,从而导致转座酶的逐渐失活,转座子的拷贝数也随之降低[24]。IS200C与IS200F较高的序列一致性(95.3%)表明其很有可能由同一个祖先进化而来(图 7)。相较IS200C,IS200F在转座酶区域更为保守(图 8),这一定程度上解释了IS200F的基因组拷贝数量高于IS200C的现象。IS200转座子在转座酶区域较为保守,这一点可以作为IS200是活性转座子的依据之一。相比而言,IS605累积了较多的突变、缺失和重组。IS609和ISSen6分别是E. coli和S. ente两个物种中含量最高的IS605转座子,但它们的残缺拷贝数量甚至都超过了完整拷贝(表 2),推测是IS605转座子在进化过程中的不断突变与非法重组导致的。
进一步研究单编码框IS200家族和双编码框IS605家族,有利于了解细菌转座元件进化过程、转座及调控机制。本研究将E. coli和S. ente中Y1ATs共分为10类,IS200仅编码Y1转座酶,是已知最小的自主IS。IS605则编码Y1和TnpB。研究表明,Y1催化IS的裂解、连接等转座活动,而TnpB对转座活动的作用机制尚不清楚,甚至可能起抑制作用[12],这可能与IS605低拷贝特性相关。研究表明TnpB具有RNA导向的核酸酶活性,本研究对其序列结构的遗传进化进行了初步分析,为挖掘靶向整合转座子提供参考。
在序列的Mafft多重比对中,发现E. coli中的ISEc94, ISEc95高度相似,但这些序列在ISfinder的Blast比对中难以找到相应的注释序列。将这部分序列翻译为蛋白质,在Hmmscan网站上进行转座酶蛋白序列的比对,比对结果显示这些序列编码>100 aa的Y1蛋白,属于IS200家族成员。由此可见,一些Y1关联转座子难以被插入序列注释软件精准识别,有着未经注释的独特序列片段,此现象一定程度上表明了序列间可能正在发生着重组[25]。
4 结论本研究在E. coli和S. ente两个菌属中挖掘到大量Y1关联转座子,其在进化关系上隶属于IS200/IS605家族。IS200和IS605存在不同的进化模式,IS200存在较多拷贝及完整拷贝,且各亚家族间Y1转座酶序列高度保守;IS605完整拷贝数较低,各亚家族间Y1转座酶和TnpB变异较大。本研究为揭示原核生物转座子进化机制和进一步挖掘活性Y1关联转座子提供重要参考。
| [1] |
沈丹, 陈才, 王赛赛, 等. Tc1/Mariner转座子超家族的研究进展[J]. 遗传, 2017, 39(1): 13. SHEN Dan, CHEN Cai, WANG Saisai, et al. Research progress on the Tc1/Mariner transposon superfamily[J]. Genetics, 2017, 39(1): 13. DOI:10.16288/j.yczz.16-160 (  0) 0) |
| [2] |
BEUZÓN C R, CHESSA D, CASADESÚS J. IS200: An old and still bacterial transposon[J]. International Microbiology, 2004, 7(1): 3-12. DOI:10.2436/im.v7i1.9438 (  0) 0) |
| [3] |
HE S, CORNELOUP A, GUYNET C, et al. The IS200/IS605 family and "peel and paste" single-strand transposition mechanism[J]. Microbiology Spectrum, 2015, 3(4): 609-630. DOI:10.1128/microbiolspec.MDNA3-0039-2014 (  0) 0) |
| [4] |
LAM S, ROTH J R. IS200: A salmonella-specific insertion sequence[J]. Cell, 1983, 34(3): 951-960. DOI:10.1016/0092-8674(83)90552-4 (  0) 0) |
| [5] |
KERSULYTE D, AKOPYANTS N S, CLIFTON S W, et al. Novel sequence organization and insertion specificity of IS605 and IS606: Chimaeric transposable elements of Helicobacter pylori[J]. Gene, 1998, 223(1-2): 175-186. DOI:10.1016/s0378-1119(98)00164-4 (  0) 0) |
| [6] |
BEUZÓN C R, CASADESÚS J. Cloning with Mud-P22 hybrid prophages: Mapping of IS200 elements on the chromosome of Salmonella typhimurium LT2[J]. Molecular and General Genetics, 1997, 256(5): 586-588. DOI:10.1007/s004380050605 (  0) 0) |
| [7] |
ALTAETRAN H, KANNAN S, DEMIRCIOGLU F E, et al. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonu-cleases[J]. Science, 2021, 374(6563): 57-65. DOI:10.1126/science.abj6856 (  0) 0) |
| [8] |
KAPITONOV V V, MAKAROVA K S, KOONIN E V. ISC, a novel group of bacterial and archaeal DNA transposons that encode Cas9 homologs[J]. Journal of Bacteriology, 2016, 198(5): 797-807. DOI:10.1128/JB.00783-15 (  0) 0) |
| [9] |
GUERILLOT R, SIGUIER P, GOURBEYRE E, et al. The diversity of prokaryotic DDE transposases of the mutator superfamily, insertion specificity, and association with conjugation machineries[J]. Genome Biology and Evolution, 2014, 6(2): 260-272. DOI:10.1093/gbe/evu010 (  0) 0) |
| [10] |
SHMAKOV S, SMARGON A, SCOTT D, et al. Diversity and evolution of class 2 CRISPR-Cas systems[J]. Nature Reviews Microbiology, 2017, 15(3): 169-182. DOI:10.1038/nrmicro.2016.184 (  0) 0) |
| [11] |
BAO W, JURKA J. Homologues of bacterial TnpB_IS605 are widespread in diverse eukaryotic transposable elements[J]. Mobile DNA, 2013, 4(1): 12. DOI:10.1186/1759-8753-4-12 (  0) 0) |
| [12] |
PASTERNAK C, DULERMO R, TON-HOANG B, et al. ISDra2 transposition in Deinococcus radiodurans is downregulated by TnpB[J]. Molecular Microbiology, 2013, 88(2): 443-455. DOI:10.1111/mmi.12194 (  0) 0) |
| [13] |
TORRE E, THRELFALL E J, HAMPTON M D, et al. Characterization of Salmonella virchow phage types by plasmid profile and IS200 distribution[J]. Journal of Applied Bacteriology, 1993, 75(5): 435-440. DOI:10.1111/j.1365-2672.1993.tb02799.x (  0) 0) |
| [14] |
YANG Peng, CRAIG P A, GOODSELL D, et al. BioEditor-simplifying macro-molecular structure annotation[J]. Bioinformatics, 2003, 19(7): 897-898. DOI:10.1093/bioinformatics/btg103 (  0) 0) |
| [15] |
LIU Wenzhong, XIE Yubin, MA Jiyong, et al. IBS: an illustrator for the presentation and visualization of biological sequences[J]. Bioinformatics, 2015, 31(20): 3359-3361. DOI:10.1093/bioinformatics/btv362 (  0) 0) |
| [16] |
RONNING D R, GUYNET C, TON-HOANG B, et al. Active site sharing and subterminal hairpin recognition in a new class of DNA transposases[J]. Molecular Cell, 2005, 20(1): 143-154. DOI:10.1016/j.molcel.2005.07.026 (  0) 0) |
| [17] |
LEE H H, YOON J Y, KIM H S, et al. Crystal structure of a metal ion-bound IS200 transposase[J]. The Journal of Biological Chemistry, 2006, 281(7): 4261-4266. DOI:10.1074/jbc.M511567200 (  0) 0) |
| [18] |
LIU Yibing, ZONG Wencheng, DIABY M, et al. Diversity and evolution of pogo and Tc1/mariner transposons in the apoidea genome[J]. Biology, 2021, 10(9): 940. DOI:10.3390/BIOLOGY10090940 (  0) 0) |
| [19] |
CERVEAU N, LECLERCQ S, BOUCHON D, et al. Evolutionary dynamics and genomic impact of prokaryote transposable elements[M]. Berlin: Springer-Verlag Berlin Heidelberg, 2011: 291-312. DOI:10.1007/978-3-642-20763-1_17
(  0) 0) |
| [20] |
LAM S, ROTH J R. Structural and functional studies of insertion element IS-200[J]. Journal of Molecular Biology, 1986, 187(2): 157-167. DOI:10.1016/0022-2836(86)90225-1 (  0) 0) |
| [21] |
CALVA E, ORDOÑEZ L G, FERNANDEZ-MORA M, et al. Distinctive IS200 insertion between gyrA and rcsC genes in Salmonella typhi[J]. Journal of Clinical Microbiology, 1997, 35(12): 3048-3053. DOI:10.1128/jcm.35.12.3048-3053.1997 (  0) 0) |
| [22] |
VAN-VALEN L. Evolutionary Genetics[J]. Science, 1962, 138(3538): 424. DOI:10.1126/science.138.3538.424 (  0) 0) |
| [23] |
沈丹. Tc1/mariner转座子挖掘, 高活性成员鉴定及其在增强子捕获中的应用[D]. 扬州: 扬州大学, 2021. DOI: 10.27441/d.cnki.gyzdu.2020.000081. SHEN Dan. Tc1/Mariner transposon mining, high-activity member identification and its application in enhancer capture[D]. Yangzhou: Yangzhou University, 2021. DOI: 10.27441/d.cnki.gyzdu.2020.000081. (  0) 0) |
| [24] |
HE Susu, GUYNET C, SIGUIER P, et al. IS200/IS605 family single-strand transposition: mechanism of IS608 strand transfer[J]. Nucleic Acids Research, 2013, 41(5): 3302-3313. DOI:10.1093/nar/gkt014 (  0) 0) |
| [25] |
SADLER M, MORMILE M R, FRANK R L. Characterization of the IS200/IS605 insertion sequence family in halanaerobium hydrogeniformans[J]. Genes, 2020, 11(5): 484. DOI:10.3390/genes11050484 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


