癌症是人类健康的第一大杀手,自噬[1]作为一种治疗癌症的机制而得到广泛的关注。细胞自噬是真核生物中细胞保守的对细胞内物质进行周转的重要过程,自噬分为三类,巨自噬、微自噬与分子伴侣介导的自噬,在自噬过程中一些损坏的细胞器和蛋白被自噬小泡包裹后,送入溶酶体(动物)或者液泡(植物)中进行降解并循环利用,自噬作为肿瘤的促进因子[2],在使用化学疗法进行抗肿瘤治疗时对肿瘤进程的影响发挥极其重要的作用,抑制自噬能增强化疗抗恶性细胞增生的作用。而其中自噬通路PI3K/akt/mtor作为影响自噬的重要通路在自噬的发生过程中起到了不可或缺的作用[3]。PI3K/akt/mtor通路是人类癌症中最常见的失调信号通路之一[4],PI3K是一种调节细胞生长、增殖、存活, 运动和代谢的脂质激酶[5-6],根据其结构、特异性的底物和不同调节功能,大致可以分为三类[7]。其中I类PI3K又分为PI3Kα,PI3Kβ,PI3Kδ和PI3Kγ。到目前为止,已有四种PI3K抑制剂被批准用于癌症治疗,PI3Kδ抑制剂(Idelalisib)是2014年第一个被批准与利妥昔单抗联合治疗慢性淋巴细胞白血病的药物[8-9]。PI3Kα抑制剂(Alpelisib[10])是第一个被批准用于实体瘤治疗的PI3K抑制剂,它结合内分泌疗法治疗乳腺癌[11]。随着多种临床实验研究的广泛报导,PI3Kα被证实是一种有前途的肿瘤治疗靶点,新型PI3Kα抑制剂的开发也取得了重大进展。
植物骆驼蓬[12](Peganum harmala L.)是一种主要分布于中国新疆、内蒙古及西北部沙漠地区的植物,其中富含的去氢骆驼蓬碱(Harmine, HM)为β-咔啉类生物碱,被广泛应用于疾病治疗中[13]。骆驼蓬种子提取物对中风、哮喘、黄疸, 腰痛等多种疾病均有显著的治疗效果[14]。有研究表明,HM具有抗肿瘤、抗阿尔茨海默、抗炎, 抗真菌和抗病毒活性[15]。HM对肿瘤的作用源自多种机制,是一种有效的多靶点药物[1-3][16-17],能够干预细胞凋亡和自噬,作用于线粒体介导的信号通路,如PI3K通路等等[18-19]。但由于大剂量的去氢骆驼蓬碱及其衍生物能够引起神经系统毒副作用,且生物利用度低,因此严重限制了其临床应用。为了减少去氢骆驼蓬碱的毒副作用,增强抗肿瘤活性及生物利用度,本研究根据Chen等[20]对去氢骆驼蓬碱母核的研究,利用ACD/Percepta软件[21]对去氢骆驼蓬碱的可修饰部位及母核结构进行结构修饰,使其对作用通路靶点蛋白PI3Kα的作用更强。
分子对接[22](Molecular docking)是基于配体与受体的几何互补与能量匹配的原则,有效确定与靶受体活性部位空间和电性特征互补匹配的小分子化合物,为寻找配体与受体最佳构象,给寻找先导化合物提供了一种可能[23],是目前创新药物的一条重要思路[24]。基于去氢骆驼蓬碱的抗肿瘤活性作用,本研究前期从去氢骆驼蓬碱的母体结构出发,根据去氢骆驼蓬碱的构效关系[25],在去氢骆驼蓬碱的第2位、7位和9位进行了结构修饰(图 1),以自噬相关通路靶点蛋白PI3Kα为特异性靶点,通过ACD/Percepta软件对PI3K通路靶点蛋白结构进行了化学结构设计以及成药性筛选,通过分子对接方式得到了结合能力前3的化合物,并使用分子动力学模拟方法对分子对接结果稳定性进行考察,确定去氢骆驼蓬碱衍生物中结合能力与稳定性更好的化合物,利用ACD/Percepta软件中的ADMET模块对这三个化合物进行筛选,选择神经毒副作用小,生物利用度高且成药性好的化合物成为去氢骆驼蓬碱结构修饰的目标化合物,为去氢骆驼蓬碱的结构修饰提供了有力的基础。
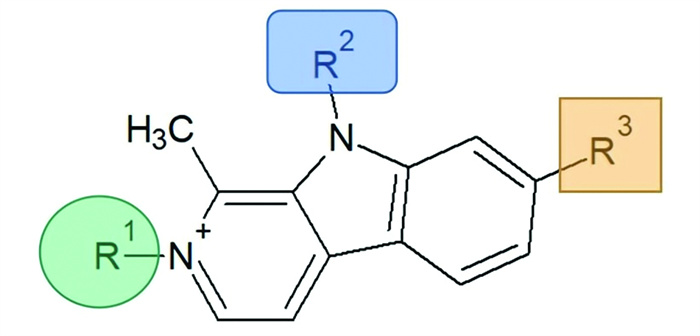
|
图 1 HM结构示意图 Figure 1 Schematic diagram of HM structure |
RCSB蛋白PDB数据库[26](罗格斯大学和加州大学圣地亚哥分校/圣地亚哥超级计算机中心)。
1.2 软件SWISS MODEL[27],在线同源建模软件,瑞士生物信息研究所。Gromacs[28]分子动力学软件,格罗宁根大学。ACD/Percepta,小分子设计软件,加拿大Advanced Chemistry Development公司。Schrdinger[29],虚拟筛选软件,德国BioLuminate公司。Discovery studio2020[30],3D-QSAR建模软件。
2 实验方法 2.1 小分子数据库生成使用PyMoL[31]分子图形系统(Schrödinger,LLC,版本2.5)提取蛋白质文件中的配体分子,并以“.pdb”格式保留以进行备份。使用ACD/Percepta软件生成有害衍生物的“.sdf”格式,并使用Schrödinger Lig-Prep模块对结构进行优化和处理,从bindingDB数据库中下载获得PI3Kα抑制剂的活性化合物,然后通过逆合成反应规则对这批化合物产生结构片段,得到片段库后,通过Percepta软件对先导化合物的取代位点进行片段组合,得到衍生物库。
2.2 虚拟筛选使用Schrödinger软件Maestro模块中的Virtual Screening Workflow进行。首先根据lipinski成药五规则,使用Qik Prop模块对小分子化合物进行初步的过滤,虚拟筛选开始之前将那些物理化学性质不具有成药性的小分子化合物过滤剔除。之后采用Glide[32]三种精度逐级进行筛选,其中高通量虚拟筛选(HTVS)保留前5%的化合物、标准精度(SP)再保留前5%的化合物、额外精度(XP)保留前两步筛选后的前5%的化合物。最后进一步应用Prime MM-GBSA方法去评估所筛选的化合物的结合自由能。
2.3 分子动力学模拟评价结合能力使用Discovery Studio 2020软件来构建PI3Kα的3D-QSAR模型,所使用的训练数据集化合物通过查阅相关文献得到。
2.4 分子动力学模拟评价结合能力用Gromacs软件对完整残基和预处理纯蛋白进行分子动力学模拟[33],蛋白力场为ff14SB。为了保持系统电中性,使用charmm gui[34]中的tleap模块将Cl-, Na+添加到基于库仑静电网格的复合系统中。体系溶解在截角八面体TIP3P水模型中,溶质周围距离设置为10 Å。将整个系统设置为500 kcal/mol, 模拟前错误的结构不能作为分子动力学模拟的起点,因此需要最小化能量,寻找能量最低点作为模拟的起点。这里用到两步能量最小化,具体方法如下:
1) 最小化水分子和抗衡离子10 000步的最速下降能量,然后最小化5 000步的共轭梯度能量。
2) 为了消除蛋白质中氨基酸的不合理结构,对整个系统进行10 000步的最速下降法,然后进行5 000步的共轭梯度能量最小化。Particle-Mesh Ewald (PME)方法用于处理周期性边界条件下的长程静电相互作用;所有涉及氢原子的共价键均受SHAKE算法约束;MD模拟的时间步长设置为2 fs,氢键距离设置为3.5 Å;在canonical ensemble(NVT)下,系统从0K缓慢升温到300 K,仿真时间为50 ps;然后在1个大气压下平衡1 ns;最后在等温等压系统(NPT)下进行100 ns模拟。
2.5 ADMET性质预测ACD/Percepta从分子结构出发,可快速、准确地预测化合物的物理化学性质、ADMET性质。用户也可以基于化合物的ADMET性质对先导化合物进行结构优化和设计,从而得到满足成药性要求的目标物,加快新药项目研发。借助于ACD/Percepta拥有独特的预测可靠性评分,避免了盲目性。本研究利用ACD/Percepta软件对所筛选出的化合物进行了ADMET预测。
3 结果 3.1 小分子数据库图 2为由ACD/Percepta生成的136 745个化合物的化合物库示意图,其中绿色为碳元素,红色为氧元素,蓝色为氮元素,白色为氢元素,黄色为硫元素。

|
图 2 化合物库分子集合 Figure 2 Compound Library Molecular Clusters |
在虚拟筛选的过程中,先由薛定谔软件中的Lig-pre模块进行了配体小分子的预处理,得到了136 745个化合物,以.sdf格式进行下一步处理;接下来对蛋白进行预处理,使用薛定谔套件2020-3版的蛋白质准备套件对蛋白质进行了预处理。以蛋白质文件4ZOP的抑制剂4Q2为中心生成对接盒子;在虚拟筛选模块中,136 745个化合物中通过HTVS(High throughput virtual screening)以5%的准入范围得到6 837个化合物进行下一步SP(Standard precision),在SP的步骤中以5%的标准从6 837中得到了341个化合物进入下一步XP(Extra precision),在XP步骤中从371个化合物中得到了17个化合物,为了从数据库中筛选配体分子,考虑了Glide ligand efficience, XP GScore, Glide ecoul, Glide emodel, Glide energy这些数据用于数据集中,进行排名按MM-GBSA ΔG从小到大排序前17的分子(表 1)。
| 表 1 根据MM GBSAΔG从小到大排列的17种化合物 Table 1 17 compounds according to mm-gbsa Δ G ordered from small to large |
为了更接近真实的情况,由XP模块产生的17个分子最终通过MM-GBSAΔG评分,筛选出MM-GBSA ΔG前3的化合物:50499, 51939和37971,蛋白质原配体与排名前3的化合物之间的相互作用残基见表 2,其化合物结构见图 3所示,其结合模式图见图 4。结合能大小分别为-77.43 kcal/mol, -77.42 kcal/mol和-75.46 kcal/mol。将准备好的蛋白质配体4Q2分别与这三个化合物进行分子对接,并对其相互作用的氨基酸残基进行分析。其中化合物37971与50499与原配体相比有共同的3个氨基酸残基建立了氢键,分别是ILE-848, ILE-932与TYR-836,而化合物51939仅与受体口袋中的TRP-780有氢键连接,见图 4,为了模拟真实环境,接下来对三个化合物进行了分子动力学评估。
| 表 2 蛋白质原配体4Q2与化合物37971, 50499, 51939的相互作用残基 Table 2 Interaction residues of protein protoligand 4Q2 and compounds 37971, 50499, 51939 |
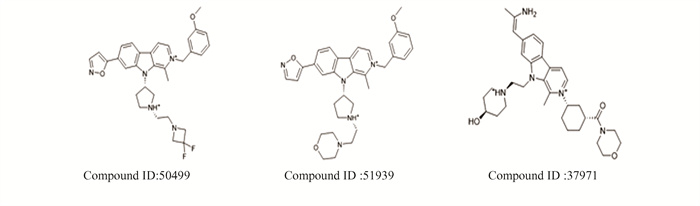
|
图 3 三种候选化合物的结构 Figure 3 Structure of three candidate compounds |
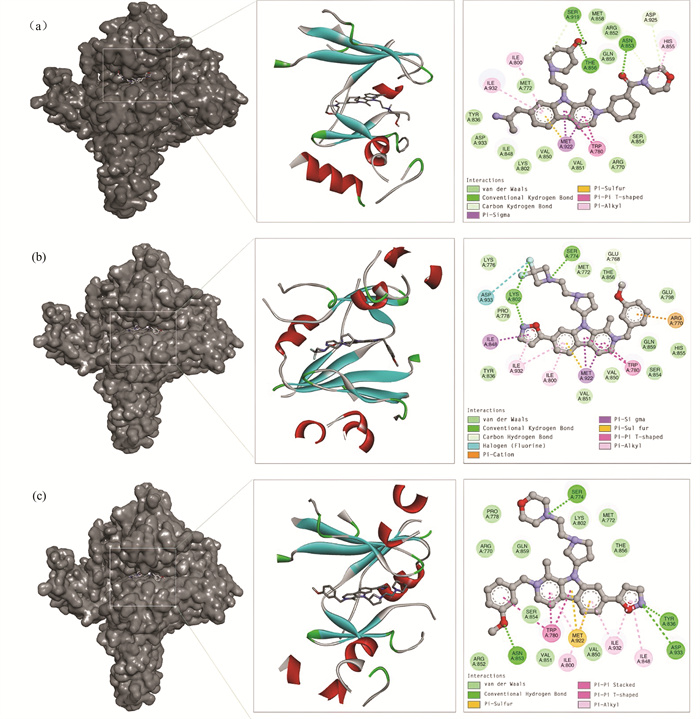
|
图 4 结合能前三对接复合物的非共价键相互作用:(a), (b)和(c)表示化合物37971, 50499和51939的相互作用 Figure 4 Non-covalent bond interactions of the first three docking complexes, where (a), (b), and (c) represent the interaction diagrams of compounds 37971, 50499, and 51939 |
分子对接模式图中衍生物37971与TYR-836, ASN-853, THR-856, SER-919四处氨基酸残基形成了氢键,同时在ILE-800, ILE-848, VAL-850, ILE-932, TYR-836附近形成了疏水键[25],在TRP-780处形成了π-π相互作用;衍生物50499则分别在ARG-770, SER-774, LYS-802等三个残基处形成了氢键,在TRP-780, ILE-800, TYR836, ILE848, VAL850, THR-856, ILE-932这七个氨基酸残基处形成了疏水键;衍生物51939在SER-774, LYS-802, ASN-853, ASP-933四个位置形成了氢键,在TRP-780, ILE-800, VAL850, THR856这四个位置上形成了疏水键。化合物库2中的衍生物7641在SER-773, SER-774, LYS-802处形成了氢键,在TRP-780, ILE-800, THR-856, ILE-932四处形成了疏水键,同时在TRP-780处形成了两处π-π相互作用;衍生物7644在SER-773, SER-774, TRP-780形成了五个氢键,同时与ILE-800, ILE-848, THR-856, ILE-932形成了四处疏水作用;衍生物7687在SER-774, LYS-802形成了两处氢键,同时在TRP-780, ILE-800, THR-856, ILE-932, ILE-848形成了六处疏水作用,在TRP-780处形成了π-π相互作用。以上衍生物均形成了较多的氢键,保证了配体与蛋白质之间结合稳定的相互作用力基础,也形成了疏水键,保证了蛋白质在疏水作用下维持着自己的三级结构。这几种化合物在分子对接中均与文献报道的PI3Kα的活性口袋重要氨基酸[35]TYR-836, VAL-851,SER774形成了相互作用,验证了虚拟筛选结果可靠性。在对接的结果中我们使用受体4ZOP的原配体4Q2进行了对接精度验证,使用Schrödinger对对接精度进行计算,重复的结果如表 3所示,图 5中碳元素为黄色的是原配体所在位置,紫色碳元素为原配体重新对接所得的RMSD最小构象,结果表明原配体与原受体之间RMSD值均小于2,说明软件对接结果可靠,图 5为glide score最低构象。
| 表 3 重复对接精度验证中RMSD值 Table 3 RMSD of redocking validate docking accuracy |
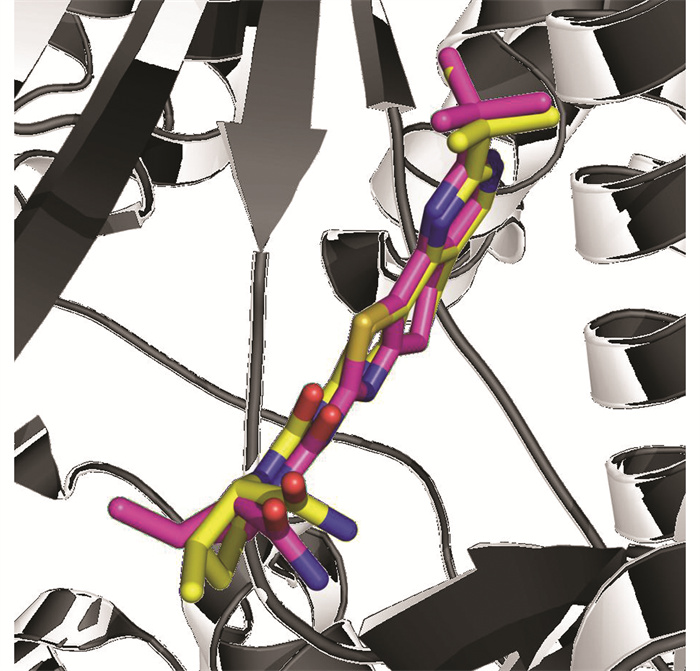
|
图 5 对接精度验证 Figure 5 Docking accuracy validation |
使用Discovery Studio 2020软件来构建PI3Kα的3D-QSAR模型,所使用的训练数据集化合物通过查阅相关文献[36]得到,并用于构建药效团模型。如图 6及表 4所示的20个化合物,用于构建Common Feature 3D-QSAR模型,其IC50范围从0.07 nM至36 700 nM不等。将化合物导入软件,在分子属性中添加IC50与PIC50属性,PIC50 = -log(IC50×10-9)。将化合物以PIC50大小排序。同时添加Principal和MaxOmitFeat属性,并根据其已知的实验活性进行定义,本实验设置为:当IC50<10 nM时为高活性化合物,Principal=2, MaxOmitFeat=0;当10 nM<IC50<1 000 nM时为中等活性化合物,Principal = 1, MaxOmitFeat = 1;当IC50>1 000 nM时为低活性化合物,Principal = 0, MaxOmitFeat =2。随机任意选择其中15个化合物作为训练集,剩余5个化合物作为测试集来验证并筛选所构建的3D-QSAR模型。根据IC50定义Principal和MaxOmitFeat属性。使用Create 3D QSAR Model模块,通过同时使用所有训练集和测试集分子,以PIC50的数据为小分子的活性数据筛选条件,使用CHARMm作为立场,得到PI3Kα的3D-QSAR模型。
|
图 6 3D-QSAR模型训练数据集化合物 Figure 6 3D-QSAR model training dataset compounds |
| 表 4 3D-QSAR模型训练数据集化合物 Table 4 3D-QSAR model training dataset compounds |
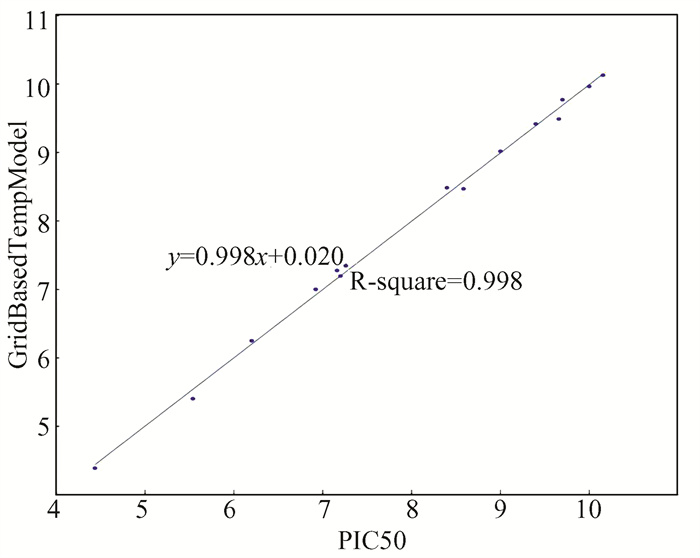
将上述筛选得到的优选化合物叠合至训练集分子,使用Calculate Molecular Properties模块以GridBasedTempModel作为评价能力,预测虚拟筛选得到化合物的PIC50预测值,从而评价其活性。通过PI3Kα训练集得到的3D-QSAR模型,结果如表 5所示,通过对比GridBasedTempModel的PIC50预测值与IC50实际值的差值(GridBasedTempModel_Residual) 可以发现,两者误差较小;3D-QSAR模型的拟合结果如图 7所示,横坐标为实际的PIC50数值,纵坐标为模型预测得到的PIC50值,该图表示训练集分子PIC50的真实值与预测值的相关性,线性关系为y=0.9998x+0.020,相关系数R方为0.998,拟合程度良好,因此该3D-QSAR训练模型较为可信。
| 表 5 3D-QSAR模型结果 Table 5 3D-QSAR model result |
|
图 7 PI3Kα训练集分子PIC50的真实值与预测值的相关性拟合线 Figure 7 Fitting line for the correlation between the true and predicted values of the PI3K training set molecule PIC50 |
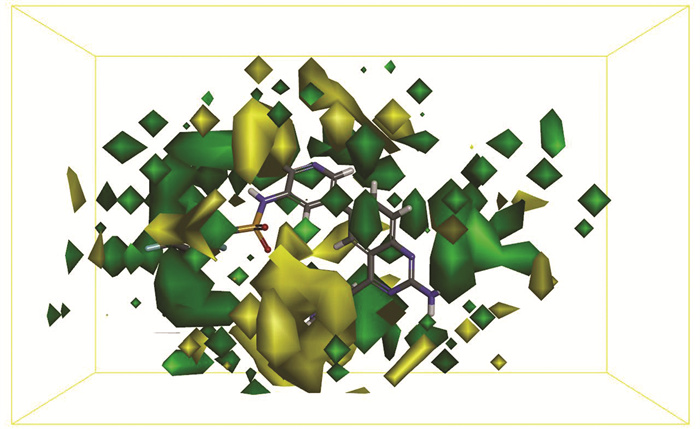
观察实际活性最好的PI3Kα训练集化合物CHEMBL3577914,其IC50=0.07 nM,PIC50=10.154 9,其预测值PIC50=10.129 4,预测值与实际差值为0.025 527。观察其静电场系数等高图(EP),如图 8所示,红色区域表示该区域取代基的负电性越强越有利于提高化合物的活性;蓝色区域表示该区域取代基的正电性越强越有利于提高化合物的活性。观察立体场系数等高图(VDW),如图 9所示,黄色区域表示该区域取代基体积的增大不利于提高化合物的活性;蓝色区域表示该区域取代基体积的增大有利于提高化合物的活性。

|
图 8 化合物CHEMBL3577914匹配至3D QSAR模型的静电场系数等高图 Figure 8 The compound chembl3577914 matched to the electrostatic field coefficient isoelectric map of the 3D QSAR model |
|
图 9 化合物CHEMBL3577914匹配至3D QSAR模型的立体场系数等高图 Figure 9 The compound chembl3577914 matches the stereo field coefficient isostere map of the 3D QSAR model |
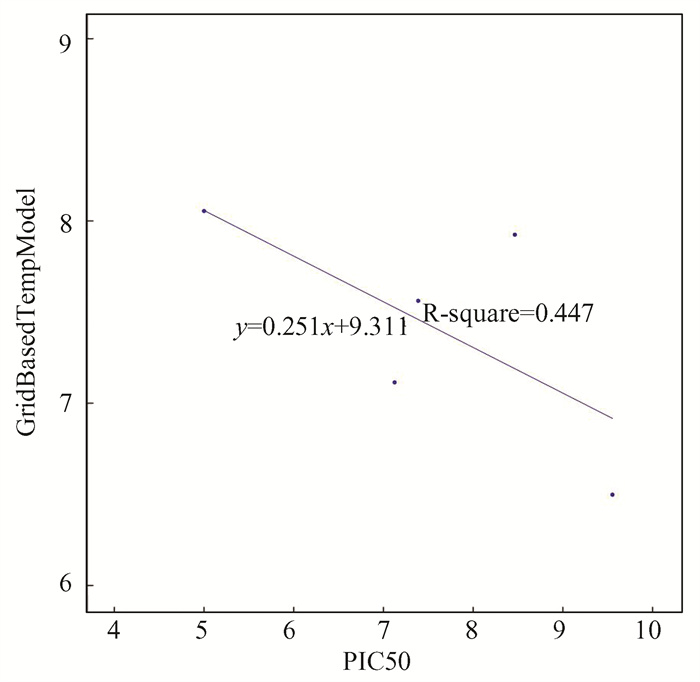
同时使用PI3Kα测试集对该3D-QSAR模型进行验证,结果如表 6所示,通过对比GridBasedTempModel的PIC50预测值与IC50实际值的差值(GridBasedTempModel_Residual) 可以发现,大部分化合物两者误差较小;3D-QSAR模型的拟合结果如图 10所示,横坐标为实际的PIC50数值,纵坐标为模型预测得到的PIC50值,该图表示训练集分子PIC50的真实值与预测值的相关性,线性关系为y=-0.251x+9.311,相关系数R方为0.447,拟合程度较好,因此选用该模型来预测上述虚拟筛选得到的化合物的活性PIC50值(表 7)。
| 表 6 PI3Kα测试集对该3D-QSAR模型得到的PIC50预测值与真实值对比 Table 6 The predicted PIC50 values obtained for this 3D-QSAR model by the PI3Kα test set were plotted against the true values |
|
图 10 PI3Kα测试分子PIC50的真实值与预测值的相关性拟合线 Figure 10 Line of fit for correlation between true and predicted values of PIC50 for PI3Kα test molecules |
| 表 7 虚拟筛选优选化合物及HM的PIC50活性预测 Table 7 prediction of PIC50 activity by virtual screening of preferred compounds and HM |
随后对上述虚拟筛选得到的优选化合物及原化合物去氢骆驼蓬碱(HM) 使用该3D-QSAR模型进行活性预测,得到结果如表 7所示。通过对比与母环结构氢骆驼蓬碱的活性预测结果其PIC50值为7.940 82,而化合物37971,50499与51939的PIC50值提高了0.7左右,活性提高了5倍,证明该虚拟筛选策略得到的优选化合物可能可以提高对PI3Kα的抑制能力。在结构上可以看出IC50为4以下的化合物结构与去氢骆驼蓬碱的结构均带有含氮杂环,且与化合物CHEMBL3774448结构具有一定相似性,表明去氢骆驼蓬碱与PI3Kα的抑制剂在结构上具有一定相似性,与其对比PIC50差距在3左右,侧面说明根据3D-QSAR所预测的生物活性值可靠,结合3D-QSAR模型对该筛选策略的验证,可以为后续去氢骆驼蓬碱的结构修饰与其他蛋白抑制剂的设计提供策略。
3.4 分子动力学模拟如图 11所示,进行了原配体和三种化合物的分子动力学分析,其RMSD值均不大于0.4 nm,表明稳定性良好,阳性化合物4Q2的稳定程度最高,波动程度最小,化合物37971次之。在最初的10 ns内,观察到RMSD的波动可忽略不计,表明复合物的初始稳定性。但是,在10 ns后,观察到RMSD的波动略大。除化合物51939外,化合物37971和50499的RMSD波动不超过0.4 nm。51939的RMSD在50-75 ns内略有增加,但在可接受的范围内,表明这些复合物的刚性构象足够稳定。复合物的RG表明蛋白质的结构是否紧密以及复合物是否紧密结合。蛋白质折叠和展开的程度取决于回转半径,可用于测量蛋白质折叠和展开的程度。RG在三种化合物的复合物中逐渐降低,表明该蛋白质和小分子的体积逐渐减小,这一点与阳性化合物4Q2表现出了一致的结果。SASA值可用于计算蛋白质和配体之间相互作用区域的大小。通过模拟轨迹来评估复合体积的变化,获得三种复合物的SASA值。SASA值可用于计算蛋白质和配体之间相互作用区域的大小。复合物的SASA值逐渐降低,表明蛋白质表面积减少,表明蛋白质结合表面积减少,这一参数与阳性化合物4Q2表现一致。此外,图 10显示在模拟过程中由三种化合物与受体组合产生的氢键的数量也改变了。复合物的稳定性和结合亲和力可能取决于氢键。观察到三种候选化合物中37971在100 ns的模拟时间内产生更多的氢键,表明组合的稳定性和结合亲和力可能取决于氢键的形成, 其中包括在100 ns内产生的氢键稳定性的占比,在氢键稳定性分析中发现,与阳性化合物4Q2对比,化合物50499与51939的稳定性均不高,仅有化合物37971还保持着高强度的稳定性。这与在虚拟筛选部分的结果中37971的结合能更高的结果表现出一致的结论,两者可互为参考。图 11还显示了模拟过程中每个残基的RMSF值。蛋白质氨基酸的平方平均数偏差(RMSF)用于分析模拟过程中单个氨基酸的波动程度,在这三个系统的100ns模拟中,其余活性口袋中氨基酸残基750-800和850-950的RMSF值波动较大,在阳性化合物中也是这部分的氨基酸拥有较大的RMSF波动,证明在分子动力学模拟中此类化合物拥有作为抑制剂的潜力,大部分产生相互作用的氨基酸相同。
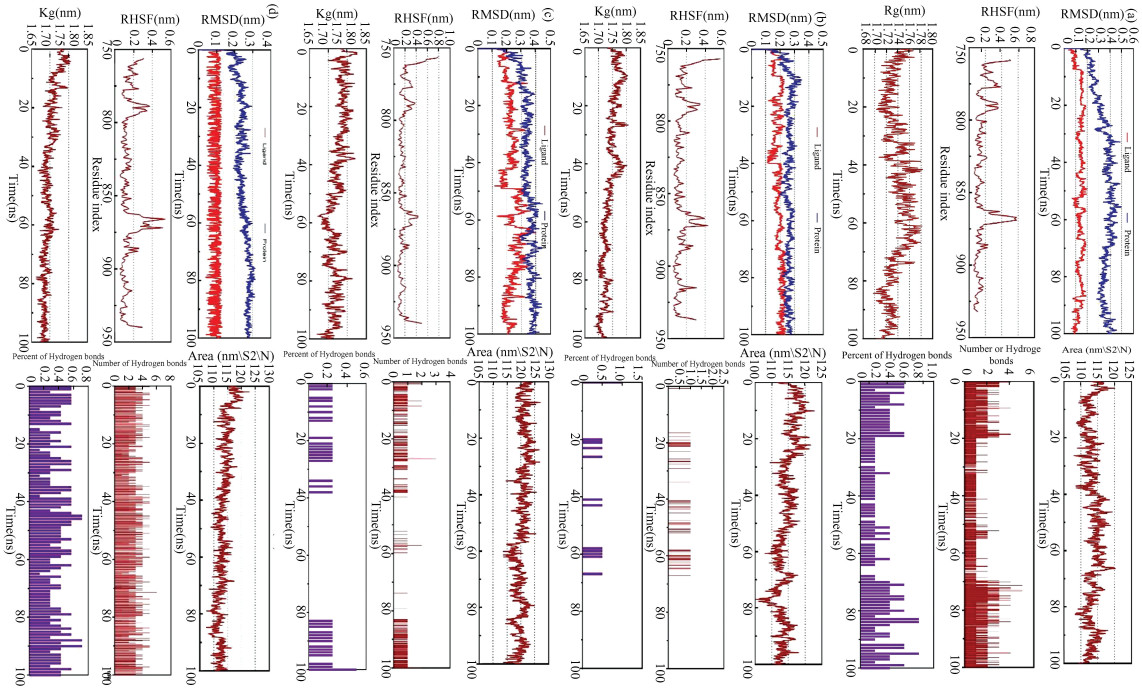
|
图 11 分子动力学模拟中,均方根偏差、均方根波动、溶剂可及面积表面积、回旋半径、氢键的数量、百分比变化 Figure 11 RMSD, SASA, RMSF, Rg, Number of H-Bonds, Percent of hydrogen bonds in MD simulation 注:a, b, c, d分别代表37971, 50499, 51939以及4Q2. |
通过ACD/Percepta对各化合物进行ADMET预测,结果如表 8所示。LogP为脂溶性参数,通常小于5比较优秀,可以看到三种衍生物在修饰后脂溶性参数良好;LogD值一版认为大于0.3比较可靠,三种化合物的LogD值分别为0.6, 0.34, 0.68,均表现为可靠;caco-2细胞膜渗透性细胞模型是一个典型的体外药物吸收模型,研究表明口服药物的吸收性与其在Caco-2细胞模型的通透系数具有良好的相关性,通常大于1表示肠道吸收良好,其中编号50499的衍生物表现出更高的肠道吸收性能,为2.43;生物利用度方面,三种化合物均超过30%,在可接受范围;P-gp抑制剂的可能性越小越好,三种化合物中,37971表现良好,为三种衍生物最小值0.38,P-gp inhibitor probability/RI表示P-gp抑制剂的可能性置信度,三者均在0.3左右,视为可靠;hERG inhibitor表示hERG通道抑制剂的可能性,与P-gp抑制剂相同的原则,也是其值越小越好,表明理论上并不是hERG通道的抑制剂,其预测值置信度大于0.3,表明结果比较可靠;Ames突变实验表示化合物是否具有致突变性,三种化合物的值均不超过0.8,其置信度分别为0.1, 0.32, 0.13,从此方面来看三种化合物的致突变性虽然不高,但是置信度也不高,需要进一步的实验;PPB%表示血浆蛋白结合率,通常与血浆蛋白结合的越多表示其在体内游离型的药物越少,转运稳定,但其缺点是药物在体内发挥作用的药量较低,三种化合物中编号为37971的衍生物血浆蛋白结合率较低,也表明其在体内游离型药物较多,可以很好地发挥药效;表观分布容积并无生理学意义,但其可以反映药物在体内的分布程度,其中编号为37971的化合物表观分布容积比其余两个化合物的值高也印证了其血浆结合率低的结果;TPSA(拓扑极性表面积)是一种常用的药物化学指标,用于优化药物渗透细胞的能力。极性表面积大于140埃平方(Å2)的分子往往难以渗透细胞膜,不易穿透血脑屏障从而作用于中枢神经系统的受体,而需要穿透血脑屏障的药物往往它的极性表面积需要小于60 Å2,在修饰后可以看到,37971的TPSA值有了显著提升,为87.84,接近90,可能因为化学结构中引入了羟基与羧基等亲水性基团导致了TPSA发生了比较大的改变,因此在理论上可以避免中枢毒性。而其他两个候选化合物的TPSA均小于60 Å2,理论上还存在着中枢毒性。
| 表 8 三种化合物的PK(药代动力学)性质预测 Table 8 Prediction of PK(Pharmacokinetics) properties of three compounds |
化合物的合成可行性与体内稳定性可从SwissADME网站中进行预测,结果见表 9,可以看到在三种候选化合物中,化合物37971的合成难度为4.99(从1到10,难度越高数字越大),合成难度中等,且为三者中最小,同时根据相关文献[37-38]可得,加速有机化合物生物降解能力的基团有-COOH, -OH, 酯、醛、酰胺等,而在37971中并无加速其生物降解性基团存在,从这一角度来说可以认为其理论上不会轻易降解。
| 表 9 合成可行性分析 Table 9 Feasibility analysis of synthesis |
化合物37971结合能为-77.43 kcal/mol,经过对接精度验证得到其对接位点准确,3D-QSAR模型中表现出的理论活性更高,且在药物动力学参数上比其他两个候选化合物更优异,拥有更好的TPSA值87.84,理论上中枢毒性更小,且合成可行性以及生物降解的预测结果在可接受范围内,可作为后续合成的目标产物,为下一步实验验证打下基础。
| [1] |
IKEDA S, ZABLOCKI D, SADOSHIMA J. The role of autophagy in death of cardiomyocytes[J]. Journal of Molecular and Cellular Cardiology, 2022, 165: 1-8. DOI:10.1016/j.yjmcc.2021.12.006 (  0) 0) |
| [2] |
LI Xiaohua, HE Shikun, MA Binyun. Autophagy and autophagy-related proteins in cancer[J]. Molecular Cancer, 2020, 19(1): 1-16. DOI:10.1186/s12943-020-1138-4 (  0) 0) |
| [3] |
XU Zhenru, HAN Xu, OU Daming, et al. Targeting PI3K/AKT/mTOR-mediated autophagy for tumor therapy[J]. Applied Microbiology and Biotechnology, 2020, 104: 575-587. DOI:10.1007/s00253-019-10257-8 (  0) 0) |
| [4] |
ALZAHRANI A S. PI3K/Akt/mTOR inhibitors in cancer: At the bench and bedside[J]. Seminars in Cancer Biology, 2019, 59: 125-132. DOI:10.1016/j.semcancer.2019.07.009 (  0) 0) |
| [5] |
FRUMAN D A, CHIU H, HOPKINS B D, et al. The PI3K pathway in human disease[J]. Cell, 2017, 170(4): 605-635. DOI:10.1016/j.cell.2017.07.029 (  0) 0) |
| [6] |
BUCHANAN C M, LEE K L, SHEPHERD P R. For better or worse: The potential for dose limiting the on-target toxicity of PI3-kinase inhibitors[J]. Biomolecules, 2019, 9(9): 402. DOI:10.3390/biom9090402 (  0) 0) |
| [7] |
TIAN Chengze, YANG Chengbin, WU Tianze, et al. Discovery of cinnoline derivatives as potent PI3K inhibitors with antiproliferative activity[J]. Bioorganic & Medicinal Chemistry Letters, 2021, 48: 128271. DOI:10.1016/j.bmcl.2021.128271 (  0) 0) |
| [8] |
YANG Qingshan, MODI P, NEWCOMB T, et al. Idelalisib: First-in-class PI3K delta inhibitor for the treatment of chronic lymphocytic leukemia, small lymphocytic leukemia, and follicular lymphoma[J]. Clinical Cancer Research, 2015, 21(7): 1537-1542. DOI:10.1158/1078-0432.CCR-14-2034 (  0) 0) |
| [9] |
FURMAN R R, SHARMAN J P, Coutre S E, et al. Idelalisib and rituximab in relapsed chronic lymphocytic leukemia[J]. New England Journal of Medicine, 2014, 370(11): 997-1007. DOI:10.1056/NEJMoa1315226 (  0) 0) |
| [10] |
ANDRÉ F, CIRUELOS E, RUBOVSZKY G, et al. Alpelisib for PIK3CA-mutated, hormone receptor-positive advanced breast cancer[J]. New England Journal of Medicine, 2019, 380(20): 1929-1940. DOI:10.1056/NEJMoa1813904 (  0) 0) |
| [11] |
NIROUMAND M C, FARZAEI M H, AMIN G. Medicinal properties of Peganum harmala L. in traditional Iranian medicine and modern phytotherapy: A review[J]. Journal of Traditional Chinese Medicine, 2015, 35(1): 104-109. DOI:10.1016/s0254-6272(15)30016-9 (  0) 0) |
| [12] |
ANDRÉ F, CIRUELOS E M, JURIC D, et al. Alpelisib plus fulvestrant for PIK3CA-mutated, hormone receptor-positive, human epidermal growth factor receptor-2-negative advanced breast cancer: Final overall survival results from SOLAR-1[J]. Annals of Oncology, 2021, 32(2): 208-217. DOI:10.1016/j.annonc.2020.11.011 (  0) 0) |
| [13] |
FROST D, MEECHOOVET B, WANG T, et al. β-carboline compounds, including harmine, inhibit DYRK1A and tau phosphorylation at multiple Alzheimer's disease-related sites[J]. Plos One, 2011, 6(5): e19264. DOI:10.1371/journal.pone.0019264 (  0) 0) |
| [14] |
ZHENG Xiyuan, ZHANG Lei, CHENG Xuemei, et al. Identification of Acetylcholinesterase Inhibitors from seeds of plants of genus peganum by thin-layer chromatography-bioautography[J]. Journal of Planar Chromatography-Modern TLC: JPC, 2011, 24(6): 470-474. DOI:10.1556/JPC.24.2011.6.3 (  0) 0) |
| [15] |
WANG Yihai, WANG Chunhua, JIANG Chenguang, et al. Novel mechanism of harmaline on inducing G2/M cell cycle arrest and apoptosis by up-regulating Fas/FasL in SGC-7901 cells[J]. Scientific Reports, 2015, 5(1): 1-10. DOI:10.1038/srep18613 (  0) 0) |
| [16] |
KIM G D. Harmine hydrochloride triggers G2/M cell cycle arrest and apoptosis in HCT116 cells through ERK and PI3K/AKT/mTOR signaling pathways[J]. Preventive Nutrition and Food Science, 2021, 26(4): 445. DOI:10.3746/pnf.2021.26.4.445 (  0) 0) |
| [17] |
CHEN Qi, MA Ke, WANG Zhuoyuan, et al. Harmine induces gastric cancer cell apoptosis through the ROSmediated PI3K/AKT signaling pathway[J]. Current Signal Transduction Therapy, 2015, 10(2): 112-118. DOI:10.2174/1574362410666150625190713 (  0) 0) |
| [18] |
LI Siwen, WANG Aqin, GU Fan, et al. Novel harmine derivatives for tumor targeted therapy[J]. Oncotarget, 2015, 6(11): 8988. DOI:10.18632/oncotarget.3276 (  0) 0) |
| [19] |
PRADIP, LEYLAND J, BRIAN, et al. PI3K-AKT-mTOR inhibitors in breast cancers: From tumor cell signaling to clinical trials[J]. Pharmacology and Therapeutics: The Journal of the International Encyclopedia of Pharmacology and Therapeutics, 2017, 175: 91-106. DOI:10.1016/j.pharmthera.2017.02.037 (  0) 0) |
| [20] |
CHEN Qi, CHAO Rihui, CHEN Hongsheng, et al. Antitumor and neurotoxic effects of novel harmine derivatives and structure-activity relationship analysis[J]. International Journal of Cancer, 2005, 114(5): 675-682. DOI:10.1002/ijc.20703 (  0) 0) |
| [21] |
RIBEIRO A R, SCHMIDT T C. Determination of acid dissociation constants (pKa) of cephalosporin antibiotics: Computational and experimental approaches[J]. Chemosphere, 2017, 169: 524-533. DOI:10.1016/j.chemosphere.2016.11.097 (  0) 0) |
| [22] |
STANZIONE F, GIANGRECO I, COLE J C. Use of molecular docking computational tools in drug discovery[J]. Progress in Medicinal Chemistry, 2021, 60: 273-343. DOI:10.1016/bs.pmch.2021.01.004 (  0) 0) |
| [23] |
ALBERICO D, SCOTT M E, LAUTENS M. Aryl-Aryl Bond formation by transition-metal-catalyzed direct arylation[J]. Chemical Reviews, 2007, 107(1): 174-238. DOI:10.1021/cr0509760 (  0) 0) |
| [24] |
ROMERO M, HARRAK Y, BASSET J, et al. Preparation of N-arylpiperazines and other N-aryl compounds from aryl bromides as scaffolds of bioactive compounds[J]. Tetrahedron, 2006, 62(38): 9010-9016. DOI:10.1016/j.tet.2006.07.011 (  0) 0) |
| [25] |
任益昌. 去氢骆驼蓬碱衍生物的设计, 合成与抗肿瘤作用机制研究[D]. 广州: 暨南大学, 2018. REN Yichang. Design, synthesis and mechanisms of action of novel harmine derivatives as potential antitumor agents[D]. Guangzhou: Jinan University, 2018. (  0) 0) |
| [26] |
BURLEY S K, BHIKADIYA C, BI C, et al. RCSB protein data bank: Powerful new tools for exploring 3D structures of biological macromolecules for basic and applied research and education in fundamental biology, biomedicine, biotechnology, bioengineering and energy sciences[J]. Nucleic Acids Research, 2021, 49(D1): D437-D451. DOI:10.1093/nar/gkaa1038 (  0) 0) |
| [27] |
SILVA M A, NASCIMENTO JU ' NIOR J C, THOMAZ D V, et al. Comparative homology of Pleurotus ostreatus laccase enzyme: Swiss model or Modeller?[J/OL]. Journal of Biomolecular Structure and Dynamics, 2022: 1-14[2022-10-30]. https://doi.org/10.1080/07391102.2022.2138975. DOI: 10.1080/07391102.2022.2138975.
(  0) 0) |
| [28] |
VAN DER SPOEL D, LINDAHL E, HESS B, et al. GROMACS: Fast, flexible, and free[J]. Journal of Computational Chemistry, 2005, 26(16): 1701-1718. DOI:10.1002/jcc.20291 (  0) 0) |
| [29] |
LEGGATT G, CHENG Guo, NARAIN S, et al. A genotype-to-phenotype approach suggests under-reporting of single nucleotide variants in nephrocystin-1 (NPHP1) related disease (UK 100, 000 Genomes Project)[J]. Scientific Reports, 2023, 13(1): 9369. DOI:10.1038/s41598-023-32169-4 (  0) 0) |
| [30] |
JEJURIKAR B L, ROHANE S H. Drug designing in discovery studio[J]. 2021, 14(2): 135-138. DOI: 10.5958/0974-4150.2021.00025.0.
(  0) 0) |
| [31] |
YUAN Shuguang, CHAN H S, HU Zhenquan. Using PyMOL as a platform for computational drug design[J]. Wiley Interdisciplinary Reviews: Computational Molecular Science, 2017, 7(2): e1298. DOI:10.1002/wcms.1298 (  0) 0) |
| [32] |
FRIESNER R A, BANKS J L, MURPHY R B, et al. Glide: A new approach for rapid, accurate docking and scoring. 1. Method and assessment of docking accuracy[J]. Journal of Medicinal Chemistry, 2004, 47(7): 1739-1749. DOI:10.1021/jm0306430 (  0) 0) |
| [33] |
蔡棣, 田浤, 姚文兵. 含有L-对硝基苯丙氨酸的BAFF改构体的分子动力学模拟与分析[J]. 中国药科大学学报, 2017, 48(2): 227-232. CAI Di, TIAN Hong, YAO Wenbing. Molecular dynamics simulation and analysis of BAFF which is incorporated with p-nitro-L-phenylalanine[J]. Journal of China Pharmaceutical University, 2017, 48(2): 227-232. DOI:10.11665/j.issn.1000-5048.20170215 (  0) 0) |
| [34] |
JO S, KIM T, IYER V G, et al. CHARMM-GUI: A web-based graphical user interface for CHARMM[J]. Journal of Computational Chemistry, 2008, 29(11): 1859-1865. DOI:10.1002/jcc.20945 (  0) 0) |
| [35] |
NEMETHY G. Hydrophobic interactions[J]. Angewandte Chemie International Edition in English, 1967, 6(3): 195-206. DOI:10.1002/anie.196701951 (  0) 0) |
| [36] |
YU Miao, GU Qiong, XU Jun. Discovering new PI3Kα inhibitors with a strategy of combining ligand-based and structure-based virtual screening[J]. Journal of Computer-Aided Molecular Design, 2018, 32: 347-361. DOI:10.1007/s10822-017-8 (  0) 0) |
| [37] |
ALEXANDER M. Biodegradation and bioremediation[M]. San Diego: Academic Press Inc, 1999.
(  0) 0) |
| [38] |
LYMAN W J, REEHL W F, ROSENBLATT D H. Handbook of chemical property estimation methods[M]. New York: McGraw-Hill, 1982.
(  0) 0) |
 2024, Vol. 22
2024, Vol. 22


