2. 山西医科大学 基础医学院,太原 030001;
3. 山西省人民医院 中医科,太原 030012
2. College of Basic Medical Sciences of Shanxi Medical University, Taiyuan 030001, China;
3. Department of Traditional Chinese Medicine, Shanxi Provincial People's Hospital, Taiyuan 030012, China
阿尔兹海默病(Alzheimer’s disease,AD)是一种与年龄相关的可导致痴呆和认知功能下降的神经退行性疾病,该病始于轻微的记忆力减退,后逐步发展为认知、语言、复杂日常活动障碍等方面[1]。全球大约有5 000万人患有阿尔茨海默病[2],给社会带来巨大负担,探索AD的有效治疗方法具有重要意义。几十年来,大多数研究都集中在脑内淀粉样蛋白-Aβ积累,tau蛋白磷酸化和神经纤维缠结等,但药物治疗效果差。最新研究发现,除突触毒性[3]、自噬[4]、神经炎症[5]外,细胞死亡的调控在AD中也起着重要作用,不同神经元细胞的异常死亡是神经退行性疾病的重要标志[6]。
失巢凋亡(Anoikis)作为一种新型的细胞死亡方式,是细胞对细胞外基质粘附丧失或损伤导致的细胞凋亡,其被认为是维持组织稳态与正常发育的重要过程。在中枢神经系统中,星形胶质细胞间的粘附连接依赖钙离子浓度,在低钙环境中,粘附连接的松动会导致细胞与基质分离,诱导失巢凋亡发生,进而导致功能神经元变性损伤,神经可塑性降低,促使AD的发生进展[7]。
本研究中,首次系统地检测了正常人群和AD个体间失巢凋亡相关基因(Anoikis-related genes,ANRGs)的差异表达水平。基于10个ANRGs的表达,将310例AD患者分为3个失巢凋亡亚型,进一步评估了亚型间的免疫微环境差异。之后,采用加权基因共表达网络分析(Weighted gene co-expression network analysis,WGCNA)分别对AD和正常人群间以及不同亚型间特异性表达的基因进行鉴定,并阐明其丰富的生物学功能。最后,采用多种机器学习算法对特异性基因进行模型构建,从而预测疾病罹患风险,为AD的分子分型提供新见解。
1 材料与方法 1.1 数据获取与差异分析通过GEO数据库(Gene Expression Omnibus,https://www.ncbi.nlm.nih.gov/geo)下载AD相关数据集,经过数据质量分析,共得到4个研究队列(GSE5281, GSE29378, GSE33000和GSE122063),详细信息见表 1。选GSE33000作为训练集,其包含157例健康个体和310例AD患者的前额叶皮层转录组测序数据,使用R语言(R 4.2.1,https://www.r-project.org/)中的“limma”包对两组进行差异分析,筛选标准:| log2FC|>0.1,校正后的P值<0.05。通过GeneCards数据库(https://www.genecards.org)下载ANRGs,根据相关性分数排序,取前10%的基因(80个)。通过Harmonizome数据库(https://maayanlab.cloud/Harmonizome)下载ANRGs,共得到137个。两个数据库取交集,共得53个ANRGs,之后与差异表达的基因取交集。
| 表 1 训练和验证集信息 Table 1 The information of training/validation datasets |
利用CIBERSORT算法对GSE33000进行打分[8],估算22种免疫细胞在每个样本中的相对丰度。CIBERSORT使用蒙特卡罗采样计算反摺积P值,只有当P值小于0.05时,打分才被认为是准确的。每个样本中22种免疫细胞的比例之和为1。同时,分析了差异表达的ANRGs与每种免疫细胞浸润丰度的相关性。
1.3 AD患者的无监督聚类基于10个差异表达的ANRGs,使用“ConsensusClusterPlus”包对训练集进行一致性聚类[9],选取最优聚类数量,鉴定不同的分子亚型。选择最大亚型数k (k=9),并根据累积分布函数曲线(Cumulative distribution function,CDF), 共识矩阵(Consensus matrix)和一致性聚类(Consistent cluster)评分>0.9,综合评估最佳聚类数。PCA主成分分析(Principal component analysis) 进一步评估各亚型与ANRGs的相关性。之后,在不同亚型间比较10个ANRGs的表达水平,以及不同免疫细胞的浸润水平。
1.4 基因集变异分析采用“GSVA”包对不同亚型的样本进行基因集变异分析(Gene set variation analysis,GSVA)[10],选取GSEA网站中“c2.cp.kegg.symbols”数据集作为分析基因集,以此来进行京都基因和基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)富集分析,进而比较不同亚型间信号通路的富集程度差异。
1.5 WGCNA分析使用“WGCNA”包进行WGCNA分析[11],鉴定关键的共表达模块。为保证结果的准确性,取方差最高的前25%基因进行后续分析。利用最优软阈值构建加权邻接矩阵,并将其转化为拓扑重叠矩阵。当最小模块大小设置为100时,使用TOM不相似度量(1-TOM)获得模块。每个模块都被随机分配了一个颜色。每个模块的特征基因代表全部的基因表达谱。模块显著性(Modular significance,MS)体现了模块与临床表型之间的关系。基因显著性(Gene significance,GS)为基因与临床表型之间的相关性。
1.6 基于多种机器学习方法构建AD风险预测模型分别对AD与正常个体,不同亚型之间进行两次WGCNA分析,选取每次分析结果的显著特征基因集,之后取交集基因进行进一步分析。基于交集基因的表达量,使用“caret”包对GSE33000进行数据预处理,之后采用随机森林(Random forest model,RF),支持向量机(Support vector machine model,SVM),广义线性模型(Generalized linear model,GLM)和极端梯度提升(Extreme gradient boosting,XGB),共4种机器学习方法进行预测模型构建,以此区分AD患者与正常个体。RF是一个包含多个决策树的分类器,并且其输出的类别是由个别树输出的类别的众数而定[12]。SVM是一类按监督学习方式对数据进行二元分类的广义线性分类器,其决策边界是对学习样本求解的最大边距超平面[13]。GLM是线性模型的扩展,通过联系函数建立响应变量的数学期望值与线性组合的预测变量之间的关系[14]。XGB是一种基于梯度增强的增强树集合,可以仔细比较分类误差和模型复杂度[15]。“caret”包通过网格搜索自动调整模型中的参数,所有机器学习模型都使用默认参数执行,并通过5倍交叉验证进行评估。“DALEX”包用于解释上述4种机器学习模型,并可视化这些模型之间的残差分布和特征重要性。执行“pROC”包以可视化受试者工作特征曲线(Receiver operating characteristic curve,ROC)下的面积(AUC值)。确定最优的机器学习模型后,发现前5个重要变量是AD的关键预测基因。最后,在GSE5281, GSE29378和GSE122063数据集进行ROC分析,验证模型的诊断价值。
1.7 构建列线图选取最优的机器学习模型,利用“rms”包建立预测AD发生率的列线图,进而可视化模型,使其更容易使用。每个预测因子均有相应的分数,“总分”表示上述预测因子的分数之和。使用校正和决策曲线评估列线图的预测能力。
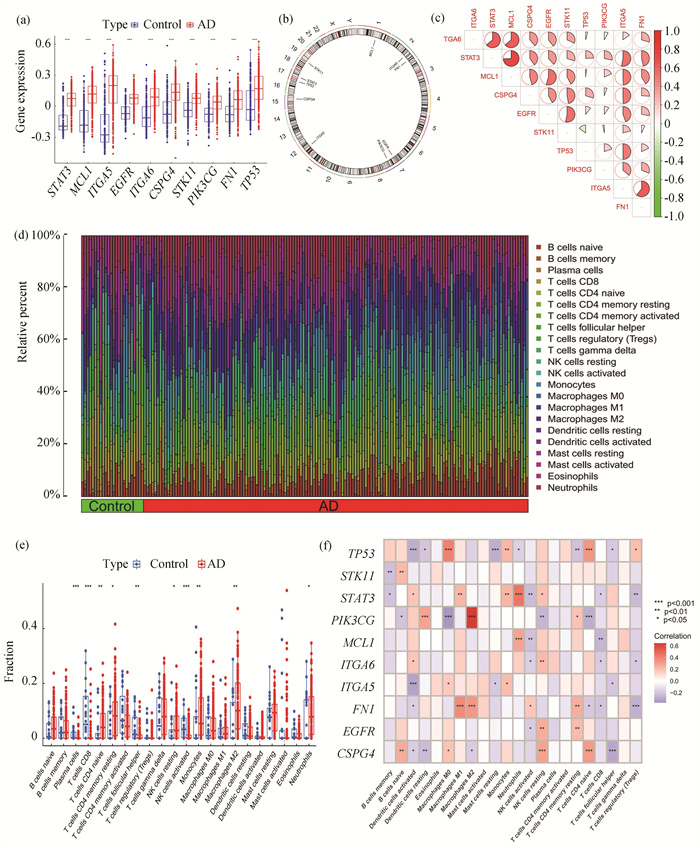
2 结果 2.1 AD患者ANRGs的异常表达与免疫微环境紊乱为了阐明ANRGs在AD发生发展中的生物学功能,首先使用GSE33000数据集进行差异分析,共得到1 666个差异表达的基因,通过与53个ANRGs取交集,共得到10个基因(图 1(a), 1(b))。相关性分析显示,多个ANRGs间存在显著的共表达关系(图 1(c))。为了阐明AD患者与正常个体组间的免疫微环境是否存在差异,进行了免疫浸润分析。基于CIBERSORT算法,分析了两组间22种免疫细胞浸润的比例差异(图 1(d))。结果显示,AD患者中幼稚CD4+ T细胞,静息记忆CD4+ T细胞,静息NK细胞,单核细胞,M2型巨噬细胞和中性粒细胞显著浸润。浆细胞,CD8+ T细胞,滤泡辅助T细胞和活化的NK细胞在正常个体中浸润水平更高(图 1(e)),这表明免疫微环境的改变可能是AD发生的主要原因。同时,相关性分析表明,多个ANRGs与不同的免疫细胞间具有相互作用(图 1(f))。这些结果表明,ANRGs可能是调节AD患者分子和免疫浸润状态的关键因素。
|
图 1 AD患者中异常表达的ANRGs:(a) 箱线图显示了10个差异表达的ANRGs;(b) 10个差异表达的ANRGs在染色体上的位置;(c) 10个差异表达ANRGs的相关性分析,红色和绿色分别代表正相关和负相关,相关系数用饼图的面积来表示;(d, e) AD与正常个体间22种免疫细胞的浸润丰度, *P<0.05, **P<0.01, ***P<0.001;(f) 10个差异表达的ANRGs与免疫细胞的相关性分析 Figure 1 Identification of dysregulated ANRGs in AD: (a) Boxplots showed 10 differentially expressed ANRGs; (b) The location of 10 ANRGs on chromosomes; (c) Correlation analysis of 10 differentially expressed ANRGs. Red and green colors represent positive and negative correlations, respectively. The correlation coefficients were marked with the area of the pie chart; (d, e) Boxplots showed the differences in immune infiltrating between AD and non-AD controls. *P < 0.05, **P < 0.01, ***P < 0.001(f) Correlation analysis between 10 differentially expressed ANRGs and infiltrated immune cells |
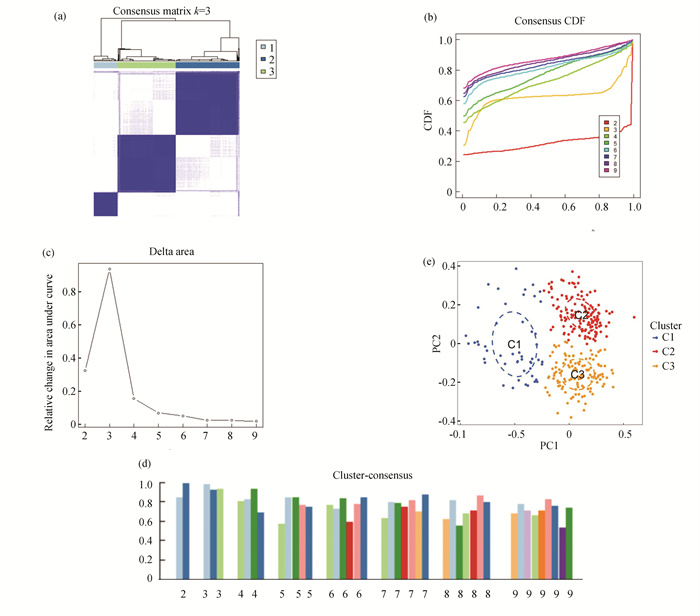
为了阐明ANRGs在AD患者中的表达模式,基于10个ANRGs的表达情况,通过一致性聚类算法对310例AD样本进行分组。当k=3时,聚类最稳定,CDF曲线在0.2~0.6的共识指数范围内波动最小(图 2(a), 2(b))。当k = 2~9时,CDF曲线下面积表现为两条CDF曲线(k和k-1)的差值(图 2(c))。仅当k = 3时,各亚型的一致性得分>0.9(图 2(d))。PCA主成分分析显示,三个亚型间具有显著差异(图 2(e))。
|
图 2 AD失巢凋亡相关分子亚型鉴定:(a)k=3时的共识聚类矩阵;(b)代表性累积分布函数曲线; (c)CDF曲线面积; (d)共识聚类得分;(e)PCA主成分分析可视化三个亚型的分布 Figure 2 Identification of anoikis-related molecular clusters in AD: (a) Consensus clustering matrix when k=3; (b) Representative cumulative distribution function (CDF) curves; (c) CDF delta area curves; (d) The score of consensus clustering; (e) PCA visualized the distribution of three subtypes |
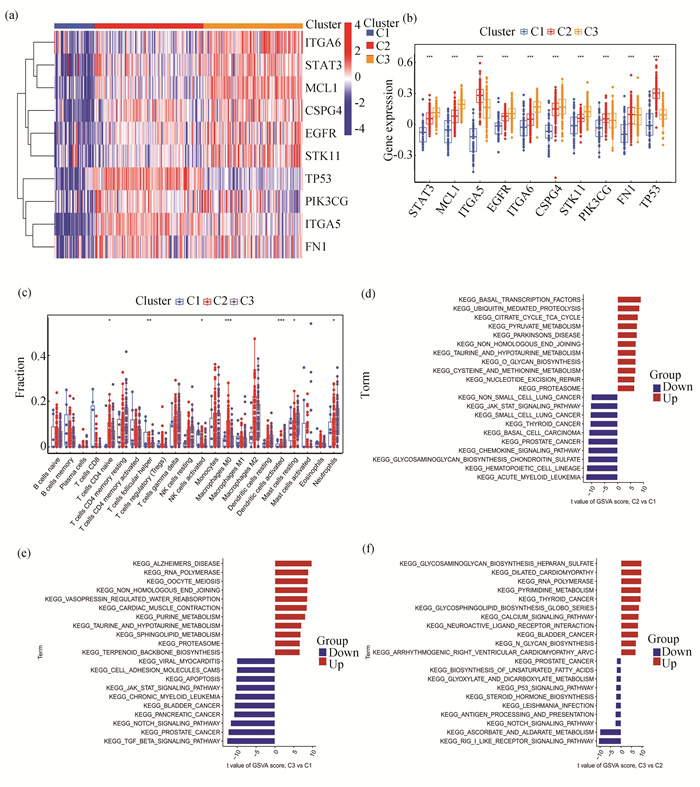
为了探究不同亚型间的分子特征,首先分析了10个ANRGs在3个亚型间的表达差异。结果显示,大多数ANRGs在cluster 3中高表达,在cluster 1中表达最低(图 3(a), 3(b))。免疫浸润分析显示,3个亚型间的免疫微环境发生显著改变,滤泡辅助T细胞和活化的NK细胞在cluster 1中显著富集,幼稚CD4+ T细胞和M0型巨噬细胞在cluster 2中显著富集,活化的树突状细胞,静息态肥大细胞和中性粒细胞在cluster 3中比例较高(图 3(c))。利用GSVA分析进一步探讨3个亚型之间信号通路的富集差异,以cluster 1和cluster 3为例,发现,相较cluster 1,cluster3中阿尔兹海默病,嘌呤代谢,牛磺酸与亚牛磺酸代谢,鞘磷脂代谢等多个通路显著上调,细胞黏附分子,细胞凋亡,JAK/STAT信号通路,Notch信号通路等多个通路则下调明显(图 3(d)-(f))。
|
图 3 三个失巢凋亡相关分子亚型间的免疫特征:(a)热图展示了三个失巢凋亡分子亚型间10个ANRGs的表达谱;(b)箱线图展示了10个ANRGs在不同分子亚型之间的差异表达;(c)22种免疫细胞在不同分子亚型之间的相对丰度;(d-f)GSVA方法的t值排序,展示不同亚型间KEGG通路的富集差异,(d):C2 vs C1,(e):C3 vs C1,(f):C3 vs C2 Figure 3 Identification of molecular and immune characteristics between the three anoikis clusters: (a) Expression patterns of 10 ANRGs between three clusters were presented in the heatmap; (b) Boxplots showed the expression of 10 ANRGs between three clusters. *P < 0.05, **P < 0.01, ***P < 0.001; (c) Boxplots showed the differences in immune infiltrating betwwen three clusters. *P < 0.05, **P < 0.01, ***P < 0.001; (d-f) t-value sequencing of GSVA method showed the enrichment difference of KEGG pathway among different clusters. (d): C2 vs C1, (d): C3 vs C1, (f): C3 vs C2 |
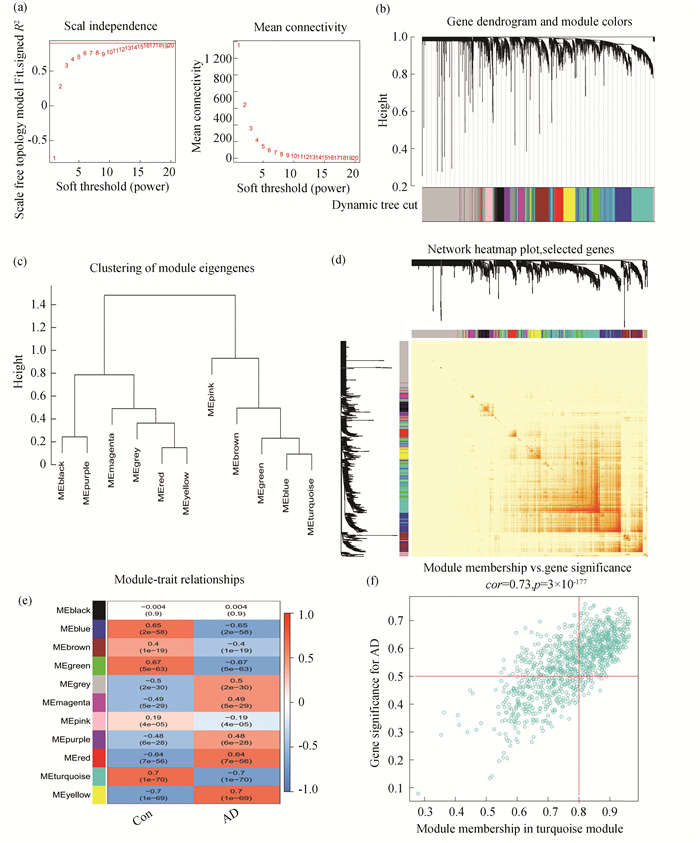
为了识别与AD发生相关的基因模块,利用WGCNA算法构建AD和正常个体间的共表达网络。计算GSE33000中每个基因表达的方差,选择方差最高的前25%基因进行分析。当软阈值为16,无标度R2=0.9时,识别共表达基因模块(图 4(a))。利用动态切割算法获得了11个不同颜色的共表达基因模块,并给出了拓扑重叠矩阵(TOM)的热图(图 4(b)-4(d))。之后,将这些基因连续应用于11个颜色模块中,分析模块-临床特征(对照组和AD组)共表达的相似性和邻接性。最终,蓝绿色模块与AD的关系最强,包括759个基因(图 4(e))。此外,我们还观察到蓝绿色模块隶属度与特征基因之间存在显著正相关(图 4(f))。
|
图 4 AD中差异表达基因的共表达网络:(a)软阈值的选择;(b)共表达模块的聚类树图,不同的颜色代表不同的共表达模块;(c)模块特征基因的聚类代表;(d)11个模块之间相关性的代表性热图;(e)模块基因与临床特征的相关性分析,每一行代表一个模块,每一列代表一种临床特征;(f)蓝绿色模块的模块隶属度与特征基因显著性之间的散点图 Figure 4 Co-expression network of differentially expressed genes in AD: (a) The selection of soft threshold power; (b) Cluster tree dendrogram of co-expression modules. Different colors represent distinct co-expression modules; (c) Representative of clustering of module eigengenes; (d) Representative heatmap of the correlations among 11 modules; (e) Correlation analysis between module eigengenes and clinical status, Each row represents a module, each column represents a clinical status; (f) Scatter plot between module membership in turquoise module and the gene significance for AD |
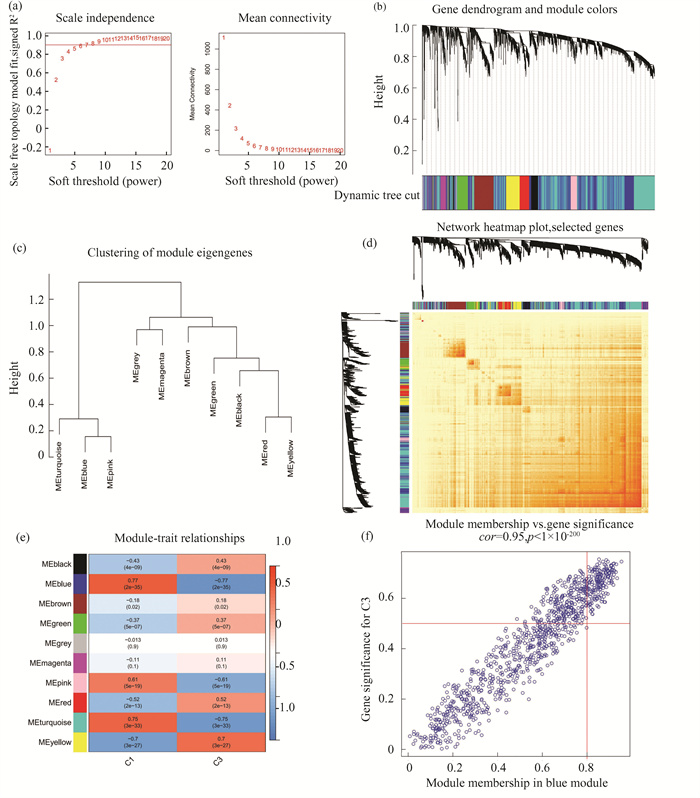
接着,分析了cluster 1和cluster 3之间的关键基因模块。将β=6和R2=0.9作为最合适的软阈值来构建无标度网络(图 5(a))。共有10个模块被确定为显著模块,热图描绘了所有模块相关基因的TOM(图 5(b)-5(d))。模块-临床特征(cluster 1和cluster 3)关系分析表明,蓝色模块(182个基因)与分子分型高度相关(图 5(e))。相关性分析表明,蓝色模块隶属度与特征基因具有显著相关性(图 5(f))。
|
图 5 分子亚型间的共表达网络:(a)软阈值的选择;(b)共表达模块的聚类树图,不同的颜色代表不同的共表达模块;(c)模块特征基因的聚类代表;(d)10个模块之间相关性的代表性热图;(e)模块基因与临床特征的相关性分析,每一行代表一个模块,每一列代表一种临床特征;(f)蓝色模块的模块隶属度与特征基因显著性之间的散点图 Figure 5 Co-expression network of differentially expressed genes between the anoikis clusters: (a) The selection of soft threshold power; (b) Cluster tree dendrogram of co-expression modules. Different colors represent distinct co-expression modules; (c) Representative of clustering of module eigengenes; (d) Representative heatmap of the correlations among 10 modules; (e) Correlation analysis between module eigengenes and clinical status. Each row represents a module; each column represents a clinical status; (f) Scatter plot between module membership in blue module and the gene significance for cluster3 |
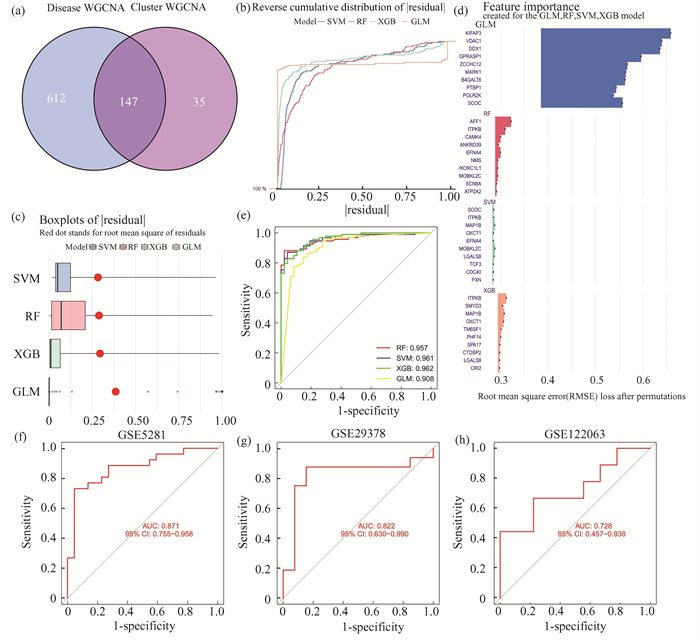
为了进一步识别具有诊断价值的AD特征基因,我们将WGCNA算法得到的两个基因模块取交集,共得到147个基因(图 6(a))。基于交集基因进行4个机器学习模型构建(RF, GLM, SVM和XGB),以此来筛选特征基因。使用“DALEX”包对模型进行解释,并绘制各模型在训练集中的残差分布(图 6(b), 6(c))。之后,根据均方根误差(Root mean square error,RMSE)对每个模型的前10个特征基因进行排序(图 6(d))。此外,计算基于5倍交叉验证的ROC曲线,评估4种机器学习模型在训练集中的诊断性能,XGB模型的AUC值最高(图 6(e))。因此,XGB模型被证明能最好地区分AD患者,选取该模型中最重要的5个基因(TM6SF1, SMYD3, OXCT1, MAP1B和ITPKB)作为预测变量进行进一步分析。最后,将XGB模型在3个验证集中进行验证。结果显示,XGB模型在验证集(GSE5281, GSE29378和GSE122063)中同样具有良好的预测能力(图 6(f)-6(h))。
|
图 6 RF、GLM、SVM和XGB机器模型的构建和评估:(a)GSE33000数据集中AD相关模块基因与分子亚型相关模块基因的交集;(b)每个机器学习模型的累积残差分布;(c)箱形图展示了每个机器学习模型的残差,红点表示残差的均方根;(d)RF、GLM、SVM和XGB模型中的特征基因;(e)基于训练集中5倍交叉验证的四种机器学习模型的ROC分析;(f-h)XGB模型在3个验证队列中的ROC分析 Figure 6 Construction and evaluation of RF, GLM, SVM and XGB machine models: (a) The intersections between module-related genes of clusters and module-related genes in the GSE33000 dataset; (b) Cumulative residual distribution of each machine learning model; (c) Boxplots showed the residuals of each machine learning model. Red dot represented the root mean square of residuals (RMSE); (d) The important features in RF, GLM, SVM and XGB machine models; (e) ROC analysis of four machine learning models based on 5-fold cross-validation in the testing cohort; (f-h) ROC analysis of the 5-gene-based XGB model based on 5-fold cross-validation in GSE5281 (f), GSE29378 (g) and GSE122063 (h) datasets |
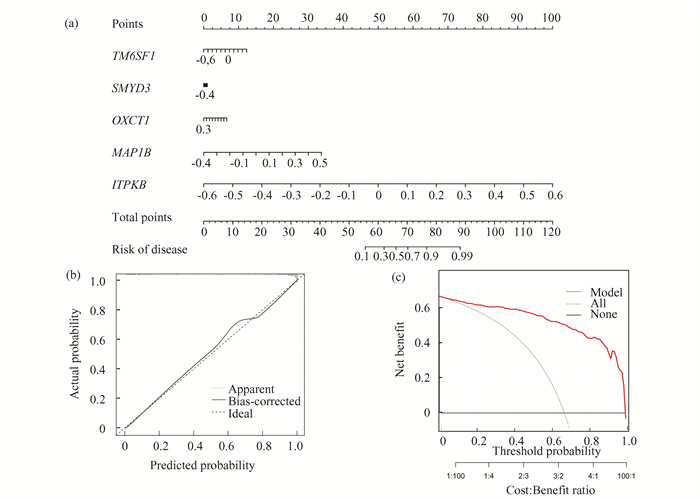
为了可视化XGB模型,构建了一个列线图来估计训练集中个体罹患AD的风险(图 7(a)),应用校正和决策曲线对列线图的预测效率进行评价。根据校正曲线,罹患AD的实际风险与预测风险之间的误差很小(图 7(b)。决策曲线表明我们的列线图具有较高的准确性,可为临床决策提供依据(图 7(c))。
|
图 7 构建列线图:(a)基于XGB模型中5个基因的AD风险预测列线图构建;(b, c)用于评估列线图模型预测效率的校准曲线(b)和决策曲线(c) Figure 7 Construction of a nomogram: (a) Construction of a nomogram for predicting the risk of AD based on the 5-gene-based XGB model; (b, c) Construction of calibration curve (b) and DCA (c) for assessing the predictive efficiency of the nomogram |
AD发病机制复杂,具有高度异质性,目前缺乏有效的治疗方法。既往研究发现,AD的抗神经退行性治疗取得一定的进展,但基于组织学与病理学的传统分类导致耐药频发。因此,识别更精准的分子亚型来指导AD的个性化治疗具有重要意义。失巢凋亡是一种新型的细胞死亡方式,是细胞对细胞外基质粘附丧失或损伤导致的细胞凋亡。然而,失巢凋亡参与AD的具体机制尚不清楚,本文对失巢凋亡相关基因在AD发病以及免疫微环境中的具体作用进行探讨,并利用失巢凋亡相关基因对AD亚型进行预测。
本研究中,首次系统分析了AD患者和正常个体脑组织中ANRGs的表达谱。与正常个体相比,AD患者中多个ANRGs存在差异表达,提示其在AD发生中承担重要作用。接着,我们计算了差异表达的ANRGs间的相关性,以阐明失巢凋亡调控因子与AD之间的关系,结果发现多个ANRGs具有显著的协同作用,这可能促进AD的进展。免疫微环境分析发现,多种免疫细胞在AD和正常个体间也存在显著差异,AD患者中的幼稚CD4+ T细胞,静息记忆CD4+ T细胞,静息NK细胞,单核细胞,M2型巨噬细胞和中性粒细胞显著浸润,而浆细胞,CD8+ T细胞,滤泡辅助T细胞和活化的NK细胞比例较低,这与既往在血液或脑组织中的研究一致[16-18]。此外,通过无监督聚类的方式,根据10个ANRGs的表达谱,鉴定了三种不同的亚型。多种免疫细胞在cluster 3中显著浸润,与阿尔兹海默症相关的多个生物学过程在cluster 3中显著上调。据此我们猜测,cluster 3患者的AD进程可能更显著,整体预后较差。
近年来,基于流行病学特征和医学影像指标的机器学习模型被逐步应用于AD患者患病的检测,这些研究表明,多因素分析相较单因素分析,具有更低的错误率,可靠性更高[19]。本研究中,通过4种机器学习模型(RF, GLM, SVM和XGB),基于两次WGCNA算法得到的交集基因表达谱进行预测模型构建,结果发现XGB模型具有最好的预测能力(AUC=0.962),并且在3个验证集中得到很好的验证。之后,选择XGB模型中的特征变量(TM6SF1, SMYD3, OXCT1,MAP1B和ITPKB),进一步构建了实用性更强的列线图。SMYD3(SET and MYND domain containing 3)是甲基转移酶家族的成员,含有SET, MYND和CTD结构域,可促进蛋白-蛋白之间相互作用、连接并结合[20]。此外,SMYD3还能促进H3K4的二甲基化和三甲基化,通过激活多个下游靶基因的转录来启动肿瘤发生,且过表达会引起细胞增殖、迁移和粘附[21]。AD的表观遗传学研究表明,组蛋白甲基转移酶和去甲基化酶之间的平衡对AD患者的大脑完整性和记忆尤为重要,而H3K4去甲基化酶与人类的记忆缺陷有关[22]。OXCT1(3-Oxoacid CoA-Transferase 1)是一种限速酶,能将肝外酮体转化为乙酰辅酶A,进入三羧酸循环以产生ATP,OXCT1基因突变引起的OXCT1缺乏症患者常表现为酮症酸中毒发作[23]。由于神经元广泛的突触连接和长距离神经投射,其对能量有极高的需求,故大多数神经退行性疾病都有代谢紊乱和特定神经元损失两大特征。当能量短缺时,神经元与胶质细胞开始利用酮体进行氧化代谢,从而为大脑活动提供能量。研究发现,生酮干预的AD患者,其记忆、语言等认知功能都出现了改善[24]。因此,OXCT1可能通过参与TCA循环途径促进AD发病[25]。MAP1B(Microtubule associated protein 1B)是编码微管结合蛋白家族的成员,主要在神经元、树突和轴突中表达,在发育过程中富集在轴突生长锥中,具有结合和稳定微管蛋白的能力[26]。MAP1B对于维持细胞骨架的稳定性具有重要的作用,该功能与其磷酸化状态有关,磷酸化MAP1B下调,则表明细胞骨架遭到破坏。经研究发现,AD患者死后脑组织的前额叶皮层出现了磷酸化的MAP1B下调[27]。而且,MAP1B蛋白还是具有低变异系数的生物标志物候选者,可以很好地区分AD和非AD认知障碍病例,并预测脑脊液中Tau/Aβ比率[28]。ITPKB(Inositol-trisphosphate 3-kinase B)是一种普遍表达的脂质激酶[29],并控制各种早期血细胞的信号转导。ITPKB作为一种调节因子,可调节ERK1/2激活,在AD患者中,ERK1/2的激活增加。在5X家族性AD小鼠模型中,神经元ITPKB过表达会显著增加ERK1/2激活和β-分泌酶1活性,导致阿尔茨海默病恶化,具体表现为星形胶质细胞增生,β淀粉样肽1-40产生和tau蛋白过度磷酸化。与健康者相比,AD患者大脑皮层中的ITPKB蛋白水平增加了3倍,并在与淀粉样斑块相关的神经元中积累。这表明,ITPKB可能是阿尔茨海默病患者的治疗新靶点[30]。
综上,本研究基于ANRGs在AD患者中的表达,对其进行更精准的分子分型,之后基于多个机器学习模型构建了可预测患者发病风险的列线图,为临床决策提供依据。然而,本研究存在一些局限性。首先,基于生物信息学的分析,尚需进一步的实验与临床队列研究来评估ANRGs在AD患者中的表达与研究价值。其次,尚需多中心队列研究,详细评估列线图的预测性能与临床意义。最后,ANRGs与AD患者免疫微环境的互作关系,仍需进一步阐明,这些将在后续的研究中不断补充。
4 结论本研究揭示了ANRGs与AD患者免疫细胞浸润的相关性,阐明不同分子亚型患者间的免疫异质性。基于5个基因的XGB模型可以准确区分AD患者与正常个体。首次确定了失巢凋亡在AD中的作用,为进一步阐明该疾病的异质性提供参考。
| [1] |
CLARK C, RABL M, DAYON L, et al. The promise of multi-omics approaches to discover biological alterations with clinical relevance in Alzheimer's disease[J]. Frontiers in Aging Neuroscience, 2022, 14: 1065904. DOI:10.3389/fnagi.2022.1065904 (  0) 0) |
| [2] |
MATTHEWS K A, XU Wei, GAGLIOTI A H, et al. Racial and ethnic estimates of Alzheimer's disease and related dementias in the United States (2015-2060) in adults aged ≥ 65 years[J]. Alzheimer's & Dementia: the Journal of the Alzheimer's Association, 2019, 15(1): 17-24. DOI:10.1016/j.jalz.2018.06.3063 (  0) 0) |
| [3] |
HAMPEL H, MESULAM M M, CUELLO A C, et al. The cholinergic system in the pathophysiology and treatment of Alzheimer's disease[J]. Brain, 2018, 141(7): 1917-1933. DOI:10.1093/brain/awy132 (  0) 0) |
| [4] |
ZHANG Zhigang, YANG Xifei, SONG Youqiang, et al. Autophagy in Alzheimer's disease pathogenesis: Therapeutic potential and future perspectives[J]. Ageing Research Reviews, 2021, 72: 101464. DOI:10.1016/j.arr.2021.101464 (  0) 0) |
| [5] |
CALSOLARO V, EDISON P. Neuroinflammation in Alzheimer's disease: Current evidence and future directions[J]. Alzheimer's & Dementia: the Journal of the Alzheimer's Association, 2016, 12(6): 719-732. DOI:10.1016/j.jalz.2016.02.010 (  0) 0) |
| [6] |
MOUJALLED D, STRASSER A, LIDDELL J R. Molecular mechanisms of cell death in neurological diseases[J]. Cell Death and Differentiation, 2021, 28(7): 2029-2044. DOI:10.1038/s41418-021-00814-y (  0) 0) |
| [7] |
CARRERAS F J. Lessons from glaucoma: rethinking the fluid-brain barriers in common neurodegenerative disorders[J]. Neural Regeneration Research, 2019, 14(6): 962-966. DOI:10.4103/1673-5374.249215 (  0) 0) |
| [8] |
NEWMAN A M, LIU C L, GREEN M R, et al. Robust enumeration of cell subsets from tissue expression profiles[J]. Nature Methods, 2015, 12(5): 453-457. DOI:10.1038/nmeth.3337 (  0) 0) |
| [9] |
SEILER M, HUANG C C, SZALMA S, et al. ConsensusCluster: A software tool for unsupervised cluster discovery in numerical data[J]. Omics: A Journal of Integrative Biology, 2010, 14(1): 109-113. DOI:10.1089/omi.2009.0083 (  0) 0) |
| [10] |
HÄNZELMANN S, CASTELO R, GUINNEY J. GSVA: Gene set variation analysis for microarray and RNA-seq data[J]. BMC Bioinformatics, 2013, 14: 7. DOI:10.1186/1471-2105-14-7 (  0) 0) |
| [11] |
LANGFELDER P, HORVATH S. WGCNA: An R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 (  0) 0) |
| [12] |
RIGATTI S J. Random Forest[J]. Journal of Insurance Medicine (New York, N.Y.), 2017, 47(1): 31-39. DOI:10.17849/insm-47-01-31-39.1 (  0) 0) |
| [13] |
WEI Xiaobin, ZHU Li, ZENG Yanyan, et al. Detection of prostate cancer using diffusion-relaxation correlation spectrum imaging with support vector machine model-a feasibility study[J]. Cancer Imaging, 2022, 22: 77. DOI:10.1186/s40644-022-00516-9 (  0) 0) |
| [14] |
LEE C S, CONWAY C. The role of generalized linear models in handling cost and count data[J]. European Journal of Cardiovascular Nursing, 2022, 21(4): 392-398. DOI:10.1093/eurjcn/zvac002 (  0) 0) |
| [15] |
YU Bin, QIU Wenying, CHEN Cheng, et al. SubMito-XGBoost: Predicting protein submitochondrial localization by fusing multiple feature information and eXtreme gradient boosting[J]. Bioinformatics, 2020, 36(4): 1074-1081. DOI:10.1093/bioinformatics/btz734 (  0) 0) |
| [16] |
DEY A, HANKEY GIBLIN P A. Insights into macrophage heterogeneity and cytokine-induced neuroinflammation in major depressive disorder[J]. Pharmaceuticals, 2018, 11(3): 64. DOI:10.3390/ph11030064 (  0) 0) |
| [17] |
DAI Linbin, SHEN Yong. Insights into T-cell dysfunction in Alzheimer's disease[J]. Aging Cell, 2021, 20(12): e13511. DOI:10.1111/acel.13511 (  0) 0) |
| [18] |
PARANJPE M. D, BELONWU S, WANG J K, et al. Sex-specific cross tissue meta-analysis identifies immune dysregulation in women with Alzheimer's disease[J]. Frontiers in Aging Neuroscience, 2021, 13: 735611. DOI:10.3389/fnagi.2021.735611 (  0) 0) |
| [19] |
FALAHATI F, WESTMAN E, SIMMONS A. Multivariate data analysis and machine learning in Alzheimer's disease with a focus on structural magnetic resonance imaging[J]. Journal of Alzheimer's Disease: JAD, 2014, 41(3): 685-708. DOI:10.3233/JAD-131928 (  0) 0) |
| [20] |
BERNARD B J, NIGAM N, BURKITT K, et al. SMYD3: A regulator of epigenetic and signaling pathways in cancer[J]. Clinical Epigenetics, 2021, 13(1): 45. DOI:10.1186/s13148-021-01021-9 (  0) 0) |
| [21] |
BINH M T, HOAN N X, GIANG D P, et al. Upregulation of SMYD3 and SMYD3 VNTR 3/3 polymorphism increase the risk of hepatocellular carcinoma[J]. Sciences Reports, 2020, 10(1): 2797. DOI:10.1038/s41598-020-59667-z (  0) 0) |
| [22] |
NIKOLAC P M, VIDETIC P A, KONJEVOD M. Epigenetics of alzheimer's disease[J]. Biomolecules, 2021, 11(2): 195. DOI:10.3390/biom11020195 (  0) 0) |
| [23] |
DONG Yina, MESAROS C, XU Peining. Frataxin controls ketone body metabolism through regulation of OXCT1[J]. PNAS Nexus, 2022, 1(3): pgac142. DOI:10.1093/pnasnexus/pgac142 (  0) 0) |
| [24] |
JENSEN N J, WODSCHOW H Z, NILSSON M, et al. Effects of ketone bodies on brain metabolism and function in neurodegenerative diseases[J]. International Journal of Molecular Sciences, 2020, 21(22): 8767. DOI:10.3390/ijms21228767 (  0) 0) |
| [25] |
ZHANG Fan, ZHONG Siran, YANG Siman, et al. Identification of potential therapeutic targets of alzheimer's disease by weighted gene co-expression network analysis[J]. Chinese Medical Sciences Journal, 2020, 35(4): 330-341. DOI:10.24920/003695 (  0) 0) |
| [26] |
HALPAIN S, DEHMELT L. The MAP1 family of microtubule-associated proteins[J]. Genome Biology, 2006, 7(6): 224. DOI:10.1186/gb-2006-7-6-224 (  0) 0) |
| [27] |
CHEN Mei, LEE H K, MOO L, et al. Common proteomic profiles of induced pluripotent stem cell-derived three-dimensional neurons and brain tissue from Alzheimer patients[J]. Journal of Proteomics, 2018, 182: 21-33. DOI:10.1016/j.jprot.2018.04.032 (  0) 0) |
| [28] |
ZHOU Maotian, HAQUE R U, DAMMER E B, et al. Targeted mass spectrometry to quantify brain-derived cerebrospinal fluid biomarkers in Alzheimer's disease[J]. Clinical Proteomics, 2020, 17: 19. DOI:10.1186/s12014-020-09285-8 (  0) 0) |
| [29] |
APICCO D J, SHLEVKOV E, NEZICH C L, et al. The Parkinson's disease-associated gene ITPKB protects against α-synuclein aggregation by regulating ER-to-mitochondria calcium release[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(1): e2006476118. DOI:10.1073/pnas.2006476118 (  0) 0) |
| [30] |
STYGELBOUT V, LEROY K, POUILLON V, et al. Inositol trisphosphate 3-kinase B is increased in human Alzheimer brain and exacerbates mouse Alzheimer pathology[J]. Brain, 2014, 137(Pt 2): 537-552. DOI:10.1093/brain/awt344 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


