2. 宁夏医科大学总医院 医学科学研究院,银川 750004;
3. 宁夏医科大学基础医学院 医学遗传学与细胞生物学系,银川 750004
2. Medical Science Research Institute, General Hospital of Ningxia Medical University, Yinchuan 750004, China;
3. Department of Cell Biology and Medical Genetics, Ningxia Medical University, Yinchuan 750004, China
自身免疫性疾病是一类机体对自身抗原发生免疫反应而导致自身组织损害的疾病,其病因复杂,涉及到遗传、表观遗传和环境等众多因素[1]。系统性红斑狼疮(Systemic lupus erythematosus,SLE)是最常见的慢性自身免疫性疾病,好发于年轻或中年女性,男女发病率约为1∶9。SLE主要临床特征是机体产生大量自身抗体,导致多个器官和组织受损[2]。尽管SLE发病机制的研究有了很大的进展,但由于SLE病因复杂,同时,疾病的异质性较大,其发病机制尚不明确,针对性的诊断和治疗十分有限[3]。近年来,随高通量测序技术的发展,已经发现了许多与SLE发病过程相关的风险基因,然而,这些基因表达调控的分子机制并未被完全阐明。
近年来,表观遗传修饰与SLE的关系越来越引起人们的关注,特别是RNA的化学修饰。目前,已经发现了100多种RNA修饰,包括5-甲基胞苷(5-methylcytosine, m5C)、N6-甲基腺苷(N6-methyladenosine, m6A)、假尿苷(Ψ)、N1-甲基腺苷(N1-methyladenosine, m1A)等[4],可修饰mRNA、tRNA、lncRNA和miRNA[5]。其中,m6A被认为是真核细胞中最普遍的mRNA修饰方式[6],主要分布在G(m6A)C (70%)或者A(m6A)C (30%)保守序列中[7],具有典型的DRACH(例如: [AGU][AG]AC[ACU])基序(Motif)特征,而且,在mRNA 3’ UTR和转录起始位点(Transcription start Site, TSS)丰度最高[8]。目前的研究表明,m6A修饰在调控基因转录、翻译以及mRNA剪切、稳定性等生物学过程中发挥重要作用[9]。m6A修饰是一个动态可逆的过程,由3组主要酶共同参与驱动,包括甲基化转移酶进行目标甲基化、去甲基化酶实现去甲基化以及甲基化阅读蛋白对甲基化RNA标签进行解码,分别被称为"写入器(Writers)"、"擦除器(Erasers)"和"读取器(Readers)"[10]。
越来越多的证据表明,m6A修饰在SLE发病过程中具有重要的调控作用[11]。通过对SLE发病关键信号通路基因的m6A修饰,影响免疫细胞的分化[12]、增殖[13], 凋亡[14]和免疫反应[15]等过程。目前,对于SLE中差异表达风险基因mRNA的m6A修饰,在整体分布、调控机制及其与SLE病理生理过程之间的关系仍不清楚。因此,深入探讨m6A对SLE中风险基因mRNA的修饰作用和机制,对于揭示SLE发病机制、发现新的治疗靶点具有重要的基础和临床意义。
本研究利用ADEx(Autoimmune diseases explorer)转录组数据集和高通量基因表达数据库(Gene expression omnibus, GEO)中的m6A高通量测序数据集,比较SLE患者和健康对照组中的差异表达基因mRNA的m6A修饰,筛选差异表达m6A修饰化的mRNA,采用生物信息学预测其m6A位点;通过Pathway注释分析、互作网络分析和表达数据集,验证其在SLE发病机制中的作用和意义,以期为SLE的诊断、治疗和预防提供新的思路和方法。
1 材料与方法 1.1 数据集使用关键词“(SLE) or (Systemic lupus erythematosus) and (m6A)”在GEO数据库中检索,获取了1项SLE患者外周血单个有核细胞(PBMC)的Methylated RNA immunoprecipitation-seq(MeRIP-seq)数据集(GSE173312);同时,在ADEx(https://adex.genyo.es)数据库[16]中搜索SLE患者转录组数据集,并对差异表达基因进行RRA(Robust rank aggregation) Meta分析,筛选出前50个基因,进行无监督聚类可视化。
1.2 差异表达分析首先,从GEO数据库下载SLE患者外周血单个有核细胞(Peripheral blood mononuclear cells,PBMC)的MeRIP-seq数据集(GSE173312);使用Hisat2,将洁净Reads与人类参考基因组(hg38)比对,利用FeatureCounts获得表达矩阵;建立SLE-HC样本分组,采用t-SNE和无监督垂直聚类分析对样本和基因进行分析。为了进一步分析差异表达基因(Differentialy expressed genes, DEGs),分别采用了DESeq2, Limma和EdgeR三种工具进行差异表达分析并选择交集;设定Log2FC绝对值大于1和调整后P-value小于0.05为筛选阈值,获得SLE患者PBMCs发生m6A修饰的差异表达基因数据集,使用热图和火山图进行可视化分析。
1.3 m6A修饰相关基因的获得检索m6A2Target数据库,纳入22个m6A系统基因,包括7个写入因子, 13个阅读因子和2个擦除因子;其中,写入因子基因包括METTL3, METTL14, WTAP, RBM15, RBM15B, CBLL1和ZC3H13;擦除因子基因包括FTO和ALKBH5;阅读因子基因包括YTHDF1, YTHDF2, YTHDF3, YTHDC1, YTHDC2, HNRNPC, HNRNPA2B1, IGF2BP1, IGF2BP2, IGF2BP3, FMR1, ELAVL1和LRPPRC。通过将SLE差异表达基因、m6A测序差异表达基因和m6A组分进行交叉,得到发生m6A修饰的SLE风险基因的集合。
1.4 m6A靶mRNA基因GO和PPI网络互作分析为了识别m6A修饰的SLE调控网络中的核心基因,所有基因被输入到STRING (https://string-db.org)数据库中,以获得基因与蛋白质相互作用(Protein-protein interaction,PPI)网络;采用MCL算法(n=3),对所获网络进行聚类分析;用Cytoscape软件中的ClueGO对PPI网络进行GO-gene可视化;采用MCODE和CytoHubba插件,通过PPI网络划分模块(节点分数阈值为0.2,K-core=2),获得核心干扰素刺激基因(Interferon-stimulated genes,ISGs)m6A修饰互作网络。
1.5 统计分析数据处理和分析采用R软件(4.2.0版)、SPSS(26.0版)和GraphPad Prism(9.0.2版)。通过统计分析和直方图获得数据分布特征;连续非正态分布变量的组间差异,采用Mann-Whitney U检验(Wilcoxon秩和检验)分析;多组间的分类数据,采用Chi检验;利用ANOVA检验比较多组间的变量;多重比较采用错误发现率(False discovery rate, FDR)校正。以P<0.05为显著性水平。
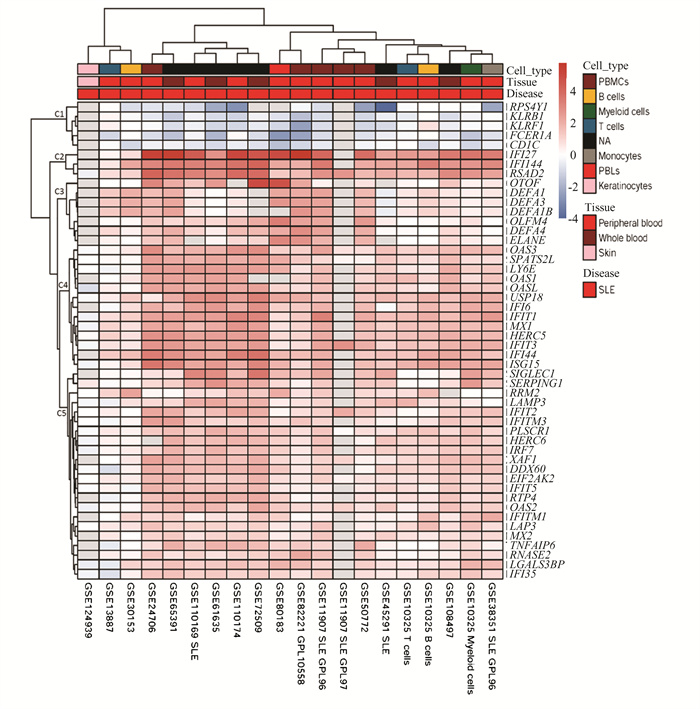
2 结果 2.1 SLE患者PBMCs一致性差异表达基因ADEx数据库纳入20个SLE芯片数据集,通过GEO RRA Meta分析,共获得1 703个差异表达的基因,以Top50基因进行聚类分析(图 1),共有5簇基因集,其中C2-C5簇中大部分基因已报道为Ⅰ型干扰素信号通路基因,在SLE患者不同免疫细胞中显著聚类且表达升高。
|
图 1 SLE患者差异表达基因Meta分析(Top50) Figure 1 Meta-analysis of differentially expressed genes in SLE patients (Top50) |
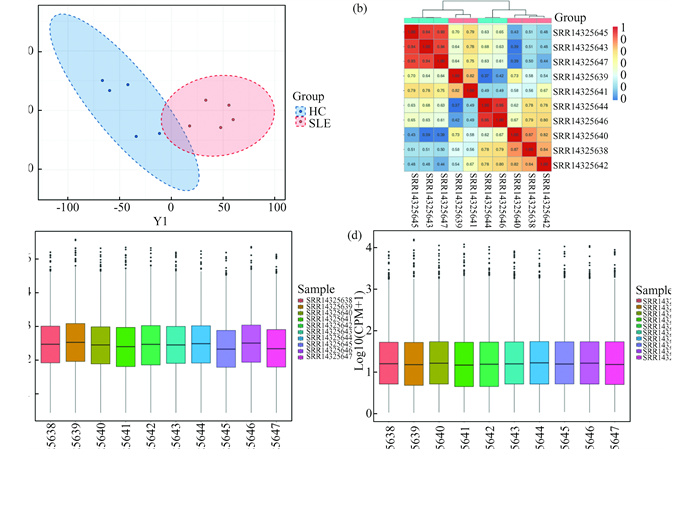
以至少在重复样本数量内的表达量(Counts)大于1为标准,过滤表达矩阵。tSNE样本聚类分析显示,MeRIP富集基因能够区分SLE患者和健康对照(图 2(a))。以表达中位值前1 000的基因对样本CPM进行相关性聚类分析,结果显示,SLE组SRR14325639和SRR14325641样本,健康对照组SRR14325644和SRR14325646,未能完全聚类。SLE组样本SRR14325638、SRR14325640和SRR14325642,健康对照组SRR14325643, SRR14325645和SRR14325647,共6个样本一致性大于0.85 (图 2(b))。结合tSNE降维结果, SLE个体异质性及与SLE差异表达基因的交叉分析,本研究未去除聚类不一致的样本。m6A测序原始数据集(Reads)经CPM标准化转换后,分布均一,可进行下游差异表达分析(图 2(c)-2(d))。
|
图 2 m6A RNA-seq数据质控结果:(a)tSNE样本聚类分析; (b)10个样本的PCC相关系数热图; (c)未标准化测序洁净reads的分布箱式图; (d)CPM标准化后的表达分布箱式图 Figure 2 Quality control results of m6A RNA-seq data : (a) Cluster analysis of samples using tSNE; (b) Heat map of PCC correlation coefficients of 10 samples; (c) Box plot of distribution of unstandardized sequenced clean reads; (d) Box plot of expression distribution after CPM normalization |
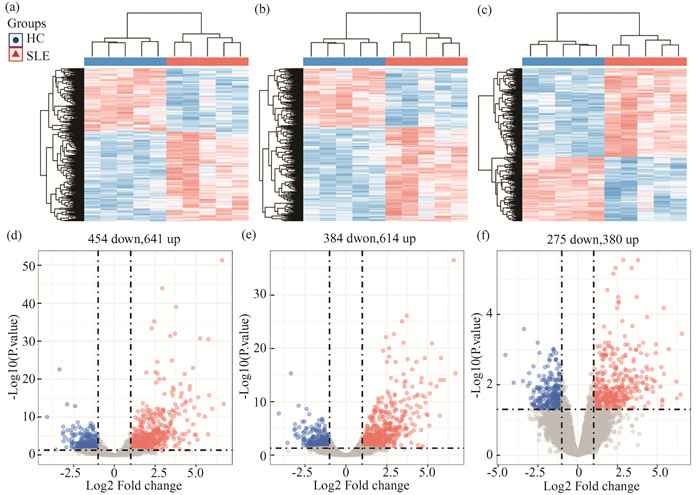
分别采用三种差异表达分析方法,获取SLE患者m6A修饰差异表达基因(表 1),其中DEseq2共获得1 095个差异表达基因,包括641个上调基因和454个下调基因(图 3(a)-3(b))。edgeR共获得998个差异表达基因,包括614个上调基因和384个下调基因(图 3(c)-3(d))。Limma Voom共获得665个差异表达基因,包括380个上调基因和275个下调基因(图 3(e)-3(f))。上述鉴定到的差异表达基因均能良好区分疾病组和对照组。
| 表 1 差异表达m6A修饰基因结果统计 Table 1 Statistics of the results of differentially expressed m6A modified genes |
|
图 3 三种不同分析方法差异表达基因可视化:(a-c)DEseq2, edgeR和Limma Voom差异表达基因聚类热图及(d-f)火山图 Figure 3 Visualization of differentially expressed genes by three different analysis methods: (a-c) Heat map of DEseq2, edgeR and Limma Voom differentially expressed genes clustering and (d-f) volcano map |
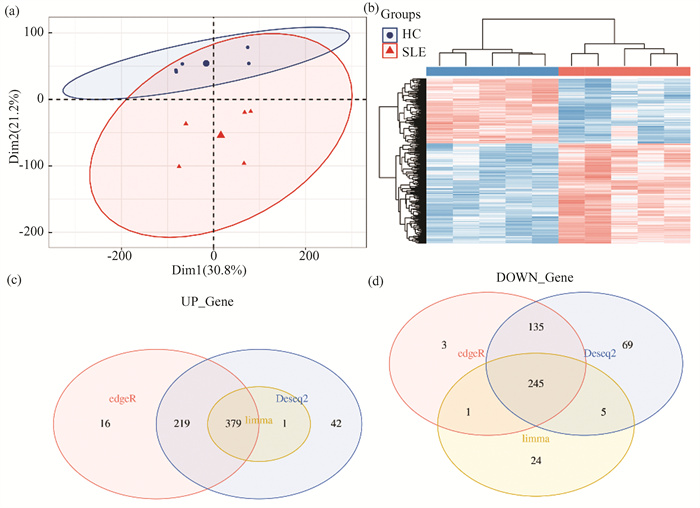
PCA结果显示,三种不同算法交叉获得的差异表达基因,能够很好的将样本进行聚类,其中第一主成分占比30.8%, 第二主成分占比21.2%,累计可解释52%的基因变异(图 4(a))。Venn分析共获得724个,包括上调基因379个(图 4(b)),下调表达基因245个(图 4(c)),m6A修饰基因聚类分析结果表明,在SLE患者组和健康对照组,表达具有显著性差异(图 4(d))。
|
图 4 SLE患者PBMCs m6A修饰差异表达分析结果:(a)差异表达基因PCA聚类分析; (b)无监督聚类热图; (c)上调差异表达基因交集; (d)下调差异表达基因交集 Figure 4 Results of differential expression analysis of m6A modifications in PBMCs from SLE patients: (a) PCA clustering analysis of differentially expressed genes; (b) Unsupervised clustering heat map; (c) Intersection of up-regulated differentially expressed genes; (d) Intersection of down-regulated differentially expressed genes |
SLE患者具有较强的异质性,为获得显著差异表达且发生m6A修饰的基因数据,将SLE差异表达基因Meta数据集(SLE_DEGs), m6A组分基因集和SLE PBMCs MeRIP-seq数据集(m6A_Down和m6A_Up)作为输入,经Venn分析,结果表明,m6A组分基因与MeRIP-seq基因之间没有交集,但与SLE差异表达基因集之间,共有3个组分基因,分别是RBM15B, FTO和IGF2BP3。SLE差异表达基因与MeRIP-seq靶标共有181个差异表达且发生m6A修饰的基因,包含上调表达123个和下调表达58个(图 5)。
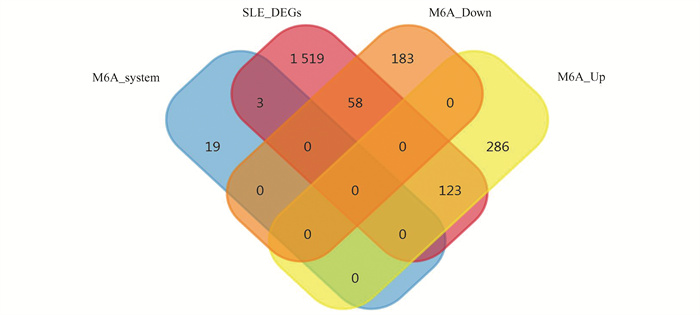
|
图 5 SLE患者m6A修饰差异表达基因Venn分析 Figure 5 Venn analysis of m6A-modified differentially expressed genes in SLE patients |
纳入分析的m6A组分共有22个,经差异表达Meta无监督聚类分析,结果显示,与健康对照组比较,SLE患者甲基化系统组分基因异常表达(图 6(a))。在FDR<0.01的筛选阈值条件下,校正差异表达(图 6(b))结果显示,IGF2BP3基因在3个数据集中均显著上调表达(P<0.01);ALKBH5和FTO分别在5个和2个数据集中显著下调表达(P<0.01)。整体上,SLE患者m6A系统组分基因在纳入的数据集中,未表现出完全一致的表达趋势,表明SLE患者中m6A修饰状态存在异质性,可能和群体及疾病的活动状态密切相关。对基因进行组织细胞位点差异表达分析,结果显示,差异表达基因主要分布在血浆(83.6%)、骨髓(67.2%), 淋巴结(57.4%)以及CD8 B细胞(38.5%)(图 6(c))。进一步对181个发生m6A修饰的异常表达基因进行KEGG Pathway分析,结果显示,主要富集于免疫系统(32.1%)、干扰素信号通路(22.6%)、细胞因子网络(20.2%), 细胞周期和有丝分裂(27.4%)及DNA复制和延伸相关过程(21.4%)(图 6(d))。
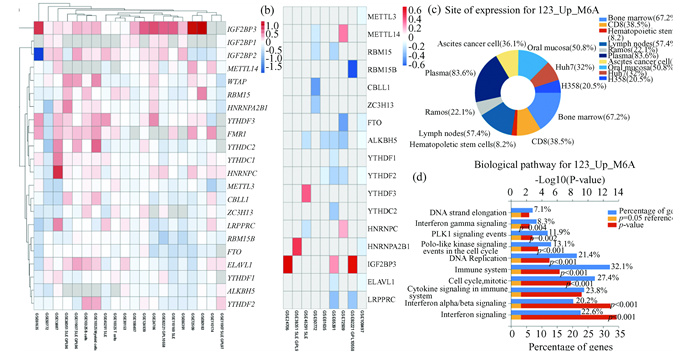
|
图 6 m6A组分基因Meta分析及SLE患者m6A修饰差异表达基因KEGG和组织位点差异表达分析:(a) 未经FDR校正的m6A组分分析热图; (b) FDR校正的m6A组分分析热图; (c) m6A修饰异常表达基因组织细胞位点差异表达分析; (d) m6A修饰异常表达基因KEGG Pathway分析 Figure 6 Meta-analysis of m6A component genes and Analysis of differentially expressed gene KEGG and tissue locus differential expression in SLE patients with m6Amodifications: (a) Heatmap of m6A component analysis without FDR correction; (b) Heatmap of m6A component analysis with FDR correction; (c) Differential expression analysis of m6A-modified aberrantly expressed genes at tissue cell sites; (d) KEGG Pathway analysis of m6A-modified aberrantly expressed genes |
以181个发生m6A修饰的基因为对象,构建PPI网络,进行MCL聚类分析,共获得4个Cluster,其中,Cluster1主要与细胞周期和DNA甲基化及DNA损伤修复密切相关(图 7(a)),Cluster2主要与Ⅰ型干扰素密切相关,其中已报道[17]的11个干扰素应答的核心基因,均被MeRIP-seq捕获,表明,这些干扰素应答基因均有m6A修饰(图 7(b))。进一步对ISG网络降维分析,结果表明,异常表达基因主要参与Ⅰ型干扰素信号通路, γ干扰素信号通路和MDA-5信号通路(图 7(c))。采用MONCODE降维分析,Cluster3主要与染色体组装修饰有关(图 7(d)),Cluster4主要与B细胞MHC Ⅱ抗原呈递过程有关(图 7(e))。进一步,对ISGs网络降维进行分析,结果显示,经m6A修饰的ISG15, IFI44L, USP18, IFIT1, RSAD2, IFI27位于ISG信号网络中的核心节点(图 7(f))。

|
图 7 m6A基因PPI网络MCL聚类及核心基因分析:(a) 细胞增殖与细胞周期m6A修饰基因互作网络; (b) Ⅰ型干扰素m6A修饰基因互作网络; (c) KEGG-gene通路分析; (d) DNA甲基化与染色体组装m6A修饰基因互作网络; (e) B细胞MHC Ⅱ抗原分子呈递m6A修饰相关基因网络; (f) Ⅰ型干扰素m6A修饰基因互作网络权重分析 Figure 7 MCL clustering and core gene analysis of m6A gene PPI network: (a) Cell proliferation and cell cycle m6Amodifier gene interaction network; (b) Type Ⅰ interferon m6A modifier gene interaction network; (c) KEGG-gene pathway analysis; (d) DNA methylation and chromosome assembly m6A modifier gene interaction network; (e) B-cell MHC Ⅱ antigen molecule presentation m6A modifier-related gene network; (f) Type Ⅰ interferon m6A modifier gene interaction Network weighting analysis |
SLE是一种自身免疫性疾病,其特征是机体产生攻击自身组织和器官的抗体,导致全身多个器官的炎症反应和组织损伤。近年来,越来越多的研究表明,RNA修饰在SLE的发病机制中发挥了重要作用,特别是m6A修饰。m6A修饰是一种常见的RNA修饰方式,可通过调节RNA的代谢、出核以及翻译,从转录后水平调控基因表达[18]。最近的研究表明,在SLE患者中mRNA, miRNA和lncRNA等多种RNA出现m6A修饰异常,与SLE患者中免疫细胞的增殖、分化, 凋亡以及免疫功能密切相关。例如,m6A修饰异常可激活B细胞和T细胞,进而改变其免疫功能,导致自身抗体的产生和免疫细胞的异常反应[19]。筛选和鉴定m6A修饰异常基因,探讨其在SLE发病机制中可能的作用,可为SLE的治疗提供新的思路和策略。
本研究首先系统分析了SLE患者中差异表达的编码基因,共筛选获得了1 703个异常表达的基因,大部分基因在SLE病理过程中的作用还不清楚。由于m6A修饰对基因表达具有重要的调控作用,可以推测,或许这些基因mRNA的m6A修饰在SLE发病中扮演者重要角色,因此,本文基于数据库,通过生物信息学,分析了SLE患者差异表达基因中m6A修饰的分布,发现SLE患者PBMCs中存在大量RNA免疫共沉淀捕获基因,表明在SLE发病过程中,差异表达基因的m6A修饰,可能会改变B细胞、T细胞等免疫细胞的功能,引发自身免疫反应。
此外,本研究在SLE患者PBMCs中,筛选出了编码参与m6A修饰调节因子的基因,包括写入因子FTO, ALKBH5和阅读因子IGFBP3。迄今为止,尚未发现ALKBH5和FTO与m6A RNA底物的共晶结构,对于ALKBH5和FTO靶分子的预测仍依赖于生物信息学。通过对SLE全转录组进行Meta分析,发现,ALKBH5和FTO在mRNA水平上均显著下调,提示SLE患者外周血PBMCs中的m6A写入水平可能会下降,这与Wu等[20]的研究结果一致。此外,一项基于RT-qPCR技术,分析了54例SLE患者和42例健康对照METTL14, ALKBH5和YTHDF2的差异表达。结果表明,ALKBH5等写入因子的基因表达显著下调[21],影响T细胞的凋亡和增殖,但其具体机制仍需进一步研究。本研究结果提示,SLE患者中细胞周期相关基因发生了m6A修饰,这可能与ALKBH5有关,具体机制需要进一步证实。同时,一项基于生物信息学研究发现,IGFBP3基因是GSE49454, GSE61635, GSE110169和GSE72509共同鉴定到的SLE的m6A组分基因[15],提示IGFBP3可能参与了SLE的病理过程。
为了进一步研究SLE差异表达基因中m6A修饰的特征,我们对公共数据进行了转录组范围的m6A修饰筛查,筛选出287个可能在SLE PBMCs中发生m6A修饰的基因。基因本体分析表明,这些基因与一些免疫细胞的富集相关,涉及到干扰素信号通路。有趣的是,我们发现,在SLE患者PBMCs中,FTO和ALKBH5等擦除酶基因表达下调,可导致m6A水平下降和干扰素刺激基因表达的增加,提示ALKBH5和FTO参与了与SLE发病相关的干扰素信号通路基因m6A的修饰。有研究表明,m6A去甲基化酶ALKBH5可通过IKKε/TBK1/IRF3通路抑制RIG-I表达和干扰素α的产生,促进头颈鳞状细胞癌进展[22-23], 但在SLE中, m6A修饰与ISGs基因的表达调控的关系尚不清楚。
筛选发生m6A修饰的SLE差异表达基因,可为SLE的发病机制的研究提供新的视角,同时,也为针对差异表达基因m6A途径的治疗策略,提供一定的理论基础。但本研究还有一定的局限性,尚未在体外与体内进行验证,后续尚需在SLE患者、细胞以及模型动物进一步研究,以期阐明m6A修饰在SLE发病过程中的作用机制。
| [1] |
WANG Lifeng, WANG Fusheng, GERSHWIN M E. Human autoimmune diseases: A comprehensive update[J]. Journal of Internal Medicine, 2015, 278(4): 369-395. DOI:10.1111/joim.12395 (  0) 0) |
| [2] |
方心宇, 冷瑞雪, 范引光, 等. 自身免疫性疾病流行病学研究进展[J]. 补充杂志名称, 2021, 25(8): 5. FANG Xinyu, LENG Ruixue, FAN Yinguang, et al. Research advances in the epidemiology of autoimmune diseases[J]. Chinese Journal of Disease Control & Prevention, 2021, 25(8): 869-873. DOI:10.16462/j.cnki.zhjbkz.2021.08.001 (  0) 0) |
| [3] |
AMEER M A, CHAUDHRY H, MUSHTAQ J, et al. An Overview of systemic lupus erythematosus (SLE) pathogenesis, classification, and management[J]. Cureus, 2022, 14(10): e30330. DOI:10.7759/cureus.30330 (  0) 0) |
| [4] |
ZHANG Zijun, PARK E, LIN Lan, et al. A panoramic view of RNA modifications: exploring new frontiers[J]. Genome Biology, 2018, 19(1): 11. DOI:10.1186/s13059-018-1394-4 (  0) 0) |
| [5] |
BOCCALETTO P, STEFANIAK F, RAY A, et al. MODOMICS: A database of RNA modification pathways. 2021 update[J]. Nucleic Acids Research, 2022, 50(D1): D231-d235. DOI:10.1093/nar/gkab1083 (  0) 0) |
| [6] |
LUO Qing, GAO Yujie, ZHANG Lu, et al. Decreased ALKBH5, FTO, and YTHDF2 in peripheral blood are as risk factors for rheumatoid arthritis[J]. BioMed Research International, 2020, 2020: 5735279. DOI:10.1155/2020/5735279 (  0) 0) |
| [7] |
ZHANG Hui, SHI Xinrui, HUANG Tao, et al. Dynamic landscape and evolution of m6A methylation in human[J]. Nucleic Acids Research, 2020, 48(11): 6251-6264. DOI:10.1093/nar/gkaa347 (  0) 0) |
| [8] |
MEYER K D, SALETORE Y, ZUMB P, et al. Comprehensive analysis of mRNA methylation reveals enrichment in 3' UTRs and near stop codons[J]. Cell, 2012, 149(7): 1635-1646. DOI:10.1016/j.cell.2012.05.003 (  0) 0) |
| [9] |
TATENO M, FUKUNISHI Y, KOMATSU S, et al. Identification of a novel member of the snail/Gfi-1 repressor family, mlt 1, which is methylated and silenced in liver tumors of SV40 T antigen transgenic mice[J]. Cancer Research, 2001, 61(3): 1144-1153. (  0) 0) |
| [10] |
SHI Hailing, WEI Jiangbo, HE Chuan. Where, When, and How: Context-dependent functions of RNA methylation writers, readers, and erasers[J]. Molecular Cell, 2019, 74(4): 640-650. DOI:10.1016/j.molcel.2019.04.025 (  0) 0) |
| [11] |
吴俊. m6A修饰在系统性红斑狼疮发病中的作用研究[D]. 合肥: 安徽医科大学, 2021. DOI: 10.26921/d.cnki.ganyu.2022.000291. WU Jun. Study on the role of N6-methyladenosine demethylase in systemic lupus erythemat-osus[D]. Hefei: Anhui Medical University, 2022. DOI: 10.26921/d.cnki.ganyu.2022.000291. (  0) 0) |
| [12] |
LEE H, BAO Suying, QIAN Yingzhi, et al. Stage-specific requirement for Mettl3-dependent m(6)A mRNA methylation during haematopoietic stem cell differentiation[J]. Nature Cell Biology, 2019, 21(6): 700-709. DOI:10.1038/s41556-019-0318-1 (  0) 0) |
| [13] |
王晓雨. m6A修饰长链非编码RNA在系统性红斑狼疮中的作用研究[D]. 合肥: 安徽医科大学, 2022. DOI: 10.26921/d.cnki.ganyu.2022.001195. WANG Xiaoyu. Study on the role of N6-methyladenosine (m6A) modificated long noncoding RNA in systemic lupus erythematosus[D]. Hefei: Anhui Medical University, 2022. DOI: 10.26921/d.cnki.ganyu.2022.001195. (  0) 0) |
| [14] |
LIU Shuiping, ZHUO Lüjia, WANG Jianjun, et al. METTL3 plays multiple functions in biological processes[J]. American Journal of Cancer Research, 2020, 10(6): 1631-1646. (  0) 0) |
| [15] |
ZHAO Xingwang, GE Lan, WANG Juan, et al. Exploration of potential integrated models of N6-Methyladenosine immunity in systemic lupus erythematosus by bioinformatic analyses[J]. Frontiers in Immunology, 2021, 12: 752736. DOI:10.3389/fimmu.2021.752736 (  0) 0) |
| [16] |
MARTORELL-MARUGÁN J, LOPEZ-DOMINGUEZ R, GARCIA-MORENO A, et al. A comprehensive database for integrated analysis of omics data in autoimmune diseases[J]. BMC Bioinformatics, 2021, 22(1): 343. DOI:10.1186/s12859-021-04268-4 (  0) 0) |
| [17] |
SHEN Mengjia, DUAN Congcong, XIE Changhao, et al. Identification of key interferon-stimulated genes for indicating the condition of patients with systemic lupus erythematosus[J]. Frontiers in Immunology, 2022, 13: 962393. DOI:10.3389/fimmu.2022.962393 (  0) 0) |
| [18] |
LV Xinyi, LIU Xiaomin, ZHAO Ming, et al. RNA methylation in systemic lupus erythematosus[J]. Frontiers in Cell and Developmental Biology, 2021, 9: 696559. DOI:10.3389/fcell.2021.696559 (  0) 0) |
| [19] |
LU Shuang, WEI Xingyu, ZHU Huan, et al. m(6)A methyltransferase METTL3 programs CD4(+) T-cell activation and effector T-cell differentiation in systemic lupus erythematosus[J]. Molecular Medicine, 2023, 29(1): 46. DOI:10.1186/s10020-023-00643-4 (  0) 0) |
| [20] |
WU Jun, DENG Lijun, XIA Yuanrui R, et al. Involvement of N6-methyladenosine modifications of long noncoding RNAs in systemic lupus erythematosus[J]. Molecular Immunology, 2022, 143: 77-84. DOI:10.1016/j.molimm.2022.01.006 (  0) 0) |
| [21] |
LUO Qing, RAO Jiayue, ZHANG Lu, et al. The study of METTL14, ALKBH5, and YTHDF2 in peripheral blood mononuclear cells from systemic lupus erythematosus[J]. Molecular Genetics and Genomics, 2020, 8(9): e1298. DOI:10.1002/mgg3.1298 (  0) 0) |
| [22] |
JIN Shufang, LI Mingyu, CHANG Hanyue, et al. The m6A demethylase ALKBH5 promotes tumor progression by inhibiting RIG-I expression and interferon alpha production through the IKKε/TBK1/IRF3 pathway in head and neck squamous cell carcinoma[J]. Molecular Cancer, 2022, 21(1): 97. DOI:10.1186/s12943-022-01572-2 (  0) 0) |
| [23] |
DENG Lijun, FANG Xinyu, WU Jun, et al. ALKBH5 expression could affect the function of t cells in systemic lupus erythematosus patients: A case-control study[J]. Current Pharmaceutical Design, 2022, 28(27): 2270-2278. DOI:10.2174/1381612828666220617154204 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


