2. 福建省海洋酶工程重点实验室,福州 350116;
3. 福建福大百特生物科技有限公司,福州 350100
2. Fujian Key Laboratory of Marine Enzyme Engineering, Fuzhou 350100, China;
3. Fujian Fuda Baite Biotechnology Co., Ltd. Fuzhou 350100, China
在过去,业界对药物分子成药性机理的理解往往滞后于相应药物实体的发现,特别是一些自然界中天然存在依靠经验而发现的药物。近些年来,随着生物分子组学技术的快速发展,这给生物医药研发和相应药物作用机理的深入解释带来了良好的契机。基因组学和蛋白质组学方法近些年已经在不同物种测序中的得到广泛的应用,各种物种高质量的测序数据得以产生,这给学术界带来了海量的生物分子层面的数据,其中不仅仅包括DNA和mRNA数据,还包括蛋白质序列及其翻译后修饰数据(例如,甲基化、乙酰化、磷酸化和糖基化等功能位点数据),蛋白质突变和稳定性数据,以及蛋白质的亚细胞定位等信息,这些组学以及相关功能数据为基于分子的药物设计提供了良好的数据支撑以及发展机会。2018年诺贝尔化学奖颁发给弗朗西丝、乔治和格雷戈里,奖励他们提出的酶定向进化方法和噬菌体展示技术。伴随着酶定向进化、基因合成,测序技术以及计算机辅助的分子模拟技术的发展,使得基于蛋白质受体的药物分子设计技术得到极大的发展。从技术性上来说,有效的药物分子在研发过程主要存在两大主题需要深入探究:一是与疾病相关的靶标生物大分子的计算模拟进行虚拟筛选和实验验证;另外一个主题是具有生物活性的小分子药物的筛选、发现和进一步优化改进等。本文聚焦在确定靶标受体基础上进行计算机辅助药物设计(Computer aided drug design)。计算机辅助药物设计是以计算化学为基础,通过计算机的模拟、计算和分析药物与受体生物大分子之间的关系,设计和优化先导化合物的一种方法。计算机辅助药物设计一般可以提高药物研发的可行性、效率和准确度。通过药物分子设计方法的有效实施,可以减少不必要的试验次数,从而大幅度缩短了研发周期。基于蛋白质受体的药物分子设计,一般可以先分析药物分子在相应受体蛋白上的作用靶点,这个过程分析的数据一般为蛋白质的四级结构数据,即靶标受体与其他配体分子(例如,蛋白质抑制剂小分子、金属离子、DNA、RNA、多肽、蛋白质等分子)或者辅酶(Coenzyme)形成的复合物结构以及相互作用信息。蛋白质四级结构一般也包括那些独立的三级结构单元聚集形成的多聚体,其中每个独立三级结构一般称为亚基,也称为单体(Monomer)。一个复合物如果包含两个亚基,则称为为二聚体(Dimer),含三个亚基则称三聚体(Trimer);类似地,一个复合物也可能是四聚体(Tetramer)、五聚体(Pentamer)或者六聚体(Hexamer)等,特别是大部分通道蛋白(例如钙离子通道蛋白)是以多聚体的形式存在。经过数十年的发展,计算机辅助技术设计出来的药物在多种疾病治疗中都有着广泛的应用,例如在癌症以及肺炎等疾病的治疗中都有相应文献报道[1-4]。同时,基于蛋白质受体的药物分析也有一定程度的进展。例如,Lu等[5]发现人体中钙离子通道TRPC6相关蛋白突变体(N338S)可以降低对相应药物的毒性。Lu等通过采用同源建模、Autodock分子对接以及Namd分子动力学模拟等方法系统分析相应数据,并对相应的潜在机理进行解释,同时采用方差分析(Anova)等统计学方法分析不同突变组和野生型TRPC6蛋白之间的差异性。伴随着组学数据积累的推进因素之外,特别是近年来随着机器学习、统计预测方法和深度学习算法的稳步发展,这使得基于计算模拟的算法近来在药物分子设计中也有一定程度上的应用[6-7]。在药物发现和设计中,机器学习和深度神经元网络已经成为一个非常活跃的研究方向[8]。例如Rezaei等[9]开发了一种基于深度学习网络的蛋白质与配体亲和力预测算法。近些年,机器学习算法在蛋白质-配体打分函数以及结合自由能计算中也有一定的应用[10]。基于机器学习和深度学习的模型一般利用数据驱动的模型来计算,预测结果也有较好的参考价值。目前,机器学习和深度学习算法已被应用于多种药物发现过程中,如蛋白质-配体打分、肽合成、基于结构的虚拟筛选、基于配体的虚拟筛查、毒性预测、药效团建模、定量构效关系、药物重新定位等[11]。药物设计中虚拟组合库的构建一般通过计算机模拟的方法,建立拥有上万个化合物的数据,并通过一些算法来表征分子结构并优化化合物库。另外,深度学习算法在虚拟组合库的构建方法的应用也有报道[12]。另外,近年来金属离子(例如Ca+,Mg2+,Cu2+等离子)[13]以及活性多肽[14]等相关内容,在对药物分子设计的作用机理理解上也有相应的进展和运用。随着分子生物学、生物化学,化学信息学和生物信息学等学科的持续发展,在现代微观生物学研究中,合理药物分子的理性设计一般是基于对疾病的分子病理以及生理学的深入理解。同时根据受体靶点的分子结构,并参考效应分子的化学结构特征设计出针对该疾病的药物分子,从而让药物分子的设计更加合理化与科学化。在此框架下设计出的药物分子往往活性强,作用专一以及副作用较低,故称为理性药物分子设计。较早的信息也许可以追溯到Hansch等[15]于20世纪60年代就已经提出了与现代理性药物分子设计理念一致的相似观点:“药物的生物活性与药物分子的一些理化参数(例如电子效应和空间效应等相关的参数)有较强的关系,且大多数理化参数都具有加和性,可以通过统计学方法推导出这些理化参数与生物活性的关系”。定量构效关系研究是应用数学模式来表达药物的化学结构因素与特定生物活性强度的相互关系。通过定量解析药物与靶点特定的相互作用,从而寻找药物的化学结构与生物活性间的量变规律,从而为潜在药物分子的结构优化提供理论依据。在药物分子设计时,需要考虑药物分子的结构对蛋白质活性的影响,即药物分子与蛋白质之间的构效关系。在本文后续的篇幅中,将按照如下框架对相关方面内容进行讨论:①药物分子设计背景;②计算机辅助药物分子设计常用方法;③分子力场;④基于分子对接的药物分子虚拟筛选;⑤基于分子动力学的药物分子设计;⑥基于药效基团模型的药物分子设计方法;⑦从头药物设计方法;⑧药物分子设计相关软件、数据库资源和经典案例。
1 药物分子设计背景在计算机上进行潜在药物分子分析和设计,是目前科研机构对药物分子进行早期筛选中所采用的主流方法之一。潜在药物分子设计一般分为潜在药物分子先导化合物的发现(Lead discovery)以及候选先导化合物的优化(Lead optimization)两个阶段。先导化合物,又称为原型物(Prototype),简称先导物,是通过各种途径和方法得到的可能具有某种生物活性的化学结构。例如,通过一系列方法筛选得到的潜在药物小分子,即为先导化合物。除了先导化合物和受体的亲和力之外,先导化合物的物理化学性质的优选也是影响后续是否采用的考虑因素。先导化合物未必是可直接应用的优良药物分子,其可能存在一些因素而不能直接使用,例如药效不强、特异性不高、或者存在毒性较大等一些潜在问题而不能直接药用,因此需要对其进行优化。先导化合物作为一种初步有效的结构,对其进行进一步的结构优化和改进,例如一些特定功能基团的修饰和改造等,从而实现其优化和实际应用。因此,先导化合物的分析是新药研发的开端。在药物分子研发过程中,先导化合物的发现和优化是药物分子研发过程中的一个重要步骤。先导化合物发现的途径主要包括:①通过一些偶然的机遇而随机发现;②筛选天然产物有效的活性成分,并进行验证和确定;③从体内生命分子和生物活性物质中筛选发现;④基于体内生物转化的代谢产物而发现;⑤在医学治疗上观察临床作用而发现;⑥基于蛋白质受体分子的结构特点而发现,并经过后续实验验证。在本文中,主要论述基于蛋白质受体特点进行计算机辅助药物分子设计的相关内容。
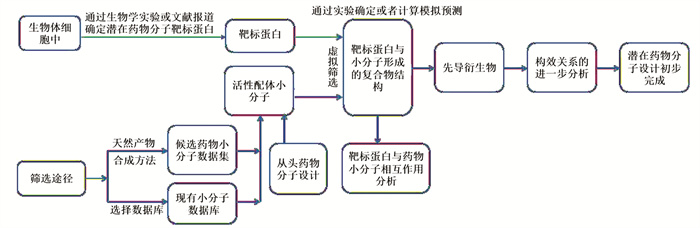
图 1中列出了药物分子设计的一般流程。目前,在以蛋白质受体为基础的新药分子的设计中一般集中在两个体系,一是以受体蛋白质为靶点的体系,设计受体蛋白的小分子调节剂(包括激动剂或者抑制剂等);二是以转运蛋白(包括载体蛋白和通道蛋白)为靶点的体系[16],设计转运蛋白的小分子调节剂。转运蛋白是重要的受体,计算机模拟设计过程中一般是选取其中一部分重要受体蛋白作为药物作用的靶标。一些小分子调节剂能增强蛋白质分子活性,促进某种反应。同时,也存在另外一些小分子调节剂,能降低蛋白质分子活性或者抑制某种反应。根据已知信息的不同,采用的药物分子设计的具体方法是不一样的(表 1)。
|
图 1 药物分子设计的一般流程 Figure 1 The general flowchart of drug molecular design |
| 表 1 不同条件下采用的药物分子分析方法 Table 1 Drug molecular analysis methods used under different conditions |
一般而言,如果已经获得蛋白质晶体结构或者可以找到高相似性同源结构模板,那么可以相对准确地预测出蛋白质的三维结构。这样则可以直接从受体蛋白质的三维结构出发,设计出能与其结合的小分子化合物(即潜在药物分子)[17]。在这个步骤中用到的主要理论研究方法包括分子对接、分子动力学模拟以及从头药物设计(官能团分析)等。初步分析一个蛋白质受体结构时,一般首先把蛋白质结构以表面(Surface)的形式显示,这样易于从表面构象中找出结构空腔(Cavities)。之后,在这些空腔中筛选出其可能的活性结合口袋(Active binding pocket)。随后将已知小分子配体放置于受体蛋白质的活性口袋内,并通过计算方法找出合理的对接构象。这个过程一般是寻找一种复合物构象,使得潜在药物配体和受体蛋白的相互作用能量函数打分数值最佳[18]。特别是随着近些年来计算机三维图形显示技术的快速发展,可以更加细致地观察和分析受体蛋白的活性空腔和结合位点,这为更加精细化的药物分子设计提供了便利。
药物分子与受体结合之后才能产生药效,因此药物分子必须具有高度的选择性和特异性。例如,以离子通道为靶点的药物分子研究,一般通过调节细胞内外的金属离子(例如Na+和Ca2+)跨膜内流而产生效应[19]。合理以及有效的药物分子设计通常离不开良好的计算机算法和软件进行辅助设计。通常来说,药物分子设计中选择对合适的算法和软件也是成功的关键因素之一。
2 计算机辅助药物分子设计常用方法在对未知结构(未通过实验方法解析出结构)的受体蛋白进行分析时,序列数据库搜索和结构数据库比对搜索是生物学家研究受体分子功能的重要手段,这在一个研究项目的起始阶段显得尤为重要。基于生物信息学和结构生物学的药物分子设计方法,这类方法利用大规模的蛋白质序列和结构数据来预测靶标蛋白结构。常用的同源模板搜索的软件包括NCBI Blast[20],MUSTER[21]以及FFAS-3D[22]等。找到合适的结构模板是准确构建受体蛋白结构模型的关键。在FFAS-3D等方法中,使用PSI-BLAST搜索大规模序列数据库(一般为NCBI NR数据库),之后进行序列谱之间的比对(Profile-profile alignment)以此找到合适的结构模板进行建模。药物分子设计方法一般会生成大量的候选药物小分子结构,而如何从这些候选小分子中挑选出潜在的药物小分子是一个非常重要的步骤。无论采用何种设计方法构建出来的潜在药物分子,往往在后续的计算验证步骤中还需要对其进行合理性评估打分。常用的筛选方法包括经验势能函数、经验结合自由能函数、基于知识的平均势能函数、以及依靠研究人员的经验判断进行筛选等策略。此外,遗传算法等统计方法在从头药物设计中也有一定的应用[23]。已有实验研究表明,药物小分子在生物体内不一定都采用能量最低的构象,往往采用的是能量较低的构象,但是这个构象与最低能量值不会差距太大。从筛选出合适小分子构象的角度来说,常用的方法包括构象结构的最优化以及构象的采样分析等。
分析药物分子和受体蛋白质之间关系的方法通常包括:①距离几何法;②分子形状法;③能量分析法等方法。距离几何法的观点是蛋白质和药物小分子配体之间的结合是通过相应的活性空腔相互作用直接决定的,这就要求这些相互作用的部位需要在空间几何位置上相对较近,一般是两个原子之间距离为3-5 Å(这里的单位1 Å等于10-10 m,即纳米的十分之一)之间,同时在能量项(例如静电作用)上相关时则可能产生一定的相互作用。距离几何法一般先确定蛋白质受体中药物分子中可能的作用位点,之后通过配体分子结合位点以及靶点分子活性位点的距离矩阵来确定最佳结合模式以及靶点活性位点的空间分布。简单来说,距离几何法是通过配体分子结合位点以及靶点分子活性位点的距离来确定最佳结合模式。分子形状法是通过寻找药物小分子在三维构象上的相似和差异点,并进行相应的统计从而获得构象和活性之间的关系模型。构象和活性之间的关系模型一般也称为定量构效关系(QSAR),这是一种借助分子的理化性质参数或结构参数,以数学和统计学手段定量研究小分子与生物大分子相互作用、小分子在生物体内吸收、分布、代谢、排泄等生理相关性质的方法。这种方法广泛应用于药物、农药、化学毒剂等生物活性分子的理性设计。对同一个小分子,因为一些区域存在一定的柔性,从而会导致小分子本身会有多种能量比较接近构象的存在。但是,一般来说药物小分子和受体蛋白质相互作用时的活性构象的三维空间构象是基本确定的,这可能和药物小分子在蛋白质活性空腔内受到的空间适应性、以及包括疏水、静电、范德华力以及氢键等一系列分子相互作用力的综合作用有关。因此,可以用一些结构和能量参数来系统性构建小分子和蛋白质活力之间的关系模型。分子形状法是距离几何法的进一步延伸,是在考虑原子之间距离的基础上考虑不同分子之间的互补性。分子形状法一般通过分子对接这种具体的方式进行分析。
设计好的药物分子一般可以保存为PDB或者SMILES等结构信息文件格式进行存储[24]。在药物分子设计过程中,一般也需要考虑靶标受体蛋白在基因组中的编码信息。学术界就有学者提出判断一个基因编码的受体蛋白是否适合作为靶标的一些参考信息[25],例如需要考虑基因的以下一些性质:基因对宿主的存活或者感染是否重要、相同或者同源的基因在其他真菌以及人体中的分布情况、可用以发展高通量筛选的生物化学或者功能信息。另外,生物芯片能够用于疾病组织的转录表达的整体分析,通过比较病变组织与正常组织的基因信息,可以确定疾病相关靶标。与基因组学不同,蛋白质组学方法则从整体上研究正常细胞的蛋白质以及病变组织细胞的蛋白质的差异。这些因素都是药物分子设计过程中需要考虑的。
从蛋白质受体的角度来看,找出一个受体蛋白质活性口袋中相互作用较为合适药物小分子的方法一般可以归为两类:①对数据库进行高通量分子对接筛选;②从头分子设计。高通量分子对接方法一般是从一个具有一定体量的药物分子数据库中进行搜索并根据对接亲和力能量数值和几何构型筛选出符合条件的小分子[26]。从头分子设计方法是通过分析活性口袋的特征,根据活性的亲疏水性基团、电荷基团以及形成氢键等情况设计出一些可以相互作用的官能基团,并把这些设计出来的基团通过一定的顺序连接起来构成一个小分子的方法。认为药物经过结构改造成为其衍生物时,其生物活性的改变主要与结构改变后引起的电子效应、疏水性、以及空间效应的变化相关。当每一因素对生物活性具有独立的、加和性的作用时,可通过统计学方法导出这些理化参数与生物活性的关系式,即Hansch方程。从头设计方法可以全新地构建一个小分子,并很好地与结合位点(Binding site)相互作用[27]。另外,从头设计方法也可以基于一个已有的结构进行优化得到新的有效结构。简言之,基于蛋白质受体的药物分子设计方法是根据靶标生物大分子的结构直接设计出能与其结合的小分子化合物,最常用的是数据库搜寻法(即分子对接)以及从头药物设计,在实际的药物分子设计过程中一般是综合应用多种方法系统性地进行。下面分别介绍这些相关的方法。
3 分子力场分子力学(Molecular mechanics)是建立在经典力学理论基础上,借助经验和半经验参数计算分子结构和能量的方法,又称力场方法(Force field method)。分子力场根据量子力学的波恩-奥本海默近似,一个分子的能量可以近似看作构成分子的各个原子的空间坐标的函数,简单地讲就是分子的能量随分子构型的变化而改变,而描述这种分子能量和分子结构之间关系的就是分子力场函数。分子力场是能量分析法的基础内容,是分子结构相互作用特性的一种简化模型,可以用于相对准确地计算原子之间的相互作用。分子力场包括组成同一分子的各个原子之间的成键相互作用,和不同分子间的范德华相互作用,大部分分子间还有氢键相互作用。学术界目前常用的分子力场包括Amber、CHARMM,CVFF以及MMX等[28-29]。能量分析法也是药物分子设计和分析中常用的方法之一。在药物研发中,模拟的体系包含多种不同的组分,包括蛋白靶标、药物候选配体分子、溶剂以及离子等,对这些组分需要选择合适的力场。其中Amber和CHARMM力场在基于受体的药物分子设计中得到广泛应用[30]。共价与非共价键相互作用是分子力场中的重要元素。共价相互作用是两个原子之间共用电子对。非共价键相互作用是两个分子之间的相互作用或大分子内部的紧密接触。不同类型的非共价键交互作用强度不同,但即使是较弱类型的交互作用,其累积效应是会比较显著的。相互作用只发生在某些原子类型之间,并且必须在一定距离、角度约束和其他限制条件内。对于两个原子之间产生的氢键,氢键供体原子必须与另一个分子上的氢键受体原子保持一定距离(通常为3-5 Å之间)。此外,相关原子群之间形成的角度也需要在特定范围内。传统的氢键相互作用可以存在于氢键供体原子和受体原子之间。元素类型为N,O,P和S的原子通常被视为经典的氢键供体原子,如果与这些原子相连,则氢原子被视为氢键供体。如果存在至少一个电子孤对(例如,NH3而非NH4+),则元素类型N,O,P和S的原子也是氢键受体原子。此外,元素类型F,Cl,Br和I等原子也被认为是氢键受体原子。通过非键相互作用来识别和优化配体和蛋白质之间的相互作用通常是基于结构的药物设计的方法之一。基于力场的能量分析法是综合考虑距离以及各种分子作用力等各种因素,并把这些因素整合为一个能量函数计算得分的分析方法。
4 基于分子对接的药物分子虚拟筛选分子对接方法广泛地应用于潜在药物分子虚拟筛选、蛋白质-药物分子相互作用的研究中。虚拟筛选是药物分子发现和开发过程中的一个重要步骤。在虚拟筛选中,分子对接通常以靶标蛋白质的晶体结构(或者通过结构模拟方法得到的预测结构)作为起始数据,之后利用分子对接方法来预测小分子与受体靶标的结合构象和结合自由能。当受体蛋白的活性口袋确定时,对接程序算法内部使用旋转和平移矩阵(Rotation and translation matrices)让底物分子的各个原子与活性口袋中的结合位点靠近以及相互作用。从而确定底物分子在活性口袋中的位置并完成分子对接过程。分子对接不仅仅用于药物分子设计,在相应生物功能作用机理分析中也有一定的应用。分子对接有时不仅仅考虑药物靶标蛋白质和小分子之间的相互作用,多肽和蛋白质之间的相互作用也是近年来学术界的研究内容之一[31-32]。
药物分子设计中最基本的原理是“锁钥原理”,即药物分子在体内与特定的靶标分子(例如,蛋白质等大分子受体)相互作用,并引起靶标分子的结构和功能的变化,这通常可以通过分子对接的方法进行研究。分子对接结果的分析一般需要同时结合理论化学、计算机和信息科学、分子生物学等相关知识综合进行。分子对接中的“诱导契合”学说认为蛋白质作为受体和药物小分子之间发生相互作用时,蛋白质结构也同时会发生一定程度的变化,特别是蛋白质的活性中心口袋区域,会受到药物小分子的诱导从而在三维结构构象上发生一定程度的变化。与此同时,在相互作用中药物小分子的结构也可能会发生一定程度的变化。因此,在这个过程中需要考虑结构可旋转的分子键和柔性区域等性质信息。在这个相互作用过程中,蛋白质作为受体结构发生了变化,从而有可能关闭或者开启其与其他分子结合的活性空腔。从生物物理学的角度来说,这个对接过程需要综合地考虑各种分子作用力,包括静电、范德华力、氢键以及疏水相互作用等。同时,还需要考虑药物分子在人体中所处的溶剂环境,例如一些脂肪代谢相关的蛋白质在疏水环境中会打开活性口袋从而提升活力[33]。分子对接技术有助于探索蛋白质靶点的功能。基于分子对接的虚拟筛选方法可以用于寻找蛋白质的小分子调节剂。小分子调节剂可以特异性地作用于蛋白质的某些基团,降低或者提升蛋白质的活性。例如,一些小分子调节剂通过限制蛋白质催化底物的反应能力,使底物浓度增高或代谢产物浓度降低,达到改善症状的目的。而另外一些小分子调节剂则可以提高蛋白质的活性,促进某种反应的进行。
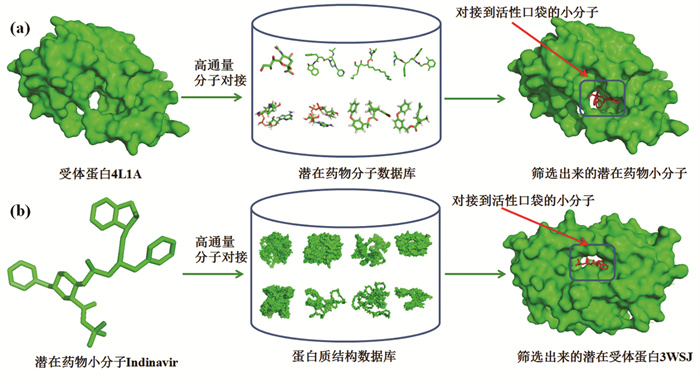
图 2(a)展示了基于受体蛋白搜索小分子数据库进行潜在药物小分子的筛选过程。一般进行搜索时需要确定受体蛋白的活性口袋,搜索结果是根据亲和力的优良进行排序来显示的。图 2(b)展示了基于小分子搜索蛋白质结构数据库过程。这一过程可以在理论上筛选小分子可以作用于哪些受体蛋白,这时的搜索一般数据库中的每一个蛋白质都需要事先知道活性口袋或者结合位点,搜索结果同样是根据亲和力的优良进行排序。
|
图 2 基于高通量分子对接的潜在药物分子筛选 Figure 2 Screening of potential drug molecules based on high-throughput molecular docking 注:(a) 药物小分子的筛选过程;(b) 基于小分子搜索蛋白质结构数据库过程. |
分子对接既可以用于一个蛋白质对一个药物小分子之间的对接,也可以用于一个蛋白质对一整个数据库中的所有潜在药物小分子(例如一个存储超过数万个小分子的数据库)进行大规模的系统性虚拟筛选,并把筛选结果根据对接的亲和力能量数值从低到高进行排序。随着高性能计算机服务器的普遍推广,可搜索的数据库规模也较之前显著增大,这样高通量分子对接的应用将越来越广泛。药物分子设计时也需要考虑辅酶的因素,这与受体的最终性质有一定的影响,这个过程也可以使用分子对接方法进行分析。与底物不同,辅酶是可以与受体蛋白质结合并成为受体的一部分。图 3为辅酶NADPH(烟酰胺腺嘌呤二核苷酸磷酸)和NADH(烟酰胺腺嘌呤二核甘酸)分别和蛋白质3D3F[34]进行相互作用的结果。其中,NADH和NADPH结构是从PubChem(https://pubchem.ncbi.nlm.nih.gov/)[35]数据库中下载得到。NADPH和蛋白质3D3F的复合物结构为实验结构[34]。NADH和蛋白质3D3F相互作用的图是采用分子对接软件Autodock Vina(http://vina.scripps.edu/)[36]进行分子对接,之后使用Discovery Studio2019软件进行相互作用显示。图 3(a)为NADPH在蛋白质3D3F构象中的位置,图 3(b)为NADPH和蛋白质3D3F中氨基酸残基的相互作用图。图 3(c)为NADH在蛋白质3D3F构象中的位置,图 3(d)为NADH和蛋白质3D3F中氨基酸残基的相互作用图。从图 3中可以看出,与NADH相比NADPH和蛋白质3D3F产生更多的分子相互作用,从而增加了复合物结构总体的刚性和稳定性。因此,NADPH-蛋白质的复合物与NADH-蛋白质的复合物相比,很可能在稳定性上会更好一些。在实际的药物分子设计中,除了需要考虑受体蛋白的活性口袋特点之外,还需要考虑受体蛋白相互作用的其他成分,包括金属离子、辅酶等,这些综合因素的考虑是最终药物分子是否可以设计成功的一些参考。
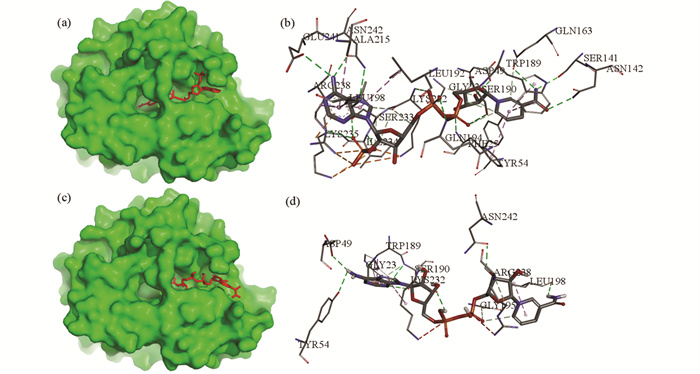
|
图 3 辅酶(Coenzyme)NADPH和NADH分别与蛋白质3D3F结构的相互作用图 Figure 3 Interaction diagrams of coenzymes NADPH and NADH with the protein 3D3F structure, respectively 注:(a) NADPH在蛋白质3D3F构象中的位置; (b)NADPH和蛋白质3D3F中氨基酸残基的相互作用; (c)NADH在蛋白质3D3F构象中的位置;(d)NADH和蛋白质3D3F中氨基酸残基的相互作用. |
高通量分子对接即虚拟筛选,也称计算机筛选,是进行生物活性筛选之前在计算机上对化合物分子进行预筛选,以降低实际筛选化合物的数目,同时提高先导化合物的发现效率。目前高通量筛选中的主要方法之一是分子对接。分子对接的主要作用包括研究活性小分子配体和生物大分子受体之间的相互作用模式、比较不同小分子与同一个大分子之间的不同作用模式、进行虚拟筛选寻找新的先导化合物结构、提供多种小分子结合构象以辅助药物分子设计和分析等[37]。
分子对接以靶标生物大分子的三维结构作为基础,研究小分子与受体分子的相互作用关系,设计出能从空间形状和化学性质两方面都能很好地与靶标分子“结合口袋”相匹配的药物分子。根据受体结合部位的特性,在小分子化合物数据库进行三维结构搜索,然后以搜索到的结构为出发点,进行结构改造,使其与受体的结合位点充分匹配。先建立大量化合物的三维结构数据库,然后将库中的小分子逐一与靶标大分子进行分子对接。主流数据库中的小分子一般为现存已知的化合物,可以从各个商业平台购置得到,或者至少可以在一些相关文献中查阅到其合成方法,因而可以较为快速地进行后续的药理性质测试。Autodock Vina软件用于高通量潜在药物的虚拟筛选中,其运行速度相对较快(例如对比Autodock4.5版本软件)。
5 基于分子动力学的药物分子设计蛋白质的动态结构分析有助于药物分子设计[38]。分子动力学模拟是分析蛋白质动态状况的一种重要方法。分子动力学模拟一般是在分子对接之后进行的一个分析,可以用于分子动态的机理解释和功能分析。因此,分子动力学在药物分子设计中也有着广泛的应用[39-40]。分子动力学的结果可以用于观测和分析蛋白质结构的动态情况,特别是酶活性口袋的开闭以及蛋白质和药物分子相互作用的动态情况。拉伸分子动力学(Steered molecular dynamics simulation)是分子动力学的一种特殊应用,近年来在药物分子设计中也有一定的使用[41]。在通常情况下,在分子动力学进行模拟的基础上,有时还会采用更加精准的量子力学和分子力学(Quantum mechanics and molecular mechanics,简称QM/MM模拟)相结合的模拟[42]。QM/MM模拟方法计算分子结构和原子、分子间相互作用比较准确,但是得到结果的过程较长、速度相对较慢。因此,考虑到速度限制的原因,往往仅仅在活性口袋中选取几个关键的氨基酸进行QM/MM模拟以深入地增强对相应机理的理解。分子动力学模拟中的一个重要步骤是对复合物的总能量进行分解分析。以Amber分子动力学模拟软件为例,其中采用的能量分解模块为MM/PBSA。能量分解其实就是计算配体和受体之间结合的亲和力,并将总能量分解到每一个氨基酸残基上,这样可以清楚地看到每个氨基酸残基对配体结合的具体能量贡献,包括范德华能、溶剂化能以及静电能等。药物小分子与受体分子有效结合之后,在立体空间上互补以及电荷分布上相互匹配,从而会一定程度上引起小分子以及受体蛋白质构象变化,促进受体和配体之间发生相互作用。
分子动力学模拟用于潜在药物分子设计过程中,对模拟时间有一定的要求,一般至少为10 ns以上对分子动态变化的分析才具有统计学上较高的显著性。一般来说,一定时长的分子动力学模拟,需要有良好的计算机硬件设备作为支撑。而且随着模拟蛋白质结构的序列长度增长,这个计算模拟过程所需要的时间则也会越长。可以通过给计算机服务器增加高性能的CPU,GPU(https://ambermd.org/GPUSupport.php)以及高容量的内存等硬件来加快分子动力学的模拟速度以缩短分子动力学的模拟时间。
药物分子和受体分子之间相互作用的过程一般是一个动态的过程,分子动力学方法正好可以很好地模拟这个相互作用的动态过程,以此来更好地确定药物分子作用机制和相互作用力的强弱,在这个过程中可以进一步优化药物分子的设计。Amber和Gromacs可能是药物设计中比较常用的两个分子动力学模拟软件。因为分子动力学运行的命令以及程序参数较多,所以对使用人员的背景知识要求可能较高。分子动力学进行药物分子和靶标蛋白相互作用模拟时,一般需要比较长的时间。
6 基于药效基团模型的药物分子设计方法药效团(Pharmacophore)又称药效基团,其提出源于活性化合物的一部分结构发生变化时,生物活性也发生相应改变,而其余部分结构发生变化时其生物活性发生却很小。这些对化合物的活性有重要作用的基团,称为药效团。药效团通常也指那些可以与受体结合位点形成氢键、静电和疏水等分子作用力相互作用的原子、官能团及其空间关系的共同特征。药效团一般假设具有同类活性的一系列化合物与靶点相互作用的活性部位是一致的。药效团方法通常认为都是基于配体的药物设计方法。虽然现在解析的蛋白质三维结构也越来越多,但对于许多与药物开发有关的重要靶蛋白或受体却进展较慢。其原因主要在于这些靶标常常是存在于细胞膜的蛋白,很难培养晶体并对其进行解析。在这种情况下,只能通过“基于配体的药物设计”这种间接的方法,从研究配体的三维结构信息入手,推测配体与靶点的作用方式,并以此指导我们的药物分子设计。研究表明,相同类型药物的化学结构具有一些相同的部分,或者具有一些有规律性的部分。药效团也可以从可以作用于同一受体蛋白的不同小分子中发现规律后提取出来。具体来说,目前药效团的范围一般包括结构构象中具有相同的疏水、电荷性和立体性质上具有相似的部分。计算出可以用于识别药物基团的各种电性性质、疏水性质和体积性质描述符。药效基团通常是那些可以与受体结合位点形成氢键、静电相互作用、范德华相互作用,或疏水相互作用的原子或官能团。研究人员一般通过构建药效团模型,得到三维结构搜索的提问结构。提问结构一般可以用于后续的数据库搜索。一般是综合的应用,例如可以将两个有效药效基团合并在一个分子中,使形成的药物或兼具两者的性质,强化药理作用,减少各自相应的毒副作用,或使两者取长补短,发挥各自的药理活性,协调地完成药物作用过程。
通常,一些重要原子或极性官能团常被选为药效团元素,例如氧原子、氮原子、羧基、氨基和羟基。药物分子中的芳杂环系统能够和靶点的芳香环侧链发生强烈的π-π相互作用,因而,芳香环也常被选为药效团元素。除此之外,还有一些假原子如氢键供体、氢键受体、疏水中心,正电基团和负电基团等。药效团方法也称为基于药物小分子结构的药物设计方法。识别出属于同一活性级别所有化合物的共同结构模式,并统计其在各个活性级别化合物中出现的频率,由此确定药效团的统计参数。分子对接和从头设计通常认为是基于受体的药物设计方法。分子对接和药效团模型都可以用于药物分子的虚拟筛选。
药效团方法是药物分子设计中常用的方法之一,其利用三维数据库搜索方法从大型化合物数据库中寻找符合药效团要求的化合物,并通过药理测试和检测进一步确证该化合物的生物活性。骨架跃迁(Scaffold hopping)[43]也是一种基于药效团的数据库搜索方法,其策略是以药效团模型为起始基础,采用计算方法在已知的小分子数据库中,寻找与测试化合物完全不同的拓扑骨架,但仍然保持原有的生物分子活性。骨架跃迁一般可以从以下几个方面进行开展:①增加药物的溶解度,将亲脂性的骨架用极性骨架替换;②改变药物的分配性,调整骨架亲水/亲脂的相对程度;③提高药物的稳定性,将容易发生代谢作用的骨架用代谢稳定性的毒性低的骨架替换;④改善药代动力学性质;⑤降低分子的柔性;⑥提高对受体的亲和力,有的骨架不只是对药效团起支撑作用,而且也参与同受体的结合,改变骨架可以提高对受体的亲和力;⑦从知识产权的角度考虑,中心骨架的改变,产生了新的结构,能够获得专利保护。进行药效团和骨架跃迁分析,一般可以尝试寻找活性更好和副作用更少的候选药物分子。基于药效团的潜在药物分子筛选流程已成为发现先导化合物的有效方法,可以显著缩短先导化合物的发现周期。ZINCPharmer[44]网站(http://zincpharmer.csb.pitt.edu)提供了直接从分子结构构建和完善药效团的相关工具,同时提供搜索ZINC小分子数据库的界面。
7 从头药物设计方法从头药物分子设计(De novo drug molecular design)方法,是基于蛋白质受体结构直接设计出与蛋白质结构在形状和性质上配合的药物配体的三维结构,或者基于一个已有的结构进行理论进化和优化得到新的药物分子结构,这可以通过取代基团或侧链的变化,使得潜在药物分子活性、生物利用度提高或者毒性降低等。这种方法有时可以设计出全新的药物小分子结构,因此称为从头药物分子设计法。从头药物分子设计方法依赖于研究人员对蛋白受体靶标结构的深入理解以及对药物分子性质知识的掌握。从头药物设计方法一般有两种策略。第一种策略为根据蛋白质活性空腔的特征产生一系列的结构片段,并通过一定的顺序连接这些结构片段从而产生候选的药物小分子。因此,第一种策略一般也称为原子生长法直接药物分子设计;第二种策略是在蛋白质活性空腔内对一个基本符合要求的已知药物小分子的结构骨架进行改造和优化。骨架跃迁[43]是从头药物分子设计的一种重要的优化方法。从头药物分子设计法也称为基于受体生物大分子结构的设计方法。
从头药物分子设计方法一般是先在目标靶点用模板构建出一个形状互补的三维分子骨架,再根据受体的其他性质如静电、氢键,范德华力和疏水性质,把分子骨架转化为具体的分子结构,这个过程中就可以生成一个基础的潜在药物小分子结构。这个过程一般需要详细深入分析受体蛋白质的特性。例如,在受体的疏水区的药物小分子的片段也为疏水基团(例如苯环、脂肪链等);在氢键供体区附近提供相应氢键受体,例如羟基等;在氢键受体区附近提供相应的氢键供体,例如氨基等。之后通过片段连接的方式来构建出药物小分子的结构[45-46]。药物经过结构改造成为其衍生物时,其生物活性的改变主要与结构改变后引起的疏水性、电子效应以及空间效应的变化相关。当每一因素对生物活性具有独立的、加和性的贡献时,可通过统计学方法导出这些理化参数与生物活性的关系式。
在这个药物分子设计过程中,一般需要采用的片段结构需要具有一定的刚性,柔性不宜过强,若柔性过强最终设计出来的药物小分子的构象变化类型样本太多而不利于选择。另外,设计的药物小分子一般不能含有已知特定毒性的基团,除非是在农业中用作农药等一些特定的场景中使用。在药物分子设计中将杂环,特别是含有氮杂环加入到药物小分子的结构中是设计农药分子结构的常用策略之一,这样的设计可以同时具有两个优点:一是提高农药的效用,二是提高其选择性。
8 药物分子设计相关软件、数据库资源和经典案例表 2中列出一些常见的药物分子设计中使用到的数据库和软件资源,还有不少其他实用的数据库和软件资源由于篇幅原因没有一一列出。对于基于蛋白质受体的药物设计方法,首先的第一步是获取其三维空间结构[47]。获得蛋白质受体结构的方法一般可以通过实验方法,例如X射线晶体衍射(X-ray crystallography)、核磁共振波谱(NMR spectroscopy)和电子显微镜技术(Electron microscopy)等方法获得其三维空间结构。这些实验获得的蛋白质受体结构一般存储在PDB数据库(https://www.rcsb.org[48]中。其中,NMR方法解析出来的结构一般为一个结构的多种状态,即动态结构的一种捕捉,这些动态构象信息对药物设计非常有参考价值[38]。PDB数据库中的一些蛋白质复合物(例如GPCR复合物[49-50])是药物分子设计的重要靶标。
| 表 2 药物分子设计相关数据库资源 Table 2 Drug molecular design relative database resources |
另外,PDB数据库中还存储着部分蛋白的突变位点数据,这些数据为药物分子设计提供一定的参考信息。若通过实验方法目前还无法获得其结构,可以通过计算模拟等方法(例如学术界经典的I-TASSER[51],Rosetta[52]以及AlphaFold[53]等方法)预测出蛋白质受体的三维空间结构。目前,主流的蛋白质结构预测算法只要在能找出合适结构模板的基础上,一般都能较为准确地预测出其三维空间结构。
Yildirim等[54]通过统计发现现有的药物靶标中有不少是膜蛋白。但是,对于膜蛋白(例如钠离子通道蛋白[16])一般较难准确地建立其三维空间结构。主要原因在于:部分膜蛋白的序列长度较长,直接找到全序列高相似的模板较难。另外,膜蛋白所处的溶剂环境与球蛋白不同,所以模拟其结构的力场也较为不同。因为目前通过实验方式解析出的膜蛋白三维空间结构较少,所以通过其统计出来的力场参数也有较大的提高空间。因此,目前直接通过计算的方法准确预测膜蛋白的三维空间结构还存在一定的困难,不过这也是学术界科研人员不断努力的一个方向,也是未来较有可能在结构模拟领域得到突破的一个方向。
一般来说,通过这些主流的结构预测方法预测出来的蛋白质受体,经过评估为比较可靠的预测结果后,是可以作为后续的功能以及药物设计分析的。AlphaFold数据库(https://alphafold.com)中存储了通过其算法预测出来的一些重要的蛋白质结构,目前其存储量超过了数亿个的预测结构。DIP,BioGRID以及STRING等提供了蛋白质相互作用的数据库以及分析算法等。另外,通过PubChem,ChEMBL和DrugBank等数据库可以获得与药物分子设计相关的小分子以及其他相关数据。这些数据库以及网络其他的各种资源为药物分子的设计提供了良好的数据和软件支持。
表 3中列出了几种PDB数据库存储的蛋白质和其抑制剂的信息。这些抑制剂是目前市面上可以公开获得到的药物。
| 表 3 一些蛋白质复合物和其底物 Table 3 Several protein complexes and ligands |
例如,在4L1A这个蛋白质中,Lopinavir是其抑制剂[55]。Lopinavir现在主要作为一种有效的HIV蛋白酶抑制剂,适用于与其他抗反转录病毒药物联合用药。在2PX6这个蛋白质中,奥利司他(Orlistat)是一种脂肪酶抑制剂,可抑制胃肠道中胰脂酶的活性,并使脂肪吸收率减少[56]。由于该药抑制肠道脂酶活性,使脂肪不能吸收而直接排出,因而容易引起油样便与腹泻,并可造成脂溶性维生素缺乏,这使其应用效果受到一定限制。但总体说来,其全身性副作用仍明显低于一般其他的减肥药。在3WSJ复合物中,Indinavir是一种蛋白酶抑制药[57],具有抗HIV-1和HIV-2蛋白酶作用,但对HIV-1的选择性偏高。Indinavir可与蛋白酶的活性部位可逆性结合,发挥竞争性抑制效应,从而阻止病毒前体多聚蛋白质的分裂并干扰新的病毒颗粒的成熟。
药物研发的一般流程包括靶标选定,先导化合物发现及其优化,临床试验,以及上市药物持续调研等步骤,其中每一个步骤的详细内容如图 4所示。药物分子设计一般来说需要综合考虑图 4中的所有步骤,其中靶标的选定、先导化合物发现和优化常用的方法包括分子对接、从头设计、以及分子动力学模拟等。在实际的药物分子设计过程中,不同的方法之间一般会交叉综合使用,所以不同方法之间的没有明显的界限。这里介绍几种学术界公开报道的潜在药物分子设计实例,其中阿格列汀(Alogliptin)已经在市面上公开销售。
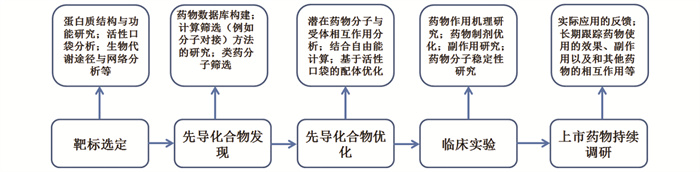
|
图 4 基于蛋白质受体的药物从设计到应用一般流程 Figure 4 A general flowchart of protein receptor-based drug development from design to application |
这是一个基于同源建模和分子对接进行潜在药物分子筛选的实例。γ-氨基丁酸氨基转移酶(GABA-AT)是人体内一种重要药物靶点蛋白质。在学术界中,设计和研发抑制GABA-AT酶的新型有效药物,直到目前仍然是一项非常重要的工作。Alobaidi等[58]采用计算机辅助药物设计工具发现一种新型可能有效的GABA-AT抑制剂。其具体研究流程如下:根据序列相似性进行同源建模,采用猪肝酶的晶体结构为模板构建了人类GABA-AT的三维空间结构。在预测模型的基础上,使用Autodock Vina等分子对接工具判断抑制剂和GABA-AT之间的结合模式和抑制效果,同时通过分子动力学模拟的方法检查不同配体和GABA-AT复合物随时间变化的稳定性。上述工作中筛选出几个很可能在实际应用中有效的药物分子。作者还进一步在靶点结构的基础上,对已知活性小分子物质进行修饰尝试来提高其活性,筛选出可作为γ-氨基丁酸氨基转移酶潜在抑制剂的小分子。
一种特效二型糖尿病治疗药物DPP-IV抑制剂阿格列汀(Alogliptin)的开发是一个从头设计的药物分子研发案例。在这个案例中,在蛋白质活性空腔内对一个符合要求的已知药物小分子的结构骨架进行改造。由于全世界糖尿病患者众多,因此开发有效的降糖药物的开发成为各个制药公司的研究目标之一。2006年,雅培的科学家发布了一个抑制剂与DPP-IV酶复合物的晶体结构,两者间通过共价结合[59]。这个复合物的结构进一步启发了相关药物研发的科学家,并进行类似非共价键结合抑制剂的尝试。该课题组科学家又解析出了一个相关结构2ONC和其抑制剂SY1的复合物结构[59],并基于SY1作为初始的先导物构象(图 5中为红色小分子),不断优化相应官能团最终得到一个有效抑制剂药物分子阿洛格列汀(图 5中为桔色小分子)。后续实验进一步表明:阿洛格列汀是一种有效、选择性的丝氨酸蛋白酶二肽基肽酶IV(DPP-IV)抑制剂。口服一定剂量阿洛格列汀后,这些非共价抑制剂可在糖尿病动物模型中持续降低血浆DPP-IV的活性,从而降低血糖。根据表 4中底物和受体的相互作用能量可推导出结果,Alogliptin相比SY1与目标蛋白的相互作用的亲和力更好,进而产生更强的抑制作用。
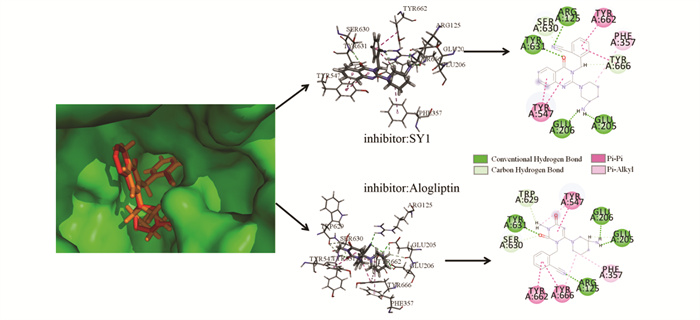
|
图 5 从头药物分子设计的药物实例 Figure 5 A de novo-based drug molecular design example |
| 表 4 受体蛋白与底物相互作用的能量分解 Table 4 Energy decomposition of interactions between protein receptor and ligands |
阿洛格列汀已于2010年批准上市,并已在药物市场得到广泛使用。图 5中的蛋白质结构和Alogliptin以及SY1相互作用图是使用Pymol以及我们实验室购置的正版Discovery Studio 2019等软件(福州大学仪器平台)绘制的。
这是一个结合分子对接和分子动力学并综合应用进行潜在药物分子筛选的例子。Rajasekhar等[40]采用基于结构的药物设计评估了一系列噻唑烷衍生物的有效抑制作用。噻唑烷是一种五元环结构,在相应位置上有硫醚和氨基。噻唑烷可与多种蛋白质靶点结合,而且是药物化学中的一个主要杂环核心。在这个工作中Rajasekhar等[40]采用的分子模拟工具包括AutoDock Vina,Glide和MM/GBSA,来分析与筛查潜在的抑制分子。通过分子动力学模拟结核分枝杆菌蛋白PrpR与三种新型支架之间的动态相互作用,验证和评估了复合物结合的亲和力。所有潜在抑制分子都分别与Arg169,Arg197,Tyr248,Arg308和Gly311等重要氨基酸残基之间存在明显和稳定的相互作用。此外,作者还通过基于机器学习的算法预测和评估了分子骨架的抑制活性,并对上述化合物进行抑制性作用力排序。这项研究揭示了治疗结核病领域中,抑制剂分子作为有效候选药物相应的潜力。
这里再回顾和追溯一个上一世纪就已经开展的潜在药物分子筛选案例:基于分子对接的药物分子设计方法。DesJarlais等[60]筛选出了非肽类HIV-1蛋白酶(HIV-PR)的抑制剂。DesJarlais等根据HIV-PR的X射线解析出实验晶体学的三维结构,采用DOCK分子对接算法搜索剑桥小分子结构数据库数据库(Cambridge structure database),并用程序产生一个覆盖结合位点的分子表面和活性空腔,通过蛋白质的表面形式可以较为容易找到合适的活性空腔,然后建立受体点的结构,产生近似圆柱体的空腔,之后用中心匹配算法来确定每个小分子能否置于其中,通过这种方式DesJarlais等筛选出了可以抑制非肽类HIV-1蛋白酶(HIV-PR)的潜在药物分子。
9 总结和展望早在上个世纪,基于蛋白质受体结构的药物分子计算机辅助设计就已经持续开展开来了。直至近几年,其仍然为学术和工业界研究的热点之一。随着近些年来越来越多高精度蛋白质复合物晶体结构的获得以及多种有效机器学习、统计预测以及深度学习新方法的提出和进展,基于结构的药物分子设计方法变得更加精准。药物分子设计过程中一般需要综合地考虑受体蛋白质和药物小分子在结构上的柔性和相互作用等关系,同时还需要考虑药物分子的作用机制和药物分子在体内转运等问题。在以蛋白质受体为基础的药物分子设计中常用的计算辅助方法包括同源建模、分子对接、药效团分析、分子动力学以及量子力学和分子力学结合的综合模拟方法等。另外,设计出来的药物分子经过实验和实际验证之后,还需要考虑稳定性以及是否易于生产等问题。
如何提高药物分子设计方法的准确性和效率等问题是一个药物是否可以研制成功的关键因素之一。基于蛋白质受体的药物分子设计方法有多种,不同的方法一般适用于不同情况。虽然基于蛋白质受体药物分子设计方法持续发展,但也存在的不少机遇和潜在挑战。在过去的几十年中,许多包含数以百万计分子数据的公共数据库的出现,例如ChEMBL和PubChem,这些为深度学习模型提供输入训练数据,从而可以更加准确地预测药物的各种生物学活性或理化性质。但是,合理药物设计只是开发新药的重要环节之一,还需要化学合成和药理、毒理测试等的密切配合。理想的开发新药之路应是“理论计算—药物设计—化学合成—药理测试”,这几个环节缺一不可,一般还要经过多次循环过程,才能达到开发新药的目的。药物的研发还需要考虑药物在体内的转运和产生效应等问题,这些对现有算法也提出了一定的挑战。伴随着计算机硬件、深度学习算法、药物相关实验数据库的持续发展和优化,这些因素可能会在未来持续促进药物设计更加合理化。本文论述的内容是仅仅在计算机上进行潜在药物分子设计的过程。实际药物分子在生物体内的基本过程是给药、吸收、转运、分布并到达作用部位,从而产生药理作用。这些综合的因素都导致了快速进行一个成功药物分子设计是一个十分有挑战性的工作。
另外,在计算机和理论上研究和分析中药也是近年来的研究趋势之一。中药复方因其多成分、多靶点的特点,近年来在国际医学界及药物发现领域获得了较高的关注度。随着各种技术的快速发展,采用计算机模拟方法研究中药也许成为中药现代化的重要机遇和方法之一。
| [1] |
ALKAFF A H, SARAGIH M, IMANA S N, et al. Identification of DNA methyltransferase-1 inhibitor for breast cancer therapy through computational fragment-based drug design[J]. Molecules, 2021, 26(2): 375. DOI:10.3390/molecules26020375 (  0) 0) |
| [2] |
BRANDT G S, NOVAK W R P. SARS-CoV-2 virtual biochemistry labs on bioinformatics and drug design[J]. Biochemistry and Molecular Biology Education, 2021, 49(1): 26-28. DOI:10.1002/bmb.21480 (  0) 0) |
| [3] |
PATRICIO R P S, VIDEIRA P A, PEREIRA F. A computer-aided drug design approach to discover tumour suppressor p53 protein activators for colorectal cancer therapy[J]. Bioorganic & Medicinal Chemistry, 2022, 53: 116530. DOI:10.1016/j.bmc.2021.116530 (  0) 0) |
| [4] |
CHEN Xuehui, YAN Huiwen, LIU Dan, et al. Structure basis for AA98 inhibition on the activation of endothelial cells mediated by CD146[J]. iScience, 2021, 24(5): 102417. DOI:10.1016/j.isci.2021.102417 (  0) 0) |
| [5] |
LU Tong, SUN Xiaojing, NECELA B M, et al. TRPC6 N338S is a gain-of-function mutant identified in patient with doxorubicin-induced cardiotoxicity[J]. Biochimica et Biophysica Acta (BBA)-Molecular Basis of Disease, 2022, 1868(11): 166505. DOI:10.1016/j.bbadis.2022.166505 (  0) 0) |
| [6] |
BORN J, MANICA M. Trends in deep learning for property-driven drug design[J]. Current Medicinal Chemistry, 2021, 28(38): 7862-7886. DOI:10.2174/0929867328666210729115728 (  0) 0) |
| [7] |
YANG Yuyao, ZHENG Shuangjia, SU Shimin, et al. SyntaLinker: Automatic fragment linking with deep conditional transformer neural networks[J]. Chemical Science, 2020, 11(31): 8312-8322. DOI:10.1039/d0sc03126g (  0) 0) |
| [8] |
XU Youjun, LIN Kangjie, WANG Shiwei, et al. Deep learning for molecular generation[J]. Future Medicinal Chemistry, 2019, 11(6): 567-597. DOI:10.4155/fmc-2018-0358 (  0) 0) |
| [9] |
REZAEI M A, LI Yanjun, WU Dapeng, et al. Deep learning in drug design: Protein-ligand binding affinity prediction[J]. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2022, 19(1): 407-417. DOI:10.1109/TCBB.2020.3046945 (  0) 0) |
| [10] |
SIVAKUMAR D, WU S. Classical and machine learning methods for protein- ligand binding free energy estimation[J]. Current Drug Metabolism, 2022, 23(4): 252-259. DOI:10.2174/1389200223666220315160835 (  0) 0) |
| [11] |
GUPTA R, SRIVASTAVA D, SAHU M, et al. Artificial intelligence to deep learning: machine intelligence approach for drug discovery[J]. Molecular Diversity, 2021, 25(3): 1315-1360. DOI:10.1007/s11030-021-10217-3 (  0) 0) |
| [12] |
PODLEWSKA S, CZARNECKI W M, KAFEL R, et al. Creating the new from the old: Combinatorial libraries generation with machine-learning-based compound structure optimization[J]. Journal of Chemical Information and Modeling, 2017, 57(2): 133-147. DOI:10.1021/acs.jcim.6b00426 (  0) 0) |
| [13] |
PALERMO G, SPINELLO A, SAHA A, et al. Frontiers of metal-coordinating drug design[J]. Expert Opinion on Drug Discovery, 2021, 16(5): 497-511. DOI:10.1080/17460441.2021.1851188 (  0) 0) |
| [14] |
CAO Longxing, GORESHNIK I, COVENTRY B, et al. De novo design of picomolar SARS-CoV-2 miniprotein inhibitors[J]. Science, 2020, 370(6515): 426-431. DOI:10.1126/science.abd9909 (  0) 0) |
| [15] |
SELASSIE C D, MEKAPATI S B, VERMA R P. QSAR: Then and now[J]. Current Topics in Medicinal Chemistry, 2002, 2(12): 1357-1379. DOI:10.2174/1568026023392823 (  0) 0) |
| [16] |
DU Yuzhe, NOMURA Y, SATAR G, et al. Molecular evidence for dual pyrethroid-receptor sites on a mosquito sodium channel[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(29): 11785-11790. DOI:10.1073/pnas.1305118110 (  0) 0) |
| [17] |
BAGCHI A. Latest trends in structure based drug design with protein targets[J]. Advances in Protein Chemistry and Structural Biology, 2020, 121: 1-23. DOI:10.1016/bs.apcsb.2019.11.008 (  0) 0) |
| [18] |
GAPSYS V, HAHN D F, TRESADERN G, et al. Pre-exascale computing of protein-ligand binding free energies with open source software for drug design[J]. Journal of Chemical Information and Modeling, 2022, 62(5): 1172-1177. DOI:10.1021/acs.jcim.1c01445 (  0) 0) |
| [19] |
O'REILLY A O, KHAMBAY B P, WILLIAMSON M S, et al. Modelling insecticide-binding sites in the voltage-gated sodium channel[J]. Biochemical Journal, 2006, 396(2): 255-263. DOI:10.1042/BJ20051925 (  0) 0) |
| [20] |
MOUNT D W. Using the basic local alignment search tool (BLAST)[J]. Cold Spring Harbor Protocols, 2007, 6(2): 55-63. DOI:10.1101/pdb.top17 (  0) 0) |
| [21] |
WU Sitao, ZHANG Yang. MUSTER: Improving protein sequence profile-profile alignments by using multiple sources of structure information[J]. Proteins, 2008, 72(2): 547-556. DOI:10.1002/prot.21945 (  0) 0) |
| [22] |
XU Dong, JAROSZEWSKI L, LI Zhanwen, et al. FFAS-3D: Improving fold recognition by including optimized structural features and template re-ranking[J]. Bioinformatics, 2014, 30(5): 660-667. DOI:10.1093/bioinformatics/btt578 (  0) 0) |
| [23] |
DEVI R V, SATHYA S S, COUMAR M S. Multi-objective genetic algorithm for de novo drug design (MoGADdrug)[J]. Current Computer-Aided Drug Design, 2021, 17(3): 445-457. DOI:10.2174/1573409916666200620194143 (  0) 0) |
| [24] |
ARUS-POUS J, PATRONOV A, BJERRUM E J, et al. SMILES-based deep generative scaffold decorator for de-novo drug design[J]. Journal of Cheminformatics, 2020, 12(1): 38. DOI:10.1186/s13321-020-00441-8 (  0) 0) |
| [25] |
JOHNSON J A, CAVALLARI L H. Cardiovascular pharmacogenomics[J]. Experimental Physiology, 2005, 90(3): 283-289. DOI:10.1113/expphysiol.2004.028506 (  0) 0) |
| [26] |
WANG Mingyang, WANG Zhe, SUN Huiyong, et al. Deep learning approaches for de novo drug design: An overview[J]. Current Opinion in Structural Biology, 2022, 72: 135-144. DOI:10.1016/j.sbi.2021.10.001 (  0) 0) |
| [27] |
PALAZZESI F, POZZAN A. Deep learning applied to ligand-based de novo drug design[J]. Methods in Molecular Biology, 2022, 2390: 273-299. DOI:10.1007/978-1-0716-1787-8_12 (  0) 0) |
| [28] |
CASE D A, CHEATHAM T E, DARDEN T, et al. The Amber biomolecular simulation programs[J]. Journal of Computational Chemistry, 2005, 26(16): 1668-1688. DOI:10.1002/jcc.20290 (  0) 0) |
| [29] |
MIKHAILOVSKⅡ O, XUE Y, SKRYNNIKOV N R. Modeling a unit cell: Crystallographic refinement procedure using the biomolecular MD simulation platform Amber[J]. International Union of Crystallography, 2022, 9(1): 114-133. DOI:10.1107/S2052252521011891 (  0) 0) |
| [30] |
COLE D J, HORTON J T, NELSON L, et al. The future of force fields in computer-aided drug design[J]. Future Medicinal Chemistry, 2019, 11(18): 2359-2363. DOI:10.4155/fmc-2019-0196 (  0) 0) |
| [31] |
CAPORALE A, ADORINNI S, LAMBA D, et al. Peptide-protein interactions: From drug design to supramolecular biomaterials[J]. Molecules, 2021, 26(5): 1219. DOI:10.3390/molecules26051219 (  0) 0) |
| [32] |
MASUDA Y. Bioactive 3D structures of naturally occurring peptides and their application in drug design[J]. Bioscience, Biotechnology, and Biochemistry, 2021, 85(1): 24-32. DOI:10.1093/bbb/zbaa008 (  0) 0) |
| [33] |
KHAN F I, LAN D, DURRANI R, et al. The lid domain in lipases: structural and functional determinant of enzymatic properties[J]. Frontiers in Bioengineering and Biotechnology, 2017, 5: 16. DOI:10.3389/fbioe.2017.00016 (  0) 0) |
| [34] |
LEI Jian, ZHOU Yanfeng, LI Lanfen, et al. Structural and biochemical analyses of YvgN and YtbE from bacillus subtilis[J]. Protein Science, 2009, 18(8): 1792-1800. DOI:10.1002/pro.178 (  0) 0) |
| [35] |
KIM S, CHEN Jie, CHENG Tiejun, et al. PubChem in 2021: New data content and improved web interfaces[J]. Nucleic Acids Research, 2021, 49(D1): D1388-D1395. DOI:10.1093/nar/gkaa971 (  0) 0) |
| [36] |
PHAM T N H, NGUYEN T H, TAM N M, et al. Improving ligand-ranking of AutoDock Vina by changing the empirical parameters[J]. Journal of Computational Chemistry, 2022, 43(3): 160-169. DOI:10.1002/jcc.26779 (  0) 0) |
| [37] |
HANWARINROJ C, THONGDEE P, SUKCHIT D, et al. In silico design of novel quinazoline-based compounds as potential Mycobacterium tuberculosis PknB inhibitors through 2D and 3D-QSAR, molecular dynamics simulations combined with pharmacokinetic predictions[J]. Journal of Molecular Graphics and Modelling, 2022, 115: 108231. DOI:10.1016/j.jmgm.2022.108231 (  0) 0) |
| [38] |
MANGLIK A, KIM T H, MASUREEL M, et al. Structural insights into the dynamic process of beta2-Adrenergic receptor signaling[J]. Cell, 2015, 161(5): 1101-1111. DOI:10.1016/j.cell.2015.04.043 (  0) 0) |
| [39] |
ALZAIN A A, ELBADWI F A, ALSAMANI F O. Discovery of novel TMPRSS2 inhibitors for COVID-19 using in silico fragment-based drug design, molecular docking, molecular dynamics, and quantum mechanics studies[J]. Informatics in Medicine Unlocked, 2022, 29: 100870. DOI:10.1016/j.imu.2022.100870 (  0) 0) |
| [40] |
RAJASEKHAR S, DAS S, KARUPPASAMY R, et al. Identification of novel inhibitors for Prp protein of Mycobacterium tuberculosis by structure based drug design, and molecular dynamics simulations[J]. Journal of Computational Chemistry, 2022, 43(9): 619-630. DOI:10.1002/jcc.26823 (  0) 0) |
| [41] |
DO P C, LEE E H, LE L. Steered molecular dynamics simulation in rational drug design[J]. Journal of Chemical Information and Modeling, 2018, 58(8): 1473-1482. DOI:10.1021/acs.jcim.8b00261 (  0) 0) |
| [42] |
BORBULEVYCH O Y, MARTIN R I, WESTERHOFF L M. The critical role of QM/MM X-ray refinement and accurate tautomer/protomer determination in structure-based drug design[J]. Journal of Computer-Aided Molecular Design, 2021, 35(4): 433-451. DOI:10.1007/s10822-020-00354-6 (  0) 0) |
| [43] |
SUN Hongmao, COUSSENS N P, DANCHIK C, et al. Discovery of small-molecule VapC1 nuclease inhibitors by virtual screening and scaffold hopping from an atomic structure revealing protein-protein interactions with a native VapB1 inhibitor[J]. Journal of Chemical Information and Modeling, 2022, 62(5): 1249-1258. DOI:10.1021/acs.jcim.1c01188 (  0) 0) |
| [44] |
KOES D R, CAMACHO C J. ZINCPharmer: Pharmacophore search of the ZINC database[J]. Nucleic Acids Research, 2012, 40(Web Server issue): W409-W414. DOI:10.1093/nar/gks378 (  0) 0) |
| [45] |
BANCET A, RAINGEVAL C, LOMBERGET T, et al. Fragment linking strategies for structure-based drug design[J]. Journal of Medicinal Chemistry, 2020, 63(20): 11420-11435. DOI:10.1021/acs.jmedchem.0c00242 (  0) 0) |
| [46] |
ARYA H, COUMAR M S. Design of novel ROCK inhibitors using fragment-based de novo drug design approach[J]. Journal of Molecular Modeling, 2020, 26(9): 249. DOI:10.1007/s00894-020-04493-3 (  0) 0) |
| [47] |
HENIKOFF S, PIETROKOVSKI S, HENIKOFF J G. Superior performance in protein homology detection with the Blocks Database servers[J]. Nucleic Acids Research, 1998, 26(1): 309-312. DOI:10.1093/nar/26.1.309 (  0) 0) |
| [48] |
VELANKAR S, BURLEY S K, KURISU G, et al. The protein data bank archive[J]. Methods in Molecular Biology, 2021, 2305: 3-21. DOI:10.1007/978-1-0716-1406-8_1 (  0) 0) |
| [49] |
SANTOS R, URSU O, GAULTON A, et al. A comprehensive map of molecular drug targets[J]. Nature Reviews Drug Discovery, 2017, 16(1): 19-34. DOI:10.1038/nrd.2016.230 (  0) 0) |
| [50] |
HAUSER A S, ATTWOOD M M, RASK-ANDERSEN M, et al. Trends in GPCR drug discovery: new agents, targets and indications[J]. Nature Reviews Drug Discovery, 2017, 16(12): 829-842. DOI:10.1038/nrd.2017.178 (  0) 0) |
| [51] |
ZHENG Wei, ZHANG Chengxin, BELL E W, et al. I-TASSER gateway: A protein structure and function prediction server powered by XSEDE[J]. Future Generation Computer Systems, 2019, 99: 73-85. DOI:10.1016/j.future.2019.04.011 (  0) 0) |
| [52] |
ANISHCHENKO I, BAEK M, PARK H, et al. Protein tertiary structure prediction and refinement using deep learning and Rosetta in CASP14[J]. Proteins, 2021, 89(12): 1722-1733. DOI:10.1002/prot.26194 (  0) 0) |
| [53] |
JUMPER J, HASSABIS D. Protein structure predictions to atomic accuracy with AlphaFold[J]. Nature Methods, 2022, 19(1): 11-12. DOI:10.1038/s41592-021-01362-6 (  0) 0) |
| [54] |
YILDIRIM M A, GOH K I, CUSICK M E, et al. Drug-target network[J]. Nature Biotechnology, 2007, 25(10): 1119-1126. DOI:10.1038/nbt1338 (  0) 0) |
| [55] |
LIU Zhigang, YEDIDI R S, WANG Yong, et al. Crystallographic study of multi-drug resistant HIV-1 protease lopinavir complex: mechanism of drug recognition and resistance[J]. Biochemical and Biophysical Research Communications, 2013, 437(2): 199-204. DOI:10.1016/j.bbrc.2013.06.027 (  0) 0) |
| [56] |
PEMBLE C W T, JOHNSON L C, KRIDEL S J, et al. Crystal structure of the thioesterase domain of human fatty acid synthase inhibited by Orlistat[J]. Nature Structural & Molecular Biology, 2007, 14(8): 704-709. DOI:10.1038/nsmb1265 (  0) 0) |
| [57] |
KUHNERT M, STEUBER H, DIEDERICH W E. Structural basis for HTLV-1 protease inhibition by the HIV-1 protease inhibitor indinavir[J]. Journal of Medicinal Chemistry, 2014, 57(14): 6266-6272. DOI:10.1021/jm500402c (  0) 0) |
| [58] |
AL-OBAIDI A, ELMEZAYEN A D, YELEKCI K. Homology modeling of human GABA-AT and devise some novel and potent inhibitors via computer-aided drug design techniques[J]. Journal of Biomolecular Structure and Dynamics, 2021, 39(11): 4100-4110. DOI:10.1080/07391102.2020.1774417 (  0) 0) |
| [59] |
FENG Jun, ZHANG Zhiyuan, WALLACE M B, et al. Discovery of alogliptin: a potent, selective, bioavailable, and efficacious inhibitor of dipeptidyl peptidase IV[J]. Journal of Medicinal Chemistry, 2007, 50(10): 2297-2300. DOI:10.1021/jm070104l (  0) 0) |
| [60] |
DESJARLAIS R L, DIXON J S. A shape- and chemistry-based docking method and its use in the design of HIV-1 protease inhibitors[J]. Journal of Computer-Aided Molecular Design, 1994, 8(3): 231-242. DOI:10.1007/BF00126742 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22