中心法则由Crick于1958年提出,叙述了遗传信息的传递过程,是所有的细胞结构生物均需遵循的法则。遗传信息从DNA传递到RNA为转录过程,从RNA传递到蛋白质为翻译过程。转录过程是以双链DNA中的一条链作为模板,在RNA聚合酶的催化作用下合成RNA[1]。转录是基因表达的调节控制和生物遗传信息传递中的一个重要环节,而且过程比较复杂,通常需要转录因子的协同帮助才可以完成。转录因子在分子生物学中具体是指一种能够结合在基因上游的特定位置,起正调节或负调节的蛋白质因子。正调节可以激活转录过程,提高转录效率,促进基因表达,在调节过程中起到正向推动作用;反之则为负调节,阻碍转录过程,降低转录效率,抑制基因表达,在调节过程中起到反向抑制作用,甚至会直接抑制转录过程的启动。在转录的起始过程,转录因子便可与RNA聚合酶形成一种复合体蛋白质,两者共同在转录的起始过程起作用。根据转录因子的作用特点不同,可以将其分为普遍转录因子和组织细胞特异性转录因子[2]。
1983年,人类在非洲爪蟾卵母细胞的转录因子中第一次发现锌指蛋白,作为转录因子其中的一类,经常会在DNA结合蛋白中出现,能够对特定的碱基序列起识别作用。锌指蛋白是真核生物基因组中最广泛分布的蛋白质之一,在人类基因组里有大约1%的序列中含有锌指蛋白。锌指蛋白的具体结构是由氨基酸环和锌离子组成,形状类似于人类的手指,故称这样的结构为锌指结构。锌指蛋白是一类含有锌指结构且必须与锌离子结合配位才能发挥作用的蛋白质[3-5]。
锌指蛋白的具体空间结构,是由半胱氨酸(Cys)残基和组氨酸(His)残基两者根据不同的数量和方式围绕锌离子所构成。根据具体空间结构的不同,Krishna等[6]把锌指蛋白分为C2H2 like, Gag knuckle, Treble clef, Zinc ribbon, Zn2/Cys6, TAZ2 domain like,Short zinc binding loops和Metallothionein,共计8种不同的折叠群(Fold group)。每一种折叠群都包含很多类型,但大部分类型的锌指蛋白都属于前三类折叠群[7]。具体来说,存在范围最广且生物功能最为重要的锌指蛋白有如下几种类型: C2H2型锌指蛋白、RING型锌指蛋白、PHD型锌指蛋白以及LIM型锌指蛋白。C2H2型锌指蛋白通常是指包含[C-x-C-x-H-x-H]结构域的锌指蛋白,作用机理是与DNA结合为结合蛋白,进而起到促进或抑制标靶基因表达的作用。C2H2型锌指蛋白约占目前已知全部锌指蛋白的45%,是锌指蛋白中数量最多的一类[8]。RING型锌指蛋白通常是指包含[C-x-C-x-C-x-H-x-C-x-C-x-C-x-C]结构域的锌指蛋白,可以对转录过程中锌指蛋白的作用对象和作用活性产生影响[9]。PHD型锌指蛋白通常是指包含[C-x-C-x-C-x-C-x-H-x-C-x-C-x-C]结构域的锌指蛋白,能够对染色质起到重塑作用,修饰表观遗传,调控识别核小体。LIM型锌指蛋白通常是指包含[C-x-C-x-H-x-C-x-C-x-C-x-C-x-(C.H, D)]结构域的锌指蛋白,能够在肌动蛋白锚定过程中起到重要作用,也可对细胞骨架之间的相互作用产生影响。除上所述之外,锌指蛋白在基因翻译、mRNA运输、细胞骨架组装、上皮细胞发育、细胞粘附、蛋白质折叠以及锌离子感应等方面均发挥重要的作用。
本文基于最新版本UniProt数据库建立了锌指蛋白家族的数据集,包含目前已知且通过实验验证的所有类型。最终选取C2H2型锌指蛋白和数量相近的非锌指蛋白共同构成预测数据集。通过输入单个特征信息和融合特征信息进行预测实验,最终结果是氨基酸二肽组分信息的预测成功率最高,达到87.86%。在此基础上,对氨基酸二肽组分信息使用多种方式的降维,降维处理后利用支持向量机算法进行预测,最终得到最高预测成功率为90.21%。预测结果表明: 特征信息的降维处理对预测结果有较好的提升。在此基础上进行融合三类特征信息,利用支持向量机算法进行预测,融合特征信息最高预测成功率为92.55%,预测结果表明: 特征信息的融合对预测结果有一定的提升作用。
1 材料与方法 1.1 数据集的构建利用机器学习方法对蛋白质进行预测是如今生物信息学研究的重点方法,建立一个客观的、有代表性的数据集对于后续工作至关重要。UniProt(https://legacy.uniprot.org/)是一个信息丰富、资源广泛的蛋白质数据库,由Swiss-Prot, TrEMBL和PIR-PSD三大数据库整合而成。本文基于UniProt数据库,严格按照以下标准构建了锌指蛋白数据集:
1) 在UniProt数据库,高级检索中输入关键词“zinc”及“finger”所得蛋白质序列,包含经过实验验证的序列2 597条,未经过实验验证的序列469 483条。
2) 在高级检索选项“advanced”里添加限制条件“AND Reviewed”,选择经过实验验证的蛋白质序列,共得到序列2 597条。
3) 去除“By similarity”, “Probably”等含糊不确定的关键字后,共得到序列2 597条。
4) 在“Sequence ”信息中选择蛋白质序列完整,去除片段序列后,共得到序列2 479条。
5) 去除含有不确定氨基酸Z, X, B, O, J, U的蛋白质序列后,共得到序列2 227条。
6) 根据蛋白质序列名称代码删除重复的蛋白质序列后,共得到序列2 183条。
由以上步骤最终得到各类锌指蛋白序列共2 183条,由于种类数量分布不平衡,C2H2型锌指蛋白序列共有1 601条,占到了总数的73.34%,其它类型只占有极少数,故将数据集中的C2H2型锌指蛋白序列筛选出来。为避免存在同源性误差,采用CD-HIT程序对数据集进行相似比对,序列相似性阈值设定为25%。完成上述步骤后,共得到363条C2H2型锌指蛋白质序列。在UniProt数据库,高级检索中输入不含关键词“zinc”及“finger”的蛋白质序列,从中随机挑选679条非锌指蛋白序列。阈值设定为25%,经过CD-HIT软件对数据集进行相似比对,最终获得362条非锌指蛋白序列。最终预测数据集由363条锌指蛋白序列和362条非锌指蛋白序列,共计725条蛋白质序列构成,详见表 1。
| 表 1 C2H2型锌指蛋白及非锌指蛋白数据集中序列数目 Table 1 Number of sequences in the C2H2 zinc finger protein and non-zinc finger protein dataset |
人体内有很多种类的蛋白质,它们性质有所不同,功能也各有差异,但组成成分都是20种氨基酸,分别为:A, C, D, E,F, G, H, I, K, L, M, N, P, Q, R, S, T, V, W和Y。本文选取氨基酸单肽组分信息(Amino acid composition, AAC)作为特征参数之一,氨基酸单肽组分信息是蛋白质研究中最普遍应用的一类特征信息,具体是计算蛋白质序列中20种氨基酸出现的频率,也可以用20维特征向量表示[10]。定义为
| $ P=\left[x_1, x_2, x_3, \cdots, x_i, \cdots, x_{20}\right] $ | (1) |
| $ x_i=\frac{m_i}{L} $ | (2) |
上述公式中:L指的是蛋白质序列长度,mi指的是蛋白质序列中第i个氨基酸出现的次数。
1.2.2 氨基酸二肽组分信息由蛋白质序列的组成可知,氨基酸对共有20×20等于400种组合。本文选取氨基酸二肽组分信息(Dipeptide composition,DC)作为特征参数之一,具体是计算两个相邻氨基酸残基的出现频率,也可以用400维特征向量表示[11]。氨基酸二肽组分信息相较于氨基酸单肽组分信息的优点是考虑了蛋白质序列中的氨基酸组合顺序。具体定义为
| $ P=\left[f_1, f_2, f_3, \cdots, f_i, \cdots, f_{400}\right] $ | (3) |
| $ f_i=\frac{n_i}{L-1} $ | (4) |
上述公式中:L指的是蛋白质序列长度,ni指的是蛋白质序列中第i个二肽出现的次数。
1.2.3 平均化学位移在对蛋白质的研究中,核磁共振技术发挥着重要作用,它可以对蛋白质在多个时间尺度上内部运动的相关信息进行跟踪记录。由于质子存在化学环境敏感性,所以在核磁共振技术手段中,质子会因为处于不同的化学环境而导致受到不同的磁场作用并产生不同的吸收频率。平均化学位移(Auto-covariance average chemical shift, acACS)即可以用各类不同质子相对于标准值的共振频率表示[12]。具体研究表明,蛋白质的平均化学位移与其二级结构有很大的相关性[13]。本文通过将蛋白质序列提交到PSIPRED(PSIPRED workbench(ucl.ac.uk))网站获得数据集中蛋白质的二级结构,然后利用python程序将化学位移的结果提取出来。具体过程可表示为
| $ A C S_i^k(j)=\frac{1}{N} \sum w_i^k(j) $ | (5) |
上述公式中:i指的是四种骨架原子
对于蛋白质P,序列中的每个氨基酸都被其平均化学位移取代,可以表示为
| $ P=\left[A_1^i, A_2^i, A_3^i, \cdots, A_L^i\right]\left(i={ }^{15} N, { }^{13} C_\alpha, { }^1 H_\alpha, { }^1 H_N\right) $ | (6) |
该化学位移的自相关协方差可表示为
| $ \begin{aligned} & \varphi_i^\lambda=\frac{1}{L-\lambda} \sum\nolimits_{k=1}^{L-\lambda}\left[A_k^i-A_{k+\lambda}^i\right]^2 \\ & \left(i={ }^{15} N, { }^{13} C_\alpha, { }^1 H_\alpha, { }^1 H_N ; \lambda<L\right) \end{aligned} $ | (7) |
最终PacACS可表示为
| $\begin{aligned} P_{a c A C S}= & {\left[\varphi_i^0, \varphi_i^1, \varphi_i^2 \cdots \varphi_i^\lambda\right] } \\ & \left(i={ }^{15} N, { }^{13} C_\alpha, { }^1 H_\alpha, { }^1 H_N\right) \end{aligned} $ | (8) |
上述公式中:λ指的是相关常数,L指的是蛋白质序列的总长度。对于不同的蛋白质,为了预测可以取得更好的结果,应选择最优组合的骨架原子组合及λ值。
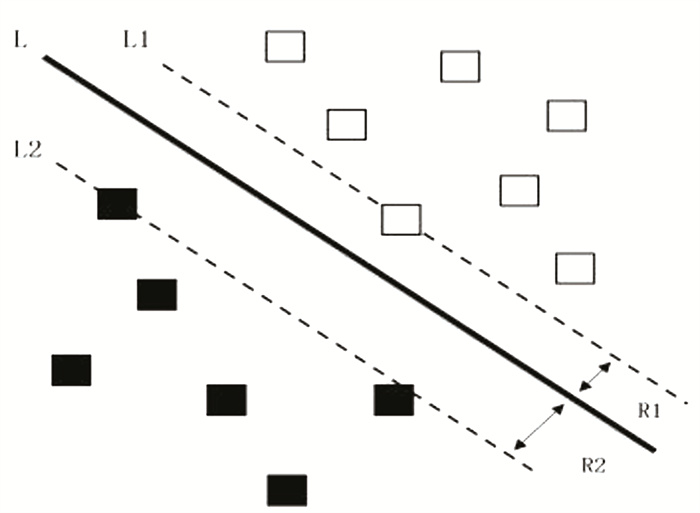
1.3 预测算法选用支持向量机(Support vector machine, SVM)作为预测算法,1955年Vapnik等[14]最早提出支持向量机算法,它是一种基于统计学理论的机器学习方法,后来也广泛用于蛋白质结构预测和功能预测。尽可能的利用最大间隔思想去降低分类器的置信风险,这是支持向量机的核心思想。将数据从低维向量映射到高维向量,使得结构风险达到最小化,使正集和负集之间的距离最大化。具体原理见图 1。
|
图 1 支持向量机原理图 Figure 1 Schematic diagram of SVM |
图 1中的黑色实心矩形和空心矩形各自代表着一类样本,黑色实线代表两类样本的分界线,黑色虚线代表距离分界线最近的样本,两条黑色虚线之间的距离为分类间隔。分类间隔的大小和误差成反比,通过调节分类间隔的大小来控制误差,得到的最优结果平面称为最优超平面。近年来,SVM作为机器学习领域的热点工具,在处理小样本、高维度和非线性类的样本具有一定的优势,被广泛应用于生物学各个研究的领域中[15]。本文使用LibSVM支持向量机算法软件包进行预测。
1.4 降维去冗余方法 1.4.1 F-score降维在预测的过程中,不相关的特征向量和特征信息的冗余现象,都会导致增加不必要的计算过程,降低预测的准确率。为了消除特征信息的冗余,去除不相关特征向量,就需要用到特征选择技术。实现特征信息在空间维数上的压缩,获得最佳维数,在众多的特征中选择对分类识别最有效的特征,即为特征选择。目前具有代表性的特征选择技术有F-score、最大相关-最小冗余(mRMR)、最大相关-最大距离(MRMD)、二项分布(BD)、递归特征消除法(RFE),主成分分析(PCA)和方差分析(ANOVA)等。本文选用F-score作为降维去冗余方法之一,F-score最早是由Chen Yi-Wei[16]提出,它的执行思路是先按照设定逻辑对各个特征向量打分,所有的特征向量都按F-score值分数排序,分数越高表明该特征越具有区别性。将得分最高的作为第一个特征向量进行预测。然后根据分数高低,把其余的特征向量陆续加入到上一个特征向量后面,再进行预测,直到添加完所有的特征向量为止[17]。F-score是一种衡量两类特征向量之间分辨能力的方法,可以实现对特征信息冗余的消除,获得最佳的维数,选择有效的特征向量 [18]。具体可以表示为
| $ F_i=\frac{\left(\bar{x}_i^{(+)}-\bar{x}_i\right)^2+\left(\bar{x}_i^{(-)}-\bar{x}_i\right)^2}{\frac{1}{n^{+}-1} \sum\nolimits_{k=1}^{n^{+}}\left(x_{k, i}^{(+)}-\bar{x}_i^{(+)}\right)^2+\frac{1}{n^{-}-1} \sum\nolimits_{k=1}^{n-}\left(x_{k, i}^{(-)}-\bar{x}_i^{(-)}\right)^2} $ | (9) |
上述公式中:
选用最大相关-最小冗余(Maximal relevance and minimal redundancy, mRMR)作为第二个特征选择算法。为了消除特征信息冗余产生的不良影响,去除不相关特征向量,改善预测模型的稳定性和有效性,提高预测结果的可靠性和准确率,有很多特征选择方法已被提出[19]。在众多特征选择方法中,选用最大相关-最小冗余方法是因为它在基于不同的分类算法下,均可以显著改善特征选择结果可靠性和分类准确率,被广泛用于许多学科研究领域中。本文使用的mRMR程序由彭等[20]开发(http://home.penglab.com/proj/mRMR/),mRMR原理[21]如下所示:
| $ I(x ; y)=\iint p(x, y) \log \frac{p(x, y)}{p(x) p(y)} \mathrm{d} x \mathrm{~d} y $ | (10) |
上述公式中:x和y是随机变量,
最大相关和最小冗余的测度指标分别定义为:
| $ \max D(S, c), D=\frac{1}{|S|} \sum\nolimits_{x_i \in S} I\left(x_i ; c\right) $ | (11) |
| $ \min R(S), R=\frac{1}{|S|^2} \sum\nolimits_{x_i, x_j \in S} I\left(x_i ; x_j\right) $ | (12) |
上述公式中:S指的是特征集,|S|指的是特征集包含的特征数目;c指的是目标类别;
为了对预测的结果进行检验和总结,很多检验方法被提出,例如:留一法(Jackknife检验)、K折交叉检验和自洽检验等。本文最终选用了Jackknife检验方法。Jackknife检验方法中,数据集中的每个样本都将作为一个独立的测试样本,数据集中测试样本之外的其余样本作为训练集,依次将所有的样本都进行检验,使得到的结果可靠、客观和严谨。对预测结果的评估同样重要,本文选取以下指标作为对结果的评价:敏感性(Sensitivity,Sn),特异性(Specificity,Sp),马修斯相关系数(Matthew’s correlation coefficient,MCC)和成功率(Accuracy,Acc)。Sn可以表明预测结果的准确性;Sp可以表明预测结果的可靠性;MCC可以表明预测结果与实际数据的相关性;Acc可以表明整个数据集的预测正确率[23]。具体表示为
| $ S n=\frac{T P}{T P+F N} $ | (13) |
| $ S p=\frac{T N}{T N+F P} $ | (14) |
| $ \begin{aligned} & M C C= \\ & \frac{T P \times T N-F P \times F N}{\sqrt{(T P+F P) \times(T N+F N) \times(T P+F N) \times(T N+F P)}} \\ & \end{aligned} $ | (15) |
| $ A c c=\frac{T P+T N}{T P+T N+F P+F N} $ | (16) |
上述公式中:TP指的是锌指蛋白序列中被正确预测的个数,TN指的是非锌指蛋白序列中被正确预测的个数,FP指的是非锌指蛋白序列中被错误预测为锌指蛋白的个数,FN指的是锌指蛋白序列中被错误预测为非锌指蛋白的个数。
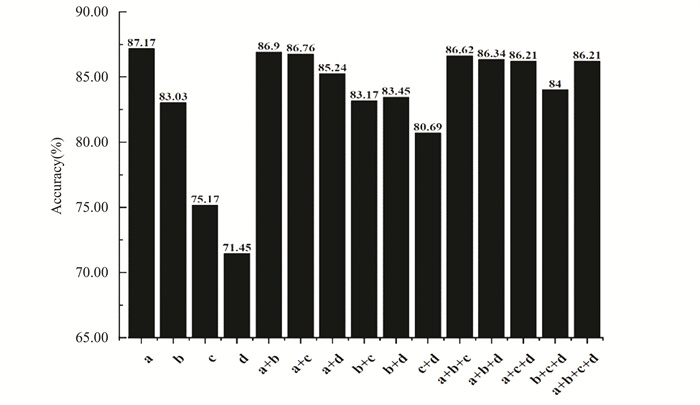
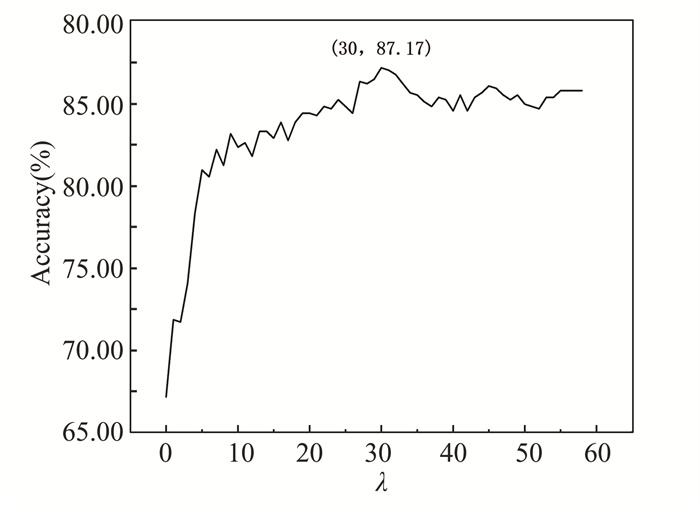
2 结果与分析 2.1 平均化学位移特征信息的最优组合选取为更好预测锌指蛋白,需要对平均化学位移特征信息的两个主要参数进行最优选择,一个参数是四种骨架原子的最优组合,另一个参数是相关常数λ的最优选择。图 2列出了平均化学位移的四种骨架原子不同组合方案预测成功率,包括单个骨架原子的预测成功率和多个骨架原子组合的预测成功率,预测结果表明,当骨架原子为15N时的成功率最高为87.17%。图 3表明在最优骨架原子15N的选择下,相关常数λ为30时的成功率最高。故本文平均化学位移的相关常数λ选择30,四种骨架原子选择单骨架原子15N。
|
图 2 平均化学位移的不同骨架原子组合方案预测成功率 Figure 2 Accuracy of different skeleton atom combination schemes of auto-covariance Average Chemical Shift 注:字母a代表15N,b代表13Cα,c代表1Hα,d代表1HN. |
|
图 3 最优原子结合模式下不同λ对应的预测成功率 Figure 3 Accuracy of different λ in the optimal atoms combination mode |
本文基于支持向量机算法,Jackknife检验方法,提取了氨基酸单肽组分信息、氨基酸二肽组分信息、平均化学位移三类特征信息,然后对数据集进行预测,详细结果见表 2。
| 表 2 不同特征参数的预测结果 Table 2 Prediction results of different feature parameters |
从表 2总体来看,单个特征信息预测成功率都在85%以上,马修斯相关系数都在0.75左右,其它各项评价指标也取得较好的结果,说明本文的预测具有较好的可靠性及参考价值。具体来看,氨基酸单肽组分信息和氨基酸二肽组分信息取得的成功率高且相近,预测结果都为87.86%。氨基酸单肽组分信息的敏感性高于其它两个参数的敏感性,说明其更适合对锌指蛋白的预测。
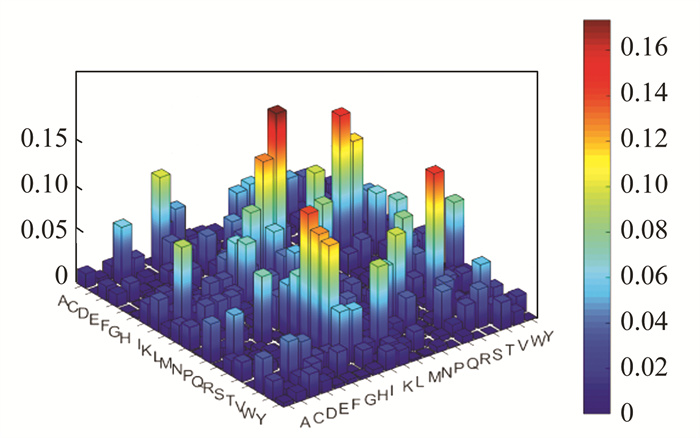
2.2.2 氨基酸二肽组分信息特征降维结果由于特征参数存在冗余现象,降低预测的准确率且增加不必要的计算过程,为了实现特征参数在空间维数上的压缩,获得最佳维数,本文选用F-score和最大相关-最小冗余(以下称mRMR)两种方法对氨基酸二肽组分信息特征参数进行降维处理。使用F-score降维方法,如图 4所示,颜色越趋于深红色,则说明相邻的氨基酸残基越具有更高的F值,并且更具有区别性,将作为第一个特征向量,依次再加入F值由高到低的其它特征向量进行预测。相反,颜色越趋于深蓝色,则不容易被区分识别。具体预测结果见表 3。
|
图 4 氨基酸二肽组分信息的F值三维热图 Figure 4 Three dimensional heat map of DC's F-score value |
| 表 3 氨基酸二肽组分信息特征参数降维和不降维处理下的预测结果 Table 3 Prediction results of Dipeptide Composition after reduced and non-reduced dimension treatment |
由表 3可以看出,使用两种降维方法对氨基酸二肽组分信息特征处理后,预测成功率明显提升,各项评价指标也取得了更好的结果。其中F-score降维处理后,特异性变化不大,敏感性和马修斯相关系数均取得了提升,说明F-score降维后的特征参数对数据集中锌指蛋白的预测更加精准,结果更具可靠性。mRMR降维后,各项评价指标的提升幅度明显,成功率达到了90.21%,表明mRMR方法更适合对氨基酸二肽组分信息特征进行降维处理。
由图 5可以看出,两种降维方法都明显提高了预测成功率,氨基酸二肽组分信息特征参数在F-score降维处理后,最终获得84维特征,成功率达到89.52%;在mRMR降维后,最终获得55维特征,成功率达到90.21%。总体而言,两种降维方法都起到了良好效果,实现了特征参数在空间维数上的压缩,一定程度上消除了特征参数的冗余现象,增加了预测的准确率。
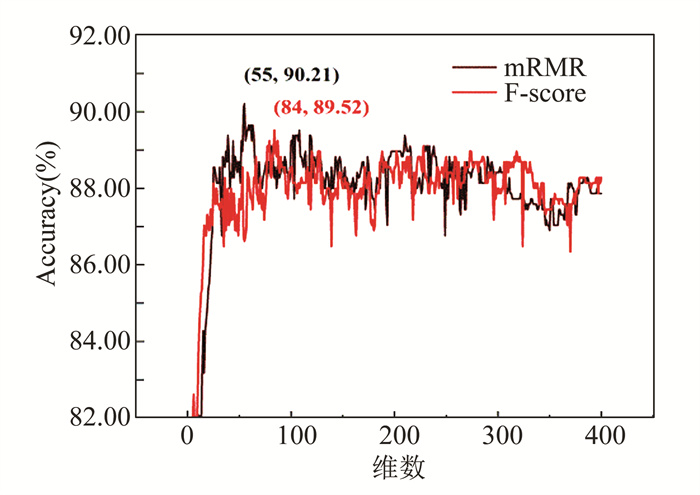
|
图 5 氨基酸二肽组分信息特征参数在不同的降维方法处理后的成功率 Figure 5 Accuracy of Dipeptide Composition after different dimension reduction methods |
在单特征信息预测的基础上,对平均化学位移、氨基酸单肽组分信息、氨基酸二肽组分信息三类特征信息进行融合,其中,氨基酸二肽组分信息采用mRMR降维后的结果。基于支持向量机算法,利用融合特征信息对数据集进行预测,结果见表 4。
| 表 4 融合特征参数的预测结果 Table 4 Prediction results of fusion feature parameters |
由表 4可以看出,氨基酸单肽组分信息和平均化学位移两类特征信息融合、氨基酸二肽组分信息和平均化学位移两类特征信息融合和三类特征信息融合的预测成功率均高于三类单特征信息的预测成功率,说明特征信息融合方法对提升锌指蛋白的预测成功率具有一定的作用。具体来看,在两类特征信息融合后,氨基酸单肽组分信息和平均化学位移两类单特征信息融合后取得了最高的预测成功率,达到92.55%,高于三类单特征信息的预测成功率。敏感性和马修斯相关系数结果也均高于三类单特征信息的结果和其它的两类特征信息融合的结果。说明氨基酸单肽组分信息和平均化学位移两类特征信息的融合对锌指蛋白的预测更具有优势,更加精确可靠。氨基酸二肽组分信息和平均化学位移两类特征信息融合后取得92.41%的预测成功率,其它各项评价指标也都取得较好结果。氨基酸单肽组分信息和氨基酸二肽组分信息两类特征信息融合后的预测成功率未高于mRMR降维后的氨基酸二肽组分信息单特征信息预测成功率,具体的原因可能是氨基酸单肽组分信息包含的蛋白质信息不够全面和充分,而且本文中并未对氨基酸单肽组分信息进行降维处理,只对氨基酸二肽组分信息特征进行了两种方式的降维处理,氨基酸单肽组分信息和氨基酸二肽组分信息两类特征信息融合后加大了数据的冗余,影响最终预测结果。在三类特征信息融合后,预测结果取得92%的成功率,各项评价指标结果较好,均高于三类单特征信息的预测评价指标结果,说明本文选取的三类特征信息对锌指蛋白的预测是有一定作用和意义的。
3 结论对C2H2型锌指蛋白的预测,可以深入了解锌指蛋白的结构和功能,为生物遗传、表观特征、医疗等方面的研究提供帮助。本文构建了C2H2型锌指蛋白和非锌指蛋白数据集,提取了三类特征信息,采用F-score和mRMR两种降维方法,利用支持向量机算法在Jackknife检验方法下对数据集进行预测。预测结果表明:
1) 利用F-score和mRMR方法对氨基酸二肽组分特征信息进行降维,mRMR降维方法好于F-score降维方法的预测结果。
2) 对特征信息进行适当融合有助于提高预测成功率。在后期研究中,进一步选取蕴含C2H2型锌指蛋白结构特征的特征参数对锌指蛋白进行预测,力争得到更高的预测成功率。
| [1] |
WEBSTER M W, WEIXLBAUMER A. The intricate relationship between transcription and translation[J]. Proceedings of the National Academy of Sciences of the United States of America, 2021, 118(21): e2106284118. DOI:10.1073/PNAS.2106284118 (  0) 0) |
| [2] |
刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用[J]. 科学通报, 2000, 45(14): 1465-1474. LIU Qiang, ZHANG Guiyou, CHEN Shouyi. Structure and regulation of plant transcription factors[J]. Chinese Science Bulletin, 2000, 45(14): 1465-1474. DOI:10.3321/j.issn:0023-074X.2000.14.002 (  0) 0) |
| [3] |
黄骥, 王建飞, 张红生. 植物C2H2型锌指蛋白的结构与功能[J]. 遗传, 2004, 26(3): 414-418. HUANG Ji, WANG Jianfei, ZHANG Hongsheng. Structure and function of plant C2H2 zinc finger protein[J]. Hereditas(Beijing), 2004, 26(3): 414-418. DOI:10.16288/j.yczz.2004.03.030 (  0) 0) |
| [4] |
MILLER J, MCLACHLAN A D, KLUG A. Repetitive zinc-binding domains in the protein transcription factor IMA from xenopus oocytes[J]. Journal of Trace Elements in Experimental Medicine, 2001, 14(2): 157-169. DOI:10.1002/j.1460-2075.1985.tb03825.x (  0) 0) |
| [5] |
LEE M S, GIPPERT G P, SOMAN K V, et al. Three-dimensional solution structure of a single zinc finger DNA binding domain[J]. Science(New York, N.Y.), 1989, 245(4918): 635-637. DOI:10.1126/science.2503871 (  0) 0) |
| [6] |
KRISHNA S S, MAJUMDAR I, GRISHIN N V. Structural classification of zinc fingers: survey and summary[J]. Nucleic Acids Research, 2003, 31(2): 532-550. DOI:10.1093/nar/gkg161 (  0) 0) |
| [7] |
赵楠, 赵飞, 李玉花. 锌指蛋白结构及功能研究进展[J]. 生物技术通讯, 2009, 20(1): 131-134. ZHAO Nan, ZHAO Fei, LI Yuhua. Advances in research on zinc finger protein[J]. Letters in Biotechnology, 2009, 20(1): 131-134. DOI:10.3969/j.issn.1009-0002.2009.01.037 (  0) 0) |
| [8] |
沈磐, 杨冬, 贺福初. C2H2型锌指蛋白结合的DNA序列预测方法的研究进展[J]. 生物化学与生物物理进展, 2017, 44(7): 573-579. SHEN Pan, YANG Dong, HE Fuchu. The advancement of the prediction methods for DNA-binding preferences of C2H2 zinc finger proteins[J]. Progress in Biochemistry and Biophysics, 2017, 44(7): 573-579. DOI:10.16476/j.pibb.2017.0047 (  0) 0) |
| [9] |
孙燕, 苟德明, 李文鑫. C2H2型锌指蛋白研究进展[J]. 生命的化学, 2001, 21(6): 473-475. SUN Yan, GOU Deming, LI Wenxin. Advances in research on C2H2 zinc finger protein[J]. Chemistry of Life, 2001, 21(6): 473-475. DOI:10.3969/j.issn.1000-1336.2001.06.012 (  0) 0) |
| [10] |
张振慧. 蛋白质分类问题的特征提取算法研究[D]. 长沙: 国防科学技术大学, 2006. DOI: 10.7666/d.y1101790. ZHANG Zhenhui. Research on algorithm in feature extraction of protein classification[D]. Changsha: National University of Defense Technology, 2006. DOI: 10.7666/d.y1101790. (  0) 0) |
| [11] |
AHMAD K, WARIS M, HAYAT M. Prediction of protein submitochondrial locations by incorporating dipeptide composition into chou's general pseudo amino acid composition[J]. The Journal of Membrane Biology, 2016, 249(3): 293-304. DOI:10.1007/s00232-015-9868-8 (  0) 0) |
| [12] |
FAN Guoliang, LI Qianzhong. Predict mycobacterial proteins subcellular locations by incorporating pseudo-average chemical shift into the general form of Chou's pseudo amino acid composition[J]. Journal of Theoretical Biology, 2012, 304: 88-95. DOI:10.1016/j.jtbi.2012.03.017 (  0) 0) |
| [13] |
姜燕. 基于多信息融合预测单定位和多定位凋亡蛋白质亚细胞位置[D]. 呼和浩特: 内蒙古大学, 2015. DOI: 10.7666/d.Y2840196. JIANG Yan. Predicting the single and multiple subcellular location of apoptosis proteins based on multi-features fusion[D]. Hohhot: Inner Mongolia University, 2015. DOI: 10.7666/d.Y2840196. (  0) 0) |
| [14] |
JING Xiaoyang, Li Fengmin. Predicting cell wall lytic enzymes using combined features[J]. Frontiers in Bioengineering and Biotechnology, 2021, 8(1): 1-8. DOI:10.3389/fbioe.2020.627335 (  0) 0) |
| [15] |
李明俊, 李凤敏. 基于多信息融合识别核定位蛋白[J]. 内蒙古农业大学学报(自然科学版), 2020, 41(1): 87-92. LI Mingjun, LI Fengmin. Identification of nucleoprotein based on different features[J]. Journal of Inner Mongolia Agricultural University (Natural Science Edition), 2020, 41(1): 87-92. DOI:10.16853/j.cnki.1009-3575.2020.01.016 (  0) 0) |
| [16] |
CHEN Yiwei, LIN Zhiren. Combining SVMs with various feature selection strategies[M]. Berlin: Springe, 2006: 315-324. DOI:10.1007/978-3-540-35488-8_13
(  0) 0) |
| [17] |
景晓洋. 热休克蛋白家族、细胞壁裂解酶的预测算法研究[D]. 呼和浩特: 内蒙古农业大学, 2021. DOI: 10.27229/d.cnki.gnmnu.2021.000384. JING Xiaoyang. The study on predictive algorithm for heat shock proteins and cell wall lytic enzymes[D]. Hohhot: Inner Mongolia Agricultural University, 2021. DOI: 10.27229/d.cnki.gnmnu.2021.000384. (  0) 0) |
| [18] |
谢娟英, 王春霞, 蒋帅, 等. 基于改进的F-score与支持向量机的特征选择方法[J]. 计算机应用, 2010, 30(4): 993-996. XIE Juanying, WANG Chunxia, JIANG Shuai, et al. Feature selection method combing improved F-score and support vector machine[J]. Journal of Computer Applications, 2010, 30(4): 993-996. (  0) 0) |
| [19] |
赖洪燕. 基于序列顺序与位置信息的启动子预测[D]. 成都: 电子科技大学, 2018. LAI Hongyan. Based on sequence-order and position-correlation information recognizing promoters[D]. Chengdu: University of Electronic Science and Technology of China, 2018. (  0) 0) |
| [20] |
DING C, PENG Hanchuan. Minimum redundancy feature selection from microarray gene expression data[J]. Journal of Bioinformatics and Computational Biology, 2005, 3(2): 185-205. DOI:10.1142/s0219720005001004 (  0) 0) |
| [21] |
PENG Hanchuan, LONG Fuhui, DING C. Feature selection based on mutual information: criteria of max-dependency, max-relevance, and min-redundancy[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2005, 27(8): 1226-1238. DOI:10.1109/TPAMI.2005.159 (  0) 0) |
| [22] |
李扬, 顾雪平. 基于改进最大相关最小冗余判据的暂态稳定评估特征选择[J]. 中国电机工程学报, 2013, 33(34): 179-186. LI Yang, GU Xueping. Feature selection for transient stability assessment based on improved maximal relevance and minimal redundancy criterion[J]. Proceedings of the CSEE, 2013, 33(34): 179-186. DOI:10.13334/j.0258-8013.pcsee.2013.34.024 (  0) 0) |
| [23] |
张松, 黄波, 夏学峰, 等. 蛋白质亚细胞定位的生物信息学研究[J]. 生物化学与生物物理进展, 2007, 34(6): 573-579. ZHANG Song, HUANG Bo, XIA Xuefeng, et al. Bioinformatics research in subcellular localization of protein[J]. Progress in Biochemistry and Biophysics, 2007, 34(6): 573-579. DOI:10.3321/j.issn:1000-3282.2007.06.004 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


