2. 大连交通大学 软件学院,辽宁 大连 116028
2. School of Software, Dalian Jiaotong University, Dalian 116028, Liaoning, China
微小核糖核酸(microRNA, 简称miRNA)是一种长度约为20-24个核苷酸的内源性非编码单链RNA分子,存在于真核生物中,包括植物和动物,可以在转录后水平调节基因表达[1]。许多研究表明,miRNA在各种复杂的生物过程中发挥着关键作用,包括细胞生长[2]、细胞分化[3]、细胞增殖[4]、细胞死亡[5]。此外,一系列研究证实,miRNA与人类疾病的发生和发展密切相关,如食管癌、结肠癌、肝癌和肺癌[6-10]。因此,采用适当的实验或计算方法来探索miRNA与疾病之间的联系,可以帮助医务人员从分子角度深入了解各种复杂疾病的病理机制并开发相关的新药[11]。一般来说,传统的生物学实验方法往往效率低下,需要投入大量的时间和金钱。然而,由于实验方法的可靠性,研究人员已经建立了许多权威的生物信息学数据库来存储实验证实的miRNA-疾病相关性。因此,miRNA-疾病关联预测的计算方法作为传统实验的辅助工具出现。通过对计算模型预测的高概率关联进行实验验证,可以有效地缩短传统实验的时间和成本。
在过去几年中,基于功能相似的miRNA往往与相似疾病相关[12],研究人员开发了多种miRNA-疾病关联预测模型,这些模型可以分为三类[7]。第一种类型的预测模型是基于打分函数的模型,它使用概率分布或统计分析来建立打分函数。例如,Mørk等[13]提出了miRNA-蛋白质-疾病关联预测模型miRPD来预测潜在的miRNA-疾病关联。他们基于miRNA-蛋白质和蛋白质-疾病关联得分定义了miRNA-疾病关联得分函数。其中,蛋白质被引入作为miRNA-疾病预测的中介。Chen等[14]提出了一种miRNA-疾病关联预测内得分和外得分的计算模型WBSMDA。他们定义了两种不同类型的函数来计算miRNA-疾病对的内得分和外得分,并将这两个得分整合以获得最终的关联分数。
第二种类型的预测模型是基于网络算法的模型,它从不同角度利用miRNA和疾病的相似性。例如,Xuan等[15]提出了一种新的预测miRNA-疾病关联的模型MIDP。对于有已知关联的疾病,MIDP在miRNA相似性网络中采用随机游走算法来预测与疾病有潜在关联的miRNA。对于没有任何已知关联的疾病,他们利用miRNA相似性网络、疾病相似性网络和已知的miRNA-疾病关联来构建miRNA-疾病双层网络。然后,他们在这个双层网络上进行随机游走,因此该模型可以用于没有任何已知关联信息的疾病。此外,Chen等[16]提出了基于异构图推断的miRNA-疾病关联预测模型HGIMDA。他们通过总结miRNA-疾病异构网络中所有边数为3的miRNA节点和疾病节点间的路径,定义了未标记的miRNA-疾病对的关联分数。在此基础上,Chen等[17]进一步提出了基于矩阵分解和异构图推断的miRNA-疾病关联预测模型MDHGI。首先,使用稀疏学习方法重构一个新的miRNA-疾病关联邻接矩阵。然后,基于重构的邻接矩阵、miRNA相似矩阵和疾病相似矩阵构建异构图。最后,建立一个迭代方程来预测miRNA-疾病对的关联概率。此外,You等[18]提出了基于路径的miRNA-疾病关联预测模型PBMDA。首先,在miRNA-疾病异构网络中搜索miRNA节点和疾病节点间所有长度小于等于3的路径。然后,基于路径的数量和每个路径的长度来计算所研究的miRNA和疾病之间的关联分数。Chen等[19]进一步提出了一个基于三层异构网络推断的miRNA-疾病关联预测模型TLHNMDA。该模型构建了一个包含miRNA、疾病和lncRNA节点的三层异构网络。基于这个三层网络,构建了一个迭代方程来获得miRNA-疾病对的关联概率。
第三种类型的预测模型是基于机器学习的模型。例如,Chen等[20]提出了预测miRNA-疾病关联的正则化最小二乘模型RLSMDA。他们在正则化最小二乘(RLS)的框架下,分别在miRNA和疾病空间构造半监督分类器,然后将两个不同空间的最优分类器组合在一起,得到miRNA-疾病对的概率。Li等[21]使用奇异值阈值(SVT)算法建立了MCMDA模型。采用矩阵填充算法更新miRNA-疾病邻接矩阵,得到最终的miRNA-疾病关联矩阵。此外,Chen等[22]提出miRNA-疾病关联预测模型RKNNMDA。他们利用KNN算法获得被研究miRNA的K个最近邻居,并使用支持向量机对K个邻居进行重新排序。然后,通过检查K个邻居与候选疾病之间的关联信息,可以计算出被研究的miRNA与候选疾病之间的关联分数。同样,作者还从疾病的角度计算了关联分数。最后,他们从两个不同的角度综合了关联得分,用于预测潜在的miRNA-疾病的关联。此外,Chen等[23]还提出了基于决策树学习的miRNA-疾病关联预测模型EGBMMDA。EGBMMDA构造了三种不同类型的特征,并将它们连接起来构建特征向量作为输入。在梯度增强的框架下,通过训练回归树获得潜在的miRNA-疾病关联的概率。
由于深度学习技术可以更好地学习数据的表示形式,并且近年来已经在基因组学和药物研发等许多领域成功应用[24],我们考虑将其应用于miRNA与疾病关联的预测。本文提出了一种端到端的深度学习模型MDAGAC来预测miRNA-疾病关联关系。首先从HMDD v2.0数据库中收集了人类miRNA-疾病关联数据,然后计算了疾病语义相似性、miRNA功能相似性、疾病和miRNA的高斯相互作用谱核相似性并整合疾病和miRNA的相似性。然后,通过图自编码器和协同训练来改善标签传播的效果。该模型分别在miRNA图和疾病图上建立了两个图自编码器,并对这两个图自编码器进行了协同训练。miRNA图和疾病图上的图自编码器能够通过初始关联矩阵重构得分矩阵,这相当于在图上传播标签。miRNA-疾病关联的预测概率可以从得分矩阵得到。最后通过五折交叉验证评估了方法的性能,结果显示模型MDAGAC在五折交叉验证中获得0.960 3±0.003 0的平均AUC和标准差。
1 材料 1.1 数据集从HMDD v2.0数据库中获得人类miRNA-疾病关联数据[25]。具体来说,有495个miRNA、383种疾病和5 430个经实验验证的miRNA-疾病关联关系。用nd和nm分别表示疾病和miRNA的数量,利用大小为nm×nd的邻接矩阵A来表示所有的miRNA-疾病对。如果miRNAm(i)与疾病d(j)有关联,则A(i, j)等于1,否则为0。
1.2 疾病语义相似性1使用MeSH[26]数据库计算疾病的语义相似性1。在MeSH数据库中,多种疾病间的关联由有向无环图(DAG)[27]表示,其中节点表示疾病,边表示关联。对于疾病D,我们可以用DAG(D)=(D, T(D), E(D))来表示该疾病,其中T(D)表示节点集,E(D)表示边集。定义疾病D的语义值DV1(D)如下:
| $ D V 1(D)=\sum\nolimits_{d \in T(D)} D 1_D(d) $ | (1) |
| $ D 1_D(d)=\left\{\begin{array}{l} 1 \text {, if } d=D \\ \max \left\{\rho * D 1_D(d^{'}) \mid d^{'} \in \text { childrenofd }\right\}, \\ \text { if } d \neq D \end{array}\right. $ | (2) |
其中,
| $ \operatorname{SS} 1_d\left(d_i, d_j\right)=\frac{\sum\nolimits_{t \in T\left(d_i\right) \cap T\left(d_j\right)}\left(D 1_{d_i}(t)+D 1_{d_j}(t)\right)}{D V 1\left(d_i\right)+D V 1\left(d_j\right)} $ | (3) |
在语义相似性1中,假设在同一层的不同节点对疾病的语义值贡献相同。但它忽略了DAG中疾病数量的因素,对于不常见的疾病d,对疾病D应该有更高的贡献值[28-29]。因此,使用等式(4)来计算D2D(d):
| $ D 2_D(d)=-\log \frac{n_d}{n_{dis}} $ | (4) |
其中,nd表示包含节点d的疾病DAG的数目,ndis表示所有疾病DAG的数目。
类似地,定义疾病D的语义值DV2(D)如下:
| $ D V 2(D)=\sum\nolimits_{d \in T(D)} D 2_D(d) $ | (5) |
因此,对于疾病di和疾病dj,使用公式(6)来计算语义相似度2SS2d(di, dj)。
| $ S S 2_d\left(d_i, d_j\right)=\frac{\sum\nolimits_{t \in T\left(d_i\right) \cap T\left(d_j\right)}\left(D 2_{d_i}(t)+D 2_{d_j}(t)\right)}{D V 2\left(d_i\right)+D V 2\left(d_j\right)} $ | (6) |
一般认为功能相似的miRNA往往与语义相似的疾病相关,Wang等[30]提出了计算miRNA功能相似性的方法。从http://www.cuilab.cn/files/images/cuilab/misim.zip中可以获得miRNA的功能相似性,用矩阵FS来表示。
1.5 疾病和miRNA的高斯相互作用谱核相似性由于不能得到所有疾病的DAG,对于没有DAG的疾病,不能通过DAG计算疾病语义相似性。所以为了获得更全面的疾病相似性,基于已知miRNA与疾病之间的关联,构建了高斯相互作用谱核相似性[31]。疾病di和疾病dj的高斯相互作用谱核相似性计算如下:
| $ G D\left(d_i, d_j\right)=\exp \left(-\gamma_d\left\|A\left(*, d_i\right)-A\left(*, d_j\right)\right\|^2\right) $ | (7) |
其中,
| $ \gamma_d=\frac{\gamma_d'}{\frac{1}{n d} \sum\nolimits_{i=1}^{n d}\left\|A\left(*, d_i\right)\right\|^2} $ | (8) |
其中,γd'设为1。类似地,为了能够得到所有miRNA的相似性,计算miRNAm(i)和miRNAm(j)的高斯相互作用谱核相似性如下:
| $ G M\left(m_i, m_j\right)=\exp \left(-\gamma_m\left\|A\left(m_i, *\right)-A\left(m_j, *\right)\right\|^2\right) $ | (9) |
| $ \gamma_m=\frac{\gamma_m'}{\frac{1}{n m} \sum\nolimits_{i=1}^{n m}\left\|A\left(m_i, *\right)\right\|^2} $ | (10) |
疾病语义相似性SS是一个稀疏矩阵,单独使用这个矩阵很难达到很好的预测效果。此外,高斯相互作用谱核相似性GD是通过已知的miRNA-疾病关联来计算的,这不够准确。因此,有必要将疾病语义相似性SS和高斯相互作用谱核相似性GD结合起来,以达到良好的预测效果。我们通过一个加权参数将SS和GD整合为一个疾病相似性矩阵SD [32]。疾病di和疾病dj整合后的疾病矩阵如下:
| $S D\left(d_i, d_j\right)=\alpha S S\left(d_i, d_j\right)+(1-\alpha) G D\left(d_i, d_j\right) $ | (11) |
| $ S S\left(d_i, d_j\right)=\frac{S S 1_d\left(d_i, d_j\right)+S S 2_d\left(d_i, d_j\right)}{2} $ | (12) |
其中α是权重,范围在0和1之间。类似地,miRNAm(i)和miRNAm(j)之间的整合相似性矩阵SM通过以下公式计算:
| $ S M\left(m_i, m_j\right)=\beta F S\left(m_i, m_j\right)+(1-\beta) G M\left(m_i, m_j\right) $ | (13) |
在这项研究中,提出MDAGAC模型来预测miRNA-疾病关联关系。MDAGAC的流程图如图 1所示。MDAGAC的第一步,数据准备,如前节所述,构建了miRNA-疾病对的关联矩阵
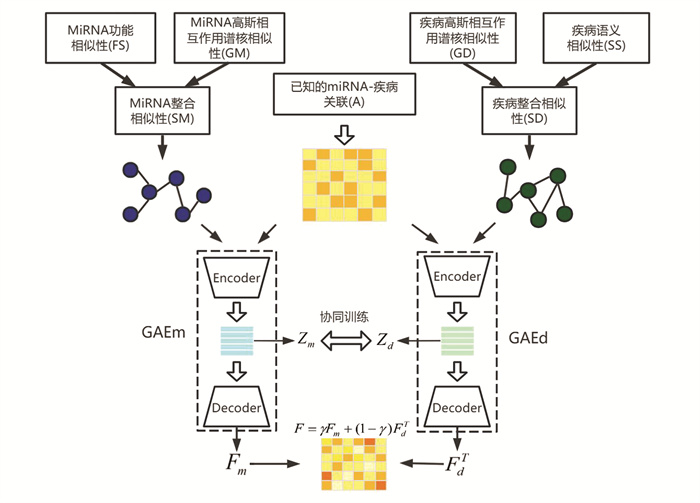
|
图 1 MDAGAC算法流程图 Figure 1 Flowchart of MDAGAC |
相似性矩阵可以看作是miRNA图或疾病图的邻接矩阵,因为每个miRNA或疾病表示该图的一个节点。根据前人的研究[33],相似性图可以构造如下。以疾病的相似性图构造为例,把
| $ S_d=C_d^T \otimes C_d+I $ | (14) |
其中
| $ S_m=C_m^T \otimes C_m+I $ | (15) |
图自编码器[34]是一个具有图卷积层[35]的自编码器。Shi等[33]证明了以A为输入,F为输出的图自编码器可以模拟标签的传播过程。在MDAGAC中,分别在miRNA图和疾病图上提出了图自编码器(GAE)[36],用GAEm和GAEd表示。
首先通过2层图卷积编码器学习特征表示Zm和Zd:
| $ Z_m=\tanh \left(N_m \cdot \text { Hardswish }\left(N_m A \theta^{(0)}\right) \theta^{(1)}\right) $ | (16) |
| $ Z_d=\tanh \left(N_d \cdot \text { Hardswish }\left(N_d A^T \varphi^{(0)}\right) \varphi^{(1)}\right) $ | (17) |
其中θ,φ表示神经网络的权重。Nm和Nd分别表示miRNA图和疾病图的归一化邻接矩阵。
| $ N_m=D_m^{-1 / 2} S_m D_m^{-1 / 2} $ | (18) |
其中Dm是Sm的度矩阵。Dm是通过公式
| $ N_d=D_d^{-1 / 2} S_d D_d^{-1 / 2} $ | (19) |
然后分别对特征表示Zm和Zd进行解码,得到分数矩阵
| $ F_m=\operatorname{sigmoid}\left(N_m \cdot \text { Hardswish }\left(N_m Z_m \theta^{(2)}\right) \theta^{(3)}\right) $ | (20) |
| $ F_d=\operatorname{sigmoid}\left(N_d \cdot \text { Hardswish }\left(N_d Z_d \varphi^{(2)}\right) \varphi^{(3)}\right) $ | (21) |
GAEm的重构误差是预测值和真实标签之间的交叉熵Lm,类似的GAEd的重构误差是Ld。图自编码器可以通过最小化重构误差来训练:
| $ L_r=\gamma L_m+(1-\gamma) L_d $ | (22) |
| $ L_m=-\sum\nolimits_{i, j} A_{i j} \log F_{m i j} $ | (23) |
| $ L_d=-\sum\nolimits_{i, j} A_{i j} \log F_{d i j} $ | (24) |
其中
最小化式(22)相当于分别在miRNA图和疾病图上训练图自编码器。以往的研究表明,整合双方信息的协同训练可以提高预测生物实体关联[33, 37]的精度。采用图自编码器学习到的特征表示Zm和Zd来定义协同训练损失[36]:
| $ L_c=\frac{1}{2}\left\|A-Z_m Z_d^T\right\|_F^2 $ | (25) |
为了避免过拟合, 加入正则化的Frobenius范数,总损失可定义为:
| $ \min _{\theta, \varphi} L_c+\mu L_r+\lambda\|\theta\|_F^2+\lambda\|\varphi\|_F^2 $ | (26) |
其中μ是参数,λ设为10-7。最后,通过Fm和Fd的线性组合得到最优分数矩阵
| $ F=\gamma F_m+(1-\gamma) F_d^T $ | (27) |
其中
| 表 1 MDAGAC算法 Table 1 MDAGAC algorithm |
首先从HMDD v2.0[25]中获得包含495个miRNA与383种疾病之间的5 430个已知关联的训练数据,然后采用五折交叉验证评估模型MDAGAC的准确性。所有已知的miRNA-疾病关联被随机分为五个大小相等的子集。每个子集依次用作测试集,而其他四个子集用作训练集。如图 2所示,MDAGAC的平均AUC和标准差为0.960 3±0.003 0,这是五折交叉验证0.961 7, 0.955 8, 0.959 1, 0.965 0和0.959 8的平均值和标准差。
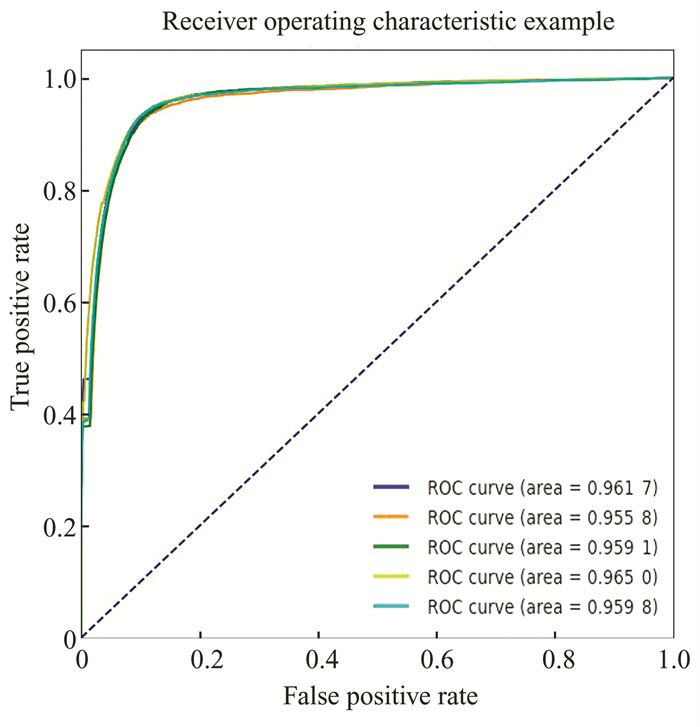
|
图 2 五折交叉验证中MDAGAC的ROC曲线 Figure 2 ROC curves of MDAGAC in 5-fold cross-validation |
为了进一步证明该模型的优越性能,比较了MDAGAC模型与八个最先进模型的预测性能,它们分别是PBMDA (0.917 2), SAEMDA(0.910 2), EGBMMDA (0.904 8), MDHGI (0.879 4), TLHNMDA (0.879 5), MCMDA (0.876 7), MaxFlow (0.857 9)和RLSMDA (0.856 9)。为了公平比较,上述模型均基于HMDD v2.0[25]进行了五折交叉验证评估。此外,由于上述模型采用了多种不同的评价指标,在此仅利用AUC值来综合衡量这些模型的预测性能。比较结果总结在表 2中。可以看到,我们的模型在这九个模型中实现了最高的AUC值。MDAGAC的优越性能得益于基于图卷积的编码器和端到端的训练方式。
| 表 2 五折交叉验证中MDAGAC与其他模型的性能对比 Table 2 Performance comparison between MDAGAC and other models in 5-fold cross-validation |
MDAGAC中的参数会影响预测性能。在本节中,通过五折交叉验证选择具有最佳平均AUC的超参数。为了验证参数α和β对疾病和miRNA的整合相似性矩阵SD和SM的有效性,首先在0到1.0的区间内定义了11个等间距的值,并将这些值应用在α和β上来训练模型。然后通过五折交叉验证来计算每个模型的AUC值。如表 3所示,当α=0.3和β=0.3时,模型的预测性能最好。
| 表 3 不同α和β值的五折交叉验证结果 Table 3 Results of 5-fold cross-validation for different values of α and β |
在MDAGAC中,通过参数γ平衡miRNA空间和疾病空间。选择
| 表 4 不同γ值的五折交叉验证结果 Table 4 Results of 5-fold cross-validation for different values of γ |
采用pytorch (https://pytorch.org/) 构建MDAGAC,并应用Adam优化器训练模型。然后,将神经网络的随机失活率设置为0.5,并通过改变学习率lr来评估MDAGAC的预测性能。结果显示在表 5中,其中最佳学习率值是0.01。此外,还对不同的隐藏层维度进行模型训练。结果如表 6所示,我们的模型预测性能随着隐藏层维度的增加而增强。但当维数大于144时,AUC不再有显著提高。因此,将隐藏层的维数设置为144,以节省模型的计算成本。
| 表 5 不同lr值的五折交叉验证结果 Table 5 Results of 5-fold cross-validation for different values of lr |
| 表 6 不同隐藏层维度的五折交叉验证结果 Table 6 Results of 5-fold cross-validation for different hidden layer dimensions |
预测潜在的miRNA-疾病关联使研究人员能够更好地了解疾病的机制,并促进复杂疾病的诊断、治疗和预防。本研究提出了一种端到端的深度学习模型来预测miRNA-疾病关联关系,称为MDAGAC。基于五折交叉验证的实验结果表明:
1) MDAGAC方法可靠有效,优于现有的几种方法。
2) 与现有的miRNA-疾病关联预测方法相比,MDAGAC采用端到端的神经网络模型来协同训练两个GAE。这种数据驱动的端到端的深度学习模型不仅提高了预测潜在miRNA-疾病关联的精度,而且为生物信息学的其它领域提供了一种通用的方法。
| [1] |
AMBROS V. The functions of animal microRNAs[J]. Nature, 2004, 431: 350-355. DOI:10.1038/nature02871 (  0) 0) |
| [2] |
BARTEL D P. MicroRNAs: Genomics, biogenesis, mechanism and function[J]. Cell, 2004, 116: 281-297. DOI:10.1016/s0092-8674(04)00045-5 (  0) 0) |
| [3] |
XIAO C, CALADO D P, GALLER G, et al. MiR-150 controls B cell differentiation by targeting the transcription factor c-Myb[J]. Cell, 2016, 165(4): 1027. DOI:10.1016/j.cell.2016.04.056 (  0) 0) |
| [4] |
JOHNNIDIS J B, HARRIS M H, WHEELER R T, et al. Regulation of progenitor cell proliferation and granulocyte function by microRNA-223[J]. Nature, 2008, 451(7182): 1125-1129. DOI:10.1038/nature06607 (  0) 0) |
| [5] |
KIM J H, WOO H R, KIM J, et al. Trifurcate feed-forward regulation of age-dependent cell death involving miR164 in Arabidopsis[J]. Science, 2009, 323(5917): 1053-1057. DOI:10.1126/science.1166386 (  0) 0) |
| [6] |
AMBROS V. microRNAs: Tiny regulators with great potential[J]. Cell, 2001, 107(7): 823-826. DOI:10.1016/s0092-8674(01)00616-x (  0) 0) |
| [7] |
CHEN Xing, XIE Di, ZHAO Qi, et al. MicroRNAs and complex diseases: from experimental results to computational models[J]. Briefings in Bioinformatics, 2019, 20(2): 515-539. DOI:10.1093/bib/bbx130 (  0) 0) |
| [8] |
CHOU C H, CHANG N W, SHRESTHA S, et al. miRTarBase 2016: Updates to the experimentally validated miRNA-target interactions database[J]. Nucleic Acids Research, 2016, 44(D1): D239-D247. DOI:10.1093/nar/gkv1258 (  0) 0) |
| [9] |
邹小龙, 董雪松, 孙学溥. 结肠癌中核内miRNA的激活调控作用研究[J]. 生物信息学, 2019, 17(2): 111-115. ZOU Xiaolong, DONG Xuesong, SUN Xuepu. Activation regulation of nuclear miRNA regulation in colon cancer[J]. Chinese Journal of Bioinformatics, 2019, 17(2): 111-115. DOI:10.12113/j.issn.1672-5565.201903009 (  0) 0) |
| [10] |
赵燕伟, 王振兴, 王熙梓, 等. 转录因子及miRNA调控食管癌耐药机制研究[J]. 生物信息学, 2022, 20(1): 56-63. ZHAO Yanwei, WANG Zhenxing, WANG Xizi, et al. Role and mechanisms of miRNA and transcription factorsregulating drug resistance of esophageal cancer[J]. Chinese Journal of Bioinformatics, 2022, 20(1): 56-63. DOI:10.12113/202009005 (  0) 0) |
| [11] |
CHEN Changzheng. microRNAs as oncogenes and tumor suppressors[J]. New England Journal Medicine, 2005, 353(17): 1768-1771. DOI:10.1056/NEJMp058190 (  0) 0) |
| [12] |
JIANG Qinghua, HAO Yangyang, WANG Guohua, et al. Prioritization of disease microRNAs through a human phenome-microRNAome network[J]. BMC Systems Biology, 2010, 4: S2. DOI:10.1186/1752-0509-4-S1-S2 (  0) 0) |
| [13] |
MØRK S, PLETSCHER-FRANKILD S, PALLEJA CARO A, et al. Protein-driven inference of miRNA-disease associations[J]. Bioinformatics, 2014, 30: 392-397. DOI:10.1093/bioinformatics/btt677 (  0) 0) |
| [14] |
CHEN Xing, YAN C C, ZHANG Xu, et al. WBSMDA: Within and between score for miRNA-disease association prediction[J]. Scientific Reports, 2016, 6(1): 21106. DOI:10.1038/srep21106 (  0) 0) |
| [15] |
XUAN Ping, HAN Ke, GUO Yahong, et al. Prediction of potential disease-associated microRNAs based on random walk[J]. Bioinformatics, 2015, 31(11): 1805-1815. DOI:10.1093/bioinformatics/btv039 (  0) 0) |
| [16] |
CHEN Xing, YAN C C, ZHANG Xu, et al. HGIMDA: Heterogeneous graph inference for miRNA-disease association prediction[J]. Oncotarget, 2016, 7(40): 65257-65269. DOI:10.18632/oncotarget.11251 (  0) 0) |
| [17] |
CHEN Xing, YIN Jun, QU Jia, et al. MDHGI: Matrix decomposition and heterogeneous graph inference for miRNA-disease association prediction[J]. PLoS Computational Biology, 2018, 14(8): e1006418. DOI:10.1371/journal.pcbi.1006418 (  0) 0) |
| [18] |
YOU Zhuhong, HUANG Zhian, ZHU Zexuan, et al. PBMDA: A novel and effective path-based computational model for miRNA-disease association prediction[J]. PLoS Computational Biology, 2017, 13(3): e1005455. DOI:10.1371/journal.pcbi.1005455 (  0) 0) |
| [19] |
CHEN Xing, QU Jia, YIN Jun. TLHNMDA: Triple layer heterogeneous network based inference for miRNA-disease association prediction[J]. Frontiers in Genetics, 2018, 9: 234. DOI:10.3389/fgene.2018.00234 (  0) 0) |
| [20] |
CHEN Xing, YAN Guiying. Semi-supervised learning for potential human microRNA-disease associations inference[J]. Scientific Reports, 2014, 4: 5501. DOI:10.1038/srep05501 (  0) 0) |
| [21] |
LI Jianqiang, RONG Zhihao, CHEN Xing, et al. MCMDA: Matrix completion for miRNA-disease association prediction[J]. Oncotarget, 2017, 8(13): 21187-21199. DOI:10.18632/oncotarget.15061 (  0) 0) |
| [22] |
CHEN Xing, WU Qiaofeng, YAN Guiying. RKNNMDA: Ranking-based KNN for miRNA-disease association prediction[J]. RNA Biology, 2017, 14(7): 952-962. DOI:10.1080/15476286.2017.1312226 (  0) 0) |
| [23] |
CHEN Xing, HUANG Li, XIE Di, et al. EGBMMDA: Extreme gradient boosting machine for miRNA-disease association prediction[J]. Cell Death & Disease, 2018, 9: 3. DOI:10.1038/s41419-017-0003-x (  0) 0) |
| [24] |
LECUN Y, BENGIO Y, HINTON G. Deep learning[J]. Nature, 2015, 521(7553): 436-444. DOI:10.1038/nature14539 (  0) 0) |
| [25] |
LI Yang, QIU Chengxiang, TU Jian, et al. HMDD v2.0: A database for experimentally supported human microRNA and disease associations[J]. Nucleic Acids Research, 2014, 42(D1): D1070-D1074. DOI:10.1093/nar/gkt1023 (  0) 0) |
| [26] |
SCHEIBLE R, STRECKER P, YAZIJY S, et al. A multilingual browser platform for medical subject headings[J]. Studies in Health Technology and Informatics, 2022, 289: 384-387. DOI:10.3233/SHTI210939 (  0) 0) |
| [27] |
LI Yu, KUWAHARA H, YANG Peng, et al. PGCN: Disease gene prioritization by disease and gene embedding through graph convolutional neural networks[J]. bioRxiv, 2019, 532226. DOI:10.1101/532226 (  0) 0) |
| [28] |
WANG Lei, YOU Zhuhong, HUANG Y A, et al. An efficient approach based on multi-sources information to predict circRNA-disease associations using deep convolutional neural network[J]. Bioinformatics, 2020, 36(13): 4038-4046. DOI:10.1093/bioinformatics/btz825 (  0) 0) |
| [29] |
WANG Lei, YOU Zhuhong, LI Yangming, et al. GCNCDA: A new method for predicting circRNA-disease associations based on graph convolutional network algorithm[J]. PLOS Computational Biology, 2020, 16(5): e1007568. DOI:10.1371/journal.pcbi.1007568 (  0) 0) |
| [30] |
WANG Dong, WANG Jun, LU Ming, et al. Inferring the human microRNA functional similarity and functional network based on microRNA-associated diseases[J]. Bioinformatics, 2010, 26(13): 1644-1650. DOI:10.1093/bioinformatics/btq241 (  0) 0) |
| [31] |
VAN LAARHOVEN T, NABUURS S B, MARCHIORI E. Gaussian interaction profile kernels for predicting drug-target interaction[J]. Bioinformatics, 2011, 27(21): 3036-3043. DOI:10.1093/bioinformatics/btr500 (  0) 0) |
| [32] |
JI Cunmei, GAO Zhen, MA Xu, et al. AEMDA: Inferring miRNA-disease associations based on deep autoencoder[J]. Bioinformatics, 2021, 37(1): 66-72. DOI:10.1093/bioinformatics/btaa670 (  0) 0) |
| [33] |
SHI Zhuangwei, ZHANG Han, JIN Chen, et al. A representation learning model based on variational inference and graph autoencoder for predicting lncRNA-disease associations[J]. BMC Bioinformatics, 2021, 22: 136. DOI:10.1186/s12859-021-04073-z (  0) 0) |
| [34] |
KIPF T N, WELLING M. Variational graph auto-encoders[J]. arXiv preprint arXiv: 1611.07308, 2016. DOI: 10.48550/arXiv.1611.07308.
(  0) 0) |
| [35] |
KIPF T N, WELLING M. Semi-supervised classification with graph convolutional networks[J]. arXiv preprint arXiv: 1609.02907, 2016. DOI: 10.48550/arXiv.1609.02907.
(  0) 0) |
| [36] |
JIN Chen, SHI Zhuangwei, ZHANG Han, et al. Predicting lncRNA-protein interactions based on graph autoencoders and collaborative training[C].//2021 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). IEEE, 2021: 38-43. DOI: 10.1109/BIBM52615.2021.9669316.
(  0) 0) |
| [37] |
HAN Peng, YANG Peng, ZHAO Peilin, et al. GCN-MF: Disease-gene association identification by graph convolutional networks and matrix factorization[C].//Proceedings of the 25th ACM SIGKDD international conference on knowledge discovery & data mining, 2019: 705-713. DOI: 10.1145/3292500.3330912.
(  0) 0) |
 2024, Vol. 22
2024, Vol. 22


