长链非编码RNA(Long non-coding RNA, lncRNA)是一种长度超过200个核糖核苷酸的具有重要生物学功能的非编码RNA。研究发现,lncRNA是参与包括转录、剪接、翻译,DNA修复和调控微小RNA(microRNA, miRNA)表达水平在内的细胞过程的关键调节因子,近年来,越来越多的研究表明,lncRNA在肿瘤增殖、血管生成、细胞凋亡和转移中发挥重要作用[1-5]。例如:lncRNA MALAT1在胰腺癌、前列腺癌和三阴性乳腺癌中过度表达,影响细胞增殖和凋亡[4];在肝细胞癌中,MALAT1的过度表达与患者的不良预后和肿瘤复发相关[5]。此外,研究发现lncRNA可以作为药物的靶点,例如:在尤文肉瘤细胞中,lncRNA HULC充当miRNA海绵吸附miR-186,促进癌基因TWIST1的表达,而小分子YK-4-279可以通过抑制HULC、上调miR-186来治疗尤文肉瘤[6]。因此,靶向lncRNA的药物设计在肿瘤治疗方面有巨大的应用潜力。然而,当前已知的药物和lncRNA关联相对较少,且通过实验来检测新的药物-lncRNA关联耗时、费力,因此,迫切需要开发计算方法来高通量识别新的药物-lncRNA关系。
当前预测药物与靶点关系的计算方法大致可分为三类:即基于网络传播的方法、机器学习方法和深度学习方法。基于网络传播的方法通常通过网络随机游走等方法传播信息来将药物与靶点联系起来。例如:TL-HGBI[7]通过整合药物、疾病和靶点三个网络进行随机游走识别新的药物靶点,具有良好的可解释性,但性能并不令人满意。基于机器学习的方法将药物-靶点关系对作为样本,将药物和靶点的信息编码到特征向量中以表征样本,然后训练分类器来识别关系是否存在。例如:矩阵分解和矩阵补全[8-9]在特征信息融合和预测药物与靶点关系方面效果不错,但这种分类方法严重依赖特征的提取和阴性样本的选择;deepDTnet[9]对药物和靶点提取多方面的相似性特征并加以整合预测,但这些特征只考虑同类型之间(药物-药物、靶点-靶点)的特征,没有从药物影响靶点表达这一方向思考。深度学习方法[10-13]在药物-靶点关系预测中也有成功的应用,例如:DeepDTI[11],LAGCN[12]等。DeepDTI通过知识图谱提取药物靶点信息再进行预测,LAGCN充分利用药物疾病网络的拓扑信息进行编码解码,但在特征的质量和可解释性方面有一定的不足。
因此,本研究拟基于药物结构信息和lncRNA序列信息利用图卷积网络(Graph convolution network)和归纳矩阵补全(Inductive matrix completion)[14]解码方法,系统地识别潜在的药物-lncRNA关系。具体地,本研究从D-lnc[15],DrugBank[16],PubChem[17]和GENCODE[18]等数据库提取药物和lncRNA信息,结合深度学习有关知识提出了一个用于预测药物-lncRNA关系的图卷积网络DLGCN(Drug-LncRNA graph convolution network)。通过效能评估发现,DLGCN的受试者工作特性曲线下面积(Area under receiver operating characteristic, AUROC)为0.843 1,性能优于其他的经典机器学习和常用的深度学习方法。因此,本研究提出的DLGCN模型能够准确识别新的药物-lncRNA关系,将有助于癌症新靶点的发现和抗癌药物的筛选。
1 材料与方法 1.1 数据集整理本研究对D-lnc数据库中实验证实的药物调控lncRNA表达的数据进行了整理,筛选出小分子药物和有序列信息的lncRNA,总共包含了35种药物和50种lncRNA组成的共89条关联关系。药物的结构信息来源于DrugBank和PubChem,lncRNA的基因组位置信息则来自GENCODE,序列由注释工具BEDTools提取得到。
1.2 图卷积网络为了高通量预测新的药物-lncRNA关联,我们提出了一种基于异质图卷积网络模型来识别潜在药物-lncRNA关联的方法。首先,构建药物-lncRNA异质网络,并构造图卷积网络训练所需的图矩阵和特征矩阵(图 1(a));然后,特征矩阵经过三层带注意力机制的图卷积层压缩成低维特征向量(图 1(b));接下来,低维特征向量再通过归纳矩阵补全解码得到重构的药物-lncRNA预测矩阵(图 1(c)),实现对关系矩阵的链路预测(Link prediction,指通过已知的网络节点以及网络结构等信息预测网络中尚未产生连边的两个节点之间产生链接的可能性)。图 1展示了本研究图卷积网络DLGCN的整个工作流程。
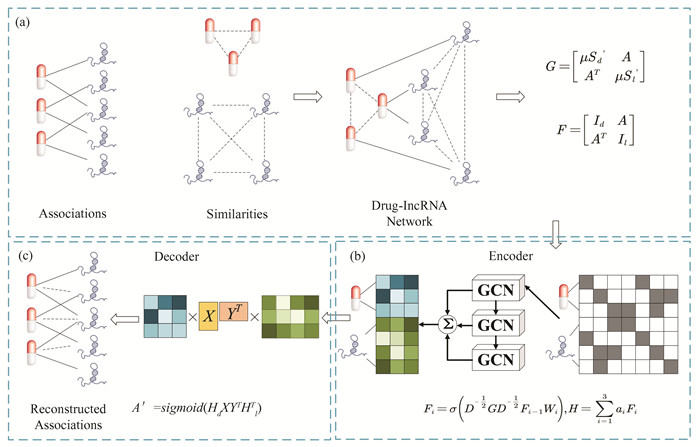
|
图 1 DLGCN工作流程 Figure 1 DLGCN Workflow 注:(a) 使用药物-lncRNA关联矩阵、药物相似性矩阵和lncRNA相似性矩阵构建异构图卷积网络,并生成图矩阵和特征矩阵; (b) 图卷积网络的编码器部分,将药物和lncRNA节点特征通过注意力机制编码为低维特征; (c) 图卷积网络的解码器部分,用药物和lncRNA节点的低维特征重构关联矩阵. |
图卷积网络是一种多层连接的神经网络架构,用于从图结构中学习节点和自身邻居节点的特征。一般的药物和靶点信息提取都只考虑同类型特征中与自己高相似度的节点所携带的信息,而药物和lncRNA不仅自身之间存在相似性,同时,药物还能够影响lncRNA的表达,因此,需要同时考虑药物、lncRNA的特征信息和药物-lncRNA关联网络中的结构信息。如图 1(a)所示,本文采用了一种整合药物-药物相似性、lncRNA-lncRNA相似性和药物-lncRNA关联的异质性网络的图卷积网络方法。
本研究将药物-lncRNA关系表示为二元矩阵A∈{0, 1}N×M,其中N,M分别表示药物和lncRNA的数量,如果药物i与lncRNA j相关,则Aij = 1;否则Aij = 0。N种药物之间和M种lncRNA之间的相似性分别表示为相似性矩阵Sd和Sl。
对于药物来说,本研究先根据药物的SMILES结构式提取分子指纹,再把分子指纹作为特征计算谷本系数。对于lncRNA来说,本研究用ENSG ID和注释文件提取lncRNA转录本序列,根据k-mer特征[19]计算lncRNA的谷本系数,本文取k = 5。谷本系数的计算公式如下:
| $ T\left(f_a, f_b\right)=\frac{f_a * f_b}{\left\|f_a\right\|^2+\left\|f_b\right\|^2-f_a * f_b} $ | (1) |
其中,fa和fb代表lncRNA或者药物a和b的特征向量,fa*fb表示a和b向量乘积,‖fa‖2和‖fb‖2表示a和b特征向量的模。
药物相似性矩阵Sd和lncRNA相似性矩阵Sl是由[0, 1]内的实数组成的密集矩阵,元素较小时表示药物或lncRNA之间的相似性较弱。本文通过保留前5%的最大元素来设置适当的阈值稀疏这两个相似性矩阵[20],具体地,矩阵里前5%的最大元素设置为1,其余元素设置为0。稀疏化矩阵不仅很好地保留了两个相似性矩阵中的重要相似性关系,同时显著降低了图卷积网络的计算复杂性。
图卷积网络计算所需的图邻接矩阵G和特征矩阵F通过下面的公式(2)和(3)得到:
| $ G=\left[\begin{array}{cc} \mu S_d^{\prime} & A \\ A^{\mathrm{T}} & \mu S_l^{\prime} \end{array}\right] $ | (2) |
| $ F=\left[\begin{array}{cc} I_d & A \\ A^{\mathrm{T}} & I_l \end{array}\right] $ | (3) |
其中,Sd’和Sl’分别表示稀疏化的药物相似性矩阵和lncRNA相似性矩阵,μ是控制图卷积传播过程中同质节点贡献的惩罚因子,Id和Il分别是N,M维的单位矩阵,T是矩阵的转置。
1.2.2 图卷积编码如图 1(b)所示,本文使用了一个三层加注意力机制的图卷积网络来编码。图卷积网络的每一层通过图聚合邻居节点的信息,整合自身和邻居节点的特征作为下一层的输入特征。对于给定邻接矩阵G的网络,图卷积网络的单层传播公式为
| $ F_i=\sigma\left(D^{-\frac{1}{2}} G D^{-\frac{1}{2}} F_{i-1} W_i\right) $ | (4) |
| $ D_{i i} \sum\limits_j\left(G_{i j}+I_{N+M}\right) $ | (5) |
其中,Fi是第i层节点的低维特征,对角矩阵D是G的度矩阵,Wi为第i层的可训练权重矩阵,σ(*)为非线性激活函数Relu。
基于上述公式,图卷积网络的第一层编码器可以表示为
| $ F_1=\sigma\left(D^{-\frac{1}{2}} G D^{-\frac{1}{2}} F_0 W_1\right) $ | (6) |
其中,W1∈R(N+M)×k是第1层的权重矩阵的输入,F1∈R(N+M)×k是图卷积网络的药物和lncRNA节点的第一层映射低维特征向量,k是低维特征的维度,F0=F是初始输入特征。在I次迭代之后,本研究可以从不同的图卷积层获得I个k维特征,这些图卷积层的计算遵循公式,其权重矩阵为Wi∈Rk×k,i= 2, 3。
不同层的低维特征捕获了图网络中不同结构的信息。第一层获取直接邻居信息,而更高层通过迭代更新低维特征来捕获多阶邻居信息。考虑到不同层的低维特征对最终特征的贡献是不一致的,本研究引入注意力机制来组合这些低维特征,并获得药物和lncRNA的最终特征:
| $ H=\left[\begin{array}{l} H_d \\ H_l \end{array}\right]=\sum a_i F_i $ | (7) |
其中,Hd∈RN×k是药物的最终特征,Hl∈RM×k是lncRNA的最终特征,ai是可学习参数并初始化为1/(i+1),i = 1, 2, 3。
1.2.3 归纳矩阵补全解码如图 1(c)所示,本研究利用一个解码器来重构药物-lncRNA关系矩阵A′进行链路预测。矩阵补全可以视为矩阵形式的链路预测,其目的是为了预测矩阵中缺失的部分,用矩阵A′近似矩阵A,然后用A′中的元素作为矩阵A中不可观察部分的元素的估计。
而由于图卷积网络中节点之间和节点自身特征的稀疏性,导致即使多次迭代之后,每个节点的最终低维特征还是比较稀疏,没有太多可用信息。大部分图深度学习链路预测只是对得到的节点特征进行数量积计算,这与传统的矩阵分解一样,但特征信息质量低甚至不可获取时,这种方法就无法准确预测缺失项。
基于上述的矩阵补全方法易受到关联矩阵的极端稀疏性和冷启动问题的影响,为了解决这一问题,本研究采用一种归纳矩阵补全方法设计了一个解码器,该方法已被用于预测基因与疾病的关联[21]。归纳矩阵补全是通过将药物和靶点特征向量与一个低秩矩阵XYT[22]相乘来生成关联矩阵。解码器的公式可以表示为
| $ A^{\prime}=sigmoid\left(H_d X Y^{\mathrm{T}} H_l^{\mathrm{T}}\right) $ | (8) |
其中,sigmoid表示sigmoid函数,X∈Rk×b和Y∈Rk×b为可训练参数,T表示矩阵的转置,且b≪k。
1.2.4 模型训练优化本研究把Aij = 1的药物-lncRNA关系作为正样本训练集,把Aij = 0的药物-lncRNA关系作为负样本训练集。然而,训练时负样本训练集是有噪声的,因为本文只有验证的药物-lncRNA调控关系,却没有明确证实不存在调控的关系,不能因为调控关系没有实验验证就全部认为是负样本。因此,为解决这个问题,本研究参考正样本和无标记样本学习(Positive and unlabled learning)里正样本和无标记样本在损失函数中受到不同的惩罚[23],采用加权交叉熵作为损失函数,如公式(9)和(10)所示:
| $ loss =-\left(\frac{\lambda}{N \times M} \times \sum\limits_{A_{i j=1}} \log A_{i j}^{\prime}+\frac{1}{N \times M} \times \sum\limits_{A_{i j=0}} \log \left(1-A_{i j}^{\prime}\right)\right) $ | (9) |
| $ \lambda=\frac{\left|A_{i j}=0\right|}{\left|A_{i j}=1\right|} $ | (10) |
其中,Aij’代表预测矩阵A’中对应的药物i与lncRNA j两者之间存在关联的可能性,越接近1时可能性越大,反之越小,|Aij = 1|和|Aij = 0|分别表示正样本和无标记样本药物-lncRNA关系的个数,权重因子λ强调了正样本的重要性,以减少无标记样本的影响。
所有可训练权重矩阵Wi都通过Xaiver方法[24]初始化,接着,本文使用Adam优化器来最小化损失函数,Adam优化器可以基于训练数据迭代更新神经网络的权重。为了防止过度拟合,本文在图卷积层中引入了节点dropout和特征dropout。此外,本文在优化时使用循环学习率[25],循环学习率使学习率(Learning rate, lr)在最大学习率和最小学习率之间发生变化,帮助平衡训练速度。
1.3 开发运行环境图卷积网络是基于Python3.7和TensorFlow1.15编写的,集成开发环境为Rycharm,操作系统为Windows10,处理器为Intel Core i5-7300HQ,图形处理器为NVIDIA GeForce GTX 1050 Ti。
2 结果 2.1 训练设置采用留一法交叉验证评估模型的预测性能,所有1 750个药物-lncRNA关系在1 750次训练时依次抽取一个作为测试集,剩下的全部作为训练集。每次训练中,基于训练集中的已知关系构建预测模型,把测试集中的关系对应的Aij改成0,并用于预测测试集中的关系。用AUROC作为效能评估的主要指标。
DLGCN中超参数主要有低维特征维度k,低秩矩阵维数b,优化器的初始学习率lr,总训练时间α,节点dropout和特征dropout率β和γ以及惩罚因子μ, 考虑这些参数在k∈(16, 32, 64),b∈(4, 8, 16),lr∈(0.001, 0.005, 0.01, 0.4),α∈(1 000, 2 000, 4 000),β, γ∈(0.1, 0.2, 0.3, 0.4)和μ∈(0.5, 1, 1.5, 2, 2.5)范围内的不同组合。根据模型达到最佳指标时参数的组合,在以下实验中为图卷积网络设置k = 64,b = 16,lr = 0.001,α= 4 000,β= 0.1,γ= 0.1和μ= 1.5。
2.2 图卷积层数和注意力机制的影响本文研究了图卷积层数对模型预测性能的影响。图卷积网络的第i卷积层捕获i阶邻居的信息,第3卷积层低维特征往往包含比第1和第2卷积层低维特征更多的信息。DLGCN-1, DLGCN-2, DLGCN, DLGCN-4分别代表卷积层数为1, 2, 3, 4的带注意力机制的图卷积网络模型的ROC曲线。从图 2(a)中可以看出,AUROC在3层时达到最高为0.843 1。这个结果可能是由于图卷积的目的是使相邻节点更接近,而增加太多层数导致节点特征收敛到一个点,引起过度拟合。同时,层数的增加也导致模型参数的数量和训练时间的逐渐增加。因此,当图卷积层的数量设置为3时,可以获得更好的模型预测性能。
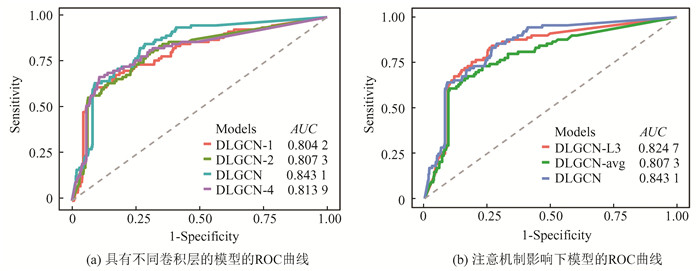
|
图 2 图卷积层数和注意力机制的影响 Figure 2 Influence of convolution layers and attention mechanism |
本文还研究了注意力机制对模型预测性能的影响。DLGCN-L3, DLGCN-avg, DLGCN分别代表卷积层数为3时只用第3层低维特征、3层低维特征算术平均、3层低维特征带注意力机制的图卷积网络模型。如图 2(b)所示,DLGCN比DLGCN-L3和DLGCN-avg产生更好的结果,表明注意力机制可识别并量化不同卷积层的重要性,提取重要卷积层的特征信息。
2.3 方法比较为进一步评估模型的效能,本研究比较了DLGCN和经典机器学习模型的性能,例如:支持向量机(SVM)、随机森林(RF)、弹性网(ENet)和K近邻(KNN)。本文把每个关系对作为一个样本,对于关系对中的药物i与lncRNA j,提取药物相似性矩阵Sd的第i行和lncRNA相似性矩阵Sl的第j行,组成N+M维特征作为关系对的特征来进行训练。本文使用Python sklearn库来实现SVM, RF, ENet, KNN算法,其中,SVM的惩罚系数c = 1,核函数选择高斯核;KNN中k = 5;随机森林的树的数目包括{120, 200, 500},树的最大深度包括{5, 10, 25, 40};弹性网中α包括{0.01, 0.05, 0.1, 0.5, 1, 5, 10},l1_ratio包括{0.01, 0.05, 0.1, 0.5, 1},随机森林和弹性网都用网格搜索法确定最佳参数。图 3(a)展示了SVM、RF、ENet、KNN的ROC曲线。可以看出,DLGCN的预测效果明显比机器学习模型更好,相比于表现最好的机器学习模型,DLGCN的AUROC值提高了0.101 6。
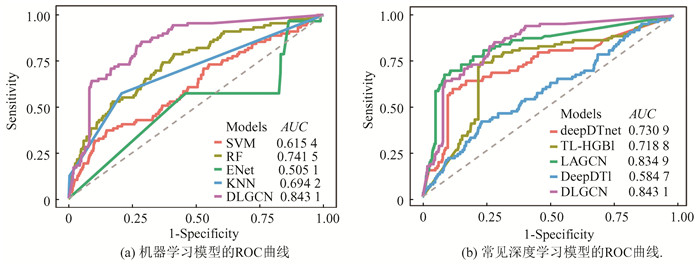
|
图 3 方法比较 Figure 3 Methods comparison |
此外,本研究将DLGCN与常见深度学习模型的预测性能进行比较,根据公开的程序代码,利用本文的数据进行预测效果评估。deepDTnet、TL-HGBI,DeepDTI和LAGCN的特征都为药物相似性矩阵,lncRNA相似性矩阵和药物与lncRNA的关联矩阵。deepDTnet的低维特征维度为64,低秩矩阵维度为16;DeepDTI舍弃了卷积层,组合特征用3层深度神经网络输出预测结果,隐藏层节点数为{84, 16, 16},dropout = 0.1;LAGCN的低维特征维度为64,惩罚系数μ=1.5,dropout = 0.1。所有方法的性能如图 3(b)所示,从中可以发现,DLGCN优于所有参与比较的方法,相比于表现最好的深度学习模型,DLGCN的AUROC值提高了0.008 2。与只基于归纳矩阵补全的deepDTnet和网络传播的TL-HGBI方法相比性能更好,DeepDTI的预测效果相对最差,LAGCN与DLGCN表现差不多,这大概因为LAGCN采用了类似的解码方法,而DLGCN的解码能力更好。
2.4 预测的影响lncRNA表达的药物D-lnc数据库记录了2019年7月以前实验验证的药物-lncRNA调控关系,因而本研究从PubMed数据库里检索并整理了2019年7月至2022年3月最新的药物干扰非编码RNA的文献,筛选出其中药物-lncRNA的关联信息。本研究还对整理的35种药物、50种lncRNA和89条实验证实的药物调控lncRNA表达的关联关系进行不抽样训练。其输入输出、训练的数据集合和参数设置与前面一致,输入包括35种药物的相似性矩阵、50种lncRNA的相似性矩阵和药物与lncRNA的关联矩阵,输出为重构的关联得分矩阵,得分越高表明预测的关系越可信。如图 4所示,本研究选取得分最高的Top 60药物-lncRNA关系对,Top 60药物-lncRNA关系对中共包含22种药物和26种lncRNA。其中,被DLGCN预测出的两个姜黄素(Curcumin)调控lncRNA的关系(Curcumin-MALAT1和Curcumin-HOTAIR)在最近的研究中得到验证。Yoshida等[26]人通过实时定量PCR(Quantitative RT-PCR, qRT-PCR)和细胞存活率分析(MTT assay)分别测定姜黄素用药前后胰腺癌细胞系Panc1和BxPC3中MALAT1的表达和细胞活性以及细胞系异种移植(Cell derived xenograft, CDX)小鼠模型中肿瘤的大小,发现姜黄素在Panc1细胞系中上调MALAT1,下调HOTAIR和PVT1的表达,吉西他滨耐药细胞系BxPC3的MTT assay结果和无胸腺裸鼠的CDX模型证明姜黄素在体内和体外都能增强BxPC3细胞对吉西他滨的敏感性。
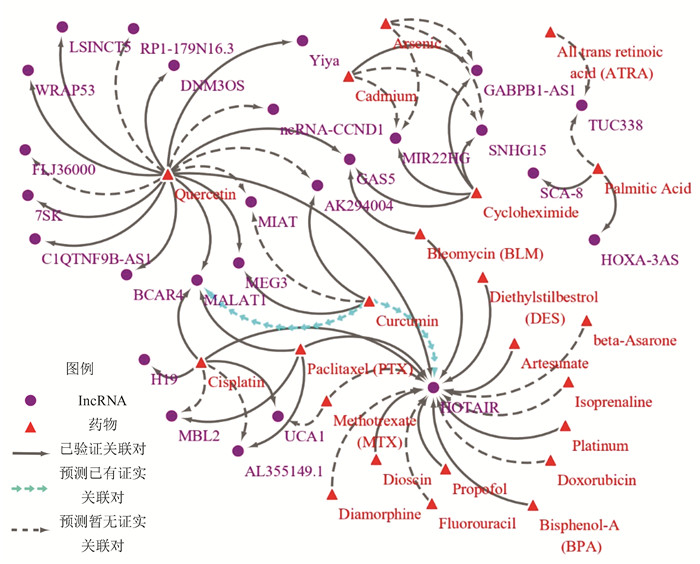
|
图 4 预测得分Top 60的药物-lncRNA关系对 Figure 4 Top 60 predicted drug-lncRNA associations |
此外,本研究也通过一些间接证据验证了预测结果,例如:全反式维甲酸(All-trans retinoic acid, ATRA)与lncRNA TUC338的关联以及多柔比星(Doxorubicin)与lncRNA HOTAIR的关联。ATRA是维生素A的活性代谢产物,在细胞增殖、细胞分化、凋亡和胚胎发育中起着重要作用。体外或体内研究表明ATRA对急性早幼粒细胞白血病、肺癌,乳腺癌和宫颈癌具有治疗作用[27]。Zhang等[28]人发现TUC338在肺癌组织中的表达高于癌旁组织,而TUC338过度表达后,肺癌细胞活性以及侵袭能力增加,表明TUC338可以促进肺癌的发展。因此,ATRA可能通过抑制TUC338的表达发挥抗癌作用。Sun等[29]人研究发现HOTAIR能显著下调miR-15b和p53的表达,进而抑制肿瘤细胞增殖、侵袭,促进肿瘤细胞凋亡。多柔比星等蒽环类药物可以诱导肿瘤细胞凋亡,因此,多柔比星-HOTAIR的关联可能是一种潜在的药物作用机制。但是,这些假设还需要进一步的生物学实验进行验证。
2.5 数据可用性DLGCN的代码以及需要的输入数据已上传至https://github.com/pengpengzjc/DLGCN,TL-HGBI的代码已上传至https://github.com/pengpengzjc/DLGCN/tree/main/TL-HGBI;而deepDTnet、DeepDTI和LAGCN的代码可在https://github.com/ChengF-Lab/deepDTnet、https://github.com/hkmztrk/DeepDTA和https://github.com/storyandwine/LAGCN中获得。
3 讨论为了准确识别潜在的药物-lncRNA关系对,本文利用图卷积网络、自编码和归纳矩阵补全技术,提出了一个图卷积网络模型DLGCN,该模型从药物-lncRNA异质网络学习低维特征来预测药物和lncRNA之间相互作用关系。带有注意力机制的图卷积网络可以有效从异质网络中学习到特征信息和网络的结构信息,并且把整合的信息编码映射到低维空间中。此外,为了防止异质网络中度很小的节点低维特征质量过低影响预测性能,DLGCN采用归纳矩阵补全方法来提高关系矩阵中缺失项预测的准确性。通过效能评估,DLGCN的AUROC结果优于其他的经典机器学习和常用深度学习方法,且预测的一些潜在的药物-lncRNA关系对已被最近的研究证实。因此,DLGCN能有效预测潜在的药物-lncRNA关联关系,将为肿瘤治疗新靶点的识别和抗癌药物的筛选提供重要参考。
DLGCN的方法也存在一定的局限性。首先,为了保证训练数据的可靠性,本研究仅保留了低通量实验验证的药物与lncRNA的关联,共89条关系对,涉及35种药物和50个lncRNA。接下来,在当前的模型中,网络节点获取邻域特征时,所有邻域节点信息对该节点的贡献相同,实际上节点和邻域节点的关联程度也是不完全一样的,需要考虑为不同的邻域节点分配不同的权重。此外,模型目前只是预测药物-lncRNA关系存在的可能性,对于关系对中具体药物是上调还是下调lncRNA不能判断。最后,该方法只能预测网络中的药物和lncRNA之间潜在的新的关联关系,不能预测网络中没有的药物或lncRNA的关联,因为加入新节点后,图矩阵G和特征矩阵F也需要更新,同时节点的低维特征也会有所改变,原模型的权重参数不能直接用于预测新的重构关联矩阵,需要再一次地训练网络。在今后的工作中,我们将进一步研究图注意力网络、知识图谱和动态图问题的解决方案,这也将有助于识别lncRNA靶点治疗肿瘤和筛选抗癌药物。
4 结论1) 本文利用图卷积网络、自编码和归纳矩阵补全技术,提出了一个图卷积网络模型DLGCN来识别潜在的药物-lncRNA关系对。
2) 通过效能评估,DLGCN的预测结果优于其他的经典机器学习和常用深度学习方法,且预测的一些潜在的药物-lncRNA关系对已被最近的研究证实。
3) DLGCN将为肿瘤治疗新靶点的识别和抗癌药物的筛选提供重要参考。
| [1] |
BARDHAN A, BANERJEE A, BASU K, et al. PRNCR1: A long non-coding RNA with a pivotal oncogenic role in cancer[J]. Human Genetics, 2022, 141(1): 15-29. DOI:10.1007/s00439-021-02396-8 (  0) 0) |
| [2] |
LIN R, MAEDA S, LIU C, et al. A large noncoding RNA is a marker for murine hepatocellular carcinomas and a spectrum of human carcinomas[J]. Oncogene, 2007, 26(6): 851-858. DOI:10.1038/sj.onc.1209846 (  0) 0) |
| [3] |
REIS E M, NAKAYA H I, LOURO R, et al. Antisense intronic non-coding RNA levels correlate to the degree of tumor differentiation in prostate cancer[J]. Oncogene, 2004, 23(39): 6684-6692. DOI:10.1038/sj.onc.1207880 (  0) 0) |
| [4] |
JIN Chuan, YAN Bingchuan, LU Qin, et al. Reciprocal regulation of Hsa-miR-1 and long noncoding RNA MALAT1 promotes triple-negative breast cancer development[J]. Tumour Biology, 2016, 37(6): 7383-7394. DOI:10.1007/s13277-015-4605-6 (  0) 0) |
| [5] |
KONISHI H, ICHIKAWA D, YAMAMOTO Y, et al. Plasma level of metastasis-associated lung adenocarcinoma transcript 1 is associated with liver damage and predicts development of hepatocellular carcinoma[J]. Cancer Sciences, 2016, 107(2): 149-154. DOI:10.1111/cas.12854 (  0) 0) |
| [6] |
MERCATELLI N, FORTINI D, PALOMBO R, et al. Small molecule inhibition of Ewing sarcoma cell growth via targeting the long non coding RNA HULC[J]. Cancer Letters, 2020, 469: 111-123. DOI:10.1016/j.canlet.2019.10.026 (  0) 0) |
| [7] |
WANG Wenhui, YANG Sen, ZHANG Xiang, et al. Drug repositioning by integrating target information through a heterogeneous network model[J]. Bioinformatics, 2014, 30(20): 2923-2930. DOI:10.1093/bioinformatics/btu403 (  0) 0) |
| [8] |
ZHONG Jiancheng, ZHOU Wubin, KANG Jiedong, et al. DNRLCNN: A cnn framework for identifying mirna-disease associations using latent feature matrix extraction with positive samples[J]. Interdiscip Science, 2022, 14(2): 607-622. DOI:10.1007/s12539-022-00509-z (  0) 0) |
| [9] |
ZENG Xiangxiang, ZHU Siyi, LU Weiqiang, et al. Target identification among known drugs by deep learning from heterogeneous networks[J]. Chemical Science, 2020, 11(7): 1775-1797. DOI:10.1039/c9sc04336e (  0) 0) |
| [10] |
COSKUN M, KOYUTURK M. Node similarity based graph convolution for link prediction in biological networks[J]. Bioinformatics, 2021, 37(23): 4501-4508. DOI:10.1093/bioinformatics/btab464 (  0) 0) |
| [11] |
TIAN Qiyuan, BILGIC B, FAN Qiuyun, et al. DeepDTI: High-fidelity six-direction diffusion tensor imaging using deep learning[J]. Neuroimage, 2020, 219: 117017-15. DOI:10.1016/j.neuroimage.2020.117017 (  0) 0) |
| [12] |
YU Zhouxin, HUANG Feng, ZHAO Xiaohan, et al. Predicting drug-disease associations through layer attention graph convolutional network[J]. Briefings in Bioinformatics, 2021, 22(4): 1-11. DOI:10.1093/bib/bbaa243 (  0) 0) |
| [13] |
ZHAO Tianyi, HU Yang, VALSDOTTIR L R, et al. Identifying drug-target interactions based on graph convolutional network and deep neural network[J]. Briefings in Bioinformatics, 2020, 22(2): 2141-2150. DOI:10.1093/bib/bbaa044 (  0) 0) |
| [14] |
JAIN P, DHILLON I S. Provable inductive matrix completion[J/OL]. arXiv preprint, 2013. https://arxiv.org/abs/1306.0626. 2013-06-04. DOI: 10.48550/arXiv.1306.0626.
(  0) 0) |
| [15] |
JIANG W, QU Y, YANG Q, et al. D-lnc: A comprehensive database and analytical platform to dissect the modification of drugs on lncRNA expression[J]. RNA Biology, 2019, 16(11): 1586-1591. DOI:10.1080/15476286.2019.1649584 (  0) 0) |
| [16] |
WISHART D S, FEUNANG Y D, GUO A C, et al. DrugBank 5.0: A major update to the DrugBank database for 2018[J]. Nucleic Acids Research, 2018, 46(D1): D1074-D1082. DOI:10.1093/nar/gkx1037 (  0) 0) |
| [17] |
KIM S, CHEN J, CHENG T, et al. PubChem in 2021: New data content and improved web interfaces[J]. Nucleic Acids Research, 2021, 49(D1): D1388-D1395. DOI:10.1093/nar/gkaa971 (  0) 0) |
| [18] |
FRANKISH A, DIEKHANS M, JUNGREIS I, et al. Gencode 2021[J]. Nucleic Acids Research, 2021, 49(D1): D916-D923. DOI:10.1093/nar/gkaa1087 (  0) 0) |
| [19] |
COMPEAU P E, PEVZNER P A, TESLER G. How to apply de Bruijn graphs to genome assembly[J]. Nature Biotechnology, 2011, 29(11): 987-991. DOI:10.1038/nbt.2023 (  0) 0) |
| [20] |
SHAO Kanghao, ZHANG Yunhao, WEN Yuqi, et al. DTI-HETA: Prediction of drug-target interactions based on GCN and GAT on heterogeneous graph[J]. Briefings in Bioinformatics, 2022, 23(3): 1-16. DOI:10.1093/bib/bbac109 (  0) 0) |
| [21] |
NATARAJAN N, DHILLON I S. Inductive matrix completion for predicting gene-disease associations[J]. Bioinformatics, 2014, 30(12): i60-i68. DOI:10.1093/bioinformatics/btu269 (  0) 0) |
| [22] |
YU H F, JAIN P, KAR P, et al. Large-scale multi-label learning with missing labels[A]. Proceedings of the International conference on machine learning, F, 2014[C]. 2014, 32: 593-601. DOI: 10.48550/arXiv.1307.5101.
(  0) 0) |
| [23] |
HSIEH C J, NATARAJAN N, DHILLON I S. PU learning for matrix completion[A]. Proceedings of the 32nd international conference on international conference on machine learning[C]. 2015: 2445-2453. DOI: 10.48550/arXiv.1411.6081.
(  0) 0) |
| [24] |
GLOROT X, BENGIO Y. Understanding the difficulty of training deep feedforward neural networks[J]. Journal of Machine Learning Research - Proceedings Track, 2010, 9: 249-256. (  0) 0) |
| [25] |
SMITH L N. Cyclical learning rates for training neural networks[A]. 2017 IEEE winter conference on applications of computer vision (WACV)[C]. 2017, 464-472. DOI: 10.1109/WACV.2017.58.
(  0) 0) |
| [26] |
YOSHIDA K, TODEN S, RAVINDRANATHAN P, et al. Curcumin sensitizes pancreatic cancer cells to gemcitabine by attenuating PRC2 subunit EZH2, and the lncRNA PVT1 expression[J]. Carcinogenesis, 2017, 38(10): 1036-1046. DOI:10.1093/carcin/bgx065 (  0) 0) |
| [27] |
NI Xiaoling, HU Guohua, CAI Xun. The success and the challenge of all-trans retinoic acid in the treatment of cancer[J]. Critical Reviews in Food Science and Nutrition, 2018, 59(3): 1-10. DOI:10.1080/10408398.2018.1509201 (  0) 0) |
| [28] |
ZHANG Y X, YUAN J, GAO Z M, et al. LncRNA TUC338 promotes invasion of lung cancer by activating MAPK pathway[J]. European Review for Medical and Pharmacological Sciences, 2018, 22(2): 443-449. DOI:10.26355/eurrev_201801_14193 (  0) 0) |
| [29] |
SUN Guan, WANG Yingyi, ZHANG Junxia, et al. MiR-15b/HOTAIR/p53 form a regulatory loop that affects the growth of glioma cells[J]. Journal of Cellular Biochemistry, 2018, 119(6): 4540-4547. DOI:10.1002/jcb.26591 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


