2. 沈阳医学院 职业卫生科,沈阳 110000
2. Department of Occupational Health, Shenyang Medical College, Shenyang 110000, China
自然界中砷分布广泛,在土壤、水和植物中都能检测出微量的砷[1]。砷可以通过饮水、食物等方式进入人体 [2]。研究发现,过量的砷暴露能够引起人体多种脏器的损伤。2017年世界卫生组织国际癌症研究机构将砷及其无机砷化合物列为一类致癌物 [3]。斑马鱼作为脊椎动物模型,与人类基因高度同源,其实验结果大多数情况下适用于人体[4]。斑马鱼的肝脏对各种毒物极其敏感,Wolfram Goessling等人研究发现,斑马鱼胚胎发育迅速,其所有的消化器官在5天时发育成熟,因此斑马鱼是肝病研究的重要工具[5-6]。因此肝脏疾病的研究中斑马鱼是重要的脊椎动物模型。美国国家卫生研究院将斑马鱼列为继大鼠和小鼠之后的第三大脊椎类模型生物,其信号传导通路与人类基本相似,在环境毒物等领域研究广泛[7]。通过运用生物信息学方法可以有助于进一步研究砷暴露对机体危害的潜在机制。
1 数据来源与方法 1.1 搜索斑马鱼和砷微阵列数据信息浏览NCBI网站(https://www.ncbi.nlm.nih.gov/),在GEO DataSets搜索框中输入斑马鱼和砷;通过搜索获得了砷处理斑马鱼的肝脏组织和斑马鱼正常肝脏组织GSE30062和GSE3048的基因表达谱[8-9]。GSE30062和GSE3048的微阵列数据均基于GPL2715平台,因此能够进行共同分析研究。其中GSE30062基因表达谱包括14个砷处理肝脏组织和10个正常肝脏组织;GSE3048的基因表达谱包括12个砷处理肝脏组织和12个正常肝脏组织。
1.2 Database of Essential Genes(DEG)的数据处理通过GEO2R在线工具筛选出GSE30062和GSE3048的基因表达谱中的差异基因。然后,在线维恩软件分析原始数据,以| logFC | >1为依据,筛选出两个数据集中的高度差异基因。(其中log FC < 0的DEG被认为是下调基因,而log FC> 0的DEG被认为是上调基因)。
1.3 通过DAVID数据库对179个交集基因进行基因本体论和KEGG途径富集分析用DAVID在线生物信息学工具(https://david.ncifcrf.gov)来分析差异基因的细胞组分(Cellular components CC)、分子功能(Molecular function MF)、生物过程(Biological process BP)和KEGG通路分析[10]。
1.4 PPI网络和模块分析筛选核心基因PPI信息可以通过在线工具STRING(用于检索相互作用基因的搜索工具)进行评估。然后,应用Cytoscape中的STRING应用程序检查这些DEGs之间的潜在相关性(最大相互作用数= 0和置信度得分≥0.4)。此外,通过Cytoscape中的MCODE应用程序用于检查PPI网络的模块(度数截止= 2,最大深度= 100,k-core = 2,节点得分截止= 0.2),运用CytoHubba应用程序模块发现复杂网络的关键基因。
1.5 10个核心基因重新进行基因本体论(Gene Ontology,GO)和KEGG途径富集分析运用蛋白质-蛋白质相互作用网络(PPI)中MCOD模块和Cytohubba模块分析获得的关键基因的交集;共10个基因分别为:PSMB3、HSP701、HSPE1、STIP1、HSPD1、HSP70、DNAJB1B、HSP90AA1.1、HSPA9H和TCP1。将筛选出的10个核心基因用DAVID在线生物信息学工具(https://david.ncifcrf.gov)来分析差异基因的细胞组分、分子功能、生物过程和KEGG通路分析。
1.6 10个核心基因在肝癌中的预后生存分析Kaplan Meier绘图仪能够评估基因对肝癌等21种癌症类型的生存率的影响。该工具可以发现和验证生存生物标志物。打开Kaplan Meier-plotter(http://kmplot.com/analysis/index.php?p=background)网站,点击start KM Plotter for liver cancer选项,将要分析的基因输入Gene symbol中,最后点击Draw Kaplan-Meier plot选项[11]。
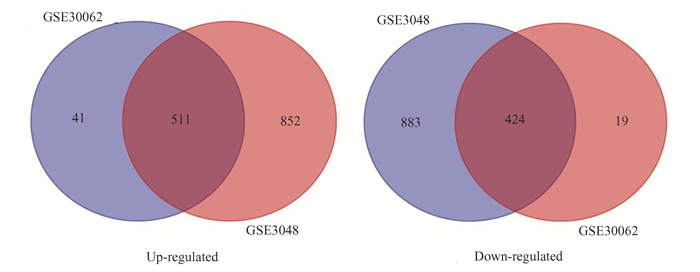
2 结果分析 2.1 筛选肝脏组织中的DEGs研究中有26个砷处理肝脏组织和22个正常肝脏组织。通过GEO2R在线工具,分别从GSE3048和GSE30062中提取了2 670和991个基因(图 1)。然后,使用维恩图软件来识别两个数据集中的常见差异基因。两个数据集的交集中有511个基因被上调(logFC> 0);有424个基因被下调(logFC < 0)(图 2)。以| logFC |>1为筛选条件,进一步筛选有显著差异的基因,上调的511个基因中有169个基因符合要求(logFC> 1);下调的424个基因中有10个基因符合要求(logFC < -1)(表 1)。

|
图 1 差异表达基因的火山图 Figure 1 Volcano diagram of DEGs |
|
图 2 差异表达基因的韦恩图 Figure 2 Venn diagram of DEGs |
| 表 1 差异表达基因 Table 1 Diagram of DEGs |
在上调的差异基因中:1)对于生物过程(BP),差异基因特别富集于蛋白质折叠、参与细胞蛋白质分解代谢过程的蛋白水解、蛋白质转运、谷胱甘肽生物合成过程等(表 2);2)对于分子功能(MF),差异基因富集于苏氨酸型内肽酶活性、内肽酶活性、ATP结合、细胞质部分、谷胱甘肽转移酶活性、肽酶活性等(表 2);3)对于GO细胞成分(CC),差异基因富集于蛋白酶体复合物、细胞质、谷氨酸-半胱氨酸连接酶复合物、内质网等(表 2)。
| 表 2 差异表达基因的基因GO分析 Table 2 Gene ontology analysis of DEGs |
在下调的差异基因中:1) 对于生物过程(BP),差异基因特别富集于细胞对异物刺激的反应、类固醇激素介导的信号通路、转录,DNA模板化等(表 2);2) 对于GO细胞成分(CC),差异基因富集于金属离子结合、氧化还原酶活性、类固醇激素受体活性、单氧化酶活性等(表 2)。
2.2.2 KEGG通路分析KEGG分析结果显示,差异基因主要富集在蛋白酶体,吞噬体,内质网中的蛋白质加工,谷胱甘肽代谢,细胞色素P450对异生物质的代谢以及氨基酸的生物合成通路(P < 0.05)(表 3);
| 表 3 差异表达基因的KEGG通路分析 Table 3 KEGG pathway analysis of DEGs |
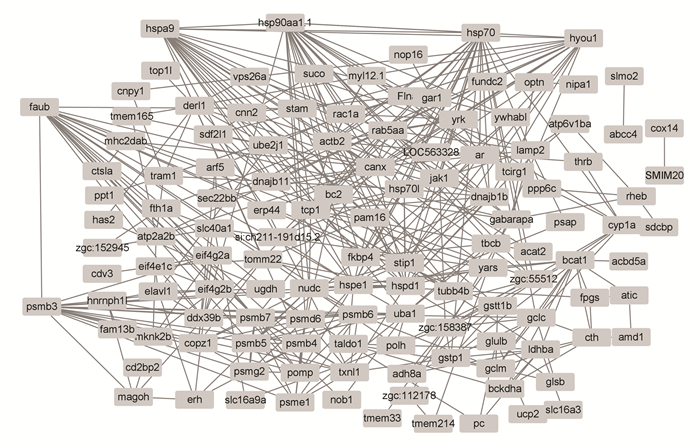
结果显示:共有126个基因入选到PPI网络综合体中,其中包括120个上调基因和6个下调基因(图 3);MCOD模块分析和CytoHubba模块分析结果取交集共有10个基因入选核心基因分别为:PSMB3、HSP701、HSPE1、STIP1、HSPD1、HSP70、DNAJB1B、HSP90AA1.1、HSPA9和TCP1(表 4)。
|
图 3 蛋白质-蛋白质相互作用网络 Figure 3 Protein-Protein interaction network |
| 表 4 MCOD模块和Cytohubba模块分析 Table 4 MCOD module and Cytohubba module analysis |
蛋白质-蛋白质相互作用网络(PPI)中MCOD模块和Cytohubba模块分析获得的关键基因的交集共10个基因分别为:PSMB3、HSP701、HSPE1、STIP1、HSPD1、HSP70、DNAJB1B、HSP90AA1.1、HSPA9和TCP1。对这10个基因从新进行GO分析(表 5)和KEGG分析(表 6)
| 表 5 核心基因GO分析 Table 5 GO analysis of core genes |
| 表 6 核心基因KEGG分析 Table 6 KEGG pathway analysis of core genes s |
生物过程(BP):核心基因特别富集于蛋白质折叠、对金属离子的反应、鳍再生见;
分子功能(MF):核心基因富集于线粒体基质和细胞质;
GO细胞成分(CC):核心基因富集于未折叠的蛋白质结合、ATP的结合和核苷酸的结合。
2.4.2 KEGG通路分析通路分析结果显示核心基因富集于内质网中的蛋白质加工通路,DNAJB1、HSP90AA1.1、HSP70L靶基因对内质网中的蛋白质加工通路的作用靶点如图 4所示。

|
图 4 内质网中的蛋白质加工通路 Figure 4 Protein processing in endoplasmic reticulum |
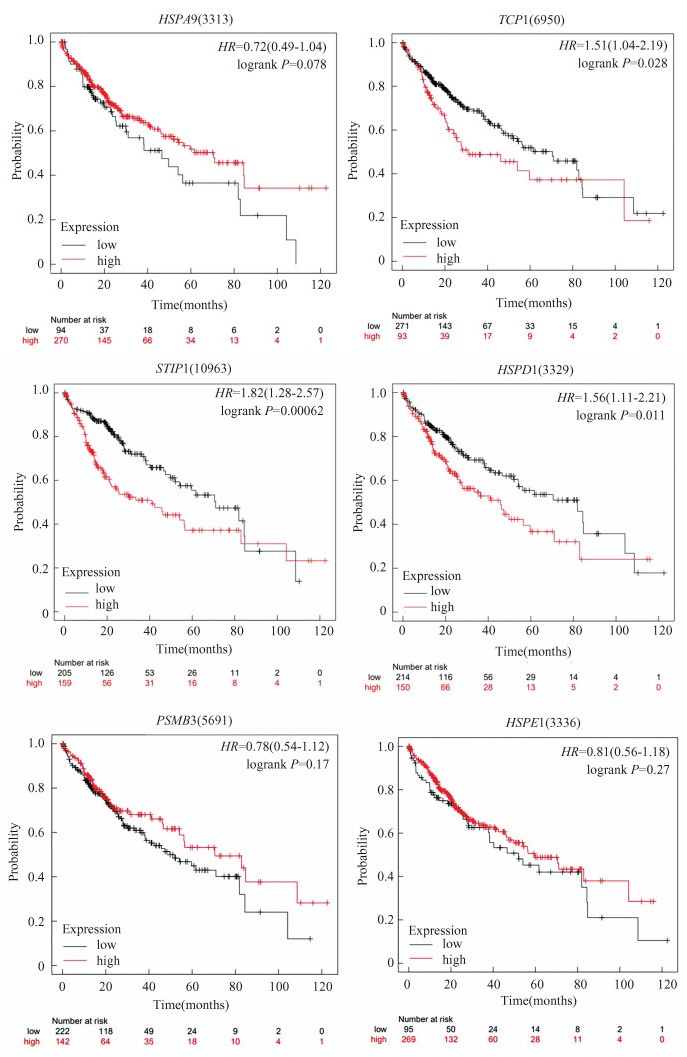
流行病学的研究结果表明,长期接触砷与肝癌的发生密切相关,对PSMB3、HSP701、HSPE1、STIP1、HSPD1、HSP70、DNAJB1B、HSP90AA1.1、HSPA9和TCP1基因的在肝癌中的预后生存分析,在10个基因中只查找出有6个基因(PSMB3、HSPE1、STIP1、HSPD1、HSPA9和TCP1)对肝癌患者的预后生存有影响,结果显示:TCP1、STIP1和HSPD1会降低肝癌的预后生存时间(P<0.05)(图 5)。
|
图 5 核心基因在肝癌患者中的预后生存分析 Figure 5 Prognostic survival analysis of core genes in patients with liver cancer |
砷暴露会对机体产生危害,研究发现砷的致病机理与氧化应激和免疫功能的损伤有关,引起皮肤、肝脏、肾脏、神经系统等损伤[12-13]。肝脏被认为是砷生物转化和甲基化的主要器官,也是砷毒性的主要靶标[14];在斑马鱼中,长时间过量的砷暴露会改变肝脏细胞中细胞周期和脂质代谢基因的表达[15];在蛋白质组学研究中,砷暴露后会改变脂质转运和代谢途径中的蛋白质表达[16]。
研究结果显示斑马鱼在急性砷暴露后会引起多种基因的改变,从GSE3048和GSE30062中提取了2 670和991个基因,经过进一步的筛选,得到179个符合条件的差异基因,这些上调的差异基因特别富集于蛋白质折叠、参与细胞蛋白质分解代谢过程的蛋白水解、蛋白质转运、谷胱甘肽生物合成过程等;差异基因富集于苏氨酸型内肽酶活性、内肽酶活性、ATP结合、细胞质部分、谷胱甘肽转移酶活性、肽酶活性等;差异基因富集于蛋白酶体复合物,细胞质、谷氨酸-半胱氨酸连接酶复合物、内质网等;下调的差异基因中,差异基因特别富集于细胞对异物刺激的反应、类固醇激素介导的信号通路、转录,DNA模板化等;差异基因富集于金属离子结合,氧化还原酶活性、类固醇激素受体活性、单氧化酶活性等。在KEGG通路分析后显示差异基因特别富集于蛋白酶体,吞噬体、谷胱甘肽代谢、细胞色素P450对异生物质的代谢通路。将176个基因用Cytoscape软件进行蛋白质- 蛋白质相互作用网络(PPI)和模块分析发现共有126个基因作用于蛋白靶点,其中有10个基因为关键基因分别为:PSMB3、HSP701、HSPE1、STIP1、HSPD1、HSP70、DNAJB1B、HSP90AA1.1、HSPA9和TCP1。在肾细胞癌和肺癌患者中PSMB3基因的高表达对应更差的预后,其机制可能是PSMB3基因沉默剪接体Sm基因的表达[17-18]。10个关键基因在肝癌中的预后生存分析发现:TCP1、STIP1和HSPD1会降低肝癌的预后生存时间。
研究表明,热休克蛋白(HSP)是一类高度保守的保护性蛋白,热休克蛋白在蛋白质分子结合过程中发挥作用,维持氨基酸链的三维结构、清除折叠错误的氨基酸链、减轻对机体的损伤[19]。通过生物信息学分析结果发现在10个核心基因的KEGG通路分析结果涉及到dre04141:内质网中的蛋白质加工通路,该通路在蛋白质的生产过程中扮演重要角色,与我们预测到的热休克蛋白的功能一致;内质网(ER)是一种亚细胞器,帮助蛋白质进行正确的折叠,将正确折叠的蛋白质被包装成运输囊泡,对错误折叠的蛋白质通过蛋白酶体定向降解,同时过量的错误折叠的蛋白质会也会引起细胞死亡[20]。在此次的研究发现的10个核心基因中,有HSP701、HSPE1、HSPD1、HSP70、HSP90AA1.1和HSPA9共6个基因属于热休克蛋白家族,已有研究表明亚砷酸钠作为热休克蛋白的诱导剂[21],该结果和我们此次生物信息学预测分析的结果一致。
4 结论1) 在砷的机制研究中HSP701、HSPE1、HSPD1、HSP70、HSP90AA1.1和HSPA9等可能会是其新的研究方向;内质网中的蛋白质加工通路以及热休克蛋白可能成为砷引起的肝损伤以及肝癌研究新的方向。
2) PSMB3、HSP701、HSPE1、STIP1、HSPD1、HSP70、DNAJB1B、HSP90AA1.1、HSPA9和TCP1这十个基因可能在砷的毒性损伤进展中起关键作用。未来需要通过一系列实验来验证,这些数据可能会为砷对肝脏损伤的潜在生物标志物和生物学机制提供一些有用的信息。
| [1] |
MIR K A, RUTTER A, KOCH I, et al. Extraction and speciation of arsenic in plants grown on arsenic contaminated soils[J]. Talanta, 2007, 72(4): 1507-1518. DOI:10.1016/j.talanta.2007.01.068 (  0) 0) |
| [2] |
ABDUL K, JAYASINGHE S S, CHANDANA E, et al. Arsenic and human health effects: A review[J]. Environmental Toxicology & Pharmacology, 2015, 40(3): 828-846. DOI:10.1016/j.etap.2015.09.016 (  0) 0) |
| [3] |
PALMA-LARA I, MARTINEZ-CASTILLO M, QUINTANA-PEREZ J C, et al. Arsenic exposure: A public health problem leading to several cancers[J]. Regulatory Toxicology and Pharmacology, 2019, 110(3): 104539. DOI:10.1016/j.yrtph.2019.104539 (  0) 0) |
| [4] |
HOWE K, CLAEK M D, TORROJA C F, et al. The zebrafish reference genome sequence and its relationship to the human genome[J]. Nature, 2013, 496(7446): 498-503. DOI:10.1038/nature12111 (  0) 0) |
| [5] |
VLIEGENTHART A D, TUCKER C S, DEL POZO J, et al. Zebrafish as model organisms for studying drug-induced liver injury[J]. British Journal of Clinical Pharmacology, 2014, 78(6): 1217-1227. DOI:10.1111/bcp.12408 (  0) 0) |
| [6] |
GOESSLING W, SADLER K C. Zebrafish: An important tool for liver disease research[J]. Gastroenterology, 2015, 149(6): 1361-1377. DOI:10.1053/j.gastro.2015.08.034 (  0) 0) |
| [7] |
WILKINS B J, PACK M. Zebrafish models of human liver development and disease[J]. Comprehensive Physiology, 2013, 3(3): 1213-1230. DOI:10.1002/cphy.c120021 (  0) 0) |
| [8] |
ZHANG Xun, UNG C Y, LAM S H, et al. Toxicogenomic analysis suggests chemical-induced sexual dimorphism in the expression of metabolic genes in zebrafish liver[J]. PLoS One, 2012, 7(12): e51971. DOI:10.1371/journal.pone.0051971 (  0) 0) |
| [9] |
LAM S H, WINATA C L, TONG Y, et al. Transcriptome kinetics of arsenic-induced adaptive response in zebrafish liver[J]. Physiological Genomics, 2006, 27(3): 351-361. DOI:10.1152/physiolgenomics.00201.2005 (  0) 0) |
| [10] |
HUANG D W, SHERMAN B T, LEMPICKI R A. Systematic and integrative analysis of large gene lists using DAVID Bioinformatics Resources[J]. Nature protocols, 2009, 4(1): 44-57. DOI:10.1038/nprot.2008.211 (  0) 0) |
| [11] |
GYORFFY B. Survival analysis across the entire transcriptome identifies biomarkers with the highest prognostic power in breast cancer[J]. Computational and Structural Biotechnology Journal, 2021, 19: 4101-4109. DOI:10.1016/j.csbj.2021.07.014 (  0) 0) |
| [12] |
WONG C P, DASHNER-TITUS E J, ALVAREZ S C, et al. Zinc deficiency and arsenic exposure can act both independently or cooperatively to affect zinc status, oxidative stress, and inflammatory response[J]. Biological Trace Element Research, 2019, 191(2): 370-381. DOI:10.1007/s12011-019-1631-z (  0) 0) |
| [13] |
NAIN S, SMITS J. Pathological, immunological and biochemical markers of subchronic arsenic toxicity in rats[J]. Environmental Toxicology, 2012, 27(4): 244-254. DOI:10.1002/tox.20635 (  0) 0) |
| [14] |
WANG H, XI S, LIU Z, et al. Arsenic methylation metabolism and liver injury of acute promyelocytic leukemia patients undergoing arsenic trioxide treatment[J]. Environmental Toxicology, 2013, 28(5): 267-275. DOI:10.1002/tox.20717 (  0) 0) |
| [15] |
CARLSON P, SMALLEY D M, VAN BENEDEN R J. Proteomic analysis of arsenic-exposed zebrafish (Danio rerio) identifies altered expression in proteins involved in fibrosis and lipid uptake in a gender-specific manner[J]. Toxicological Sciences, 2013, 134(1): 83-91. DOI:10.1093/toxsci/kft086 (  0) 0) |
| [16] |
CARLSON P, VAN BENEDEN R J. Arsenic exposure alters expression of cell cycle and lipid metabolism genes in the liver of adult zebrafish (Danio rerio)[J]. Aquatic Toxicology, 2014, 153: 66-72. DOI:10.1016/j.aquatox.2013.10.006 (  0) 0) |
| [17] |
KOBATAKE K, IKEDA K, TEISHIMA J, et al. Complexity in radiological morphology predicts worse prognosis and is associated with an increase in proteasome component levels in clear cell renal cell carcinoma[J]. Frontiers in Oncology, 2022, 12: 1039383. DOI:10.3389/fonc.2022.1039383 (  0) 0) |
| [18] |
BLIJLEVENS M, KOMOR M A, SCIARRILLO R, et al. Silencing core spliceosome sm gene expression induces a cytotoxic splicing switch in the proteasome subunit beta 3 mrna in non-small cell lung cancer cells[J]. International Journal of Molecular Sciences, 202, 21(12): 4192. DOI: 10.3390/ijms21124192.
(  0) 0) |
| [19] |
HU Chen, YANG Jing, QI Ziping, et al. Heat shock proteins: Biological functions, pathological roles, and therapeutic opportunities[J]. MedComm (2020), 2022, 3(3): e161. DOI:10.1002/mco2.161 (  0) 0) |
| [20] |
OAKES S A, PAPA F R. The role of endoplasmic reticulum stress in human pathology[J]. Annual Review of Pathology, 2015, 10: 173-194. DOI:10.1146/annurev-pathol-012513-104649 (  0) 0) |
| [21] |
TAYLOR B F, MCNEELY S C, MILLER H L, et al. Arsenite-induced mitotic death involves stress response and is independent of tubulin polymerization[J]. Toxicology and Applied Pharmacology, 2008, 230(2): 235-246. DOI:10.1016/j.taap.2008.02.030 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


