结直肠癌(Colorectal cancer, CRC)是最常见的消化道恶性肿瘤,最新的全球癌症统计报告显示,结直肠癌已居全球癌症发病谱第三位,死因谱第二位[1]。预计2022年我国和美国将分别有大约482万和237万的新发癌症病例[2],结直肠癌发病率仍将高居前三,其形势不容乐观,而我国结直肠癌患者的预后较美国差。深入了解结直肠癌的分子机制、发现新的预后相关生物分子具有重要的意义。
丝氨酸蛋白酶抑制剂(Serine protease inhibitors, SERPIN)超家族,以丝氨酸为活性中心,控制丝氨酸和半胱氨酸蛋白酶的活性,参与凝血、纤维蛋白溶解、炎症、补体激活、细胞凋亡和细胞迁移等生理病理过程[3-7]。此外,SERPIN家族编码基因在多种肿瘤组织和正常组织间存在差异表达,参与肿瘤细胞的增殖、侵袭和转移[8-9],影响患者的生存与预后[10-12]。已有研究表明SERPINs家族基因参与结直肠癌的发生、发展与迁移等生物学过程[13-14],然而,SERPINs家族基因与结直肠癌的肿瘤微环境的改变、免疫浸润情况以及生存预后的关系研究,仍存在一定的不足。
本研究通过分析TCGA数据库和GEO数据库中结肠腺癌的高通量测序数据,通过生物信息学方法探究SERPINs家族基因在结肠腺癌中的预后价值,构建SERPINs相关预后生存模型并进行验证,探讨SERPINs家族基因、预后、肿瘤微环境以及免疫浸润之间的相关性,以期通过生物信息学方法辅助结肠癌的基础研究,促进研究成果向临床方向的转化。
1 材料和方法 1.1 数据来源从TCGA数据库获得结肠腺癌(COAD)的RNA测序(RNA-seq)数据(肿瘤数据473例,正常组织数据41例)以及对应的临床信息,数据格式为标准化后的FPKM格式,并将转录组数据转换为更接近GEO数据信息的每百万读取数的转录本(Transcripts per million reads,TPM)格式,从GEO数据库下载GSE40967型文件,其中包含585例结肠腺癌转录组数据及临床数据。通过GeneCards数据库(http://www.genecards.org/)检索以及TCGA数据库和GEO数据库中现有COAD转录组测序数据,筛选出SERPINA1/2/3/4/5/6/7/9/10/12、SERPINB1/2/3/4/5/6/7/8/9/9P1/10/11/12/13、SERPINC1、SERPIND1、SERPINE1/2、SERPINF1/2、SERPING1、SERPINH1及SERPINI1/2共34个SERPIN家族成员进行分析研究。
1.2 SERPINs家族基因一致性聚类分析将TCGA数据和GEO数据进行合并,剔除其中年龄、性别、生存状态、临床病理特征等重要临床信息缺失以及正常组织的样本,最终获得1 034例COAD患者信息,利用R包“ConsensusClusterPlus”对SERPINs家族基因表达矩阵进行一致性聚类分析[15],依次选取聚类值(k)1~9,再根据聚类效果选择聚类稳定性较高的k值。
1.3 富集分析以“c2.cp.kegg.v7.4.symbols.gmt”为参考基因集利用“GSVA”包对不同聚类簇进行基因集变异分析(GSVA),P < 0.05有统计学意义,以确定两个不同聚类簇之间的生物通路差异;并利用“GSVA”包的单样本富集分析(ssGSEA)算法计算不同分型COAD患者的免疫浸润丰度。
1.4 SERPINs家族基因相关评分预后模型的建立和评估利用R包“caret”将1 034例COAD患者随机均分为两组,训练集(Train)组和验证集(Test)组各517例。训练集(Train)用于构建预后风险模型,而验证集(Test)用于验证模型。利用“limma”包筛选COAD聚类簇之间的差异表达基因(DEGs),筛选条件:| Log2FC|>0.585,FDR<0.05,然后利用单因素Cox回归分析筛选出预后相关的DEGs,再利用多因素Cox回归分析和LASSO回归分析构建预后风险模型:
| $ 风险评分=\sum\limits_{i=1}^n { Expi } * { Coefi } $ |
即:用基因的表达水平与其对应的系数相乘,根据风险评分的中位值将样本分为高风险组合低风险组,利用“survminer”R包对高、低风险组进行Kaplan-Meier分析,评估两组样本的生存率差异,并应用“timeROC”包绘制时间依赖性ROC曲线,评估模型基因的预测稳定性。结合临床数据和风险评分绘制出列线图、校准曲线以及风险曲线对模型进行评估验证。
2 结果 2.1 SERPINs家族在结肠腺癌的表达及预后关系与41例正常组织相比,在COAD患者中多个SERPIN家族成员存在异常表达,SERPINB1、SERPINB9、SERPINB5、SERPINB2、SERPIND1、SERPINE2、SERPINA7、SERPINF2、SERPINE1、SERPINH1以及SERPINA4表达显著上调(图 1(a)),SERPINA9、SERPINA6、SERPINF1、SERPING1以及SERPINA5表达显著下调,通过分析34个基因共表达关系及其对COAD的预后影响,我们构建了SERPIN家族在COAD中的预后网络图,其中SERPINB7、SERPINE1、SERPINH1和SERPINI1的表达水平与COAD具有显著的预后关系(图 1(b))。

|
图 1 SERPIN家族基因在COAD中的表达 Figure 1 Expression of SERPIN family genes in COAD |
为了进一步研究SERPIN家族基因在CAOD中的表达特点,根据34个SERPIN基因的表达情况采用一致性聚类算法对COAD患者进行分类,当k=2时分型最佳,此时分型内部相关性最高,分型之间相关性最低(图 2(a)),在此基础上,进行主成分(PCA)分析,分离结果显示,能根据SERPINs家族基因的表达量能够区分分型1与分型2(图 2(b)),不同分型之间的生存分析结果显示,分型1的生存率显著高于分型2(图 2(c)),此外,用热图展示SERPINs的表达谱与COAD分型以及临床病理特征之间的关系,包括分型、年龄、性别、肿瘤分级、T、N、M分期(图 2(d)),(同时也进一步显示出来源于GEO和TCGA两个数据库的COAD数据在分型和临床病理分期上结果的高度一致性,此处是解释两聚类簇分型较好非数据库不同造成,来源于TCGA和GEO数据库在分型结果上一致)。
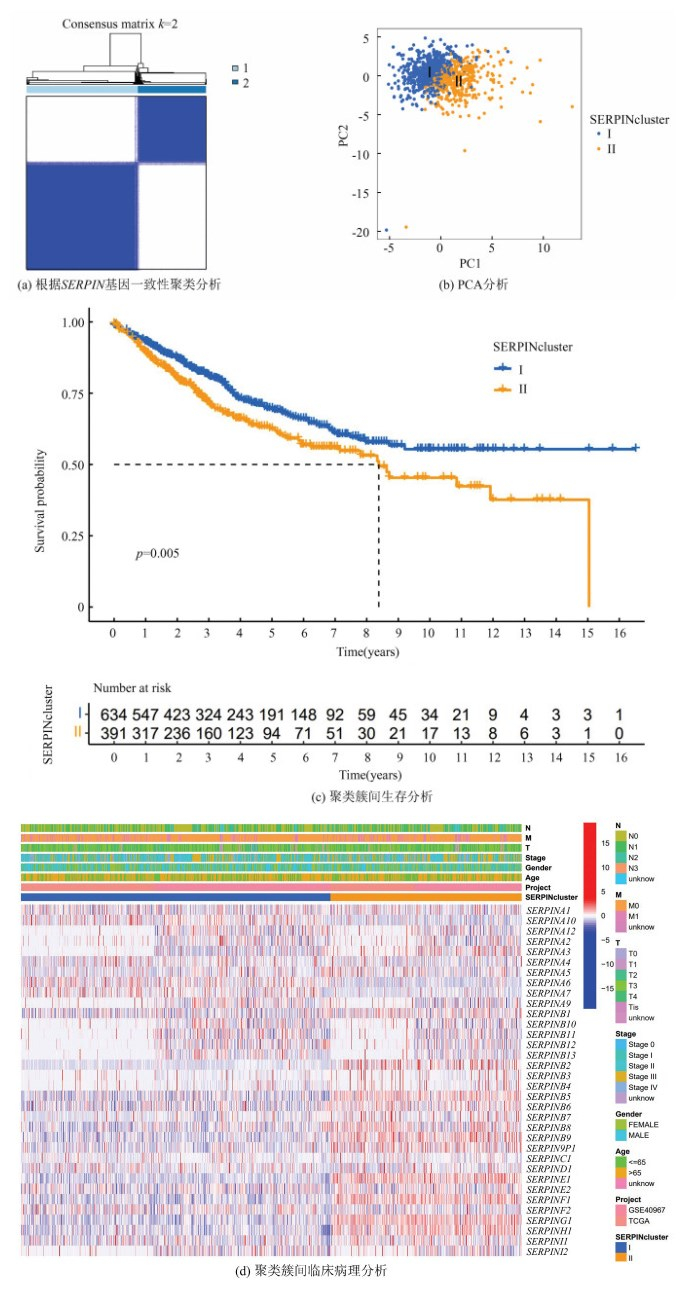
|
图 2 基于SERPIN家族基因的肿瘤分型 Figure 2 Tumor classification based on SERPIN family genes |
通过GSVA分析不同COAD分型间的信号通路差异,结果表明:分型1的COAD样本在过氧化相关通路上有显著的富集,而分型2在Toll样受体信号通路、趋化因子信号通路、细胞黏附分子通路以及MAPKA信号通路等都有显著的富集(图 3(a))。ssGSEA分析发现,分型Ⅱ较分型Ⅰ具有更高的免疫相关细胞水平,如活化型CD4+细胞、活化型B细胞、活化型树突状细胞、自然杀伤细胞、嗜酸粒细胞、中性粒细胞、巨噬细胞等(图 3(b))。

|
图 3 聚类簇的富集分析 Figure 3 Enrichment analysis of clusters |
对两种分型的COAD进行差异基因分析,确定了1 078个差异基因,运用单因素COX回归分析从这些DEGs最终筛选出435个预后相关的差异基因;在训练集(Train)数据中应用LASSO回归和多因素COX回归确定出11个预后基因进行风险评分模型的构建(图 4(a),图 4(b));预后风险评分=0.1832×TIMP1+0.2249×LAMA2-0.1505×EPHB2-0.1985×CXCL13+0.1874×PRRX2+0.1473×EGR3-0.1993× ABAT-0.1101×CXCL11+0.1545×FOXD1-0.0923×MMP3-0.0619×ITLN1。根据train组风险评分的中位值将train组分为低风险组和高风险组,高低风险组生存分析结果显示,低风险组患者具有明显的生存优势(图 4(c)),时间依赖性ROC曲线显示,训练集(Train)的1、3、5年生存率的AUC值分别是0.717、0.717、0.720(图 4(f))。通过验证集(Test)和总体(All)数据集利用模型风险评分预测COAD患者1、3和5年生存率AUC-ROC与训练集(Train)相似(图 4(d),4(e),4(g), 4(h))。
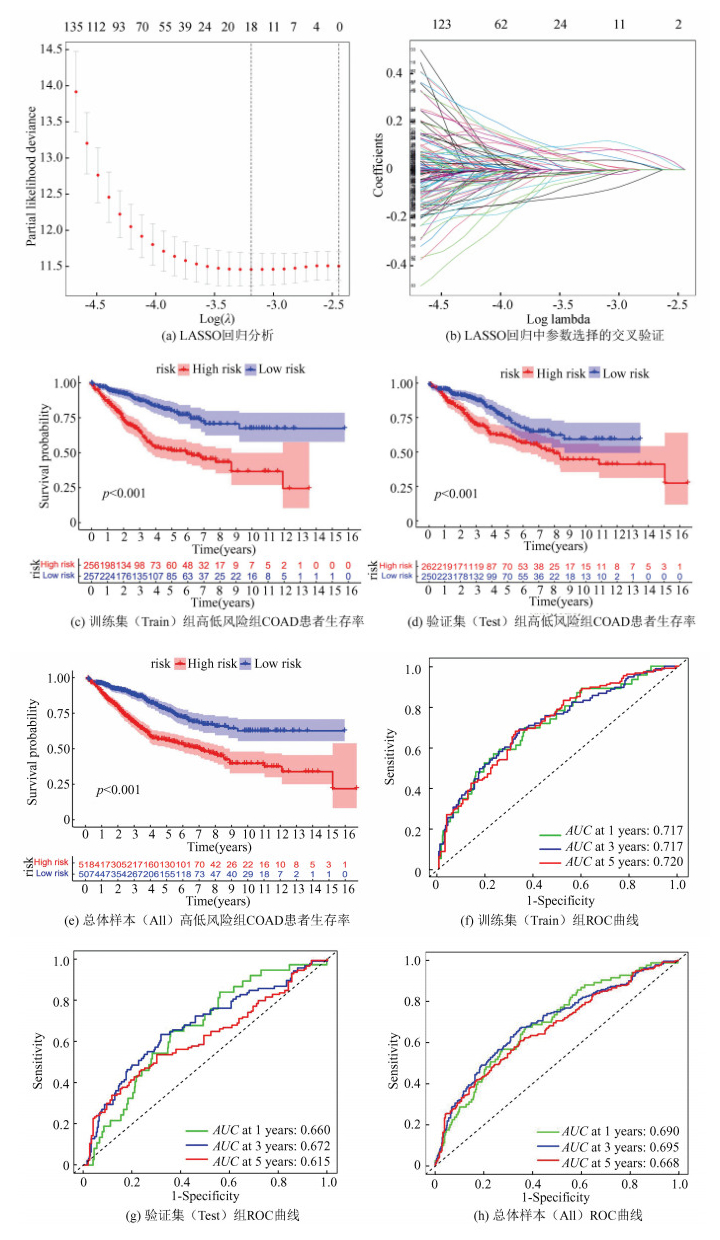
|
图 4 SERPIN家族相关基因风险评分模型的构建与验证 Figure 4 Construction and validation of SERPIN family related gene risk scoring model |
为了进一步验证风险评分模型对COAD患者的预后价值,根据多因素COX回归分析结果,构建一个包含年龄、性别、风险评分以及临床病理特征的列线图来估计COAD患者1、3、5年的总体生存期(OS)(图 5(a)),该列线图的校准曲线也显示了预期值与观测值高度一致性(图 5(b)), 与高低风险相关的SERPIN家族基因(图 5(c)),与低风险评分COAD患者相比,高风险患者中SERPINB2、SERPINE1、SERPINF1、SERPINA5、SERPIND1、SERPINB6、SERPINA3、SERPING1、SERPINB5、SERPINE2、SERPINH1表达上调,而SERPINA6和SERPINA7表达下调。

|
图 5 预测列线图的构建和验证 Figure 5 Generation and validation of predictive nomogram |
利用ESTIMATE算法[16]计算不同风险组COAD患者免疫评分、基质评分和ESTIMATE评分,根据评分结果比较不同风险组COAD患者肿瘤微环境(Tumor microenvironment,TME)特征,结果显示,高风险组基质细胞的含量高于低风险组,而免疫细胞的含量在低风险组中含量更高(图 6)。随后,利用CIBERSORT免疫浸润算法[17]评估风险评分与患者免疫细胞浸润之间的相关性,我们发现,记忆B细胞、M0巨噬细胞、M2巨噬细胞、活化型肥大细胞、中性粒细胞与风险评分呈正相关,初始B细胞、静止型树突细胞、M1巨噬细胞、活化型CD4+记忆T细胞、CD8+细胞、滤泡辅助性T细胞呈负相关(表 1)。
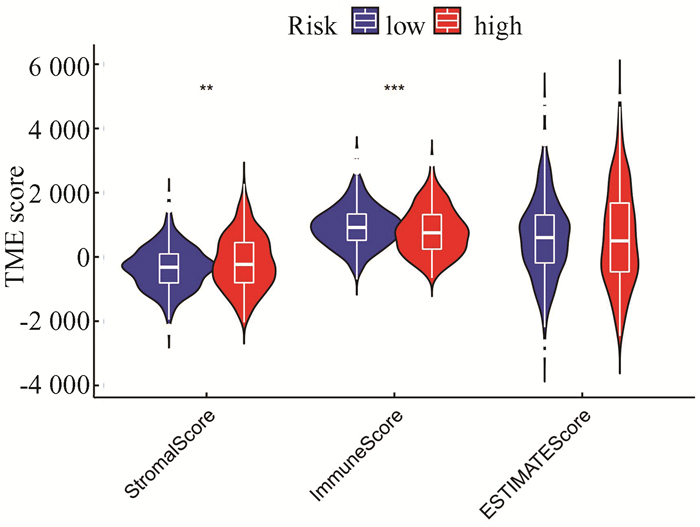
|
图 6 肿瘤微环境与风险评分关系 Figure 6 Relationship between tumor microenvironment and risk score |
| 表 1 COAD风险评分与免疫细胞浸润之间的相关性分析 Table 1 Correlation analysis between COAD risk score and immune cell infiltration |
SERPIN是目前已知亚型最多的蛋白酶抑制剂家族,参与机体血液凝固、血管生成、炎症反应、细胞迁移等多种生物学功能,行为广泛,作用复杂,影响肿瘤微环境[18],诱发免疫状态的改变[19-20],促进肿瘤的发转移与侵袭。基于目前SERPIN基因在在结直肠癌中个研究仍有不足,其在结肠腺癌中的致病意义仍未完全明确,本研究第一次利用生物信息学方法,通过SERPIN家族相关基因构建在结肠腺癌中的预后评分模型,该模型能够预测COAD患者的生存率,反映肿瘤微环境、免疫浸润与COAD的相关性。
研究发现,许多SERPIN家族成员在COAD中存在异常表达。已有研究表明SERPINE1、SERPINH1和SERPINI1在结肠癌的发生发展、侵袭转移中发挥重要作用[14, 21-22],SERPIN家族成员在COAD的作用机制是非常复杂的,我们的研究结果证实了SERPINE1、SERPINH1和SERPINI1在COAD中是高风险因子,过表达影响患者预后。此外,我们还发现SERPINB7在COAD患者中存在异常表达,且影响患者预后,但目前仍缺乏SERPINB7在COAD方面的研究,其具体作用机制有待进一步发掘。此外,依据SERPIN的表达量将COAD患者分为两个聚类簇,分型Ⅱ较分型Ⅰ预后较差,然而在免疫细胞浸润分析中发现,分型Ⅱ较分型Ⅰ具有更多的免疫细胞富集,这提示SERPIN家族可能通过影响免疫细胞的表达和功能等影响COAD的预后。
本研究筛选出11个SERPIN相关预后基因进行COAD预后模型的构建,该模型在训练集(1、3、5年AUC均>0.7) 和验证集(1、3、5年AUC均>0.6)以及总体样本(1、3、5年AUC均>0.6)中均表现出良好的预测效能。根据SERPIN家族成员在该模型中高低风险组表达情况我们发现,SERPINA6和SERPINA7在低风险组高表达,这提示SERPINA6和SERPINA7可能是一种保护基因,既往研究发现,SERPINA6编码血浆皮质类固醇结合球蛋白(CBG)[23],SERPINA7编码甲状腺素结合球蛋白(TBG)[24],参与人体的体液调节,但其目前在COAD中的作用尚不明确。SERPINB2、SERPINE1、SERPINF1、SERPINA5、SERPIND1、SERPINB6、SERPINA3、SERPING1、SERPINB5、SERPINE2、SERPINH1在高风险组表达上调,这提示这些基因高表达的COAD患者预后不良。有研究表明,SERPINB2可能通过炎症调节,参与结直肠癌的调节[25],SERPINF1的过表达增加结直肠癌抗PD-1抗体的抗性,增强肿瘤耐药性[26]。SERPINA5能通过PI3K/AKT途径发挥致癌基因作用促进胃癌的增殖和转移[27]。SERPIND1能通过PI3K/AKT途径促进卵巢癌细胞增殖、迁移、侵袭[28]。沉默SERPINA3能够减少MMP-2和MMP9的表达,抑制结肠癌侵袭和转移[29].随着研究的深入和技术的发展,越来越多的证据表明SERPIN家族通过多种途径在癌症中发挥作用,就我们研究来看,SERPIN家族在COAD中的研究仍有很大的研究空间,仍需要大量的基础实验进一步验证和发掘。
肿瘤微环境(TME)包括浸润性癌巢及其周围的基质细胞和免疫细胞,近年来,越来越多研究表明TME在肿瘤的发生和转移中起着至关重要的作用[30-31],我们的风险评分与肿瘤微环境成分相关性分析结果显示,低风险组具有更高的免疫细胞含量。此外,我们的免疫细胞浸润分析也证实了这一点,低风险评分的COAD样本具有更好的免疫细胞浸润。这提示TME和免疫状态的调节可能是SERPIN基因影响COAD患者预后的原因之一。
4 结论综上所述,SERPIN家族相关基因构建的预后模型可用于预测COAD的患者的预后,有利于进一步指导临床对COAD的诊疗。该评分模型同时也表明TME和免疫状态在COAD的预后中也发挥着重要的作用,此外,许多SERPIN家族成员在COAD中风险性也通过该模型得以展示,但其在COAD中的具体调控途径和作用方式仍需进一步研究和验证。
| [1] |
SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: Globocan estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA: A Cancer Journal for Clinicians, 2021, 71(3): 209-249. DOI:10.3322/caac.21660 (  0) 0) |
| [2] |
XIA C, DONG X, LI H, et al. Cancer statistics in China and United States, 2022: profiles, trends, and, determinants[J]. Chinese Medical Journal, 2022, 135(5): 584-590. DOI:10.1097/CM9.0000000000002108 (  0) 0) |
| [3] |
AYMONNIER K, KAWECKI C, AROCAS V, et al. Serpins, new therapeutic targets for hemophilia[J]. Thrombosis and Haemostasis, 2021, 121(3): 261-269. DOI:10.1055/s-0040-1716751 (  0) 0) |
| [4] |
BATIHA G E, AL-KURAISHY H M, AL-MAIAHY T J, et al. Plasminogen activator inhibitor 1 and gestational diabetes: the causal relationship[J]. Diabetology & Metabolic Syndrome, 2022, 14(1): 1-6. DOI:10.1186/s13098-022-00900-2 (  0) 0) |
| [5] |
SILVERMAN G A, WHISSTOCK J C, ASKEW D J, et al. Human clade B serpins (ov-serpins) belong to a cohort of evolutionarily dispersed intracellular proteinase inhibitor clades that protect cells from promiscuous proteolysis[J]. Cellular and Molecular Life Sciences : CMLS, 2004, 61(3): 301-325. DOI:10.1007/s00018-003-3240-3 (  0) 0) |
| [6] |
SPENCE M A, MORTIMER M D, BUCKLE A M, et al. A comprehensive phylogenetic analysis of the serpin superfamily[J]. Molecular Biology and Evolution, 2021, 38(7): 2915-2929. DOI:10.1093/molbev/msab081 (  0) 0) |
| [7] |
MKAOUAR H, MARIAULE V, RHIMI S, et al. Gut serpinome: Emerging evidence in IBD[J]. International Journal of Molecular Sciences, 2021, 22(11): 6088. DOI:10.3390/ijms22116088 (  0) 0) |
| [8] |
SOMAN A, ASHA NAIR S. Unfolding the cascade of SERPINA3: Inflammation to cancer[J]. Biochimica et Biophysica Acta-Reviews on Cancer, 2022, 1877(5): 188760. DOI:10.1016/j.bbcan.2022.188760 (  0) 0) |
| [9] |
FAN M, XIONG X, HAN L, et al. SERPINA5 promotes tumour cell proliferation by modulating the PI3K/AKT/mTOR signalling pathway in gastric cancer[J]. Journal of Cellular and Molecular Medicine, 2022, 26(18): 4837-4846. DOI:10.1111/jcmm.17514 (  0) 0) |
| [10] |
WANG Y, GU W, WEN W, et al. SERPINH1 is a potential prognostic biomarker and correlated with immune infiltration: A pan-cancer analysis[J]. Frontiers in Genetics, 2022, 12: 756094. DOI:10.3389/fgene.2021.756094 (  0) 0) |
| [11] |
SCHNEIDER M A, RICHTMANN S, GRVNDING A R, et al. Transmembrane serine protease 2 is a prognostic factor for lung adenocarcinoma[J]. International Journal of Oncology, 2022, 60(4): 1-12. DOI:10.3892/ijo.2022.5329 (  0) 0) |
| [12] |
PACZEK S, MROCZKO B. The role of selected serpins in gastrointestinal (GI) malignancies[J]. Journal of Clinical Medicine, 2022, 11(20): 6225. DOI:10.3390/jcm11206225 (  0) 0) |
| [13] |
KIM W T, MUN J Y, BAEK S W, et al. Secretory SERPINE1 expression is increased by antiplatelet therapy, inducing MMP1 expression and increasing colon cancer metastasis[J]. International Journal of Molecular Sciences, 2022, 23(17): 9596. DOI:10.3390/ijms23179596 (  0) 0) |
| [14] |
VYCITAL O, PITULE P, HOSEK P, et al. Expression of serpin b9 as a prognostic factor of colorectal cancer[J]. Anticancer Research, 2019, 39(11): 6063-6066. DOI:10.21873/anticanres.13813 (  0) 0) |
| [15] |
WILKERSON M D, HAYES D N. ConsensusClusterPlus: A class discovery tool with confidence assessments and item tracking[J]. Bioinformatics, 2010, 26(12): 1572-1573. DOI:10.1093/bioinformatics/btq170 (  0) 0) |
| [16] |
YOSHIHARA K, SHAHMORADGOLI M, MARTÍNEZ E, et al. Inferring tumour purity and stromal and immune cell admixture from expression data[J]. Nature Communications, 2013, 4: 1-11. DOI:10.1038/ncomms3612 (  0) 0) |
| [17] |
NEWMAN A M, LIU C L, GREEN M R, et al. Robust enumeration of cell subsets from tissue expression profiles[J]. Nature Methods, 2015, 12(5): 453-457. DOI:10.1038/nmeth.3337 (  0) 0) |
| [18] |
WANG Shaokun, PANG Li, LIU Zuolong, et al. SERPINE1 associated with remodeling of the tumor microenvironment in colon cancer progression: A novel therapeutic target[J]. BMC Cancer, 2021, 21(1): 1-13. DOI:10.1186/s12885-021-08536-7 (  0) 0) |
| [19] |
LI Fazhan, ZHOU Jun, LI Zedong, et al. Screening of immunosuppressive cells from colorectal adenocarcinoma and identification of prognostic markers[J]. Bioscience Reports, 2021, 41(4): BSR20203496. DOI:10.21203/rs.3.rs-23944/v1 (  0) 0) |
| [20] |
GATTO M, IACCARINO L, GHIRARDELLO A, et al. Serpins, immunity and autoimmunity: Old molecules, new functions[J]. Clinical Reviews in Allergy & Immunology, 2013, 45(2): 267-280. DOI:10.1007/s12016-013-8353-3 (  0) 0) |
| [21] |
MATSUDA Y, MIURA K, YAMANE J, et al. SERPINI1 regulates epithelial-mesenchymal transition in an orthotopic implantation model of colorectal cancer[J]. Cancer Science, 2016, 107(5): 619-628. DOI:10.1111/cas.12909 (  0) 0) |
| [22] |
ZHONG H, WANG Z, WEI X, et al. Prognostic and immunological role of SERPINH1 in pan-cancer[J]. Frontiers in Genetics, 2022, 13: 900495. DOI:10.3389/fgene.2022.900495 (  0) 0) |
| [23] |
TOEWS J N C, PHILIPPE T J, HILL L A, et al. Corticosteroid-binding globulin (SERPINA6) establishes postpubertal sex differences in rat adrenal development[J]. Endocrinology, 2022, 163(11): bqac152. DOI:10.1210/endocr/bqac152 (  0) 0) |
| [24] |
FANG Y, CHEN H, CHEN Q, et al. Compound hemizygous variants in SERPINA7 gene cause thyroxine-binding globulin deficiency[J]. Molecular Genetics & Genomic Medicine, 2021, 9(2): e1571. DOI:10.1002/mgg3.1571 (  0) 0) |
| [25] |
XU Y, WANG X, CHU Y, et al. Analysis of transcript-wide profile regulated by microsatellite instability of colorectal cancer[J]. Annals of Translational Medicine, 2022, 10(4): 169. DOI:10.21037/atm-21-6126 (  0) 0) |
| [26] |
DENIS M, GRASSELLY C, CHOFFOUR P A, et al. In vivo syngeneic tumor models with acquired resistance to Anti-PD-1/PD-L1 therapies[J]. Cancer Immunology Research, 2022, 10(8): 1013-1027. DOI:10.1158/2326-6066.CIR-21-0802 (  0) 0) |
| [27] |
FAN M, XIONG X, HAN L, et al. SERPINA5 promotes tumour cell proliferation by modulating the PI3K/AKT/mTOR signalling pathway in gastric cancer[J]. Journal of Cellular and Molecular Medicine, 2022, 26(18): 4837-4846. DOI:10.1111/jcmm.17514 (  0) 0) |
| [28] |
GUO Q, ZHU L, WANG C, et al. SERPIND1 affects the malignant biological behavior of epithelial ovarian cancer via the pi3k/akt pathway: A mechanistic study[J]. Frontiers in Oncology, 2019, 9: 954. DOI:10.3389/fonc.2019.00954 (  0) 0) |
| [29] |
CAO L L, PEI X F, QIAO X, et al. SERPINA3 silencing inhibits the migration, invasion, and liver metastasis of colon cancer cells[J]. Digestive Diseases and Sciences, 2018, 63(9): 2309-2319. DOI:10.1007/s10620-018-5137-x (  0) 0) |
| [30] |
ITO K, OSAKABE M, SUGIMOTO R, et al. Differential expression in the tumor microenvironment of mRNAs closely associated with colorectal cancer metastasis[J]. Annals of Surgical Oncology, 2023, 30(2): 1255-1266. DOI:10.1245/s10434-022-12574-1 (  0) 0) |
| [31] |
YAMAMOTO Y, KASASHIMA H, FUKUI Y, et al. The heterogeneity of cancer-associated fibroblast subpopulations: Their origins, biomarkers, and roles in the tumor microenvironment[J]. Cancer Science, 2023, 114(1): 16-24. DOI:10.1111/cas.15609 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


