新型冠状病毒感染(COVID-19)已在全球范围内造成大范围流行,据世界卫生组织数据,截至2023年2月1日,COVID-19全球感染病例已超过6亿7 000万例,死亡人数超过680万,对全球公共卫生构成重大威胁。COVID-19的主要症状表现为发热、干咳、乏力,重症患者会快速发展为急性呼吸窘迫综合征,甚至引发凝血病和多器官衰竭,进而威胁患者生命[1-2]。COVID-19也会对患者造成长期影响,在发病后90-150 d内仍可能出现胸痛、呼吸困难、肌肉疼痛、全身乏力等症状[3]。因此开发针对COVID-19的特效药物,在感染后尽早降低体内的病毒载量,最大程度消除病毒带来的不良影响已成为COVID-19防治的重中之重。
COVID-19由严重急性呼吸系统综合征冠状病毒2(SARS-CoV-2)引起,病毒进入人体后其表面刺突蛋白(S蛋白)的受体结合域与宿主细胞膜上的血管紧张素转化酶2(ACE2)结合,促使病毒进入宿主细胞,之后病毒裂解并将基因组RNA释放至细胞质,利用宿主核糖体翻译出两种较大的共末端多蛋白pp1a和pp1ab,再被3CL蛋白酶(主蛋白酶)水解加工成16种非结构蛋白,这些非结构蛋白可参与病毒RNA的复制。病毒颗粒在内质网和高尔基体中完成组装,并通过胞吐作用释放至宿主细胞外[4-5]。从病毒感染和病毒新颗粒形成的机制看,S蛋白是病毒进入细胞的主要蛋白,3CL蛋白酶在生成非结构蛋白中发挥关键作用,二者是病毒感染及复制过程中的关键分子,可作为抑制、杀灭病毒的重要靶标[6-7]。
本研究选择S蛋白和3CL蛋白酶两种分子作为受体,以FDA批准的2 726种小分子药物为配体,利用分子对接方法,分析受体和配体的亲和能,从中筛选出了3种(阿巴瑞克(Abarelix)、西曲瑞克(Cetrorelix)、鞣酸(Tannic acid))针对S蛋白和1种(戈舍瑞林(Goserelin))针对3CL蛋白酶的潜在抑制剂,同时对筛选药物的特征、与靶标的相互作用、与基因的相互作用进行了分析,旨在为未来开发新型冠状病毒感染的治疗药物提供参考。
1 数据和方法 1.1 数据针对S蛋白共提取了3种结构,均包含与ACE2结合的关键受体结合域(Receptor binding domain, RBD):第一种来自S蛋白受体结合域与人类ACE2嵌合的复合物(PDB ID: 6VW1);第二种为跨物种识别的S蛋白与ACE2的复合物(PDB ID: 7C8J);第三种为γ型突变SARS-CoV-2的S蛋白与ACE2的复合物(PDB ID: 7EKC)。针对3CL蛋白酶,分子对接使用的结构来自SARS-CoV-2的3CL蛋白酶与N3抑制剂复合物(PDB ID:6LU7)。
2726种用于对接的药物小分子结构信息来自数据库Drugbank[8](https://go.drugbank.com/),小分子药物均为经FDA批准上市的药物,主要为抗肿瘤、抗菌类药物。数据信息包括药物分子的名称、原子坐标、相对分子质量、上市时间、适用病症等。辉瑞公司开发的Paxlovid中的有效成分Nirmatrelvir的三维结构从PubChem中获取(PubChem CID: 155903259)。
1.2 方法药物虚拟筛选过程包括分子结构准备和分子对接两步,具体如下:
1) 受体蛋白结构准备:从PDB数据库获取相应蛋白的三维结构,先利用Pymol[9]从复合物中去除配体分子及水分子,分离出S蛋白的受体结合域及3CL蛋白酶,将上述两种结构作为对接受体,利用AutoDock软件包中的工具对受体蛋白添加极性氢原子并计算整个分子电荷,保存为用于对接的pdbqt格式。
2) 小分子配体结构准备:从Drugbank获取的小分子,其原有文件格式为sdf格式,利用OpenBabel[10]为其添加极性氢原子并将其转换为对接所需的pdbqt格式,生成小分子的三维结构坐标。
3) 对接位点(Box)选取:分子对接位点的选择以复合物的原有配体结合位点为基础,适当扩大范围作为最终的对接位点。由于3种S蛋白的序列差异主要体现在整个序列的首尾,与ACE2结合的位点保守性较高,因此针对3种S蛋白选取同样的对接位点,x、y、z轴格点数均设置为50,三个方向的中心坐标分别为-30.934、26.838、22.868。针对3CL蛋白酶,对接位点设置在其天然配体N3抑制剂附近,x/y/z轴格点数均为60,三个方向的中心坐标分别为-13.609、12.221、65.033。
4) 利用Autodock Vina[11]进行分子对接:将对接位点作为搜索区域,扫描不同类型的原子计算其格点能量,通过平移、旋转方式调整配体分子的取向,寻找使得复合物整体结合能量最低的构象。
对接计算中采用半柔性对接策略[12],固定靶点蛋白,改变受体小分子药物构象,在简化部分计算量的同时保证较好的预测能力[13]。选择对接亲和能作为经验打分函数,亲和能由氢键、离子相互作用、疏水相互作用、构象熵等分量组成,每个分量对应的系数通过拟合具有已知亲和能的配体-蛋白质复合物的测试集回归分析得到公式1[14]:
| $ \begin{align} & {{G}_{\text{bind}}}={{G}_{0}}+{{G}_{\text{hb}}}\sum{f\left( R, \alpha \right)}+{{G}_{\text{ionic}}} \\ & \sum{f\left( R, \alpha \right)}+{{G}_{\text{lipo}}}\left| {{A}_{\text{lipo}}} \right|+{{G}_{\text{rot}}}{{N}_{\text{ROT}}} \\ \end{align} $ | (1) |
式中各项含义如下:
△Gbind:最终的亲和能;
△G0:通过多元线性回归得到的45种蛋白质-配体复合物的实验结合数据;
△Ghb:氢键分量;
△Gionic:离子相互作用分量;
△Glipo:疏水相互作用分量;
△Grot:构象熵分量;
f(△R, △α):与复合物的理想距离R以及理想角度α有关的函数,决定氢键和离子相互作用;
Alipo:蛋白质受体与包含非极性原子配体之间的接触表面积;
NROT:配体中可旋转键数目。
5) 结果选择:亲和能数值越低,证明小分子与靶标蛋白的复合物构象越稳定,结合程度越好,以此作为选择候选药物的依据。把针对每种蛋白得到的受体-药物小分子复合物按照亲和能从低到高排列,选取亲和能排名前十的小分子作为候选分子,分析药物性质及与靶点的相互作用残基。
针对3CL蛋白酶,将筛选出的药物与已批准上市的Paxlovid中的有效成分Nirmatrelvir进行对比,该药物也以3CL蛋白酶为作用靶点,比较候选药物与该药物之间的结合能与相互作用残基,理论上验证筛选出的药物作为治疗COVID-19的可能性。同时利用DGIdb数据库提供的信息对候选药物和人体内基因的相互作用开展分析,总结出了药物与体内基因存在的相互作用类型及得分,随后利用NCBI数据库确定了每种基因在器官中的表达量,进一步评估候选药物在人体内的代谢情况。
2 结果 2.1 S蛋白的潜在抑制剂筛选根据受体和配体的亲和能数值,本研究对每种复合物选择了亲和能最低的前4个药物分子进行可视化,利用Pymol展示受体蛋白与小分子的结合情况、二者之间的相互作用以及发生作用的残基位点与残基名称。
结果表明,在靶向SARS-CoV-2的S蛋白嵌合受体结合域的4个小分子中(图 1、图 2),阿巴瑞克(Abarelix)显示出较好的结合能力。该药物与谷氨酸-354、精氨酸-355、丝氨酸-359、酪氨酸-396、谷氨酸-516残基形成氢键相互作用(图 1(a))。这些相互作用集中在S蛋白的中部,大部分为通过羧基形成的氢键相互作用。Abarelix为促性腺激素释放激素拮抗剂,可用于治疗前列腺癌[15],本研究发现该药物也可能具有抑制S蛋白与ACE2结合的功能。
|
图 1 药物分子与S蛋白嵌合受体结合域的小分子药物 Figure 1 Small molecular drugs targeting chimeric receptor binding domain of S protein of SARS-CoV-2 注:(a)Abarelix, ΔG=-11.0 kcal/mol; (b)Surfaxin, ΔG=-10.3 kcal/mol; (c)Simeprevir, ΔG=-9.5 kcal/mol; (d)Dihydroergotamine, ΔG=-9.4 kcal/mol (左图为分子全貌,右图为局部,下同). |

|
图 2 靶向S蛋白嵌合受体结合域四种优选药物的二维结构 Figure 2 2D structures of 4 small molecular drugs targeting receptor binding domain of S protein of SARS-CoV-2 注: (a)Abarelix; (b) Surfaxin; (c)Simeprevir; (d) Ergotamine. |
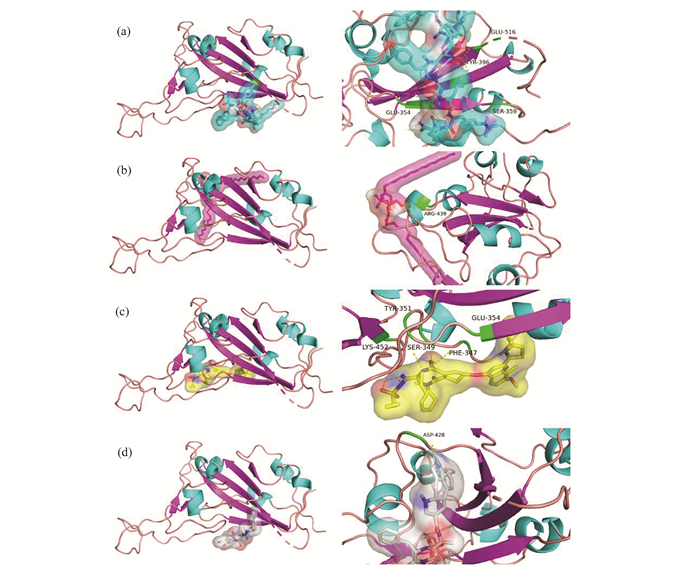
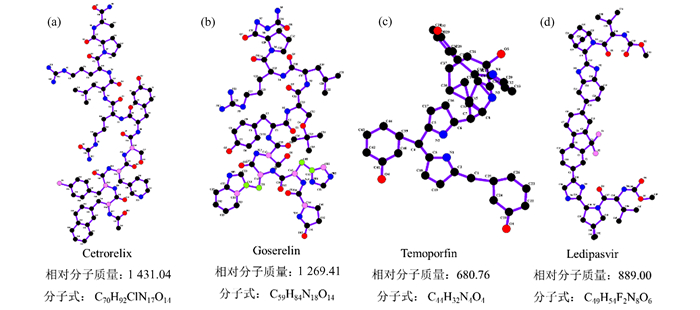
在靶向跨物种识别的S蛋白受体结合域的候选药物中(图 3、图 4),西曲瑞克(Cetrorelix)的结合情况较好,可在谷氨酸-340、精氨酸-346、丝氨酸-349、天冬氨酸-354、异亮氨酸-468、苏氨酸-470等多个残基位点形成极性相互作用(图 3(a))。该药物属于促性腺激素释放激素拮抗剂,临床上主要用于防止受控卵巢刺激期间的过早排卵[16]。

|
图 3 靶向SARS-CoV-2跨物种识别S蛋白受体结合域的小分子药物 Figure 3 Small molecular drugs targeting receptor binding domain of cross-species S protein of SARS-CoV-2 注: (a)Cetrorelix, ΔG=-9.7 kcal/mol; (b)Goserelin, ΔG=-9.6 kcal/mol; (c)Temoporfin, ΔG=-9.5 kcal/mol; (d)Ledipasvir, ΔG=-9.5 kcal/mol. |
|
图 4 靶向SARS-CoV-2跨物种识别S蛋白受体结合域四种优选药物的二维结构 Figure 4 2D structures of 4 small molecular drugs targeting receptor binding domain of cross-species S protein of SARS-CoV-2 注: (a) Cetrorelix; (b) Goserelin; (c) Temoporfin; (d) Ledipasvir. |
在靶向γ型突变的S蛋白受体结合物的候选药物中(图 5、图 6),鞣酸(Tannic acid)显示出较好的结合效果,可与谷氨酸-340和丝氨酸-349形成氢键(图 5(a)),该药物可以作为收敛剂使用以沉淀蛋白质,具有抗病毒和抗菌作用[17]。

|
图 5 靶向γ型突变SARS-CoV-2 S蛋白受体结合域的小分子药物 Figure 5 Small molecular drugs targeting receptor binding domain of γ variant S protein of SARS-CoV-2 注: (a)Tannic acid, ΔG=-12.1 kcal/mol; (b)Ganirelix, ΔG=-10.1 kcal/mol; (c)Temoporfin, ΔG=-9.2 kcal/mol; (d)Apixaban, ΔG=-9.2 kcal/mol. |

|
图 6 靶向γ型突变SARS-CoV-2 S蛋白受体结合域四种优选药物的二维结构 Figure 6 2D structures of 4 small molecular drugs targeting receptor binding domain of γ variant S protein of SARS-CoV-2 注: (a) Tannic acid; (b) Ganirelix; (c) Temoporfin; (d) Apixaban. |
经序列比对发现3种S蛋白的差异主要集中在氨基酸序列的首尾,而与ACE2结合部分的结构相对保守,上述药物与S蛋白发生相互作用的残基大部分集中于谷氨酸、苏氨酸、精氨酸等残基。因此我们也针对3种不同类型的S蛋白筛选出的结合能排名前十的药物进行了对比,发现替莫卟吩(Temoporfin)与布美诺肽(Bremelanotide)在3种S蛋白受体结合域中均展现出较好的结合能力(图 7),同时有5种药物与跨物种识别的S蛋白和γ突变型S蛋白都有很好的结合,并集中在ACE2结合的位点附近,证明3种S蛋白的受体结合域有很高的相似性,可以针对其共有的结构位点进行药物开发。
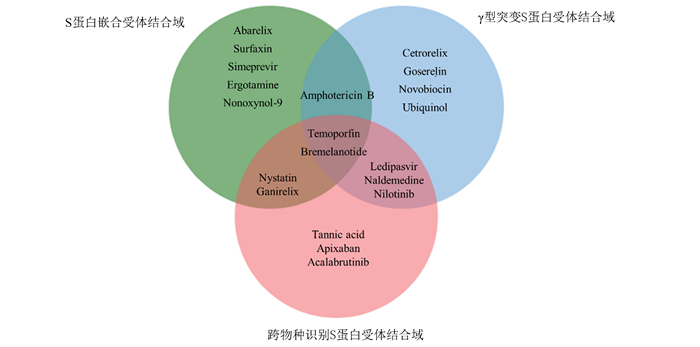
|
图 7 靶向不同种类S蛋白受体结合域的药物对比 Figure 7 Comparison of drugs targeting different S protein receptor binding domain |
在针对3CL蛋白酶的药物筛选中(图 8、图 9),戈舍瑞林(Goserelin) 拥有最低的结合能(-10.7 kcal/mol),结合效果最佳,可与3CL蛋白酶的赖氨酸-137、赖氨酸-236、天冬氨酸-238、酪氨酸-239、亮氨酸-287、谷氨酸-288残基形成相互作用。该药物临床上适用于晚期前列腺癌的姑息治疗、子宫内膜异位症的治疗及晚期乳腺癌的姑息治疗[18]。
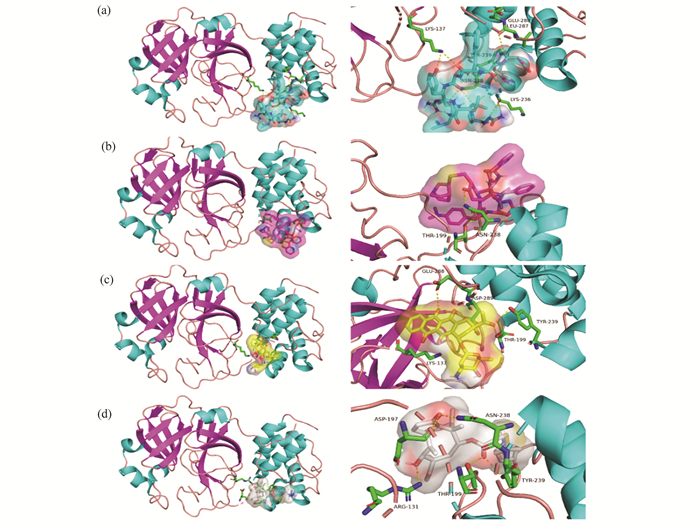
|
图 8 靶向3CL蛋白酶的小分子药物 Figure 8 Small molecular drugs targeting 3CL protease 注: (a)Goserelin, ΔG=-10.7 kcal/mol; (b)Quinupristin, ΔG=-10.3 kcal/mol; (c)Idarubicin, ΔG=-8.9 kcal/mol; (d)Retapamulin, ΔG=-8.3 kcal/mol. |

|
图 9 靶向3CL蛋白酶四种优选药物的二维结构 Figure 9 2D structures of 4 small molecular drugs targeting 3CL protease 注: (a)Goserelin; (b)Quinupristin; (c)Idarubicin; (d)Retapamulin. |
新型冠状病毒S蛋白高度易变,而3CL蛋白酶相对保守,因此新型冠状病毒的特效药主要针对3CL蛋白酶进行开发,如辉瑞公司靶向SARS-CoV-2的3CL蛋白酶开发了Paxlovid口服药物,本文也针对Paxlovid的有效成分Nirmatrelvir进行了对接验证。利用LigPlot+[19]和Pymol分别对复合物进行二维(图 10(a))和三维水平(图 10(b))的可视化,确定了Nirmatrelvir与3CL蛋白酶之间的极性和非极性相互作用。该药物与3CL蛋白酶的结合能介于-6.5~-7.0 kj/mol之间,拥有比较好的结合能力,并与3CL蛋白酶199位的苏氨酸和287位的亮氨酸发生氢键相互作用。
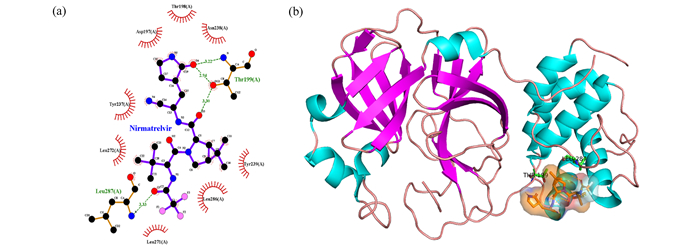
|
图 10 3CL蛋白酶与Nirmatrelvir的对接情况 Figure 10 Binding mode of 3CL protease and Nirmatrelvir 注: (a)Nirmatrelvir与3CL蛋白酶极性与非极性相互作用; (b)Nirmatrelvir与3CL蛋白酶三维结合构象与极性相互作用残基. |
在确定Nirmatrelvir与3CL蛋白酶的相互作用区域后,选定该区域作为对接位点,将Nirmatrelvir与排名前四的候选药物放在同一坐标轴下与3CL蛋白酶进行对接,直观地对比Nirmatrelvir和候选药物与3CL蛋白酶的结合位点以及结合模式(图 11)。对比发现四种候选药物与Nirmatrelvir结合的位点均集中在第130-200位的残基附近,并可与赖氨酸、天冬酰胺、苏氨酸、酪氨酸、亮氨酸等残基发生氢键相互作用以及非极性相互作用,说明这四种药物与Nirmatrelvir具有相似的作用位点及相互作用方式,与3CL蛋白酶结合的机理有一定相似性,理论上能够对3CL蛋白酶的活性起到抑制作用。
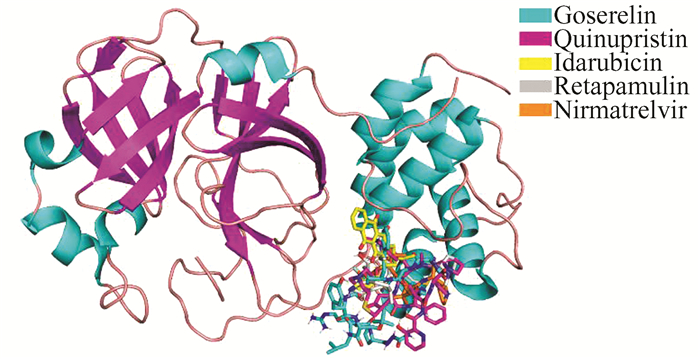
|
图 11 Nirmatrelvir与候选药物的结合情况比较 Figure 11 Binding comparison of Nirmatrelvir with candidate drugs 注:蓝绿色:Goserelin;紫红色:Quinupristin;黄色:Idarubicin;灰白色:Retapamulin;橙色:Nirmatrelvir(彩图见电子版:http://swxxx.alljournals.cn/ch/index.aspx.2024年第22卷第1期). |
为了进一步探究候选药物治疗新型冠状病毒肺炎的可能性,在PubChem中查询了与3CL蛋白酶结合能力排名前十的小分子药物的物理化学性质(表 1),包括分子质量、脂水分配系数、氢键供体/受体数目、可旋转键数目、拓扑极性表面积等参数,与Nirmatrelvir作了层次聚类分析,分析药物之间的相似性。从层次聚类图(图 12)中可以看出,Surfaxin、Quinupristin、Retapamulin、Idarubicin四种药物与Nirmatrelvir具有较高的相似性,可能具有相似的药效,后续可在此基础上针对药物的药效团改进其结构,以增强药物对3CL蛋白酶的抑制能力。
| 表 1 与Nirmatrelvir进行层次聚类的药物信息 Table 1 Drugs hierarchical clustering with Nirmatrelvir |

|
图 12 针对3CL蛋白酶小分子的层次聚类情况 Figure 12 Hierarchical clustering of small molecular drugstargeting 3CL protease |
本研究还探究了候选小分子药物与人体内基因之间的相互作用,以评估药物在器官内的富集区域及代谢情况。利用DGIdb数据库(https://dgidb.org/)查询了候选药物与基因的互作情况,选取相互作用得分高于0.5的药物-基因作用对,研究其发生相互作用的类型、基因名称及基因功能,该相互作用得分是根据已有的文献和实验数据综合计算得出的[20]。之后利用NCBI数据库(https://www.ncbi.nlm.nih.gov/)确定了与候选药物发生相互作用的基因在器官与组织中的分布情况(表 2),进一步确定了药物在器官内可能富集的区域。
| 表 2 候选药物与基因相互作用情况 Table 2 Interactions of candidate drugs and genes |
SARS-CoV-2已在全球范围内造成大规模感染,2020年3月11日,世界卫生组织正式将新型冠状病毒感染列为全球大流行病。SARS-CoV-2通过呼吸道进入患者体内,损伤呼吸道黏膜,进而对呼吸系统的正常运行造成影响,使患者出现咳嗽、呼吸困难等一系列的身体不良反应。除此之外随着病毒在体内入侵范围的扩大,可能会对患者的神经系统、消化系统、心血管造成一系列损伤,严重者可危及生命[21]。COVID-19的后遗症也不可小觑,有研究表明在感染3个月后,患者仍有不同程度的疲劳、头痛、味觉/嗅觉丧失等症状[22],且后遗症的严重程度与初次感染的病情严重程度高度相关[23]。目前国内疫苗接种率已超过90%,可有效降低重症率与致死率,但随着病毒的持续变异,其传染性逐渐增强,致使疫苗的防感染率下降,故亟需开发针对COVID-19的特效药物,在感染初期迅速降低病毒载量,最大程度上降低对健康的损害。口服小分子药物与其他药物相比具有突出优势:①给药方式简单,患者顺应性强;②储存、运输条件更易满足;③生产成本更低,因此小分子药物有望成为治疗COVID-19的关键手段,具有重要的研究价值。
在已知受体和药物结构的前提下,分子对接是虚拟筛选药物的经典策略,近年来在计算机辅助药物设计领域得到了广泛的应用,如对生物分子复合物结构的预测和针对某一特定靶点的药物筛选等[24]。虚拟筛选能够从大量的小分子中快速筛选出具有成药性的活性化合物,大大降低了通过实验筛选化合物的盲目性,并且可以缩短研究周期,降低药物开发成本。随着深度学习技术的发展,根据氨基酸序列预测蛋白质三维结构的准确性也逐步提高,而基于深度学习的模型Alphafold2[25]凭借其超过90%的准确率为蛋白质结构预测提供了更多可能性,其预测出的蛋白结构可作为分子对接的靶标,大大推动了药物开发的进程。
目前已有部分研究利用分子对接与虚拟筛选方式,以COVID-19的关键蛋白为靶点筛选潜在的治疗药物,如Linda E.等[26]利用机器学习和药效团建模两种方法从草药数据库HerbalDB中筛选出先导药物,再利用分子对接方式进一步筛选;Ahmed A.等[27]利用15种抗丙肝病毒药物与3CL蛋白酶对接并进行分子动力学模拟,发现Sofosbuvir与蛋白的活性位点结合较为稳定。
本研究筛选出的靶向不同突变类型S蛋白的药物,其结合位点大部分集中在S蛋白与ACE2结合的活性位点附近,理论上对S蛋白具有一定的抑制能力。筛选出的靶向3CL蛋白酶的药物结合位点均集中在第130-200位的残基附近,层次聚类分析也显示出候选药物与Nirmatrelvir物理化学性质的相似性,侧面验证了候选药物可对3CL蛋白酶的活性起到抑制作用。
此外本研究也对候选药物与体内基因的相互作用进行了分析,结果见表 2。与候选药物发生相互作用的基因,其编码的蛋白涉及到多种酶、细胞表面受体、转运、转录,能够参与人体的多种生化过程。与Abarelix发生相互作用的KLK3基因在前列腺中特异性高表达,该基因表达产物为在前列腺上皮细胞中合成的蛋白酶,可用于前列腺癌的诊断。Cetrorelix能够与HSPD1发生相互作用,该基因在全身范围内均有表达,在肾上腺、肾脏内均有较高的表达量,其编码的线粒体蛋白可以作为先天免疫系统中的信号分子,对于线粒体中新输入蛋白质的折叠和组装起到重要作用。Goserelin与LHCGR之间存在一定的相互作用,该基因编码促黄体激素和绒毛膜促性腺激素的受体,该受体属于G蛋白偶联受体1家族,其活性由激活腺苷酸环化酶的G蛋白介导。明确基因在体内高表达的区域可为候选药物在人体内的代谢过程提供一定参考。
4 结论本文基于分子对接方法,从2 726个经FDA批准上市的小分子药物中筛选治疗新型冠状病毒感染的潜在药物,针对不同种类的S蛋白筛选出了阿巴瑞克(Abarelix)、西曲瑞克(Cetrorelix)、鞣酸(Tannic acid)三种结合情况较好的候选药物;针对3CL蛋白酶,戈舍瑞林(Goserelin)具有较好的结合能力。此外将靶向3CL蛋白酶的候选药物与辉瑞公司开发的Nirmatrelvir做了对比,发现结合能较低的候选药物与Nirmatrelvir具有相似的结合位点,验证了虚拟筛选药物的可行性。本研究可为新型冠状病毒特效药物的快速发现提供参考。
| [1] |
SHI Yu, WANG Gang, CAI Xiaopeng, et al. An overview of COVID-19[J]. Journal of Zhejiang University Science, 2020, 21(5): 343-360. DOI:10.1631/jzus.b2000083 (  0) 0) |
| [2] |
SUN Xinjuan, WANG Tianyuan, CAI Dayong, et al. Cytokine storm intervention in the early stages of COVID-19 pneumonia[J]. Cytokine & Rowth Factor Reviews, 2020, 53: 38-42. DOI:10.1016/j.cytogfr.2020.04.002 (  0) 0) |
| [3] |
BRODIN P, CASARI G, TOWNSEND L, et al. Studying severe long COVID to understand post-infectious disorders beyond COVID-19[J]. Nature Medicine, 2022, 28(5): 879-882. DOI:10.1038/s41591-022-01766-7 (  0) 0) |
| [4] |
MORSE J S, LALONDE T, XU S, et al. Learning from the Past: Possible urgent prevention and treatment options for severe acute respiratory infections caused by 2019-nCoV[J]. ChemBioChem, 2020, 21(5): 730-738. DOI:10.1002/cbic.202000047 (  0) 0) |
| [5] |
SHEREEN M A, KHAN S, KAZMI A, et al. COVID-19 infection: Emergence, transmission, and characteristics of human coronaviruses[J]. Journal of Advanced Research, 2020, 24: 91-98. DOI:10.1016/j.jare.2020.03.005 (  0) 0) |
| [6] |
WANG Xueqing, GUAN Yuanfang. COVID-19 drug repurposing: A review of computational screening methods, clinical trials, and protein interaction assays[J]. Medicinal Research Reviews, 2021, 41(1): 5-28. DOI:10.1002/med.21728 (  0) 0) |
| [7] |
ALLAM A E, AMEN Y, ASHOUR A, et al. In silico study of natural compounds from sesame against COVID-19 by targeting M pro, PL pro and RdRp[J]. RSC Advances, 2021, 11(36): 22398-22408. DOI:10.1039/D1RA03937G (  0) 0) |
| [8] |
WISHART D S, FEUNANG Y D, GUO A, et al. DrugBank 5.0: A major update to the DrugBank database for 2018[J]. Nucleic Acids Research, 2018, 46(D1): D1074-D1082. DOI:10.1093/nar/gkx1037 (  0) 0) |
| [9] |
YUAN Shuguang, CHAN H C S, HU Zhenquan. Using PyMOL as a platform for computational drug design[J]. Wiley Interdisciplinary Reviews: Computational Molecular Science, 2017, 7(2): 1-10. DOI:10.1002/wcms.1298 (  0) 0) |
| [10] |
YOSHIKAWA N, HUTCHISON G R. Fast, efficient fragment-based coordinate generation for open babel[J]. Journal of Cheminformatics, 2019, 11(1): 1-9. DOI:10.1186/s13321-019-0372-5 (  0) 0) |
| [11] |
NGUYEN N T, NGUYEN T H, PHAM T, et al. Autodock vina adopts more accurate binding poses but autodock4 forms better binding affinity[J]. Journal of Chemical Information and Modeling, 2019, 60(1): 204-211. DOI:10.1021/acs.jcim.9b00778 (  0) 0) |
| [12] |
FAN Jiyu, FU Ailing, ZHANG Le. Progress in molecular docking[J]. Quantitative Biology, 2019, 7(2): 83-89. DOI:10.1007/s40484-019-0172-y (  0) 0) |
| [13] |
TROTT O, OLSON A J. AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading[J]. Journal of Computational Chemistry, 2010, 31(2): 455-461. DOI:10.1002/jcc.21334 (  0) 0) |
| [14] |
WANG Cunxin, XU Xianjin. Docking-based inverse virtual screening approach[J]. Journal of Beijing University of Technology, 2019, 45(11): 1164-1172. DOI:10.11936/bjutxb2019060001 (  0) 0) |
| [15] |
MOUSSA M, PAPATSORIS A, DELLIS A, et al. Current and emerging gonadotropin-releasing hormone (GnRH) antagonists for the treatment of prostate cancer[J]. Expert Opinion on Pharmacotherapy, 2021, 22(17): 2373-2381. DOI:10.1634/theoncologist.5-2-162 (  0) 0) |
| [16] |
TEPEKOY F, UYSAL F, ACAR N, et al. The effect of GnRH antagonist cetrorelix on Wnt signaling members in pubertal and adult mouse ovaries[J]. Histochemistry and Cell Biology, 2019, 152: 423-437. DOI:10.1007/s00418-019-01817-0 (  0) 0) |
| [17] |
WANG M, SEPTIER C, BRIGNOT H, et al. Astringency sensitivity to tannic acid: Effect of ageing and saliva[J]. Molecules, 2022, 27(5): 1617. DOI:10.3390/molecules27051617 (  0) 0) |
| [18] |
WANG Siyuan, PEI Lin, HU Taobo, et al. Protective effect of goserelin on ovarian reserve during (neo) adjuvant chemotherapy in young breast cancer patients: A prospective cohort study in China[J]. Human Reproduction, 2021, 36(4): 976-986. DOI:10.1093/humrep/deaa349 (  0) 0) |
| [19] |
LASKOWSKI R A, SWINDELLS M B. LigPlot+: Multiple ligand-protein interaction diagrams for drug discovery[J]. Journal of Chemical Information and Modeling, 2011, 51(10): 2778-2786. DOI:10.1021/ci200227u (  0) 0) |
| [20] |
FRESHOUR S L, KIWALA S, COTTO K C, et al. Integration of the drug-gene interaction database (DGIdb 4.0) with open crowdsource efforts[J]. Nucleic Acids Research, 2020, 49(D1): D1144-D1151. DOI:10.1093/nar/gkaa1084 (  0) 0) |
| [21] |
WENG Linman, SU Xuan, WANG Xueqiang. Pain symptoms in patients with coronavirus disease (COVID-19): A literature review[J]. Journal of Pain Research, 2021, 14: 147-159. DOI:10.2147/JPR.S269206 (  0) 0) |
| [22] |
THE LANCET. Understanding long COVID: A modern medical challenge[J]. The Lancet, 2021, 398(10302): 725. DOI:10.1016/S0140-6736(21)01900-0 (  0) 0) |
| [23] |
LOPEZ L S, WEGMAN O T, PERELMAN C, et al. More than 50 long-term effects of COVID-19: A systematic review and meta-analysis[J]. Scientific Reports, 2021, 11(1): 1-12. DOI:10.1038/s41598-021-95565-8 (  0) 0) |
| [24] |
MENG Xuanyu, ZHANG Hongxing, MEZEI M, et al. Molecular docking: A powerful approach for structure-based drug discovery[J]. Current Computer Aided-Drug Design, 2011, 7(2): 146-157. DOI:10.2174/157340911795677602 (  0) 0) |
| [25] |
CRAMER P. AlphaFold2 and the future of structural biology[J]. Nature Structural & Molecular Biology, 2021, 28(9): 704-705. DOI:10.1038/s41594-021-00650-1 (  0) 0) |
| [26] |
ERLINA L, PARAMITA R I, KUSUMA W A, et al. Virtual screening of Indonesian herbal compounds as COVID-19 supportive therapy: machine learning and pharmacophore modeling approaches[J]. BMC Complementary Medicine and Therapies, 2022, 22(1): 1-35. DOI:10.21203/rs.3.rs-29119/v1 (  0) 0) |
| [27] |
AL-KARMALAWY A A, ALNAJJAR R, DAHAB M, et al. Molecular docking and dynamics simulations reveal the potential of anti-HCV drugs to inhibit COVID-19 main protease[J]. Pharmaceutical Sciences, 2021, 27(Covid-19): S109-S121. DOI:10.3389/fchem.2021.661230 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


