识别复杂性状和疾病间遗传关联有助于确定疾病的危险因素。人类的复杂性状如体重、BMI、腰围、认知能力等对疾病的易感状态受遗传因素控制。目前已有一些软件可以识别复杂性状和疾病间的遗传相关性并探索其间是否存在因果关联。例如:ldsc (https://github.com/bulik/ldsc) [1], TwosampleMR和MR-BMA (https://github.com/verena-zuber/demo_AMD)等[2]。
ldsc是基于Linux系统设计的命令行工具,用于从GWAS汇总统计中估计遗传力和遗传相关性。目前该工具广泛应用于遗传关联相关分析[1, 3]。另外,ldsc也可用于计算LD分数。TwosampleMR是基于R语言开发的软件包,它基于孟德尔随机化理论,主要应用于表型间的因果关联分析。孟德尔随机化(MR)遵循“亲代等位基因随机分配给子代”的孟德尔遗传规律,使用基因型作为工具变量来推断表型间的因果关联。孟德尔随机化基于三个假设:1)工具变量与暴露显著相关;2)工具变量不应该与暴露的混杂因素相关;3)工具变量只通过暴露影响结局。第二个和第三个假设也被称为独立性假设[4]。更进一步地,多变量MR是标准MR框架的扩展,用于在单个模型中考虑多个潜在风险因素。然而,当前多变量MR的实现使用标准线性回归,因此在许多风险因素下表现不佳。MR-BMA是基于Bayesian model averaging方法开发的双样本多变量MR的方法,已被开发为一个R包,它可以弥补以上缺点。
虽然以上的三种工具均已在研究中广泛使用,但对于实现识别复杂性状和疾病间遗传关联仍具有一些限制性。首先,这三个工具是基于不同的系统环境,因此对于对R或Linux不熟悉的用户不够友好,在工具使用上有困难。其次,三种工具虽然功能强大,但是函数很多,对于以识别复杂性状和疾病间遗传关联为目地的分析来说,有很多函数是冗余的。最后,目前还没有工具和流程整合这些工具用于识别复杂性状和疾病间遗传关联。
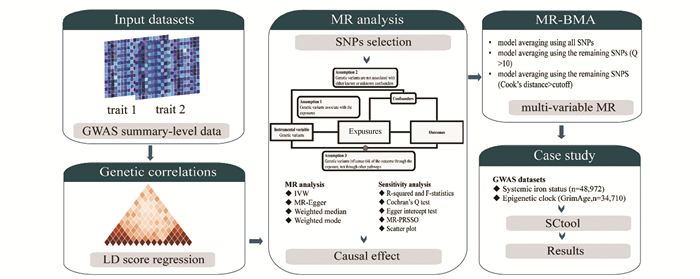
因此,针对以上存在的问题,本研究开发了SCtool,一个开源、跨平台和用户友好的软件工具,用于识别复杂性状和疾病间遗传关联的工具。本研究的流程图见图 1。
|
图 1 本研究的流程图 Figure 1 Flowchart of this study |
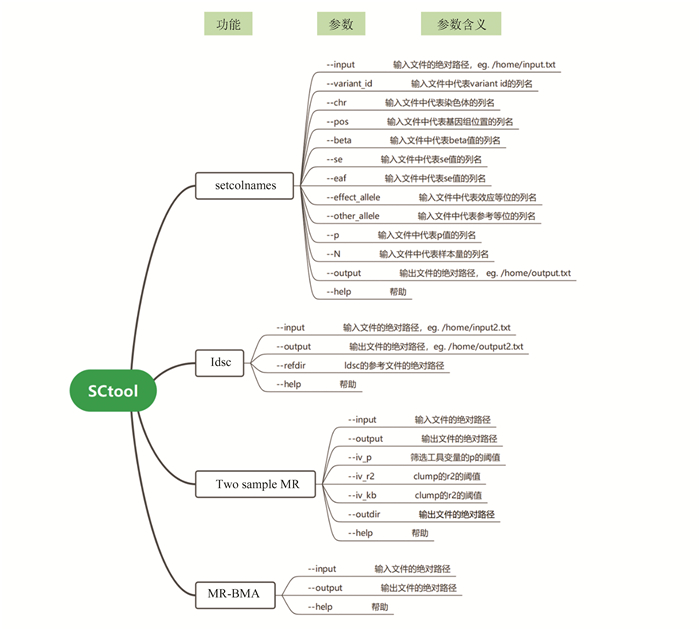
SCtool是基于Linux系统的命令行工具(图 2)。该工具的使用需要安装ldsc和R包TwosampleMR,MR-BMA已被内置在SCtool中,无需安装。该工具的输入数据为GWAS汇总统计数据,主要分为以下三个步骤:第1步,利用ldsc计算复杂性状与疾病间的遗传相关性;第2步,基于MR理论识别复杂性状与疾病间的因果关联;第3步,基于MR-BMA进行多变量MR,考虑多个变量共同作用对结局的影响,并可证明第2步是否具有稳健性。SCtool的功能、参数及参数含义见图 2。
|
图 2 SCtool工具的功能、参数及参数含义 Figure 2 SCtool Function, parameters, and parameter meanings |
利用ldsc命令行软件识别复杂性状与疾病间的遗传相关性。分为以下两个步骤:第1步,利用munge_sumstats.py检查输入的汇总统计文件中列出的等位基因是否与用于估计LD分数的数据中列出的等位基因相符。有时少数等位基因会不匹配;这通常表明SNP的标记有误。这个步骤中有一处筛选条件需要注意,在log文件中如果平均卡方小于1.02,则说明这些数据不适用于LD score回归,无需进行下一步分析[5]。另外,该步骤将输入的汇总文件转换为.sumstats.gz格式的文件作为下一步骤的输入文件;第2步,以上一步的输出文件作为这一步的输入文件,利用ldsc.py计算二者间的遗传相关性,并分别计算其遗传力。SCtool将以上步骤整合,并可通过一条命令实现,使用方法如下所示:SCtool ldsc --input ./input.txt --output ./output.txt --refdir ./refdir。
1.3 识别复杂性状与疾病间的因果关联为了确保工具变量的有效性,需要满足MR分析的三个主要假设。第1步,为了满足第一个假设,使用Bonferroni校正SNP的数量[6-8],在全基因组显着阈值(P<5.0×10-8)下过滤与暴露密切相关的SNP。该阈值已成为GWAS统计显着性的标准,特别是对于欧洲人[9]。为了尽量减少LD产生的混杂因素的影响,r2<0.01的SNP[5]。回文SNP被消除以避免偏差。最后,计算R平方统计量和F统计量以过滤弱工具变量(F<10)。作为敏感性分析,计算方法如下:R2=(2β2×MAF×(1-MAF))/(2β2×MAF×(1-MAF)+2N×MAF×(1-MAF)×SE2),F=(R2×(N-2))/(1-R2)(MAF, 最小等位基因频率; N, 样本量大小; SE, 标准误; β, SNP在GWAS暴露中的效应估计值)。第2步,进行MR分析。MR研究越来越多地用于提供证据,以评估随机临床试验中尚未确定因果关系的治疗的潜在有效性我们采用乘法随机效应逆方差加权(IVW)MR作为MR分析的主要方法。IVW MR结合了暴露通过遗传变异对结果的遗传预测影响。第3步,进行敏感性分析。采用MR-Egger、加权中位数和加权模数法作为敏感性分析,验证IVW MR的可靠性。Cochran的Q检验用于确定所选工具变量中是否存在异质性。此外,应用MR-PRESSO检测水平多效性和异常值。
SCtool将以上步骤整合,并可通过一条命令实现,使用方法如下所示:SCtool MR --input ./input.txt --ourdir ./outputdir --refdir ./refdir --iv_p 5e-8 --iv_r2 0.01 --iv_kb 5000。
1.4 MR-BMA分析为了避免当前基于线性回归的MR方法的局限性并进一步加强结果的可靠性,本研究执行了一种基于贝叶斯模型平均(MR-BMA)的新方法[2]。MR-BMA利用了一个多变量框架,当满足两个条件时,模型中可以包含多个暴露:1)它们都与模型中使用的至少一个SNP工具紧密相关;2)它们不会导致多重共线性。模型的后验概率(PP)使用独立先验和closed-form贝叶斯因子估计。此外,还计算了每个风险因素的边际纳入概率(MIP),定义为存在风险因素的情况下所有模型的PP之和。利用每个模型的模型平均因果效应(MACE)来比较风险因素或解释方向。最终,根据每个模型的PP值排名(阈值设置为0.02)优先选择最佳模型。Q统计量用于检测无效工具作为模型中的异常值[10]。库克距离(Cd)被用来量化有影响力的遗传变异[11]。本研究在过滤异常值和有影响力的变体后重新运行分析。
SCtool将以上步骤整合,并可通过一条命令实现,使用方法如下所示:SCtool MRBMA --input ./input.txt-output ./output.txt。
1.5 案例分析本研究应用的其中一组数据来自Iron Status Genetics Consortium的衡量全身性铁状态的数据,其中包括(血清铁、铁蛋白、转铁蛋白饱和度和转铁蛋白)[12]。参与研究的样本数为48 972,均为欧洲人群。
另一组数据为衡量表观遗传年龄加速的表观遗传时钟相关的数据集,该表观遗传时钟称为GrimAge[12-13]。该数据集的参与者共计34 710,均为欧洲人群。
具体将SCtool应用于该案例的代码如下所示,分别对应上述的三个分析:1)SCtool ldsc --input GrimAge.txt xqt.txt tdb.txt ztdb.txt ztdbb.txt --output ./ld_output.txt --refdir ./refdir;2)SCtool MR --input ./input.txt xqt.txt tdb.txt ztdb.txt ztdbb.txt --ourdir ./mr_outputdir --refdir ./refdir --iv_p 5e-8 --iv_r2 0.01 --iv_kb 5000;3)SCtool MRBMA --input ./input.txt xqt.txt tdb.txt ztdb.txt ztdbb.txt -output ./mrbma_output.txt。其中GrimAge.txt,xqt.txt,tdb.txt,ztdb.txt,ztdbb.txt分别代表GrimAge、血清铁、铁蛋白、转铁蛋白和转铁蛋白饱和度的文件名。三条命令即可完成分析,大大减少了命令行的使用,对用户更加友好。
2 结果与分析 2.1 遗传相关性所有数据的平均卡方均大于1.02,没有数据被过滤掉。结果如表 1所示,GrimAge与四种铁状态的遗传相关性都不显著相关(P>0.05)。
| 表 1 全身性铁状态与表观遗传时钟的遗传相关性 Table 1 Genetic correlation between GrimAge and systemic iron status |
本研究确定了与铁蛋白,血清铁,转铁蛋白和转铁蛋白饱和度相关的工具变量。为了尽量减少LD产生的混杂因素的影响,去除了一些SNP(r2<0.01)。铁蛋白暴露数据集中的rs411988在harmonized时被删除。这些SNP的所有F统计量均大于10。此外,没有一个SNP作为回文序列被过滤掉。这些SNPs被用作进一步MR分析的工具变量。
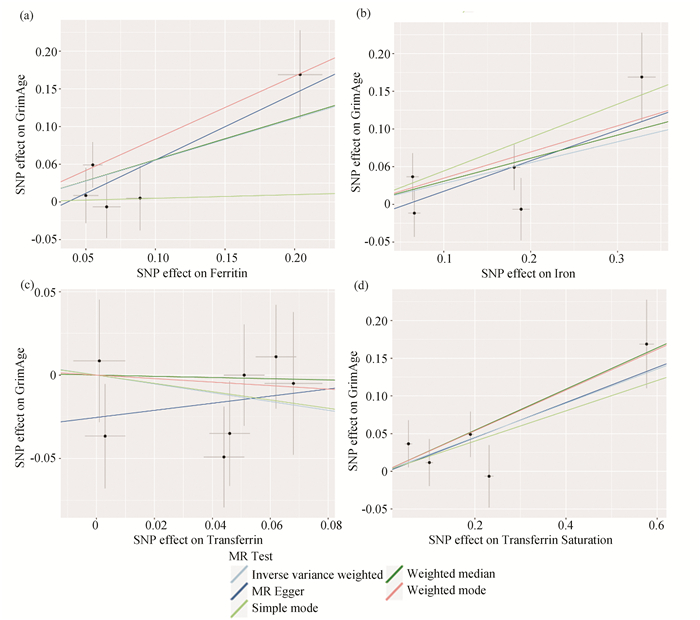
GrimAge与全身性铁状态的IVW MR结果如表 2所示。我们发现铁蛋白作为暴露会显著加速GrimAge (IVW beta = 0.56, 95% CI 0.15-0.95, p = 6.79×10-3)。总的来说,铁蛋白每增加1SD可以使GrimAge这种表观遗传时钟增加0.56年。转铁蛋白饱和度每增加1SD会使GrimaAge显著增加0.23年(IVW beta = 0.23, 95% CI 0.08-0.37, p = 2.36×10-3)。血清铁与GrimAge的因果关联也具有显著性(beta = 0.28, 95% CI 0.05-0.50, p = 1.59×10-2 > 0.012 5)。但是,对于转铁蛋白与GrimAge之间的因果关联并不显著(P>0.05)。以上的主要分析的估计效应方向与敏感性分析的方向相同(MR-Egger,加权中位数和simple mode)(图 3)。
| 表 2 GrimAge与全身性铁状态的IVW MR结果 Table 2 The results of IVW MR between GrimAge and systemic iron status |
|
图 3 MR检验:(a)铁蛋白;(b)血清铁;(c)转铁蛋白;(d)和转铁蛋白饱和度对GrimAge的影响 Figure 3 MR tests: Scatter plot visualizing the effect of genetic instruments on (a) Ferritin; (b) Serum iron; (c) Transferrin, and (d) Transferrin saturation against their effects on GrimAge 注:浅蓝色直线代表Inverse variance weighted方法; 深蓝色直线代表MR Egger方法; 浅绿色直线代表Simple mode方法; 深绿色代表Weighted median方法; 粉色直线代表Weighted mode方法.(彩图见电子版:http://swxxx, alljournals.cn/ch/index.aspx.2024年第22卷第1期). |
接下来,为了确保因果关联分析的稳健性,本研究进行了其它的敏感性分析。Cochran's Q statistic[14]和MR-PRESSO[15]显示没有异质性存在(所有P >0.05)。此外,MR-Egger截距检验显示没有水平多效性存在。总的来说,敏感性分析表明以上的IVW MR的结果是相对稳健的。
2.3 MR-BMA分析为了进一步验证给定多效性结果的稳健性,采用MR-BMA分析来评估全身铁状态与表观遗传年龄加速之间的因果效应(表 3)。最初使用12个遗传变量进行MR-BMA,这些遗传变量由四个铁状态标记的工具变量的联合集组成。没有过滤掉任何SNP,因为没有SNP的Q统计值超过10。最后,在筛选出库克距离超过阈值的SNP后,分别利用9、8、8和10个SNPs对表观遗传钟上的4个铁状态生物标志物进行MR-BMA。总的来说,MR-BMA估计的铁蛋白对GrimAge的因果效应与MR IVW结果一致,转铁蛋白饱和度对GrimAge也是如此。
| 表 3 MR-BMA估计系统铁状态与GrimAge之间的因果关系 Table 3 MR-BMA estimate of the causal relationship between systemic iron status and GrimAge |
总的来说,SCtool以GWAS汇总数据作为输入,通过整合ldsc, TwosampleMR和MR-BMA,能够识别复杂性状和疾病间的遗传关联,是一个用户友好的、跨平台的整合工具。
如案例分析所示,通过使用SCtool揭示了全身性铁状态与表观遗传时钟GrimAge之间的遗传关联。结果显示GrimAge与铁蛋白,血清铁,转铁蛋白和转铁蛋白饱和度没有显著的遗传相关性。另外,MR和MR-BMA的结果显示铁蛋白、转铁蛋白饱和度和血清铁会加速表观遗传年龄衰老。该结论与之前的研究报道的结果相符[16-21]。
SCtool也具有局限性。目前它只整合了三个工具,在未来的研究中我们将添加更多的功能,进一步识别复杂性状与疾病间的共享位点。另外,我们将会纳入更多数据集用于混杂因素的识别,例如,BMI、血压、血脂、血糖、身高和体重等常见的性状。
4 结论本研究开发了SCtool, 一个用户友好、跨平台的工具。该工具整合了ldsc, TwosampleMR和MR-BMA以识别复杂性状和疾病间的遗传关联(遗传相关性和因果关联)。
| [1] |
BULIK-SULLIVAN B K, LOH P R, FINUCANE H K, et al. LD Score regression distinguishes confounding from polygenicity in genome-wide association studies[J]. Nature Genetics, 2015, 47(3): 291-295. DOI:10.1038/ng.3211 (  0) 0) |
| [2] |
ZUBER V, COLIJN J M, KLAVER C, et al. Selecting likely causal risk factors from high-throughput experiments using multivariable Mendelian randomization[J]. Natrue Communnication, 2020, 11(1): 29. DOI:10.1038/s41467-019-13870-3 (  0) 0) |
| [3] |
BULIK-SULLIVAN B, FINUCANE H K, ANTTILA V, et al. An atlas of genetic correlations across human diseases and traits[J]. Nature Genetics, 2015, 47(11): 1236-1241. DOI:10.1038/ng.3406 (  0) 0) |
| [4] |
BURGESS S, BUTTERWORTH A, THOMPSON S G. Mendelian randomization analysis with multiple genetic variants using summarized data[J]. Genetic Epidemiology, 2013, 37(7): 658-665. DOI:10.1002/gepi.21758 (  0) 0) |
| [5] |
ALEXANDER M, CURTIS D. LD scores are associated with differences in allele frequencies between populations but LD score regression can still distinguish confounding from polygenicity[J]. Annals of Humman Genetics, 2020, 84(5): 412-416. DOI:10.1111/ahg.12370 (  0) 0) |
| [6] |
DUDBRIDGE F, GUSNANTO A. Estimation of significance thresholds for genomewide association scans[J]. Genetic Epidemiology, 2008, 32(3): 227-234. DOI:10.1002/gepi.20297 (  0) 0) |
| [7] |
THE WELLCOME TRUST CASE CONTROL C. Genome-wide association study of 14 000 cases of seven common diseases and 3 000 shared controls[J]. Nature, 2007, 447(7145): 661-678. DOI:10.1038/nature05911 (  0) 0) |
| [8] |
PE'ER I, YELENSKY R, ALTSHULER D, et al. Estimation of the multiple testing burden for genomewide associationstudies of nearly all common variants[J]. Genetic Epidemiology, 2008, 32(4): 381-385. DOI:10.1002/gepi.20303 (  0) 0) |
| [9] |
BARSH G S, COPENHAVER G P, GIBSON G, et al. Guidelines for genome-wide association studies[J]. PLoS Genetics, 2012, 8(7): e1002812. DOI:10.1371/journal.pgen.1002812 (  0) 0) |
| [10] |
HUANG L, LI L, LUO X, et al. The association between serum iron status and risk of asthma: A 2-sample Mendelian randomization study in descendants of Europeans[J]. American Journal of Clinical Nutrition, 2019, 110(4): 959-968. DOI:10.1093/ajcn/nqz162 (  0) 0) |
| [11] |
ELEDUM H. Leverage and influentialobservations on the Liu type estimator in the linear regression model with the severe collinearity[J]. Heliyon, 2021, 7(8): e07792. DOI:10.1016/j.heliyon.2021.e07792 (  0) 0) |
| [12] |
BENYAMIN B, ESKO T, RIED J S, et al. Novel loci affecting iron homeostasis and their effects in individuals at risk for hemochromatosis[J]. Nature Communnication, 2014, 5: 4926. DOI:10.1038/ncomms5926 (  0) 0) |
| [13] |
MCCARTNEY D L, MIN J L, RICHMOND R C, et al. Genome-wide association studies identify 137 genetic loci for DNA methylation biomarkers of aging[J]. Genome Biology, 2021, 22(1): 194. DOI:10.1186/s13059-021-02398-9 (  0) 0) |
| [14] |
BOWDEN J, SPILLER W, DEL GRECO M F, et al. Improving the visualization, interpretation and analysis of two-sample summary data Mendelian randomization via the Radial plot and Radial regression[J]. International Journal of Epidemiology, 2018, 47(4): 1264-1278. DOI:10.1093/ije/dyy101 (  0) 0) |
| [15] |
VERBANCK M, CHEN C Y, NEALE B, et al. Detection of widespread horizontal pleiotropy in causal relationships inferred from Mendelian randomization between complex traits and diseases[J]. Nature Genetics, 2018, 50(5): 693-698. DOI:10.1038/s41588-018-0099-7 (  0) 0) |
| [16] |
CASALE G, BONORA C, MIGLIAVACCA A, et al. Serum ferritin and ageing[J]. Age and Ageing, 1981, 10(2): 119-122. DOI:10.1093/ageing/10.2.119 (  0) 0) |
| [17] |
ZECCA L, YOUDIM M B, RIEDERER P, et al. Iron, brain ageing and neurodegenerative disorders[J]. Nature Reviews: Neuroscience, 2004, 5(11): 863-873. DOI:10.1038/nrn1537 (  0) 0) |
| [18] |
JOHNSON M A, FISCHER J G, BOWMAN B A, et al. Iron nutriture in elderly individuals[J]. FASEB Journal, 1994, 8(9): 609-621. DOI:10.1096/fasebj.8.9.8005389 (  0) 0) |
| [19] |
GARRY P J, HUNT W C, BAUMGARTNER R N. Effects of iron intake on iron stores in elderly men and women: longitudinal and cross-sectional results[J]. Journal of The American College of Nutrition, 2000, 19(2): 262-269. DOI:10.1080/07315724.2000.10718925 (  0) 0) |
| [20] |
WANG L, ZHOU Q, CHEN L, et al. Iron-mediated lysosomal-mitochondrial crosstalk: A new direction in the treatment of aging and aging-related diseases[J]. Acta Biochim Biophys Sin (Shanghai), 2020, 52(11): 1293-1295. DOI:10.1093/abbs/gmaa115 (  0) 0) |
| [21] |
SMITH M A, HARRIS P L, SAYRE L M, et al. Iron accumulation in Alzheimer disease is a source of redox-generated free radicals[J]. Proceedings of the National Academy of Sciences of the United States of America, 1997, 94(18): 9866-9868. DOI:10.1073/pnas.94.18.9866 (  0) 0) |
 2024, Vol. 22
2024, Vol. 22


