2. 哈尔滨医科大学 生物信息科学与技术学院,哈尔滨 150076
2. College of bioinformatics science and technology, Harbin Medical University, Harbin 150076, China
登革热,是登革病毒(Dengue virus, DENV)经蚊媒传播引起的急性传染病,被认为是最重要的虫媒传染病。全世界每年约有3.9亿人感染登革热,波及100多个国家,其中以菲律宾,越南,泰国,马来西亚等东南亚国家和南美洲的巴西最为严重[1]。而我国发生登革热爆发或流行及本地感染病例的地区有广东,广西,海南,浙江等,因此登革热成为了危害大众健康的传染性疾病之一[2]。在目前的研究中,登革病毒的实验室诊断方法存在着对设备要求高,操作繁琐,诊断时间长,精度差等缺点。因此,从分子层面寻找准确高效的登革热诊断标志物意义重大。
竞争性内源RNA(Competing endogenous RNA, ceRNA)网络在复杂疾病的生物学功能中起着至关重要的作用。有研究表明,通过比较正常细胞和肾透明细胞癌细胞中的ceRNA网络来检测失调的ceRNA相互作用,可有助于研究肾透明细胞癌的发病机制和识别新的生物标志物[3]。此外,也有研究表明ceRNA网络失衡以及分子之间的相互调控在骨关节炎中扮演着重要的角色。为骨关节炎的治疗提供新的见解[4]。目前登革热相关的失调ceRNA研究却很少。基因治疗是当前研究疾病的热门,也是未来的疾病治疗的趋势之一,ceRNA的存在可能影响疾病的产生和发展,并能成为初次确诊、预后评估的标准以及疾病治疗的目标。
本研究基于ceRNA调控机制,结合登革热表达数据,通过构建登革热相关ceRNA失调网络,对网络进行模块挖掘与富集分析,解析模块性质,从而发现其相关的功能。探究登革热对ceRNA调控作用的影响及其相关机制,为该疾病的诊断与治疗提供指导。
1 材料和方法 1.1 材料的获取与处理 1.1.1 表达谱数据的获取和处理基因表达芯片数据和登革热外周血样本数据从GEO数据库下载,数据编号为GSE51808, GSE96656。通过标准化和离群样本处理,选择GSE51808中18例登革热样本、19例恢复期样本、9例正常对照样本以及GSE96656中31例登革热样本、9例正常对照样本进行研究。
1.1.2 人类miRNA靶基因数据的获取StarBase数据库是一个由高通量实验数据CLIP-Seq和mRNA测序数据支持的miRNA靶标数据库[5]。本文从StarBase数据库中得到了人类的miRNA-mRNA数据,其中包括了423 976条互作对,386个miRNA和13 802个mRNA。
1.1.3 人类免疫相关基因数据的获取Immport数据库由四个组件组成:包括私有数据、共享数据、数据分析和资源。用于数据存档,传播,分析和重复使用[6]。从中下载2 498个人类相关的免疫基因。
1.1.4 登革热相关病毒蛋白数据的获取HVPPI数据库是一个综合了人类宿主—病毒蛋白质互作关系的数据库[7],能够用于解析复杂疾病和病毒感染之间的关系。从其中下载了8 410条登革热与人类宿主蛋白质之间的互作关系对,标准化后得到2 941个登革热病毒蛋白基因。
1.1.5 蛋白质互作数据的获取STRING数据库是一个蛋白质相互作用分析数据库,可用于对目的蛋白质进行检索,并绘制出相关蛋白质-蛋白质互作(Protein-Protein Interaction,PPI)的综合网络[8]。从其中下载了蛋白质网络数据和注释信息,结合HVPPI数据库中登革热病毒关联的宿主蛋白进行分析。
1.2 实验方法 1.2.1 差异表达分析对获得的登革热疾病与健康组表达数据,使用R语言中的limma包进行差异表达分析,选取阙值为|logFC|>2, FDR < 0.05。筛选出用于实验的差异基因,并用pheatmap包绘制差异热图,ggplot2包绘制火山图,以展示每个差异基因在样本中的表达情况[9]。
1.2.2 ceRNA网络的构建对从StarBase(V2.0)数据库下载的miRNA-mRNA互作数据预处理,经过去重及标准化后存入0/1矩阵进行超几何计算,得到ceRNA关系对矩阵。筛选FDR < 0.05并且共享miRNA数目大于等于3的ceRNA关系对,再与差异基因匹配,得到潜在ceRNA关系对。接着结合登革热疾病样本中的表达数据计算潜在ceRNA分子间的皮尔森相关系数并选取大于0.7以及FDR < 0.05的互作对用于构建ceRNA网络[10-11]。
1.2.3 失调ceRNA网络可视化将获得的失调ceRNA互作关系对投入Cytoscape生成网络。并依据网络中基因的差异表达方向对网络节点颜色进行绘制,接着使用Mcode方法进行模块挖掘,并选定节点数大于10的模块进行下一步分析。
1.2.4 富集分析使用WebGestalt数据库[12]对筛选出的差异表达基因以及网络模块进行富集分析。
1.2.5 ceRNA网络模块基因与登革热病毒蛋白互作分析将HVPPI中的登革热相关宿主蛋白质映射至STRING数据库的PPI网络,联合ceRNA网络模块中的基因,确定模块基因与登革热相关宿主蛋白的互作关系,并统计与各个模块基因直接互作的人类宿主蛋白数量。
2 结果 2.1 登革热差异表达的基因筛选对登革热数据外周血芯片表达数据(GSE51805)中的15例登革热样本,28例健康样本(包含19例恢复期样本和9例正常对照样本)进行差异表达分析,以|logFC|>2, FDR < 0.05进行筛选,共识别出251个差异基因,包括175个上调基因和76个下调基因(见图 1a和1b)。接着对筛选出的差异表达基因使用WebGestalt数据库进行富集分析,发现其主要富集在DNA复制,错配修复以及细胞周期等生物学通路中(见图 1c)。

|
图 1 登革热差异表达分析及富集分析 Figure 1 Dengue fever differential expression analysis and enrichment analysis |
基于miRNA靶基因数据库及共表达分析识别登革热相关失调ceRNA网络,使用Cytoscape进行可视化(见图 2),网络中红色节点表示上调基因,蓝色节点表示下调基因。其中共有179个节点,1 607条边,上调的基因有143个,下调的基因有36个。基因表达上调相关的ceRNA网络中共有143个节点,1 493条边,表达下调相关的ceRNA网络中共有36个节点,114条边。
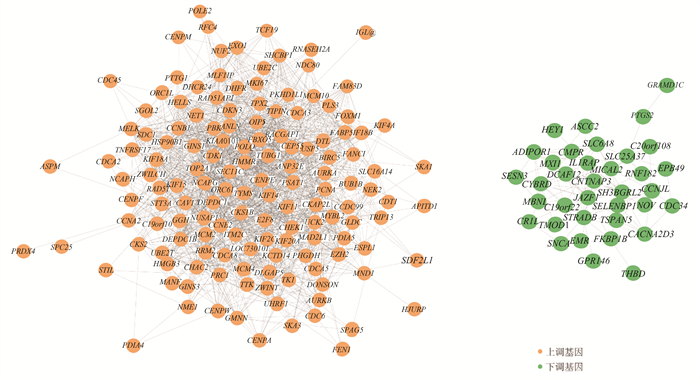
|
图 2 登革热相关ceRNA失调网络 Figure 2 Dengue-associated ceRNA dysregulation network |
在Cytoscape中利用Mcode插件对网络进行模块挖掘,总共得到9个模块,选出节点数目大于10的4个模块(见表 1)。细胞免疫和体液免疫是人体抵御病毒感染的重要途经。本文下载了免疫相关的基因集合,并对各模块和免疫基因进行筛选,发现模块2中的基因与免疫基因的重合数目最高,包括FABP5(Fatty acid binding protein 5), C19orf10(Myeloid derived growth factor), TNFRSF17(TNF Receptor superfamily member 17)等(见表 1)。因此选择模块2展示及后续分析(见图 3a)。
| 表 1 登革热失调ceRNA网络模块属性 Table 1 Attributes of dengue fever dysregulation ceRNA network module |
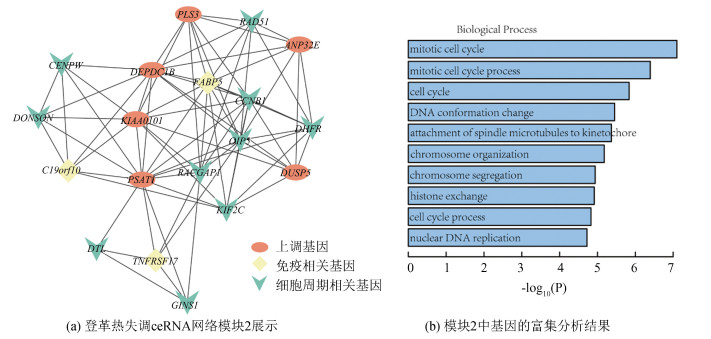
|
图 3 登革热失调ceRNA网络模块展示 Figure 3 Exhibitiaon of dengue fever dysregulation ceRNA network module |
通过WebGestalt网站对模块2基因进行富集分析,结果主要聚集在有丝分裂细胞周期等功能(见图 3b),说明了登革病毒感染人体时会对细胞的活动产生影响,与细胞周期有密切联系。
2.4 ceRNA网络模块的外部数据验证利用GSE96656的登革热外部数据对ceRNA网络模块表达进行验证,通过秩和检验发现外部数据中ceRNA网络模块基因的表达模式与训练数据集中基本保持一致(P < 0.05)。其中KIF2C, CCNB1, DUSP5, PSAT1, RAD51, DEPDC1B, DTL, RACGAP1, GINS1, KIAA0101, OIP5, DONSON, DHFR, C19orf10, FABP5, CENPW基因在感染登革热的血液样本中表达显著上调,表明这些基因具有潜在的登革热诊断效能(见图 4a,4b)。联合HVPPI和STRING数据库分析发现ceRNA网络模块基因能够通过PPI网络与登革热相关宿主基因直接互作,其中ANP32E互作的登革热病毒相关宿主蛋白基因数量最多。结果表明登革热病毒可能通过调控宿主蛋白基因的互作关系从而影响模块基因的表达(见图 4c)。
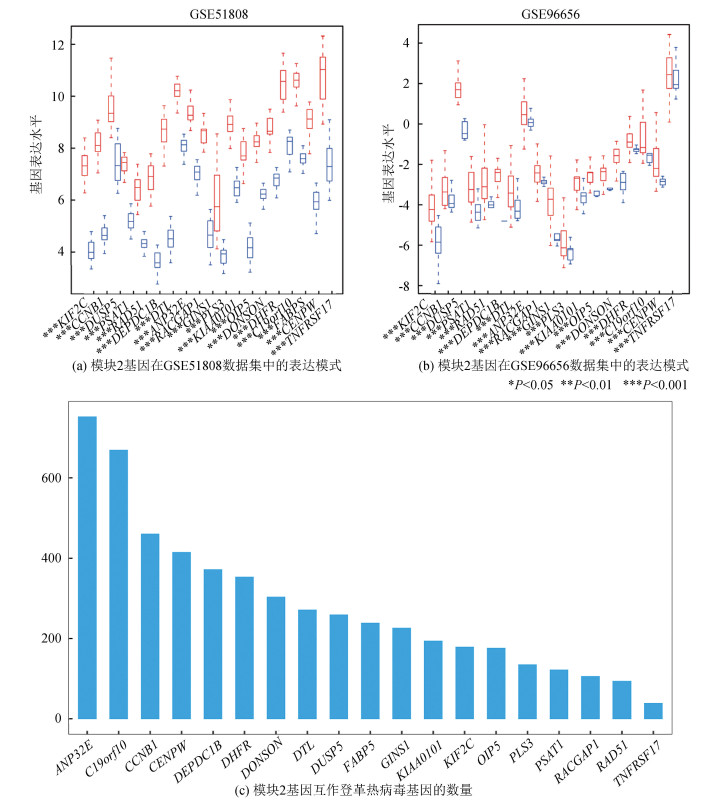
|
图 4 模块2基因在不同数据集中的表达模式及互作登革热病毒基因的数量展示 Figure 4 Module 2 expression patterns of genes in different data sets and the number of interacting dengue virus genes 注:图(a)和图(b)中红色箱式图表示模块2基因在登革热样本中的表达情况,蓝色箱式图表示模块2基因在正常样本中的表达情况。 |
使用公共数据库中的登革热样本外周血液数据,通过生物信息学分析,筛选出登革热差异表达基因并进行功能富集分析,发现DNA复制,细胞周期等生物学通路在登革热患者中显著失调。人体细胞被病毒与细菌等成功入侵后,会引起机体免疫系统应答能力下降,从而导致免疫系统功能异常[13-15]。因此在构建的登革热相关ceRNA网络中筛选出与免疫基因存在交集的网络模块,并且模块基因同时富集到与细胞周期相关的通路中。通过对网络模块的基因表达进行外部数据验证,发现模块中的大多数基因在不同的数据中表达趋势相同,表明识别获得的登革热诊断标志物具有鲁棒性。
已有研究表明,ceRNA网络模块中的差异基因在免疫调控,细胞周期等生物学过程中发挥着重要作用,并在癌症和诊断和治疗的研究中有所应用。如FABP5能通过参与细胞调节因子的产生维持T淋巴细胞的稳态[16]。青蒿琥酯(ART)可能通过抑制FABP5调节PI3K/AKT通路进而影响肝癌细胞的增殖和迁移,FABP5可能作为ART治疗肝细胞癌的新靶点基因[17]。FABP5在肾透明细胞癌组织中呈现高表达,且与预后差相关,有望成为肾透明细胞癌药物的重要治疗靶点[18]。TNFRSF17在正常和恶性浆细胞的表面以及成熟B细胞上持续表达,并且在骨髓中长寿浆细胞的存活中起重要的作用,同时也是嵌合抗原受体T细胞免疫疗法(CRT-T)的一个热门靶点[19-20]。
KIF2C则是有丝分裂相关的重要驱动蛋白,并且是Wnt/β-catenin通路的直接靶点,是介导Wnt/β-catenin和mTORC1信号串扰的关键因子[21]。GINS1参与了低等真核生物[22]和人类的DNA复制过程[23]。GINS1也是肝细胞癌,肺腺癌,肾透明细胞癌的诊断和预后的生物标志物[24]。PSAT1基因沉默可以使非小细胞肺癌细胞周期调节蛋白cyclin D1降解,细胞分裂被阻滞于G0/G1期,最终抑制细胞增殖[25]。目前关于本研究中筛选出的差异基因和登革热之间的关系还鲜有报道,通过HVPPI和STRING数据库互作分析发现ceRNA网络模块基因能够与登革热相关宿主蛋白直接互作,并且表达模式在不同登革热数据集中基本一致,说明其在登革热疾病诊断中具有潜在价值。
4 结论本研究解析登革热相关基因差异表达模式,并构建了登革热失调ceRNA网络,通过模块挖掘识别与登革热诊断关联网络模块,为寻找登革热诊断标志物提供了新思路。
| [1] |
李晋涛. 登革热防治研究进展[J]. 第三军医大学学报, 2019, 41(19): 1902-1907. LI Jintao. Progress in research on dengue fever control[J]. Journal of the Third Military Medical University, 2019, 41(19): 1902-1907. DOI:10.16016/j.1000-5404.201909095 (  0) 0) |
| [2] |
岳玉娟, 任东升, 刘起勇. 2005-2013年中国大陆登革热疫情时空分布[J]. 疾病监测, 2015, 30(7): 555-560. YUE Yujuan, REN Dongsheng, LIU Qiyong. Spatial and temporal distribution of dengue fever in mainland China from 2005 to 2013[J]. Disease Surveillance, 2015, 30(7): 555-560. DOI:10.3784/j.issn.1003-9961.2015.07.008 (  0) 0) |
| [3] |
WANG Hong, XU Dahua, HUANG Huiying, et al. Detection of dysregulated competing endogenous RNA modules associated with clear cell kidney carcinoma[J]. Molecular Medicine Reports, 2018, 18(2): 1963-1972. DOI:10.3892/mmr.2018.9189 (  0) 0) |
| [4] |
杨雷, 伍搏宇, 熊辉, 等. ceRNA调控网络在骨关节炎中的研究进展及中医药干预现状[J]. 湖南中医药大学学报, 2022, 42(8): 1376-1383. YANG Lei, WU Boyu, XIONG Hui, et al. Research progress of ceRNA regulatory network in osteoarthritis and current situation of TCM intervention[J]. Journal of Hunan University of Chinese Medicine, 2022, 42(8): 1376-1383. DOI:10.3969/j.issn.1674-070X.2022.08.024 (  0) 0) |
| [5] |
LI Junhao, LIU Shun, ZHOU Hui, et al. starBase v2.0: Decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J]. Nucleic Acids Research, 2014, 42(Database issue): D92-D97. DOI:10.1093/nar/gkt1248 (  0) 0) |
| [6] |
BHATTACHARYA S, DUNN P, THOMAS C G, et al. ImmPort, toward repurposing of open access immunological assay data for translational and clinical research[J]. Scientific Data, 2018, 5: 180015. DOI:10.1038/sdata.2018.15 (  0) 0) |
| [7] |
LI Si, ZHOU Weiwei, LI Donghao, et al. Comprehensive characterization of human-virus protein-protein interactions reveals disease comorbidities and potential antiviral drugs[J]. Computational and Structural Biotechnology Journal, 2022, 20: 1244-1253. DOI:10.1016/j.csbj.2022.03.002 (  0) 0) |
| [8] |
SZKLARCZYK D, GABLE A L, NASTOU K C, et al. The STRING database in 2021: Customizable protein-protein networks, and functional characterization of user-uploaded gene/measurement sets[J]. Nucleic Acids Research, 2021, 49(D1): D605-D612. DOI:10.1093/nar/gkaa1074 (  0) 0) |
| [9] |
GU Z, EILS R, SCHLESNER M. Complex heatmaps reveal patterns and correlations in multidimensional genomic data[J]. Bioinformatics, 2016, 32(18): 2847-2849. DOI:10.1093/bioinformatics/btw313 (  0) 0) |
| [10] |
SALMENA L, POLISENO L, TAY Y, et al. A ceRNA hypothesis: the Rosetta Stone of a hidden RNA language?[J]. Cell, 2011, 146(3): 353-358. DOI:10.1016/j.cell.2011.07.014 (  0) 0) |
| [11] |
夏天, 肖丙秀, 郭俊明. 竞争性内源RNA: 一种全新的基因表达调控模式[J]. 中国生物化学与分子生物学报, 2012, 28(12): 1075-1081. XIA Tian, XIAO Bingxiu, GUO Junming. Competitive endogenous RNA: A novel mode of gene expression regulation[J]. Chinese Journal of Biochemistry and Molecular Biology, 2012, 28(12): 1075-1081. (  0) 0) |
| [12] |
LIAO Yuxing, WANG Jing, JAEHNIG E J, et al. WebGestalt 2019: Gene set analysis toolkit with revamped UIs and APIs[J]. Nucleic Acids Research, 2019, 47(W1): W199-W205. DOI:10.1093/nar/gkz401 (  0) 0) |
| [13] |
韩莉, 林昭春, 付柏林. 带状疱疹44例细胞及体液免疫功能变化与临床分析[J]. 四川医学, 2014, 35(3): 336-338. HAN Li, LIN Zhaochun, FU Bailin. Cellular and humoral immune function changes and clinical analysis of 44 cases of herpes zoster[J]. Sichuan Medical Science, 2014, 35(3): 336-338. (  0) 0) |
| [14] |
柯江维, 陈强, 段荣, 等. EV71病毒引起手足口病患儿免疫状态变化的研究[J]. 实验与检验医学, 2014, 32(1): 5-7+40. KE Jiangwei, CHEN Qiang, DUAN Rong, et al. Changes of immune status in children with hand, foot and mouth disease induced by EV71 virus[J]. Experimental and Laboratory Medicine, 2014, 32(1): 5-7+40. DOI:10.3969/j.issn.1674-1129.2014.01.002 (  0) 0) |
| [15] |
赵晟珣, 王桦, 杨帆, 等. 高龄老年肺部感染患者免疫功能变化与免疫调节治疗临床研究[J]. 中华临床医师杂志: 电子版, 2014, 8(6): 1026-1030. ZHAO Shengxun, WANG Hua, YANG Fan, et al. Clinical study on the changes of immune function and immunomodulatory therapy in elderly patients with pulmonary infection[J]. Chinese Journal of Clinicians: Electronic Edition, 2014, 8(6): 1026-1030. DOI:10.3969/cma.j.issn.1674-0785.2014.06.009 (  0) 0) |
| [16] |
ADACHI Y, HIRAMATSU S, TOKUDA N, et al. Fatty acid-binding protein 4 (FABP4) and FABP5 modulate cytokine production in the mouse thymic epithelial cells[J]. Histochemistry and Cell Biology, 2012, 138(3): 397-406. DOI:10.1007/s00418-012-0963-y (  0) 0) |
| [17] |
王清森, 吴静, 周家伟, 等. 青蒿琥酯通过FABP5调节PI3K/AKT通路影响肝癌细胞的增殖和迁移[J]. 安徽医科大学学报, 2022, 57(9): 1367-1374. WANG Qingsen, WU Jing, ZHOU Jiawei, et al. Artesunate affects the proliferation and migration of hepatocellular carcinoma cells by regulating the PI3K/AKT pathway through FABP5[J]. Journal of Anhui Medical University, 2022, 57(9): 1367-1374. (  0) 0) |
| [18] |
高鑫, 张淑芳, 黄邓高, 等. 基于公共数据库分析FABP5在肾透明细胞癌中表达及预后的意义[J]. 天津医药, 2018, 46(11): 1145-1150. GAO Xin, ZHANG Shufang, HUANG Denggao, et al. The expression and prognostic significance of FABP5 in renal clear cell carcinoma were analyzed based on the public database[J]. Tianjin Pharmaceutical, 2018, 46(11): 1145-1150. DOI:10.11958/20180999 (  0) 0) |
| [19] |
O'CONNOR B P, RAMAN V S, ERICKSON L D, et al. BCMA is essential for the survival of long-lived bone marrow plasma cells[J]. Journal of Experimental Medicine, 2004, 199(1): 91-98. DOI:10.1084/jem.20031330 (  0) 0) |
| [20] |
VAN DE DONK N W C J, USMANI S Z, YONG K. CAR T-cell therapy for multiple myeloma: State of the art and prospects[J]. Lancet Haematology, 2021, 8(6): e446-e461. DOI:10.1016/S2352-3026(21)00057-0 (  0) 0) |
| [21] |
WEI Shi, DAI Miaomiao, ZHANG Chi, et al. KIF2C: A novel link between Wnt/β-catenin and mTORC1 signaling in the pathogenesis of hepatocellular carcinoma[J]. Protein Cell, 2021, 12(10): 788-809. DOI:10.1007/s13238-020-00766-y (  0) 0) |
| [22] |
KUBOTA Y, TAKASE Y, KOMORI Y, et al. A novel ring-like complex of Xenopus proteins essential for the initiation of DNA replication[J]. Genes & Development, 2003, 17(9): 1141-1152. DOI:10.1101/gad.1070003 (  0) 0) |
| [23] |
DE FALCO M, FERRARI E, DE FELICE M, et al. The human GINS complex binds to and specifically stimulates human DNA polymerase alpha-primase[J]. Embo Reports, 2007, 8(1): 99-103. DOI:10.1038/sj.embor.7400870 (  0) 0) |
| [24] |
AHMAD M, HAMEED Y, KHAN M, et al. Up-regulation of GINS1 highlighted a good diagnostic and prognostic potential of survival in three different subtypes of human cancer[J]. Brazilian Journal of Biology, 2021, 84: e250575. DOI:10.1590/1519-6984.250575 (  0) 0) |
| [25] |
YANG Yi, WU Jueheng, CAI Junchao, et al. PSAT1 regulates cyclin D1 degradation and sustains proliferation of non-small cell lung cancer cells[J]. International Journal of Cancer, 2015, 136(4): E39-E50. DOI:10.1002/ijc.29150 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


