2. 福建省科学技术信息研究所,福州 350300;
3. 福建省海洋酶工程重点实验室,福州 350116;
4. 福建福大百特生物科技有限公司,福州 350100
2. Scientific and Technical Information Institute of Fujian Province, Fuzhou 350300, China;
3. Fujian Key Laboratory of marine enzyme engineering, Fuzhou 350116, China;
4. Fujian Fuda Baite Biotechnology Co., Ltd., Fuzhou 350100, China
酶分子设计是以其分子结构与功能规律作为基础,通过生物化学、分子生物学以及生物信息学等手段,在基因和蛋白质层面上对现有酶分子进行改造优化,制造和设计出改良的酶分子,以满足工业生产、生活以及科研的需要。酶是生物体内一种重要的分子,在细胞内部行使着重要的生物学功能。例如,酶作为蛋白质的一种重要类型,通常是一种具有高效催化效率和底物选择特异性的天然生物催化剂。酶分子设计是在深入了解其空间结构与生物学功能关系的基础上,改造和设计酶以获得具有特定物理和化学性质的新分子。在漫长的分子进化历程中,自然界已经进化出种类多样的酶,但天然酶一般只是在自然条件下才发挥出其良好的生物学功能。在实际生活中,酶以及相关制剂在造纸、酿酒、纺织以及皮革等相关工业领域都有着广泛的应用。另外,酶制剂一般可以用可再生资源进行生产,最终又可以通过自然界的微生物进行降解,具有绿色环保的特点,在提倡可持续发展的今天有着非常重要的应用意义。但在实际工业生产中的高温高压以及非水相溶剂(例如,酿酒中用到的酶制剂在酒精以及高温环境中)等非常温环境下酶的稳定性与催化活性一般会有所下降,甚至在一些情况下酶的活性可能会消失。因此,当代酶工程的研究趋势之一就是探寻耐高温、耐酸碱、耐盐等可以适应工业需求的酶。根据实际需要,通常需要对蛋白质以及酶进行改造和优化设计,同时了解其发挥生物学功能所需要的生物环境,使其能够在工业环境或者特定条件下仍然可以发挥甚至提高其生物学功能。在自然界中也存在一些可以适应工业需求的酶,这些酶一般存在于嗜高温、嗜酸碱以及高盐的细菌中。
从工业应用的角度来讲,酶及其相关制剂的研发主要考虑两个因素。一是酶制剂的分子特性参数,包括耐热性、耐酸碱性、非水溶剂稳定性、抗氧化性、底物特异性、酶的催化活力效率以及酶表面活性剂的耐受性等;二是酶发酵水平的高低,即生产酶的工业成本的高低。本文主要讨论酶的分子特性参数。从分子层面上来看,酶分子所具有的生物学功能有很大程度上是由其三维空间结构以及其所处的溶剂环境条件共同决定的。因此,酶分子三维空间结构构象、生物学功能信息以及所处溶剂环境条件对准确的酶分子设计提供了基础性的数据支撑。近年来,随着X射线晶体衍射、冷冻电镜以及核磁共振等实验技术快速和稳定的发展,越来越多的包括蛋白质在内的一系列复杂分子的三维结构得到准确解析,这些分子结构一般可以通过在蛋白质结构数据库(https://www.rcsb.org)中检索获得。另外,在AlphaFold数据库(https://www.alphafold.ebi.ac.uk)中也提供了大量预测的蛋白质结构[1]。近些年,也有报道使用AlphaFold和机器学习算法进行分子对接的研究[2]。这些分子三维结构对相应的生物学机理解释提供了更加精细的信息,这也进一步促进了理性药物开发和蛋白质分子设计的发展。在我们课题组前期的工作中,对几种酶进行了研究,包括漆酶[3-4]和几丁质酶[5-6]等,另外我们课题组也分别在两个实例中对特定的酶进行改造,提高了酶的催化活力[7],以及提高了酶的热稳定性[8]。国内外都有不少分子设计相关的网站和工具可以使用,例如美国密歇根大学结构生物信息学课题提供的https://zhanggroup.org/research/#ProteinDesign网站上就有不少基于蛋白质受体的分子设计工具。
从分子的角度来看,酶分子可以和多种分子相互作用,包括多肽、多糖、金属离子以及小分子等,另外酶和其他蛋白质之间也会相互作用。同时,一部分的蛋白质是生物体内重要的酶[9]。酶分子的催化作用一般具有专一性、高效性、以及作用条件较为温和等特点。通常来讲,依靠结构信息的酶功能机理解释,一般可以使用数学模型来描述酶分子结构和其催化活性以及稳定性之间的相关关系。这些数学模型的基本假设是酶分子结构中包含了决定其物理、化学及生物学等方面性质的大部分信息,而这些理化性质则进一步决定了酶分子的生物活性和稳定性。简而言之,酶分子结构以及生物化学性质与其生物活性也存在一定程度上的相关性。另外,溶剂环境对酶分子功能也有影响。因此,结构和溶剂环境是构成分析酶分子生物学功能模型的重要参数[10]。目前常见的结构参数包括疏水性、电荷极性、几何构型(例如立体和拓扑信息等)、构象表面特征以及理化性质等。基于结构的分子设计的原理,就是完成结构特征的数字化或者特征特化,同时不断优化相应的结构特征,以提高酶分子催化活性和稳定性。本文从酶分子三维结构以及所处溶剂的视角对酶分子设计进行系统性总结和讨论。
1 酶分子设计简介通常来讲,酶分子设计的目的是获得特定需求的新酶分子,或者获得比已有酶具有更优良特性的新分子,或者设计出从基因层次上更容易表达的酶,或者探索可以展示出其最优生化性能的溶剂环境条件[11]。溶剂环境对酶性能有影响,简单来说酶的催化活力在非极性溶液和水性介质溶液中有显著差异,这是由于酶在非极性溶液环境中的介电常数较水介质溶液中的介电常数低。在非极性溶液环境中两个带电荷基团之间的静电作用比在极性溶液环境中显著增高。酶分子设计的原理就是让其从一种状态到另外一种状态(例如,设计出更适合特定需求的状态)转变的过程。在自然界中也存在这种酶分子在不同状态之间根据不同溶液环境自动化转变的过程,其中比较著名的例子之一是脂肪酶。脂肪酶隶属于羧基酯水解酶类,能够逐步地将甘油三酯水解成甘油和脂肪酸。脂肪酶一般存在于含有脂肪的动物、植物和微生物的组织中。同时,脂肪酶也广泛地应用于药品、食品生产以及生物化学产品等实际应用方面[12]。
来源不同的脂肪酶,在氨基酸序列、组成以及结构上存在一定的差异,但一般相同类型的脂肪酶的三维结构是比较相似的,即脂肪酶之间存在进化层面上较强的弱同源性。脂肪酶的活性部位一般由丝氨酸(S)、天冬氨酸(D)、以及组氨酸(H)等氨基酸残基组成。脂肪酶属于丝氨酸蛋白酶类,这个主要由三个氨基酸组成的活性区域一般也称为S-D-H三联体催化部位。脂肪酶的催化部位一般包埋在分子结构内部,结构表面则被一小段特定的氨基酸残基形成的螺旋盖状结构覆盖(此结构又称为脂肪酶活性口袋的“盖子”),对三联体催化部位起保护作用。脂肪酶活性影响的关键相关因素之一是活性口袋的开闭。通常认为,酶分子处于活性口袋打开的状态(即激活状态Activation state)时,才能更易于在与底物的碰撞过程中发生结合和反应。在图 1中清晰地以三维结构形式展示一种脂肪酶活性口袋的开闭状态。从PDB[13]结构数据库中下载了一种脂肪酶的两种状态结构,其PDB号分别为1DT3和1EIN[14]。在Pymol[15]软件中对两个酶结构通过使用结构比对命令进行结构重叠。其中,图 1a中红色和黑色圆分别是这个酶激活和非激活状态时活性口袋的大小。蓝色和红色箭头分别表示结构重叠的两个结构(见图 1a)分开后的两个结构(见图 1b, 1c)。从图 1a中可以看到,1DT3为酶活性口袋未激活时的种状态,颜色主要渲染为红色;1EIN为此酶活性口袋的打开状态,颜色主要渲染为绿色显示。

|
图 1 脂肪酶活性口袋的开闭状态 Figure 1 Open and closing states of the lipase activity pocket |
在这个脂肪酶中,活性结合位点为146位的丝氨酸(S146)、201位的天冬氨酸(D201)以及258位的组氨酸(H258)。这三个结合位点氨基酸残基在图 1a中渲染为黄色。通过结构重叠的结果,可以看到这个脂肪酶的开闭状态主要差异在于一个二级结构以α-螺旋为主的区域(即脂肪酶活性口袋的盖子,在图 1a中渲染为褐色),这个区域在这个酶中的位置为85到92的氨基酸片段“SIENWIGN”。当脂肪酶结构处于催化活性激活状态时,其活性口袋明显比非活性状态时更大(相应活性口袋区域在图 1中标注为黑色和红色圈)。图 1b为脂肪酶打开活性口袋状态时的脂肪酶表面图,其中黄色区域为活性氨基酸残基。图 1c为脂肪酶活性口袋关闭状态时的表面图,虽然此时活性氨基酸也标注为黄色但是因为其被包埋在结构内部所以无法观测到。当脂肪酶分子处于水溶液中时,盖子的疏水性氨基酸朝内,从而关闭活性口袋。而当脂肪酶处于油水界面时,疏水氨基酸会朝界面处油脂分子结合,从而打开了活性口袋,这个过程增强了底物和酶活性中心的催化活力。大多数脂肪酶在脂质与水的界面上时会自动打开这个盖子从而使脂肪酶的活性提高。当脂肪酶处于非活化状态时,这个盖子又可以自动关闭以保护活性部位。例如在纯水介质中,脂肪酶中盖子主要是关闭的,而在疏水层的存在下(例如油脂界面处),盖子会逐渐打开以激活催化活性。
与脂肪酶类似,自然界中的不少酶都存在这种活性口袋的开闭状态。例如,胃蛋白酶也存在生物活性激活和非激活两种状态。从生物化学的角度来看,胃酸可以激活胃蛋白酶。同时,胃蛋白酶非激活状态结构可以让其贮存在其合成部位而没有引起细胞或组织自我消化与水解的潜在危险,待细胞需要其功能时再进一步被激活。从分子三维结构的角度来看,胃蛋白酶原处于没有活性状态结构时也主要是因为活性中心未暴露出来。一般通过调整胃蛋白酶所处环境的pH值、以及对一些特定多肽链的剪切修饰等方法使酶的活性中心形成或暴露出来,进而激活其酶活性。胰蛋白酶原也有类似的活化机制。另外还有其他类似例子,例如Liu等报道昆虫中的一种酶可以在蜕皮液中被激活从而在特定时期发挥其生物学活性[16]。
一些酶在不同的溶剂环境中(例如油水界面、溶剂的酸碱性),其活性口袋区域可能处于关闭和打开的两种重要的不同状态。这个核心活性区域的关闭和激活打开现象也为酶分子设计提供了一些思路,在需要提高酶分子的活性时打开其活性区域或者在保持结构稳定性的同时尽量扩张其活性空腔区域,并探索促进酶分子打开和关闭其活性口袋的最优溶剂环境。
2 酶分子设计工具之分子对接分子对接是分子之间空间结构与能量的双重匹配过程。分子对接是酶分子设计中的重要方法之一。分子对接这一概念的历史最早可以追溯到19世纪由Fischer等学者提出的受体学说。受体学说认为,其他分子会与酶分子即受体会发生类似钥匙与锁之间的识别关系,这种分子识别关系主要依赖两者之间空间与能量的相互作用。通常情况下,可以与酶相互作用(即可对接)的分子包括小分子、金属离子、多糖以及多肽等,以及蛋白质和蛋白质之间也可以对接。不同类型的分子与酶对接一般使用不同的对接力场体系,即使用不同的对接程序完成这个对接过程。小分子(包括多糖)和酶分子对接一般使用AutoDock Vina[17]。酶分子和其他蛋白质大分子之间的对接一般使用Zdock[18]。多肽和酶分子对接一般使用Flexpepdock[19]。网络上还有不少其他可以实现类似功能的程序和网站。不同的对接体系一般所使用的能量函数不同,在通常情况下不同能量函数之间不可混用。
分子对接在酶分子设计中主要用于衡量酶分子和底物(或者酶分子和其他分子)之间的可匹配性。分子对接一般是从两个维度进行,第一个维度是计算结合亲和力的大小;第二个维度是计算分子复合物的结合模式构象。一般而言,活力更好的酶与相应底物的亲和力更强;另一方面,分子对接过程中,也可以分析活性通道中改变哪些氨基酸可以更好地减少底物对接过程中的空间位阻效应。空间位阻效应主要指底物进入酶分子活性空腔过程中的空间阻碍作用。酶分子催化反应中空间位阻效应会降低其催化活性。通常来讲,底物进入受体活性通道中的空间位阻效应越小,那么相应酶分子的活性则越高。空间位阻效应的原理是每个原子在分子中占有一定的空间。如果原子太接近了,相邻的原子就会形成重叠的电子云表现为斥力。另外,对接结果中对酶分子-底物相互作用的关键氨基酸分析,也可以对设计更强亲和力的酶分子提供一定的线索。一般可以设计不同的结构突变体,之后不同的结构突变体分别和底物进行对接,通过对接的亲和力分数大小以及结构构象判断突变是否比原始结构更加优良。
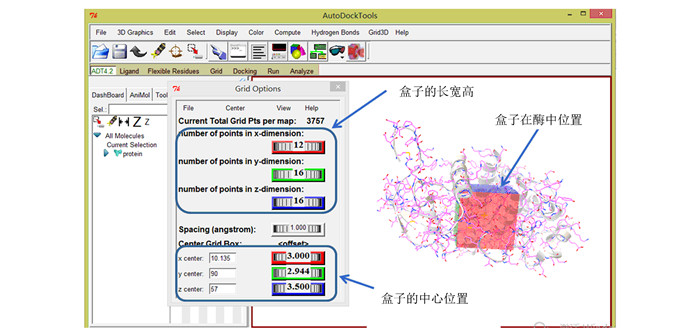
图 2为AutoDock Vina软件的使用界面。学术界通常使用AutoDock Vina等一些开源软件对酶分子和小分子进行对接。在进行分子对接前,酶分子的结构文件格式需要进行初始化,例如酶分子结构文件一般需要经过去水、加氢原子、加电荷等处理后存为pdbqt格式。类似地,小分子配体也需要经过前处理并存储为pdbqt格式。AutoDock Vina在分子对接过程中还需要指定一个对接区域盒子(Box),使盒子包括酶分子的活性口袋(见图 2),可以把一些与配体有相互作用的氨基酸残基选择出来作为设定box的参考。盒子的位置和大小一般通过指定盒子的中心位置(x, y, z)以及盒子的长宽高来确定的(见图 2)。酶活力直接相关的区域称为酶的活性口袋或者活性中心。通常又将活性中心分为结合部位和催化部位。结合部位负责与底物结合,决定酶的专一性。催化部位负责底物键的断裂和新键形成,决定酶的催化能力。酶的活性部位位于空腔之内,底物分子(或者一部分)结合到空腔内并发生催化作用。空腔是疏水性较高的区域,非极性氨基酸多,但也含有极性氨基酸,以便与底物结合并发生催化反应。其疏水性质产生一个微环境,提高与底物的结合能力有利于催化。空腔的活性位点对于不需要辅酶的酶来说就是几个氨基酸,而对于需要辅酶的酶来说辅酶或者辅酶的一部分也可能是活性部位的组成部分。酶与底物结合主要依靠氢键、盐键、范德华力、疏水作用等作用力。酶的活性中心一般具有一定的柔性和可运动性。
|
图 2 AutoDock Vina程序分子对接界面 Figure 2 Molecular docking interface of AutoDock Vina program |
图 3为酶分子1IEP(转移酶的一种)[20]分别和小分子伊马替尼(Imatinib)以及布洛芬(Ibuprofen)进行分子对接的结果。伊马替尼(图 3a上小分子)是全球首个获得批准的肿瘤发生相关信号传导抑制剂。布洛芬(图 3b上小分子)也为酶的一种抑制剂,在生物体内有镇痛、抗炎等作用。可以通过对接结果来判断两个抑制剂对蛋白1IEP作用效果的差异。这里对复合物的模型的模拟是使用AutoDock Vina[21]软件进行分子对接得到的。在图 3a中,黄色小分子为伊马替尼在对接前的初始结构,红色小分子为对接后的结果。蛋白质1IEP和布洛芬对接结果在图 3b中(图 3b中红色为对接后的布洛芬),其亲和力数值为-8.5 kcal/mol。而酶1IEP和伊马替尼对接的亲和力数值为-10.5 kcal/mol。从亲和力的数值上来说,小分子伊马替尼对酶1IEP相互作用更好一些。在这个实际例子中可以根据对接结果的能量来判断蛋白质和小分子相互作用的强弱。

|
图 3 使用AutoDock Vina软件进行分子对接例子 Figure 3 Examples of molecular docking using AutoDock Vina |
图 4是以球形对酶分子的活性空腔进行展示。此图为使用Discovery Studio 2019(福州大学海洋酶工程重点实验室购置的正版Discovery Studio软件,https://www.3ds.com)绘制而成。此酶分子的活性空腔区域可以用一个球状结构进行表示。球状结构的酶活性口袋一般用(x, y, z, r)的一组4个数值组成的向量表示。其中坐标(x, y, z)表示该球体的球心位置,以及r表示球体的半径。

|
图 4 酶活性空腔的展示 Figure 4 Demonstration of an enzyme active cavity |
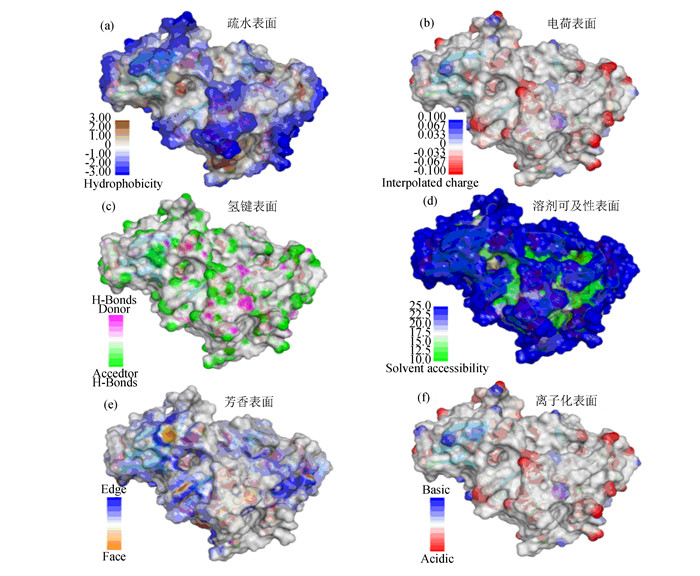
找到酶分子的空腔是分析其结合位点的关键步骤之一[22],一般通过把酶分子结构以表面的形式显示的方式寻找空腔[23]。在图 5中,使用Discovery Studio软件画出的酶分子1IEP的6种表面形式(表 1详细列出6种表面信息),包括疏水表面(见图 5a)、电荷表面(见图 5b)、氢键受体和供体表面(见图 5c)、溶剂可及性表面(见图 5d)、芳香环表面(见图 5e)以及离子化表面(见图 5f)。
|
图 5 蛋白质不同表面形式 Figure 5 Different types of protein surfaces |
| 表 1 不同类型的蛋白质表面 Table 1 Different types of protein structural surfaces |
分子对接不仅仅在酶分子设计领域有着广泛应用,也常常用于一些蛋白质功能的机理解释。例如,生物杀虫剂有效成分通常是一些化学小分子物质,那么实用的杀虫剂就需要可以抑制虫子中的一些受体分子(通常为蛋白质),但是同时需要对人体内相应的受体分子的没有抑制作用或者抑制作用相对弱一些。在这种情况下,可以对杀虫剂的有效成分小分子与昆虫以及人体内相应的蛋白质进行分子对接,并进行亲和力和构象分析。通常来讲,设计良好的杀虫剂小分子对昆虫中相应蛋白质的亲和力会更强一些。
除了计算酶分子和底物结合亲和力外,分子对接结果还可以用于详细地分析活性区域以及相互作用关键氨基酸,这在酶分子设计中可以对关键氨基酸突变体设计提供一些设计思路。分子对接是研究底物和酶的重要手段之一。某个底物能够激活生物酶、靶点、受体信号的传递,这些都可以通过分子对接方法进行相应的分析。
通常来讲,分子对接是进行酶分子设计的重要手段之一。但是,酶分子的实际应用不是简单的与配体分子结合完成的。通过生物实验的分离手段将蛋白质分离后,其体外活性状态和其在生物体里(细胞膜上或细胞液里)是不太一样的。作为酶分子设计,做完分子对接后不是结束,后面组织、动物的实验、或者工业实际应用,这一系列的问题都需要研究通了。生物体各系统是相互连贯的,而且多系统平行。因此,基于分子对接的酶分子设计或者底物设计后续都要进行详细的实验验证。
3 酶分子设计工具之动力学模拟酶分子设计中另外一个常用到的方法是分子动力学模拟(Molecular dynamics simulation)[24-25]。经典分子动力学的模拟过程一般是从一个定义好的构型开始,这个构型可以是分子对接好的复合物也可以只是一个蛋白质单体,之后使用牛顿运动方程来计算蛋白质原子的动态轨迹。对于酶分子体系中的每一原子,所受的合力根据其与其他粒子之间的相互作用来计算,这些相互作用通常以力场的形式进行描述。分子力场有时也被称为势函数。一般分子力场势函数包括以下几个部分:描述分子内成键作用的项;键伸缩能:构成分子的各个化学键在键轴方向上的伸缩运动所引起的能量变化;键角弯曲能: 键角变化引起的分子能量变化;二面角扭曲能:单键旋转引起分子骨架扭曲所产生的能量变化;交叉能量项:上述作用之间耦合引起的能量变化。一个可靠的分子力场对准确模拟出酶相应的动态变化构象具有关健性的作用。目前常用的分子力场是CHARMM和Amber等力场[26]。同时,分子力是分子动力学模拟中的参数之一,分子力以粒子的质量为加速度, 再加上粒子以前的位置和速度, 这就决定了一小段时间步长之后粒子的新位置。目前常用的分子动力学模拟软件包有Amber(http://ambermd.org)和Gromacs(http://www.gromacs.org)等。通常来讲,长时间的分子动力学模拟(例如,10纳秒以上的模拟过程)对理解酶生物学功能背后的原理以及酶分子设计非常有用。通常来讲,模拟的时间长度越长,相应的结果越可靠。近些年新版本Amber[27]软件(例如版本21)通常通过CPU多线程以及GPU等技术来提高模拟的速度进而缩短了分子模拟程序运行时间。
在分子动力学模拟中也需要考虑酶分子所处的溶剂环境因素,这样才能更准确地模拟出相应的动态结果。科学界已经证实一些酶分子变性后在去除变性因素仍然可以恢复其之前的三维结构。这表明酶分子的序列及其所处的溶剂环境也一定程度上决定其三维空间结构。所以,在动力学模拟中,常常通过调整不同的参数(例如溶剂的pH值)以及不同的溶剂化环境(例如,水溶液、有机溶液或者水和有机溶剂的混合体系)的方式来获得酶分子特定的动态三维结构。酶在有机相中催化反应过程有其特点:例如,疏水性底物或者产物的溶解度增加;可以一定程度抑制有水参与的副反应;酶一般不溶于有机容易易于回收;一定程度减少微生物污染等。因此,进行动力学模拟时,需要指明酶所处的溶剂环境。在溶剂环境和结构确定的条件下,动力学模拟一般可以模拟出酶的动态结构。
目前,经典分子动力学模拟Amber网站[27]提供软件的下载和使用教程。但可能由于动力学模拟需要的计算量较大等原因,Amber网站目前还没有提供可以直接提交数据就计算的网络服务器。在用Amber软件过程中,进行结合自由能计算与能量分解通常用MM-PBSA/GBSA方法。为了更深入地表示蛋白和氨基酸残基之间的相互作用,一般研究工作中会用相互作用能量和氨基酸残基数的一个关系图表示,可以把每个残基对结合能的贡献算出来。从使用的角度来说,分子动力学模拟一般主要分为能量最小化(Minimization)、体系加热(Heating)、平衡(Equilibrition)、数据采集(Production)以及结果分析等几个主要步骤。同时要注意动力学模拟前需要进行前体系溶剂化,并在体系中加抗衡离子等准备步骤。分子动力学模拟常用的时间单位是皮秒(符号ps)以及纳秒(符号ns)。经过一定时间长度的模拟,通常可以获得上千个甚至数量更多的酶结构构象。结果分子中,一般通过聚类算法把这些构象进行分类并筛选出具有代表性构象以此来观察酶的动态结构[28]。
在生物体内或者生物反应体系中,每个酶分子结构都不是静止的,基本都像个小机器,有一定的构象变化。但结构生物学目前的晶体衍射实验手段通常只能获得静止的三维结构图片。NMR实验方法可以获得一定范围内酶的动态结构。为了揭示相应分子机制,就要不断获得中间态的三维空间结构照片,获得的动态帧数越多则相应机制解释得越精准。动力学模拟可以用于模拟酶催化过程中的构象变化以及计算这个成果中的自由能变化,找出酶催化过程的关键氨基酸残基。动力学模拟也可以用于模拟酶与不同底物的结合状态,以及探索可能的催化机理。因此,基于分子动力学模拟得到的分子动态结构是分子设计的重要信息之一,成为现有NMR实验方法获得酶动态结构的一种重要补充方法。
4 小分子相关资源从小分子数据库PubChem[29]中(https://pubchem.ncbi.nlm.nih.gov)可以获得各种小分子的结构文件和相应分子功能信息。另外,PDB(https://www.rcsb.org)数据库中存储的虽然主要为蛋白质的结构信息,但是其中不少蛋白质为复合物结构,在这些复合物结构中也存在小分子结构,即也可以从PDB数据库的蛋白质的复合物中获得小分子的结构数据。PDB数据库中同时存储着蛋白的突变数据,这些突变体的不同突变位点对应着不同的性质,这些突变位点数据为酶分子设计提供了重要参考信息。可以用序列比对和结构叠加等方法把这些位点对应到目标酶上,从而可以根据模板的突变位点和性质信息系完成初步的酶分子设计和优化。对于未知结构和结合位点的酶,可以使用一些预测工具,例如密歇根大学的结构生物信息学实验室https://zhanggroup.org网站,进行结构和功能位点的预测,这样可以根据相应的预测结果进行分析。
除了从PubChem和PDB等数据库直接下载小分子外,小分子的结构也可以采用ChemOffice[30]以及Discovery Studio等软件进行直接绘制得到,并可通过能量最小化等步骤优化其三维结构。特别是对一些比较新颖的小分子结构,一般从数据库中难以直接获得其三维结构。这种情况下,通过绘图软件获得小分子的三维结构是可行的途径之一。配体分子与受体酶分子之间相互作用关系通常分为激活、抑制、以及拮抗三种作用关系。激活作用主要是指配体提高了酶分子相应的活性。与此相反,抑制作用是指配体抑制了酶分子的活性。受体拮抗剂,也叫阻断剂,指能与受体结合,并能阻止激动剂产生效应的一类配体物质。拮抗作用系指两种以上分子合并使用后,使激动剂作用反而减弱或消失,多数情况下有拮抗作用的分子不宜配对使用。
一般而言,直接通过分子对接和分子动力学的方式难以直接判断小分子对酶分子是激活、抑制、以及拮抗三种作用中的哪一种。为了分析具体的相互作用类型,一般是通过实际的生物实验来判断小分子和酶分子之间的作用关系。
5 酶分子设计常用策略目前,学术界关于酶分子设计通常采用定点突变(Site-directed mutagenesis)的方法进行设计的主要两个方向为提高酶分子的催化活性以及提高其稳定性。在分子设计中,提高酶分子的催化活性和提高酶分子稳定性一般采用两种不同的策略。提高酶分子催化活性一般是以酶分子-底物的复合物(可以是实验解析的复合物构象,也可以为分子对接的到的复合物结果)中距离底物3-5 Å距离范围内的酶分子氨基酸进行突变,即在酶分子的活性口袋及其附近进行位点突变;而提高酶分子稳定性则一般选取远离酶分子活性中心区域的氨基酸进行突变。有时会进行多个位点协同突变,以期同时提高酶分子的活性和稳定性。提高酶分子的稳定性是让其更好地适合工业应用。虽然目前学术界已经筛选出了数量众多以及种类多样的酶分子,但是在工业应用的环境一般都是处于高温高压以及强酸强碱条件下,大多数酶分子在这种情况下可能会失活或者不太稳定。因此,需要对酶分子进行设计和改造,使其在特定条件下仍然可以起到一定的生物学功能甚至可以提高其生物学功能。
表 2中列出了用于酶分子设计不同目标的策略。从生物物理学的角度来看,提高酶的热稳定性的分子设计策略一般是通过增加酶分子中化学键的数量,包括共价键和非共价键的数量来提高酶分子的热稳定性以及耐酸碱性等。构成酶分子的原子间通过共用电子对作用形成的化学键为共价键,共价键一般包括肽键以及二硫键等。非共价键包括主要包括氢键、范德华力、盐键、疏水作用力、芳环堆积作用、卤键等。这些共价键和非共价键的化学键的数量增多一般有利于提高酶分子的稳定性。另外,酶分子制剂也需要一定的稳定性以方便储存、运输以及在实际在复杂环境中使用。同时,降低酶分子聚集性也是提高其稳定性的一个方面。因此,提高稳定性是酶分子设计中需要考虑的一个重要因素。
| 表 2 用于酶分子设计的不同目标 Table 2 Different objects in enzyme molecular design |
酶分子一般可以和许多功能类似的小分子分别对接。在这种情况下,可以分析这些小分子之间的共性,一般可以通过药效团(Pharmacophore)[31]的方式进行的。药效团是特征化的小分子三维结构要素的组合,可以分为两种类型。一类是具有相同药理作用的类似物,它们具有某种基本结构,即相同的化学结构部分,如磺胺类药物、局麻药、受体阻断剂、拟肾上腺素药物等;另一类是一组化学结构完全不同的分子,但它们以相同或类似的机理与同一受体键合,产生同样或者类似的药理作用,如己烯雌酚的化学结构比较简单,但因其立体构象与雌二醇相似,也具有雌激素样作用。对药效团的分析可以找出底物和蛋白质结合的关键区域,发现其中的规律,这为酶分子设计提供一定的线索。同时,生物实验中一般通过易错PCR的方法可以获得酶分子的突变体并进行筛选。易错PCR一般通过调节体系中的Mg2+、Mn2+以及dNTP浓度来提高PCR过程中的突变率。在易错PCR过程中,增加突变频率可以通过在每一轮PCR反应中使用低保真的DNA聚合酶,改变dNTP浓度(例如,使其中1种dNTP浓度高于其他3种),同时可以一定程度地提高二价阳离子Mg2+、Mn2+等在溶液中的浓度来达到。同时,这是一个累计的过程,可以通过多轮的易错PCR过程调节最终的突变率。另外,随机突变也是酶分子设计的常用策略之一。
酶分子设计中常常也会考虑以增加二硫键(Disulfide bond)的方式来提高稳定性。二硫键是连接不同肽链或同一肽链中,两个不同半胱氨酸残基之间的化学键。二硫键是一种比较稳定的共价键,在酶分子中主要起着稳定肽链空间结构的作用。一般而言,一个酶分子中二硫键数目越多,则酶分子对抗外界因素影响的稳定性就愈大。在酶分子设计中,也常常以提高酶分子整体二硫键数量的方式来提高酶分子的稳定性。一个区域打算设计为其他不同的二级结构,那么可以从容易形成特定二级结构的氨基酸中选取。二级结构是分子设计中的重要内容。例如,1988年杜邦公司宣布,成功设计并合成了由四段反平行α-螺旋组成为蛋白质。另外,酶分子相应区域的亲疏水性和酸碱性也是分子设计的考虑因素。在农业科学的研究中,常常以开发酶或者蛋白质抑制剂的方式来设计农药分子。因此,抑制剂的开发也是酶学研究中的内容之一。凡是能降低酶促反应速度的物质统称为抑制剂。表 3中详细列出了酶分子设计的常见策略。
| 表 3 酶分子设计中常用策略 Table 3 Common strategies used in enzyme molecular design |
酶分子的聚合情况有时也是影响其功能的一个重要方面。一个酶分子的结构域数量一般是以其一条链中所含结构域的个数进行确定的。一般而言,酶分子的结构文件中有几条链,那么这个酶分子就是几聚体。例如,漆酶(Laccase)就有双聚体和三聚体两种类型[32]。酶分子为几聚体的情况也可以做为分子设计的内容之一。例如,陈等[33]就通过突变氨基酸阻止蛋白质的二聚化来设计阻止肿瘤和炎症发生的靶点。
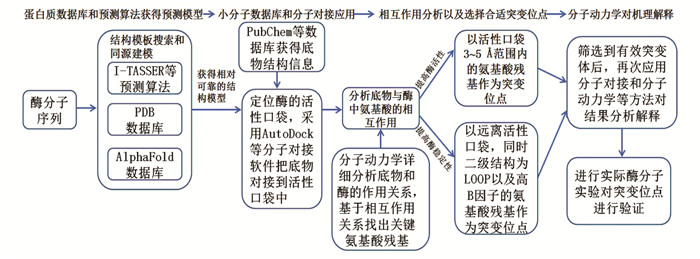
酶分子设计的过程(见图 6),首先需要通过结构预测算法(例如,Ⅰ-TASSER[34])或者搜索数据库(例如PDB和AlphaFold数据库)等方法建立所研究对象的结构模型,在此基础上进行结构-功能-应用的关系研究,然后提出设计方案,通过实验验证后进一步修正设计,这往往需要几次循环才能达到目的。一般的分子设计工作通常可以按以下几个主要步骤进行:
|
图 6 酶分子设计一般流程 Figure 6 A general flowchart for enzyme design |
1) 通过结构生物学实验或者同源建模等方法构建目标酶分子的三维结构模型;
2) 找出对所要求的性质有重要影响的位置和区域。提高酶催化活力一般是选取靠近活性口袋的氨基酸,而提高酶稳定性一般选取远离活性口袋的氨基酸。提高酶稳定性选取远离酶活位置一般是为了在提高酶稳定性的基础上不影响酶的活性,同时二级结构区域一般选择为LOOP;
3) 对上一步中所选出位点上改变残基所得到的突变体,一方面使突变体酶分子产生具有所要求的性质,同时考虑残基的体积、疏水性等性质的变化所带来的影响;
4) 预测突变体的结构。同时计算突变后的自由能的改变量ΔΔG=ΔG(天然)- ΔG(突变),从能量变化的角度来判断突变体是否符合要求,其中ΔG为折叠态和伸展态之间的吉布斯自由能差;
5) 定性或定量计算优化所得到的突变体结构是否具有所要求的性质。
一般而言,能否成功地进行酶分子设计,除了要求有较为合理的设计方案外,一般还需要在研究过程中探索相应酶所具有的结构化学和物理化学参数等,最终设计出来的结构是否符合需求需要通过实际生物学实验来进一步验证。
6 酶分子设计经典案例表 4中列出了酶分子设计相关的生物信息学软件资源。这些软件资源广泛的应用酶分子设计的研究中。近些年来国内外不少课题组都研发一些经典的酶分子设计案例。例如,Xia等通过比较三种酶结构(嗜冷酶VPR、嗜中温酶PRK和嗜热酶AQN),并进行了多副本分子动力学模拟[35]。然后进行了连续静电学计算和静电表面势的比较,实验结果表明,静电特性差异有助于三种酶对不同温度的适应,从而为进一步的酶工程、诱变以及分子设计研究提供了基础[35]。在这个案例中,Xia等直接从PDB数据库中获得蛋白质晶体结果,之后使用GROMACS软件进行多副本的分子动力学模拟,这样有助于较为准确的模拟出酶催化过程的动态过程。同时,作者采用VMD软件进行结构图形显示和分析。在多种分子作用力的比较中,静电作用为三种酶性质差异的主要因素。
| 表 4 酶分子设计相关资源 Table 4 Related resources for enzyme design |
Tang等[36]通过改进的标准化B因子指标以及二级结构的LOOP方法来定位提高酶热稳定性的关键区域,同时采用环嫁接策略提高枯草杆菌蛋白酶的热稳定性。B因子指标是晶体学中原子状态的“模糊度”,其数值越大则相应部分构象就越不稳定或者柔性越强。因此,酶结构中B高因子区域的氨基酸突变后,酶稳定性容易提高。在这个案例中,Tang等[37]采用Swiss-Model网站对目标蛋白进行结构建模,并用Pymol软件进行B因子计算,之后采用Amber软件进行分子动力学模拟。B因子法是这个工作中筛选突变区域的主要标准。筛选好相应区域后,突变刚性更强的氨基酸残基,作者通过这种增加了酶结构总体的刚性的方式进而提高了酶的热稳定性。
Lin课题组通过使用AutoDock软件进行分子对接以及Amber软件进行分子动力学模拟等方法,筛选出了谷氨酸突变为丙氨酸的突变策略,分别提高了两种酶的催化活力[38-39]。Lin课题组主要采用定位酶活性口袋附近氨基酸的策略,对底物结合以及进入活性口袋的过程进行优化,进而进行突变提高了酶活性。
Nikolaivits等[40]对多酚氧化酶进行系统性研究,对酶活性位点附近几个关键氨基酸残基进行突变,并最终筛选出其中一种有效突变体(G292N,即序列中292位的甘氨酸突变为天冬酰胺),这种突变体可以提高酶活。Lyu等[41]通过基因工程改造短乳杆菌提高谷氨酸脱羧酶的生产效率,和野生菌株相比,改造后的菌株生产效率有显著提高。Nikolaivits和Lyu这两个案例采用的策略和软件和Lin课题组的方法类似。
在以上这些例子中,分子对接、分子动力学模拟以及生物物理学等方法都可以综合地应用于酶分子设计中。另外,分子对接和分子动力学模拟除了可以用于筛选突变位点之外,也常常用于分子作用机理的解释。分子设计方法不仅仅局限于本文所论述的这些策略,也可以考虑其他不同的方法。例如Patil等采用利用超声波对微生物培养过程中形成的细胞束进行疏松,促进了菌体生长,产酶量提高以及提高了酶活[42-43]。但是使用超声波对酶机构的影响,目前还没有明确的结论。另外,通过酶固定化的方法也是提高酶活力的方法之一[44]。例如,将酶固定在纳米颗粒上可以减少酶解旋,通常可以提高酶稳定性和催化性能。酶经过固定化处理后通常稳定性可以提高。例如,Aslani等[45]通过固定化胰蛋白酶提高其活性和稳定性。一些酶需要和金属离子结合才能发挥其催化活性,例如漆酶是催化氧化反应的一种多铜离子氧化酶,漆酶通常使用氧分子和铜离子来启动催化过程。因此,在一些酶分子设计中可能还需要考虑金属离子的相互作用。
目前,开展酶分子设计的研究一般需要结合实验验证进行,所以相应的研究成果一般发表在实验类的杂志或者一些综合类的杂志中,例如这里所讨论的一些案例发表在Journal of Biological Chemistry、Enzyme and Microbial Technology以及Applied Biochemistry and Biotechnology等杂志上。
7 总结随着学术界对酶分子理解不断加深,酶分子设计逐渐成为国内外不少课题组的研究内容之一。酶分子设计是在深入了解其序列、结构以及功能关系的基础上进行的一种理性设计和改造方案,甚至可以设计出自然界中还未存在的新结构。酶的催化活性通常取决其活性口袋特点,通常而言在酶总体结构适合的范围内其活性口袋越大则酶活力可能越高。在合理范围内,扩大酶活性口袋通常有两个方向,其一是设计更好的突变体;其二,改变酶所处的溶剂环境,不同的溶剂环境会导致酶活性口袋的打开或者关闭。
酶分子活性通常受其三维结构构象的显著影响。因此,酶分子设计一般是在其结构基础上进行的改造,这是一个系统性过程。酶分子是一个复杂的体系,不少酶分子具有多靶点、整体性等特点。酶分子行使其生物学功能过程中一般由多个部分协同发挥作用,重要和不重要部分只是相对而言的。同时,也可以通过优化酶分子所处的溶剂环境以及固定化等方法提高酶的稳定性和催化活力和稳定性。通过虚拟的氨基酸突变等技术设计更高催化活力、高稳定以及选着性的酶是一种通用的方法。近些年生物信息学技术的发展,使酶分子设计方法在国内外不少课题组都有一些基于计算方法的成功案例。随着PDB以及AlphaFold等数据库提供的蛋白质结构数量越来越多,基于结构的酶分子设计在未来将很可能会得到较为广泛的应用。
简而言之,酶分子是一种重要的生物分子,目前酶分子设计的主要方向有提高热稳定性、酸碱稳定性、抗氧化性、提高酶的催化活力、提高催化底物专一性、耐非水溶剂环境以及减少副作用等。由于酶分子结构、功能以及所处溶剂环境的复杂性,特定的酶需要进行具体和详细分析后才能更好地设计出新酶。随着生物技术的不断发展,酶分子在学术研究和工业生产中已经有越来越广泛的应用。酶分子设计目的就是为蛋白质工程提供设计方案,使酶及其制剂产品更好地为生活和工业生产服务。因此,掌握酶和蛋白质的相应机理并进行合理的设计是科学研究与商业研发的一个重要方向。通常来讲,酶分子设计是在其三维结构的基础上进行结构-功能-溶剂环境-应用关系的研究。酶分子设计之后,一般通过实验验证后进一步修正和优化设计。
| [1] |
JUMPER J, EVANS R, PRITZEL A, et al. Highly accurate protein structure prediction with AlphaFold[J]. Nature, 2021, 596(7873): 583-589. DOI:10.1038/s41586-021-03819-2 (  0) 0) |
| [2] |
WONG F, KRISHNAN A, ZHENG E J, et al. Benchmarking AlphaFold-enabled molecular docking predictions for antibiotic discovery[J]. Molecular Systems Biology, 2022, 18(9): e11081. DOI:10.15252/msb.202211081 (  0) 0) |
| [3] |
WANG Zehua, HU Rongmin, YE Xiqian, et al. Laccase 1 gene from Plutella xylostella (PxLac1) and its functions in humoral immune response[J]. Journal of Insect Physiology, 2018, 107: 197-203. DOI:10.1016/j.jinsphys.2018.04.001 (  0) 0) |
| [4] |
ZHOU Zhimin, LI Renkuan, NG T B, et al. A new laccase of lac 2 from the white rot fungus cerrena unicolor 6884 and Lac 2-Mediated degradation of aflatoxin B1[J]. Toxins (Basel), 2020, 12(8): 476. DOI:10.3390/toxins12080476 (  0) 0) |
| [5] |
LI Renkuan, HU Yajuan, NG T B, et al. Expression and biochemical characterization of a novel chitinase ChiT-7 from the metagenome in the soil of a mangrove tidal flat in China[J]. International Journal of Biological Macromolecules, 2020, 158: 1125-1134. DOI:10.1016/j.ijbiomac.2020.04.242 (  0) 0) |
| [6] |
LI Renkuan, HU Yajuan, HE Yujie, et al. A thermophilic chitinase 1602 from the marine bacterium Microbulbifer sp. BN3 and its high-level expression in Pichia pastoris[J]. Biotechnology and Applied Biochemistry, 2021, 68(5): 1076-1085. DOI:10.1002/bab.2027 (  0) 0) |
| [7] |
ZHANG Yajiao, LANG Bin, ZENG Deyang, et al. Truncation of kappacarrageenase for higher kappacarrageenan oligosaccharides yield with improved enzymatic characteristics[J]. International Journal of Biological Macromolecules, 2019, 130: 958-968. DOI:10.1016/j.ijbiomac.2019.02.109 (  0) 0) |
| [8] |
SU Bingmei, XU Xinqi, YAN Renxiang, et al. Mutagenesis on the surface of a beta-agarase from Vibrio sp. ZC-1 increased its thermo-stability[J]. Enzyme and Microbial Technology, 2019, 127: 22-31. DOI:10.1016/j.enzmictec.2019.04.006 (  0) 0) |
| [9] |
HENZLER-WILDMAN K A, LEI M, THAI V, et al. A hierarchy of timescales in protein dynamics is linked to enzyme catalysis[J]. Nature, 2007, 450(7171): 913-916. DOI:10.1038/nature06407 (  0) 0) |
| [10] |
BREYDO L, SALES A E, FERREIRA L, et al. Effects of osmolytes on protein-solvent interactions in crowded environment: Analyzing the effect of TMAO on proteins in crowded solutions[J]. Archives of Biochemistry and Biophysics, 2015, 570: 66-74. DOI:10.1016/j.abb.2015.02.021 (  0) 0) |
| [11] |
JANG W D, KIM G B, KIM Y, et al. Applications of artificial intelligence to enzyme and pathway design for metabolic engineering[J]. Current Opinion in Biotechnology, 2022, 73: 101-107. DOI:10.1016/j.copbio.2021.07.024 (  0) 0) |
| [12] |
KASHYAP A, KUMAR S, DUTT R. A review on structurally diversified synthesized molecules as monoacylglycerol lipase inhibitors and their therapeutic uses[J]. Current Drug Research Reviews, 2022, 14(2): 96-115. DOI:10.2174/2589977514666220301111457 (  0) 0) |
| [13] |
MICHALSKA K, JOACHIMIAK A. Structural genomics and the protein data bank[J]. Journal of Biological Chemistry, 2021, 296: 100747. DOI:10.1016/j.jbc.2021.100747 (  0) 0) |
| [14] |
KHAN F I, LAN D, DURRANI R, et al. The lid domain in lipases: Structural and functional determinant of enzymatic properties[J]. Frontiers in Bioengineering and Biotechnology, 2017, 5: 16. DOI:10.3389/fbioe.2017.00016 (  0) 0) |
| [15] |
JANSON G, PAIARDINI A. PyMod 3: A complete suite for structural bioinformatics in PyMOL[J]. Bioinformatics, 2021, 37(10): 1471-1472. DOI:10.1093/bioinformatics/btaa849 (  0) 0) |
| [16] |
LIU Lin, ZHOU Yong, QU Mingbo, et al. Structural and biochemical insights into the catalytic mechanisms of two insect chitin deacetylases of the carbohydrate esterase 4 family[J]. Journal of Biological Chemistry, 2019, 294(15): 5774-5783. DOI:10.1074/jbc.RA119.007597 (  0) 0) |
| [17] |
TROTT O, OLSON A J. AutoDock Vina: Improving the speed and accuracy of docking with a new scoring function, efficient optimization, and multithreading[J]. Journal of Computational Chemistry, 2010, 31(2): 455-461. DOI:10.1002/jcc.21334 (  0) 0) |
| [18] |
VREVEN T, VANGAVETI S, BORRMAN T M, et al. Performance of ZDOCK and IRAD in CAPRI rounds 39-45[J]. Proteins, 2020, 88(8): 1050-1054. DOI:10.1002/prot.25873 (  0) 0) |
| [19] |
LONDON N, RAVEH B, COHEN E, et al. Rosetta FlexPepDock web server--high resolution modeling of peptide-protein interactions[J]. Nucleic Acids Research, 2011, 39(Web server issue): : W249-W253. DOI:10.1093/nar/gkr431 (  0) 0) |
| [20] |
NAGAR B, BORNMANN W G, PELLICENA P, et al. Crystal structures of the kinase domain of c-Abl in complex with the small molecule inhibitors PD173955 and imatinib (STI-571)[J]. Cancer Research, 2002, 62(15): 4236-4243. (  0) 0) |
| [21] |
KADUKOVA M, CHUPIN V, GRUDININ S. Docking rigid macrocycles using Convex-PL, AutoDock Vina, and RDKit in the D3R grand challenge 4[J]. Journal of Computer-Aided Molecular Design, 2020, 34(2): 191-200. DOI:10.1007/s10822-019-00263-3 (  0) 0) |
| [22] |
SPITZER R, CLEVES A E, JAIN A N. Surface-based protein binding pocket similarity[J]. Proteins, 2011, 79(9): 2746-2763. DOI:10.1002/prot.23103 (  0) 0) |
| [23] |
CHIKHI R, SAEL L, KIHARA D. Real-time ligand binding pocket database search using local surface descriptors[J]. Proteins, 2010, 78(9): 2007-2028. DOI:10.1002/prot.22715 (  0) 0) |
| [24] |
SELVARAJ C, DINESH D C, PANWAR U, et al. Structure-based virtual screening and molecular dynamics simulation of SARS-CoV-2 Guanine-N7 methyltransferase (nsp14) for identifying antiviral inhibitors against COVID-19[J]. Journal of Biomolecular Structure and Dynamics, 2021, 39(13): 4582-4593. DOI:10.1080/07391102.2020.1778535 (  0) 0) |
| [25] |
DO P C, LEE E H, LE L. Steered molecular dynamics simulation in rational drug design[J]. Journal of Chemical Information and Modeling, 2018, 58(8): 1473-1482. DOI:10.1021/acs.jcim.8b00261 (  0) 0) |
| [26] |
BEU T A, FARCAS A. CHARMM force field and molecular dynamics simulations of protonated polyethylenimine[J]. Journal of Computational Chemistry, 2017, 38(27): 2335-2348. DOI:10.1002/jcc.24890 (  0) 0) |
| [27] |
HOFFMANN F, MULDER F A A, SCHAFER L V. Accurate methyl group dynamics in protein simulations with AMBER force fields[J]. The Journal of Physical Chemistry B, 2018, 122(19): 5038-5048. DOI:10.1021/acs.jpcb.8b02769 (  0) 0) |
| [28] |
XIAO Xiuchan, ZENG Xiaojun, YUAN Yuan, et al. Understanding the conformation transition in the activation pathway of beta2 adrenergic receptor via a targeted molecular dynamics simulation[J]. Physical Chemistry Chemical Physics, 2015, 17(4): 2512-2522. DOI:10.1039/c4cp04528a (  0) 0) |
| [29] |
KIM S, CHEN Jie, CHENG Tiejun, et al. PubChem 2019 update: Improved access to chemical data[J]. Nucleic Acids Research, 2019, 47(D1): D1102-D1109. DOI:10.1093/nar/gky1033 (  0) 0) |
| [30] |
BUNTROCK R E. ChemOffice Ultra 7.0[J]. Journal of Chemical Information and Computer Sciences, 2002, 42(6): 1505-1506. DOI:10.1021/ci025575p (  0) 0) |
| [31] |
SCHALLER D, PACH S, BERMUDEZ M, et al. Exploiting water dynamics for pharmacophore screening[J]. Methods in Molecular Biology, 2021, 2266: 227-238. DOI:10.1007/978-1-0716-1209-5_13 (  0) 0) |
| [32] |
SLAGMAN S, ZUILHOF H, FRANSSEN M C R. Laccase-mediated grafting on biopolymers and synthetic polymers: A critical review[J]. Chemistry and Biology Chemistry, 2018, 19(4): 288-311. DOI:10.1002/cbic.201700518 (  0) 0) |
| [33] |
CHEN Xuehui, YAN Huiwen, LIU Dan, et al. Structure basis for AA98 inhibition on the activation of endothelial cells mediated by CD146[J]. iScience, 2021, 24(5): 102417. DOI:10.1016/j.isci.2021.102417 (  0) 0) |
| [34] |
ZHENG Wei, ZHANG Chengxin, BELL E W, et al. Ⅰ-TASSER gateway: A protein structure and function prediction server powered by XSEDE[J]. Future Generation Computer Systems, 2019, 99: 73-85. DOI:10.1016/j.future.2019.04.011 (  0) 0) |
| [35] |
XIA Yuanling, SUN Jianhong, AI Shimeng, et al. Insights into the role of electrostatics in temperature adaptation: a comparative study of psychrophilic, mesophilic, and thermophilic subtilisin-like serine proteases[J]. RSC Advances, 2018, 8(52): 29698-29713. DOI:10.1039/C8RA05845H (  0) 0) |
| [36] |
TANG Heng, SHI Ke, SHI Cheng, et al. Enhancing subtilisin thermostability through a modified normalized B-factor analysis and loop-grafting strategy[J]. Journal of Biological Chemistry, 2019, 294(48): 18398-18407. DOI:10.1074/jbc.RA119.010658 (  0) 0) |
| [37] |
WATERHOUSE A, BERTONI M, BIENERT S, et al. SWISS-MODEL: Homology modelling of protein structures and complexes[J]. Nucleic Acids Research, 2018, 46(W1): W296-W303. DOI:10.1093/nar/gky427 (  0) 0) |
| [38] |
SU Bingmei, WU Dongyan, XU Xinqi, et al. Design of a PL18 alginate lyase with flexible loops and broader entrance to enhance the activity and thermostability[J]. Enzyme and Microbial Technology, 2021, 151: 109916. DOI:10.1016/j.enzmictec.2021.109916 (  0) 0) |
| [39] |
XU Xinqi, ZENG Dayong, WU Dongyan, et al. Single-point mutation near active center increases substrate affinity of alginate lyase AlgL-CD[J]. Applied Biochemistry and Biotechnology, 2021, 193(5): 1513-1531. DOI:10.1007/s12010-021-03507-x (  0) 0) |
| [40] |
NIKOLAIVITS E, VALMAS A, DEDES G, et al. Considerations regarding activity determinants of fungal polyphenol oxidases based on mutational and structural studies[J]. Applied and Environmental Microbiology, 2021, 87(11): e00396-00321. DOI:10.1128/AEM.00396-21 (  0) 0) |
| [41] |
LYU C J, ZHAO W R, HU S, et al. Physiology-oriented engineering strategy to improve gamma-aminobutyrate production in lactobacillus brevis[J]. Journal of Agricultural and Food Chemistry, 2017, 65(4): 858-866. DOI:10.1021/acs.jafc.6b04442 (  0) 0) |
| [42] |
PATIL U, MOKASHE N, SHAHA J, et al. Ultrasound-assisted improvements in biocatalytic activity and production of organic-solvent stable protease from Bacillus circulans MTCC 7942[J]. Ultrasonics Sonochemistry, 2018, 40(Pt A): 201-205. DOI:10.1016/j.ultsonch.2017.07.012 (  0) 0) |
| [43] |
WANG Yang, ZHANG Zhadi, HE Ronghai, et al. Improvement in enzymolysis efficiency and changes in conformational attributes of corn gluten meal by dual-frequency slit ultrasonication action[J]. Ultrasonics Sonochemistry, 2020, 64: 105038. DOI:10.1016/j.ultsonch.2020.105038 (  0) 0) |
| [44] |
KIESERLING H, GIEFER P, UTTINGER M J, et al. Structure and adsorption behavior of high hydrostatic pressure-treated beta-lactoglobulin[J]. Journal of Colloid and Interface Science, 2021, 596: 173-183. DOI:10.1016/j.jcis.2021.03.051 (  0) 0) |
| [45] |
ASLANI E, ABRI A, PAZHANG M. Immobilization of trypsin onto Fe3O4@SiO2 -NH2 and study of its activity and stability[J]. Colloids and Surfaces B: Biointerfaces, 2018, 170: 553-562. DOI:10.1016/j.colsurfb.2018.06.022 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


