2. 聊城市人民医院 呼吸内科,山东 聊城 252000
2. Department of Respiratory Medicine, Liaocheng People's Hospital, Liaocheng 252000, Shandong, China
胃癌是最常见的恶性肿瘤之一,在全球范围内疾病致死率排名中胃癌排名第三位[1]。我国是胃癌的高发国家,胃癌的发病率高于世界平均水平。胃癌的恶性程度高,侵袭性强,据统计胃癌的五年生存率仅为10%[2]。胃癌的发生发展是一个长期复杂的过程,多种因素共同作用才导致其形成。目前,胃癌相关的一系列风险因子已经被研究者们发现,比如:饮食习惯、病毒病菌感染等。此外,越来越多的研究发现,遗传因素也是胃癌发生的一个重要风险因素,通过影响细胞进程从而导致胃癌的发生。但是胃癌的致病过程非常复杂,目前其致病机制还未能尚未研究透彻[3]。因此,迫切需要寻找到有效的胃癌生物标记物,为胃癌的诊断和治疗提供有效的靶点。传统的生物学研究往往仅以单个分子为研究对象,虽然这种研究在分子水平上揭示疾病的致病机制作用显著[4-5]。但是在复杂致病机制的研究中,不仅需要了解单个分子对疾病的影响,还要分析这些分子间是如何相互作用、相互影响的,从而全面的了解疾病的发生机制。现阶段的研究发现很多蛋白编码基因如:FGFR2、APC、CASP10、IRF1等均为已知的胃癌致病基因,这些编码基因在肿瘤患者中不但异常表达,甚至会影响患者的预后[6-8]。目前研究发现微小非编码RNA(microRNA, miRNA)可以调控mRNA,抑制mRNA表达或者降解mRNA[9]。长非编码RNA(Long non-coding RNA, lncRNA)可以作为miRNA“海绵”吸附miRNA,使得miRNA对靶基因的抑制减小,间接调控mRNA表达升高[10-11]。本研究的样本是从加利福尼亚大学圣克鲁斯大学(UCSC)xena数据库中下载的mRNA、miRNA、lncRNA表达数据以及临床数据,通过miRNA调控mRNA/lncRNA关系数据结合三者的共表达关系,识别出lncRNA-miRNA-mRNA三元组,不同的三元组相互关联形成胃癌相关的共表达网络,从网络中筛选出已知致病的且差异表达胃癌相关基因关联的lncRNA,识别出新的生物标记物,并且分析三元组关系对患者预后的影响。
1 材料与方法 1.1 数据来源本研究中的所有样本表达数据均从加利福尼亚大学圣克鲁斯大学(UCSC)xena(https://xenabrowser.net)的数据库中下载。共407个样本,其中胃癌样本375个,正常样本32个。所有样本都检测了mRNA、miRNA、lncRNA的表达数据,均使用Illumina Hiseq高通量测序平台,其中mRNA和lncRNA的表达数据为基因的Reads count值,miRNA的表达数据为Log(Reads count+1)值。使用Ensembl[12]数据库内的ENSG ID和基因类型对应关系数据,将mRNA和lncRNA的表达进行区分。使用miRBase[13]数据库将miRNA数据的ID对应为成熟miRNA名称。所有的患者样本均包含样本的生存状态、总体生存、性别、年龄及肿瘤病理学状态等信息。miRNA和mRNA/lncRNA的互作信息来源于RNA互作数据库ENCORI[14],数据库包含超过2 500 000条miRNA-mRNA互作关系,1 100 000条miRNA-lncRNA互作关系。已知胃癌(Gsatric cancer)相关的编码基因来源于MalaCards[15]数据库,共有296个基因和胃癌相关,本次研究选取关系最紧密的31个基因进行接下来的分析。
1.2 差异表达基因筛选为了全面的研究胃癌相关编码基因受哪些非编码基因的调控,不仅需要整理收集疾病已知基因,也需要整合胃癌差异表达基因。本研究通过胃癌数据筛选出差异表达的编码基因,使用R包edgeR[16]分析mRNA的差异表达情况,错误发现率(False discovery rate, FDR), 肿瘤样本和正常样本差异倍数(Fold change, FC)。然后使用DAVID对差异表达的胃癌相关基因进行生物学功能富集分析,显著的功能富集结果如图 1所示。
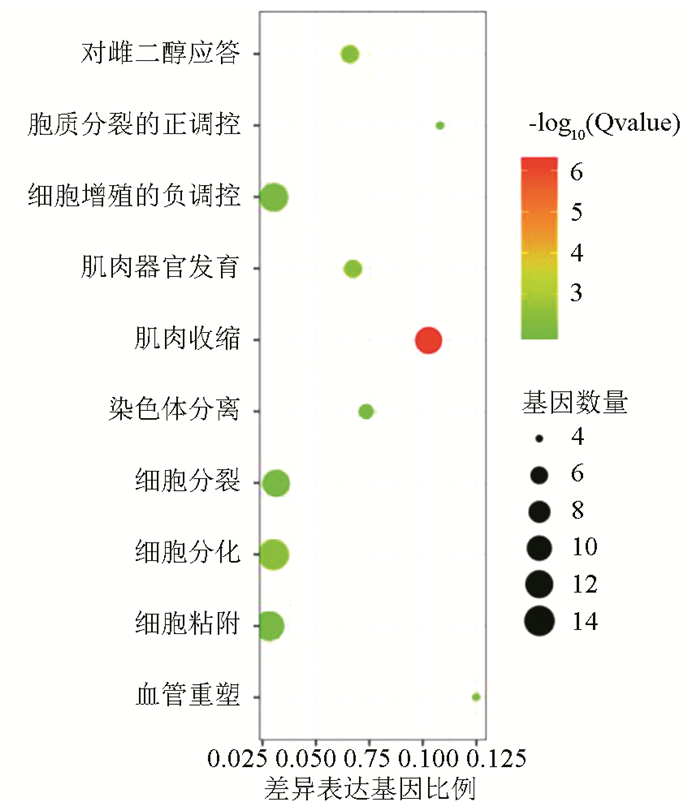
|
图 1 差异表达基因功能富集分析结果 Figure 1 Functional enrichment of differentially expressed genes |
mRNA和lncRNA共同竞争miRNA形成互作三元组。首先提取所有已知基因和筛选后的差异表达基因互作的miRNA,根据提取后的miRNA筛选与其互作的lncRNA,得到潜在的三元组互作关系对。然后使用xena下载的表达数据进行三者的相关性计算,计算方法使用的是斯皮尔曼相关,选取P < 0.01的miRNA与mRNA/lncRNA负相关(R < 0)关系对以及mRNA与lncRNA正相关(R>0)关系对,这样就从潜在的三元组中进一步计算得到了存在关联关系的三元组。然后使用软件Cytoscape 3.7.2进行三元组构建的网络进行可视化。
1.4 风险模块筛选根据得到的三元组关系对,得到多个连通的模块。所有三元组关系对均为miRNA介导,通过miR2Disease、HMDD[17]数据库以及文献检索,获取已知胃癌相关miRNA,对模块内的miRNA进行超几何检验,找到检验结果显著的模块。超几何检验公式如下:
| $ P=\frac{\left(\begin{array}{l} M \\ K \end{array}\right) \times\left(\begin{array}{l} N-M \\ n-k \end{array}\right)}{\left(\begin{array}{l} N \\ n \end{array}\right)} $ | (1) |
公式中N为网络内所有的miRNA数量,M为网络内HMDD数据库内获取胃癌相关miRNA数量,n为模块内miRNA数量,k为模块内胃癌相关miRNA数量,模块的显著性按照P值由小到大进行排序。
1.5 统计分析分析使用R语言进行,在分析过程中使用的R包分别为:ggplot2、edgeR、TCGAbiolinks、forestplot、clusterProfiler、pheatmap、survival。使用患者的基因表达的高低,将样本分为两组。使用Kaplan-Meier曲线和log-rank检验来评估两组患者生存时间的差异。
2 结果 2.1 疾病相关基因及差异表达基因收集整理目前研究已经证实的胃癌相关基因以及胃癌表达谱内显著差异表达的基因作为候选的疾病相关基因,从MalaCards数据库获得了31个目前研究最为重要的基因,包括FGFR2、APC、CASP10、IRF1等在胃癌发生发展中起作用的关键基因。根据下载数据的样本编号,把疾病和正常样本进行分类,使用R软件的edgeR方法对375个疾病样本和32个正常样本进行差异表达分析,共分析了22 686个编码基因,根据设定的差异基因型筛选尺度,获取了237个显著差异表达的基因,其中上调的基因95个,下调的基因142个。将得到的差异表达基因进行聚类,如图 2所示,从聚类结果上可以看出,差异表达基因可以有效的将正常和疾病样本区分开。
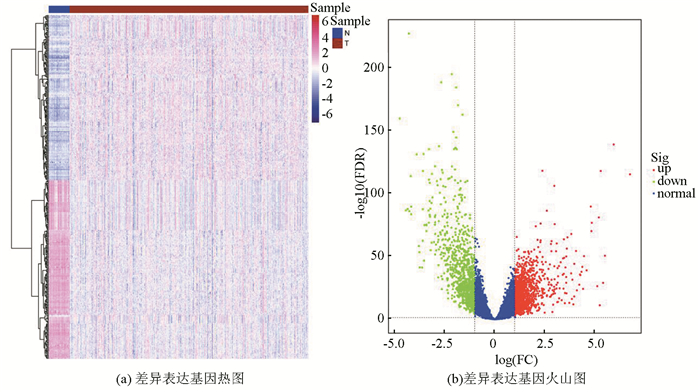
|
图 2 差异表达基因热图及火山图 Figure 2 Heatmap and volcano plot of differentially expressed genes 注:红色圆点代表高表达基因,绿色圆点代表低表达基因. |
基于RNA互作数据库ENCORI(https://starbase.sysu.edu.cn/index.php),筛选出miRNA和mRNA/lncRNA的互作关系对,然后对已疾病相关基因同miRNA关系对、miRNA和lncRNA关系对、共享miRNA的mRNA和lncRNA关系对进行相关性计算,共计算了2 201对miRNA和mRNA/lncRNA的相关性,获得了显著负相关的关系对279条,2 615对mRNA和lncRNA的相关性,获得了显著正相关关系对1 220条。整合以上显著的关系对,保留能形成互作三元组的关系对,最终共得到包含146条关系对的三元组网络,网络内包含32个mRNA,40个miRNA,44个lncRNA,如图 3所示。
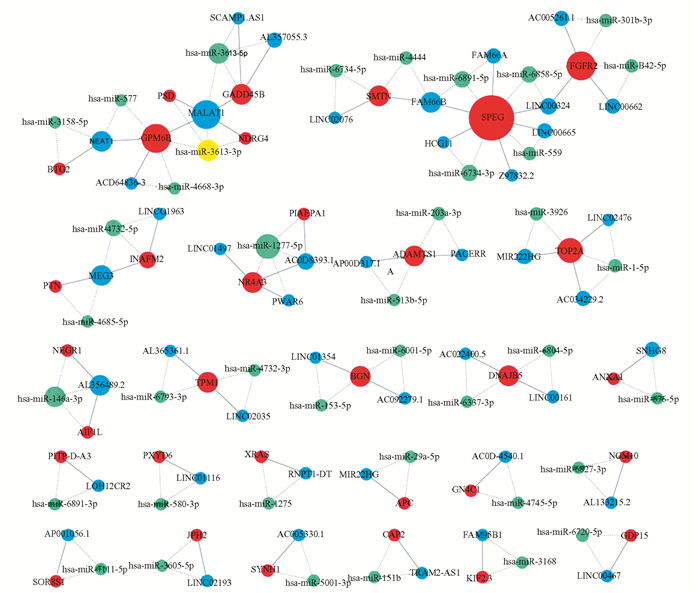
|
图 3 胃癌相关mRNA-miRNA-lncRNA共表达网络 Figure 3 Co-expression network of mRNA-miRNA-lncRNA Note: The blue circle represents lncRNA, the red circle represents mRNA, the green circle represents miRNA, the solid line denotes positive correlation of connected genes, the dotted line denotes negative correlation of connected genes. |
由三元组关系对构建的网络并不是全连通的网络,本研究想识别出哪些小的独立的模块是胃癌相关三元组网络中最为重要的模块,哪些三元组关系对在模块中起到关键作用。模块内所有的三元组关系对均通过miRNA介导,所以使用已知的疾病相关数据库,对每个模块内的miRNA进行统计学显著性检验,已知胃癌相关miRNA越能显著富集在模块内,那么模块越可能在胃癌的发生发展中起到关键作用。三元组互作关系网内共40个miRNA, 其中7个是已知胃癌相关基因,对每个模块进行超几何检验,通过检验结果发现,9号模块结果最为显著(P=0.02)。继续分析里模块内唯一的编码基因BGN,发现在很多研究中已经发现了BGN在胃癌患者中的异常表达,在胃癌发生发展中起到重要作用[18]。但以往研究并未发现BGN表达异常的原因,本研究认为BGN高表达的原因是与其形成三元组的lncRNA吸附了抑制BGN表达的miRNA,从而导致了BGN的高表达,lncRNA LINC01354和AC092279.1与BGN相关性如图 4所示。胃癌患者中BGN显著高表达,而且将胃癌患者通过BGN表达值高低分为两组,高表达患者的预后显著低于低表达的患者,如图 5所示。

|
图 4 BGN与lncRNA LINC01354、AC092279.1相关性 Figure 4 Correlation between BGN, lncRNA LINC01354, and AC092279.1 |

|
图 5 BGN基因的差异表达情况及生存曲线分析 Figure 5 Differential expression and survival analysis of BGN |
在过去的十几年中,已经发现了miRNA和lncRNA能在人类的癌症中其关键作用,而且与癌症的发生发展密切相关[19]。miRNA是最著名的非编码RNA,它参与调控编码基因的表达,主要是参与降解mRNA或者抑制其表达,从而削弱相应编码蛋白的功能[20]。近年来另一个非编码RNA,lncRNA的研究也越来越多,lncRNA是一种长度大于200 bp的非编码RNA,随着研究的不断进展发现了lncRNA的功能范围很广,最为研究人员所认可的是lncRNA通过miRNA介导与mRNA互作,调控mRNA的表达,这种相互竞争关系的互作的发现,为研究者发现新的癌症生物标记物提供了帮助[21-22]。作为人类最为常见的实体肿瘤之一,胃癌的发病机制的研究还不清晰[23],尽管研究者们大量的研究已经很大程度改善了疾病的治疗效果,但是对于晚期的胃癌患者预后还是很差[24]。胃癌早期不易被发现,因为早期没有显著症状出现,多数患者在出现厌食、消化不良、腹痛的时候才会进一步进行医学检查,一旦确诊为胃癌,大部分已经是胃癌中晚期,这时候胃癌已经开始快速发展了。胃癌能够得到更好的治疗的前提是清晰的了解胃癌的致病机制,只有明确了胃癌发生发展的详细分子机制,才能找到更好治疗方案,调整最优的治疗策略,才能帮助研发出更适合的药物[3]。
本研究中,从胃癌已知的疾病相关编码基因以及差异表达基因出发,找到在转录过程中能调控这些重要的胃癌基因表达的miRNA以及lncRNA,lncRNA通过"吸附"miRNA,从而使得miRNA对mRNA的抑制能力减弱,导致mRNA的表达上升。致癌基因的高表达以及抑癌基因的低表达都是胃癌发生发展的原因,在本研究中发现了23个mRNA-miRNA-lncRNA三元组关系对模块,通过进一步分析,发现第9组模块是研究结果中最为显著和胃癌相关的,同时也发现了,这个模块中的核心基因在胃癌患者中显著高表达,而且在胃癌患者中,表达值越高的患者预后越差。在这一模块中所有的miRNA都是以往研究已经发现的胃癌相关miRNA,基于研究发现的这一模块结构,本研究认为lncRNA LINC01354和AC092279.1很可能在胃癌发生发展中扮演了重要的角色,是新的潜在的生物标记物。研究发现了多个三元组关系模块,而且多个模块内包含目前研究已知的疾病相关非编码RNA,比如lcnRNA MALTA1及MEG3[25-26],诸多研究中已经发现这两个基因的异常表达影响胃癌患者的预后,在本研究的模块内,这两个lncRNA也是模块内的核心基因,在模块中起到关键作用。通过这些已知的胃癌相关lncRNA调控的mRNA也可能在胃癌致病过程中起到关键作用,希望在后续的研究中继续验证。
4 结论本研究从胃癌已知的疾病相关编码基因以及差异表达基因出发,构建了胃癌mRNA-miRNA-lncRNA三元组关系组成得ceRNA调控网络,通过对网络的挖掘,识别出与胃癌发生发展相关的调控lncRNA。LINC01354和AC092279.1很可能在胃癌发病机制中发挥了重要的调控作用,是新的潜在的生物标记物。
mRNA-miRNA-lncRNA三元组关系对构建的模块能够影响胃癌患者的预后,为以往研究中无法解释的胃癌患者中mRNA表达异常提供了一种理论依据。但本研究仍有不足之处,没能整理完整的胃癌相关lncRNA,没有从其它角度继续对三元组关系加以验证。后续计划将包括疾病相关非编码RNA信息在内的所有的疾病相关信息进行整合,并且将胃癌数据进行分型,研究不同亚型的胃癌患者相关的三元组关系模块,以及重要的调控基因,为胃癌治疗方法提供研究基础。
| [1] |
FERLAY J, SOERJOMATARAM I, DIKSHIT R, et al. Cancer incidence and mortality worldwide: Sources, methods and major patterns in GLOBOCAN 2012[J]. International Journal of Cancer, 2015, 136(5): E359-E386. DOI:10.1002/ijc.29210 (  0) 0) |
| [2] |
CHEN W, ZHENG R, BAADE P D, et al. Cancer statistics in China, 2015[J]. CA-A Cancer Journal for Clinicians, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 (  0) 0) |
| [3] |
VAN CUTSEM E, SAGAERT X, TOPAL B, et al. Gastric cancer[J]. Lancet, 2016, 388(10060): 2654-2664. DOI:10.1016/S0140-6736(16)30354-3 (  0) 0) |
| [4] |
PHAROAH P D, GUILFORD P, CALDAS C, et al. Incidence of gastric cancer and breast cancer in CDH1 (E-cadherin) mutation carriers from hereditary diffuse gastric cancer families[J]. Gastroenterology, 2001, 121(6): 1348-1353. DOI:10.1053/gast.2001.29611 (  0) 0) |
| [5] |
EL-OMAR E M, CARRINGTON M, CHOW W H, et al. Interleukin-1 polymorphisms associated with increased risk of gastric cancer[J]. Nature, 2000, 404(6776): 398-402. DOI:10.1038/35006081 (  0) 0) |
| [6] |
SUN X J, ZHENG Z H, FU H, et al. The I1307K mutation and protein expression of APC gene in gastric cancer[J]. Hereditas(Beijing), 2003, 25(3): 253-357. (  0) 0) |
| [7] |
DUFFY M J, LAMERZ R, HAGLUND C, et al. Tumor markers in colorectal cancer, gastric cancer and gastrointestinal stromal cancers: European group on tumor markers 2014 guidelines update[J]. International Journal of Cancer, 2014, 134(11): 2513-2522. DOI:10.1002/ijc.28384 (  0) 0) |
| [8] |
JANG J H, SHIN K H, PARK J G. Mutations in fibroblast growth factor receptor 2 and fibroblast growth factor receptor 3 genes associated with human gastric and colorectal cancers[J]. Cancer Research, 2001, 61(9): 3541-3543. (  0) 0) |
| [9] |
WAHID F, SHEHZAD A, KHAN T, et al. MicroRNAs: Synthesis, mechanism, function, and recent clinical trials[J]. Biochimica Biophysica Acta, 2010, 1803(11): 1231-1243. DOI:10.1016/j.bbamcr.2010.06.013 (  0) 0) |
| [10] |
PARASKEVOPOULOU M D, HATZIGEORGIOU A G. Analyzing MiRNA-LncRNA Interactions[J]. Methods in Molecular Biology, 2016, 1402: 271-286. DOI:10.1007/978-1-4939-3378-5_21 (  0) 0) |
| [11] |
LIU Xiao, JIAO Tongli, WANG Yinzhong, et al. Long non-coding RNA GAS5 acts as a molecular sponge to regulate miR-23a in gastric cancer[J]. Minerva Medica, 2016, 9(11): 11412-11419. (  0) 0) |
| [12] |
ZERBINO D R, ACHUTHAN P, AKANNI W, et al. Ensembl 2018[J]. Nucleic Acids Research, 2018, 46(D1): D754-D761. DOI:10.1093/nar/gkx1098 (  0) 0) |
| [13] |
KOZOMARA A, BIRGAOANU M, GRIFFITHS-JONES S. miRBase: From microRNA sequences to function[J]. Nucleic Acids Research, 2019, 47(D1): D155-D162. DOI:10.1093/nar/gky1141 (  0) 0) |
| [14] |
LI Junhao, LIU Shun, ZHOU Hui, et al. starBase v2.0: decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data[J]. Nucleic Acids Research, 2014, 42(Database issue): D92-97. DOI:10.1093/nar/gkt1248 (  0) 0) |
| [15] |
RAPPAPORT N, TWIK M, PLASCHKES I, et al. MalaCards: An amalgamated human disease compendium with diverse clinical and genetic annotation and structured search[J]. Nucleic Acids Research, 2017, 45(D1): D877-D887. DOI:10.1093/nar/gkw1012 (  0) 0) |
| [16] |
ROBINSON M D, MCCARTHY D J, SMYTH G K. edgeR: A bioconductor package for differential expression analysis of digital gene expression data[J]. Bioinformatics, 2010, 26(1): 139-140. DOI:10.1093/bioinformatics/btp616 (  0) 0) |
| [17] |
HUANG Z, SHI J, GAO Y, et al. HMDD v3.0: A database for experimentally supported human microRNA-disease associations[J]. Nucleic Acids Research, 2019, 47(D1): D1013-D1017. DOI:10.1093/nar/gky1010 (  0) 0) |
| [18] |
HU Lei, DUAN Yantao, LI Jianfang, et al. Biglycan enhances gastric cancer invasion by activating FAK signaling pathway[J]. Oncotarget, 2014, 5(7): 1885-1896. DOI:10.18632/oncotarget.1871 (  0) 0) |
| [19] |
SCHMITT A M, CHANG H Y. Long noncoding RNAs in cancer pathways[J]. Cancer Cell, 2016, 29(4): 452-463. DOI:10.1016/j.ccell.2016.03.010 (  0) 0) |
| [20] |
LIN S, GREGORY R I. MicroRNA biogenesis pathways in cancer[J]. Nature Reviews Cancer, 2015, 15(6): 321-333. DOI:10.1038/nrc3932 (  0) 0) |
| [21] |
BEERMANN J, PICCOLI M T, VIERECK J, et al. Non-coding RNAs in development and disease: background, mechanisms, and therapeutic approaches[J]. Physiological Reviews, 2016, 96(4): 1297-1325. DOI:10.1152/physrev.00041.2015 (  0) 0) |
| [22] |
SONG Yongxi, SUN Jingxu, ZHAO Junhua, et al. Non-coding RNAs participate in the regulatory network of CLDN4 via ceRNA mediated miRNA evasion[J]. Nature Communications, 2017, 8(1): 289. DOI:10.1038/s41467-017-00304-1 (  0) 0) |
| [23] |
LI Qinglin, ITO K, SAKAKURA C, et al. Causal relationship between the loss of RUNX3 expression and gastric cancer[J]. Cell, 2002, 109(1): 113-124. DOI:10.1016/s0092-8674(02)00690-6 (  0) 0) |
| [24] |
YAMASHITA K, EMA A, HOSODA K, et al. Macroscopic appearance of Type Ⅳ and giant Type Ⅲ is a high risk for a poor prognosis in pathological stage Ⅱ/Ⅲ advanced gastric cancer with postoperative adjuvant chemotherapy[J]. World Journal of Gastrointestinal Oncology, 2017, 9(4): 166-175. DOI:10.4251/wjgo.v9.i4.166 (  0) 0) |
| [25] |
QI Y, OOI H S, WU J, et al. MALAT1 long ncRNA promotes gastric cancer metastasis by suppressing PCDH10[J]. Oncotarget, 2016, 7(11): 12693-12703. DOI:10.18632/oncotarget.7281 (  0) 0) |
| [26] |
CUI Haibin, GE H E, WANG Yongsen, et al. MiR-208a enhances cell proliferation and invasion of gastric cancer by targeting SFRP1 and negatively regulating MEG3[J]. The International Journal of Biochemistry & Cell Biology, 2018, 102: 31-39. DOI:10.1016/j.biocel.2018.06.004 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


