2. 内蒙古自治区功能基因组生物信息学重点实验室,包头 014010
2. Inner Mongolia Key Laboratory of Functional Genomics and Bioinformatics, Inner Mongolia University of Science and Technology, Baotou 014010, China
减数分裂重组是细胞减数分裂过程的一个基本组成部分。它始于减数分裂Ⅰ前期,通过拓扑异构酶SPO11等蛋白质复合物作用下,诱导程序化的DNA双链断裂(DSB),进而在同源染色体之间形成交叉,发生遗传物质交换[1-3]。减数分裂重组确保了染色体正确分离、基因组的稳定性,并且能够将单倍型遗传物质进行更新,传递给下一代,产生遗传多样性[4-5]。重组异常会导致染色体的不正确分离和非整倍体,可能会导致原发性卵巢功能不全、不孕、卵巢早衰等多种疾病[6-7]。减数分裂重组不仅通过协助染色体分离在上游配子发生中发挥重要作用,而且还推动基因组进化。研究表明,重组可能通过诱导突变或选择相关方式推动基因组进化。例如,在哺乳动物中,基因组中重组率的不均匀分布可能通过GC偏向的基因转换影响碱基含量[8-10]。
全面了解哺乳动物和植物中的核苷酸变异对于揭示疾病的遗传基础至关重要。在研究基因组中的突变模式时,突变的上下文依赖性通常较少受到关注。一些研究表明,SNP位点的替换模式对上下文具有依赖关系。忽略突变对紧邻碱基的依赖性可能会影响祖先等位基因的推断和选择作用的检验结果[11]。研究表明,DNA复制错误、甲基化胞嘧啶脱氨基和偏向基因转变存在上下文依赖性,突变率强烈依赖于相邻核苷酸[12]。考虑上下文依赖的情况下,突变率的变化幅度最高可达50倍[12]。突变的上下文依赖关系中,最主要的是紧邻碱基效应[13]。在哺乳动物中,主要的紧邻碱基依赖效应发生在CpG二核苷酸上,这是由于甲基化胞嘧啶脱氨基导致突变的结果[14],因此二核苷酸CpG含量一般强烈影响核苷酸替换的比例。
据文献报道,在人的基因组当中,突变对邻近5bp之内的侧翼序列具有很强的依赖性,而且这种依赖关系在不同区域中不尽相同[15-16]。用全基因组水平的碱基含量进行归一化处理后,突变位点的上游碱基偏好为C,下游碱基偏好为G[15-16]。如果染色体上的GC含量较高,则突变位点的上游碱基为C,下游为G的几率更高[15]。在水稻中,同样存在突变位点的上游碱基为C、下游碱基为G的偏好性,但这种偏好性与GC含量呈负相关[17]。突变位点两侧C/G出现的概率总体上高于A/T碱基。另外,有研究表明,突变相邻碱基的偏好性在不同的染色体上也有一定的差异[13, 15, 17]。
小鼠和人类相邻碱基对变异的影响相似[18],相邻碱基距离变异的位置越近其影响越大。根据SNP位点的实际观测频率发现,突变位点上游紧邻碱基的偏好性由强至弱的顺序为A>T>C>G,下游紧邻碱基的偏好性顺序为T>A>G>C。这种顺序受到全基因组碱基含量的影响:用全基因组碱基含量标准化后SNP上游紧邻碱基的偏好性由强至弱的顺序为C>G≈A>T,下游紧邻碱基的偏好性顺序为G>C≈T>A(遵循碱基配对互补原则)。跟人类基因组相比,小鼠中突变对相邻碱基的偏好性比人类基因组中的偏好性弱很多[15, 18]。
在水稻基因组中,基因间区和内含子区域发生的变异,其突变上游碱基偏好为C,下游碱基偏好为G。这种效应广泛存在于转换突变中[17],这与CpG中甲基化胞嘧啶脱氨基变成T的情况一致[14-17]。在水稻基因间区和内含子区中,转换/颠换的比值与邻近碱基中的A/T值呈负相关[17]。在编码区中的变异没有类似情况。
虽然有文献报道邻近碱基影响单核苷酸多态性,但定量且有效评估这种“近邻碱基效应”的方法并不多见。对于依赖于紧邻碱基的突变偏好性如何随重组率变化也没有深入研究。我们之前的研究发现,果蝇和人类基因组中的二核苷酸组成和重组率相关,这可能与高重组区中的依赖于紧邻碱基的突变偏好性有关[19-20]。但该研究中并没有利用SNP数据对具体的突变对紧邻碱基的依赖性进行直接观测。深入探讨不同重组率下突变对紧邻碱基的依赖性,有助于我们进一步理解碱基替换模式和基因组进化。
1 数据与方法 1.1 SNP数据人类SNP数据(基于hg19版本)来源于“千人基因组计划(1 000 genomes project,1KG)”。SNP的VCF文件(基于hg37版本)从http://ftp.1000genomes.ebi.ac.uk/vol1/ftp/release/20130502/下载。该VCF文件包含基于东亚(East Asian)、南亚(South Asian)、非洲(African)、欧洲(European)、北美(North America)五个超级种群共2 504个样本的人类基因组变异信息,其中包括变异位点、变异类型、祖先等位基因(Ancestral allele)、派生等位基因(Derived allele)、每个样本中的基因型(Genotype)等详细信息。筛选出质量合格的SNP信息(不包括插入缺失突变)。
1.2 重组率图谱重组率数据来自Bherer等[21]2017年发表的文章。该数据是基于100 000多个减数分裂相关的重组事件构建的高分辨率遗传图谱。该图谱中收集了近6个人类系谱的重组事件集合,涉及总共104 246个个体。绝大多数减数分裂来自欧洲血统的个体,占所有减数分裂的93.7%,6.3%来自其他来源,包括非裔美国人(1.6%)、东亚人(1.8%)和拉丁裔美国人(1.5%)。合并后的数据集包括2 338 628个女性和999 007个男性重组事件。该重组率图谱中,每一个遗传标记位点对应一个重组率值。基于该遗传图谱计算的平均重组率为1.202 cM/Mb。每个SNP位点的重组率数据取自其邻近下游遗传标记位置的重组率值。我们将重组率大于等于1 cM/Mb的定义为高重组率,重组率小于等于0.1 cM/Mb的定义为低重组率,重组率介于0.1 cM/Mb和1 cM/Mb之间的数据定义为中重组率。低中高三组中,包含的SNP数量分别为19 325 661(24.7%)、39 149 595(50.0%)和19 838 307(25.3%)。
1.3 SNP注释用SnpEff(version 4)注释SNPs。它可以注释和预测遗传变异的影响[22]。在注释中使用了重组率图谱、SNP数据。利用人类基因组(hg19版本)的fasta序列文件和人类基因注释信息构建了SnpEff注释所需的数据库,然后分别注释三个VCF文件(对应低、中、高重组率)。
1.4 SnpSift对不同基因组区域SNP的分离SnpSift(version 4)[22]是可以用来过滤、拆分、提取带注释的文件的一个工具。SnpEff注释SNP后,利用SnpSift筛选出不同基因组区域(Intron, Intergenic, Exon, Upstream,Downstream)的SNP,分别计算不同重组率下的编码区与非编码区的SNP对紧邻碱基的依赖性。
1.5 条件互信息的定义条件互信息(Conditional mutual information, CMI)是给定第三个变量时两个随机变量互信息的期望值[23]。如果确定第三变量时另外两个随机变量的互信息增大,则认为第三变量确定的条件下,另外两个随机变量的相关性得到增强。条件互信息在特征筛选、多维时间序列图模型、miRNA调控网络分析等领域被广泛应用[24-27]。
本文定义的平均条件互信息(见图 1),用于表示紧邻碱基确定的条件下SNP位点上的突变偏好性,是考虑紧邻碱基所有可能取值的平均条件互信息。部分条件互信息(Partial conditional mutual information)是只考虑特定紧邻碱基的条件互信息。为了计算条件互信息,提取每个SNP的位置信息以及上游和下游位置,并使用bedtools(Version 2.29.1)[28]的“getFasta”选项从基因组中获得相应的核苷酸。在此基础上用图 1所示的方式计算条件互信息。
|
图 1 用于量化依赖于紧邻碱基的突变偏好性的条件互信息计算示意图 Figure 1 Schematic diagram of conditional mutual information calculation for quantifyingneighbor-dependent mutation bias 注:评估突变对上游碱基的依赖性时,p(z)表示SNP位点上游紧邻碱基的频率;评估突变对两侧紧邻碱基的依赖性时,p(z)为SNP位点两侧碱基组合类型的频率,p(x)为祖先等位基因的频率,p(y)为衍生等位基因的频率. |
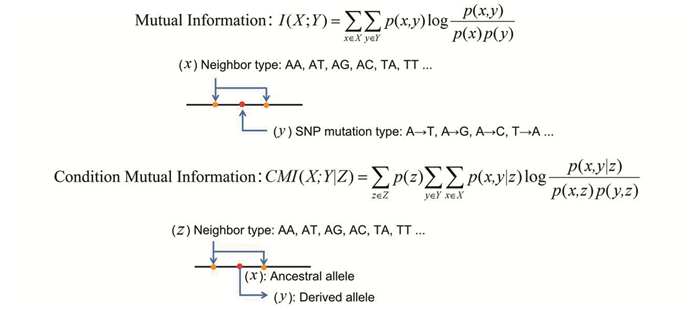
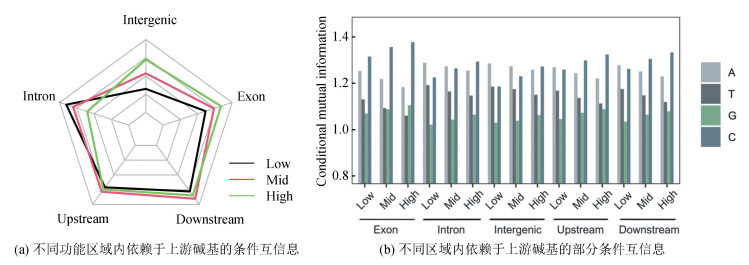
首先,根据条件互信息的定义分析了在上游紧邻碱基确定的情况下SNP位点的突变偏好性。结果表明,条件互信息随着重组率的增加而增加(见图 2a)。部分条件互信息的结果表明突变对上游位点的依赖性由强变弱的顺序为A>C>T>G。当上游紧邻碱基为A、C时,SNP位点具有非常强的突变偏好性(见图 2b)。当上游碱基为A或T时,随着重组率增加SNP突变偏好性减弱。当上游碱基为C或G时,结果正好相反,即随着重组率增加SNP的突变偏好性增强。
|
图 2 依赖于上游碱基的条件互信息和部分条件互信息 Figure 2 Upstream base-dependent conditional mutual information and partial conditional mutual information 注:图c-g中的数据表示特定上游碱基条件下SNP位点上发生特定突变的条件概率,即图 1公式中的p(x, y|z)。Low, Mid和High分别表示低、中、高三组重组率区. |
另外,对特定上游碱基条件下SNP位点上发生特定突变的条件概率进行聚类和比较分析(见图 2c-2g)。就整体而言,发现无论上游是什么碱基,SNP位点上A碱基都偏好突变成G碱基,在SNP上游碱基为T或C时,突变偏好性最强,其偏好性随着重组率升高而减弱(见图 2d);T碱基倾向突变成C碱基,当其上游碱基为A时,SNP位点有最强的突变偏好性,重组率对该突变偏好性具有抑制作用(见图 2e)。紧邻碱基为C时,SNP位点上具有最强的G到A的突变偏好性,且重组率越高,偏好性增强(见图 2f);C碱基倾向突变成T,受上游碱基的影响较小(见图 2g)。这种C>T的取代主要来自甲基化胞嘧啶的水解脱氨基作用[14~16]。
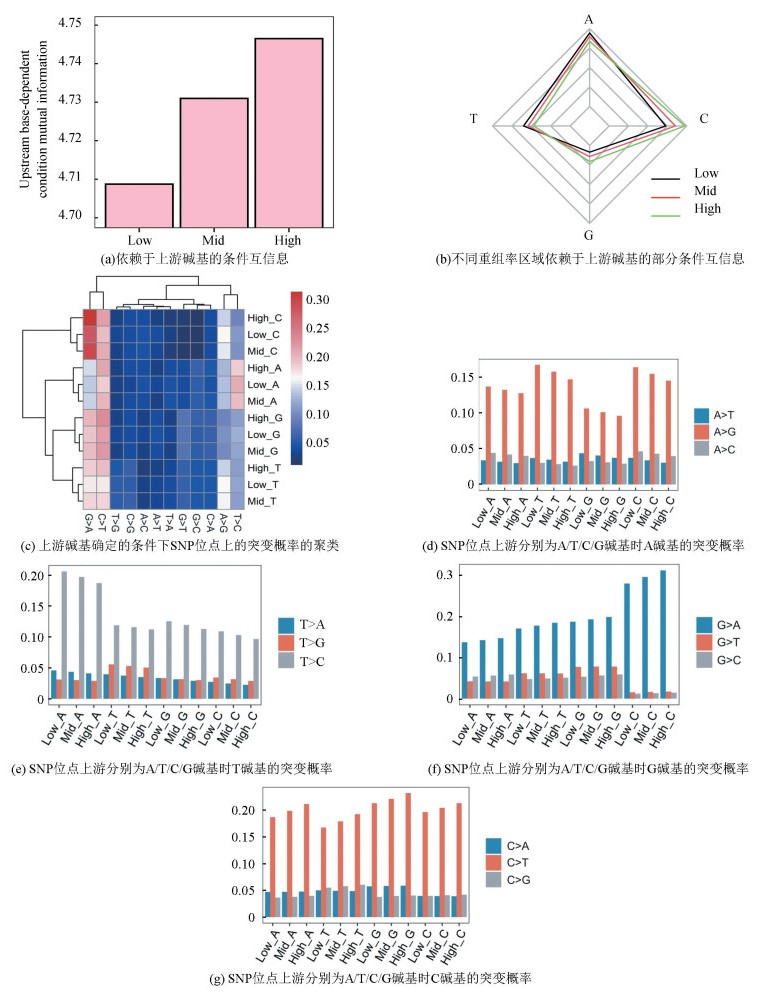
2.2 两侧碱基对SNP突变偏好性的影响SNP两侧紧邻碱基的组合如何影响突变偏向的呢?较高的重组率也会增强突变对紧邻碱基的依赖性(见图 3a)。当SNP两侧为A/A、A/T、A/G、T/T、C/T和C/G时,SNP具有较强的突变偏好(见图 3b)。关于重组与突变偏好性之间的具体关系,我们发现当SNP两侧碱基为A/G、C/G或C/T时,随着重组率的增加突变偏好性增强。相反,当SNP两侧碱基为A/A或T/T时,重组率对SNP突变偏好性产生抑制作用。
|
图 3 依赖于两侧碱基的条件互信息和部分条件互信息 Figure 3 Two-flanking-bases-dependent conditional mutual information and partial conditional mutual information 注:图c-g中的数据表示两侧碱基确定的条件下SNP位点上发生特定突变的条件概率,即图 1公式中的p(x, y|z)。Low, Mid和High分别表示低、中、高三组重组率区. |
对两侧碱基确定的条件下SNP位点上发生特定突变的条件概率进行聚类和分析(见图 3c-3g)。发现A碱基始终倾向于突变成碱基G,T碱基倾向突变成C碱基,G碱基突变成A碱基的偏向性最强,C碱基倾向突变成T碱基。当两侧碱基为AT/TT/CT时,SNP位点上A碱基倾向于突变成G碱基(见图 3d);当两侧为AA/AT/AG时,突变更倾向于T突变成C碱基,但这种偏好性随着重组率的增加而减弱(见图 3e);当两侧为AG/CG时,C碱基显示出强烈的C>T突变偏好,该突变偏好性与重组率正相关(见图 3g)。这与文中的总体互信息基调一致,说明我们定义的互信息和条件互信息均能够评估突变对紧邻碱基的依赖程度。综上所述,我们的结果表明,人类基因组中SNP位点的突变偏好与重组和相邻核苷酸类型有关。
2.3 基因组各区域内上游碱基对突变偏好性的影响在基因组不同功能区域内,变异对相邻碱基偏好性与重组率之间的相关性是否有差异?我们用条件互信息计算了不同基因组区域的突变对紧邻碱基的依赖性,并分析了其与重组率之间的相关性。
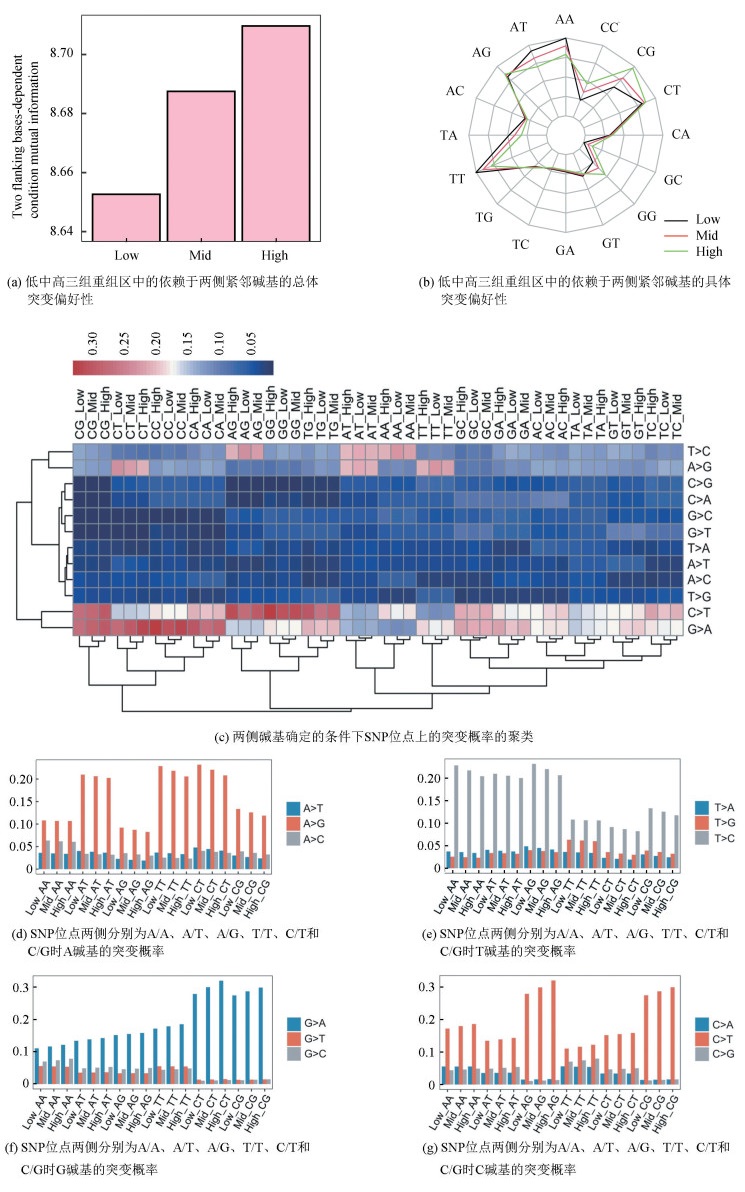
结果表明:在基因间区和外显子区域的条件互信息随重组率增加而增大,而在内含子区域的条件互信息与重组率呈负相关(见图 4a)。外显子和上下游区域内SNP突变对上游紧邻碱基的依赖性由强到弱的顺序为C>A>T>G,在内含子和基因间区内突变对上游紧邻碱基的依赖性由强到弱的顺序为A>C>T>G。上游紧邻碱基为A、C时,SNP位点的突变偏好性最强。当上游碱基为A时,随着重组率增加不同区域内SNP突变偏好性减弱。当上游碱基为C时,不同区域SNP的突变偏好性随着重组率增加而增强。
|
图 4 基因组不同功能区域内依赖于上游碱基的条件互信息和部分条件互信息 Figure 4 Upstream base-dependent conditional mutual information and partial conditional mutual information in different functional regions of the genome 注:图c-g中的数据表示特定上游碱基条件下SNP位点上发生特定突变的条件概率. |
另外,我们分析了SNP具体的突变偏好性(见图 4b)。在12种突变类型中,A碱基依然偏好突变成G碱基,T碱基偏好突变成C碱基,重组率越高其抑制作用增强。C碱基倾向于突变成T碱基,G碱基倾向突变成A碱基,二者与重组率呈正相关。因此我们重点分析了这四种SNP突变偏好性受重组率影响后的变化(见图 4c-4g)。在内含子与基因间区中,上游碱基为A的情况下T>C和C>T的突变偏好性较强,在不考虑重组率的情况下二者对上游紧邻碱基的依赖程度相似(见图 4d,4g);上游碱基为G时,G>A、C>T有强烈的突变偏好性;G>A表现出对上游碱基C的依赖性。在外显子中发现,当紧邻碱基为A和T时,表现出与内含子、基因间区不同的偏好强度。上游碱基为A时,C>T的突变偏好比内含子和基因区间都大;上游碱基为T时,G>A、C>T的突变偏好也是强于内含子和基因间区(见图 4c)。
2.4 基因组各区域内的突变对两侧碱基的依赖性与重组率的关系重组率越高,外显子与基因间区SNP的突变偏好性越强;而内含子区域SNP的突变偏好受到高重组率的抑制(见图 5a)。从部分条件互信息来看,不同区域内SNP突变对两侧碱基的依赖程度不同。外显子区域、以及基因上下游区域具有最强突变偏好性的SNP位点的两侧碱基为CG、CT、AG,而内含子、基因间区具有最强偏好性的SNP两侧碱基为TT、AA(见表 1)。由于选择压力,在编码区内发生的变异大多数发生在密码子的第三位上[80, 81],大多导致同义突变。在Zhao等[17]水稻基因组的研究中发现编码区内邻近碱基为C/G时密码子第三位点发生突变的频率较高,这与我们的结果一致。
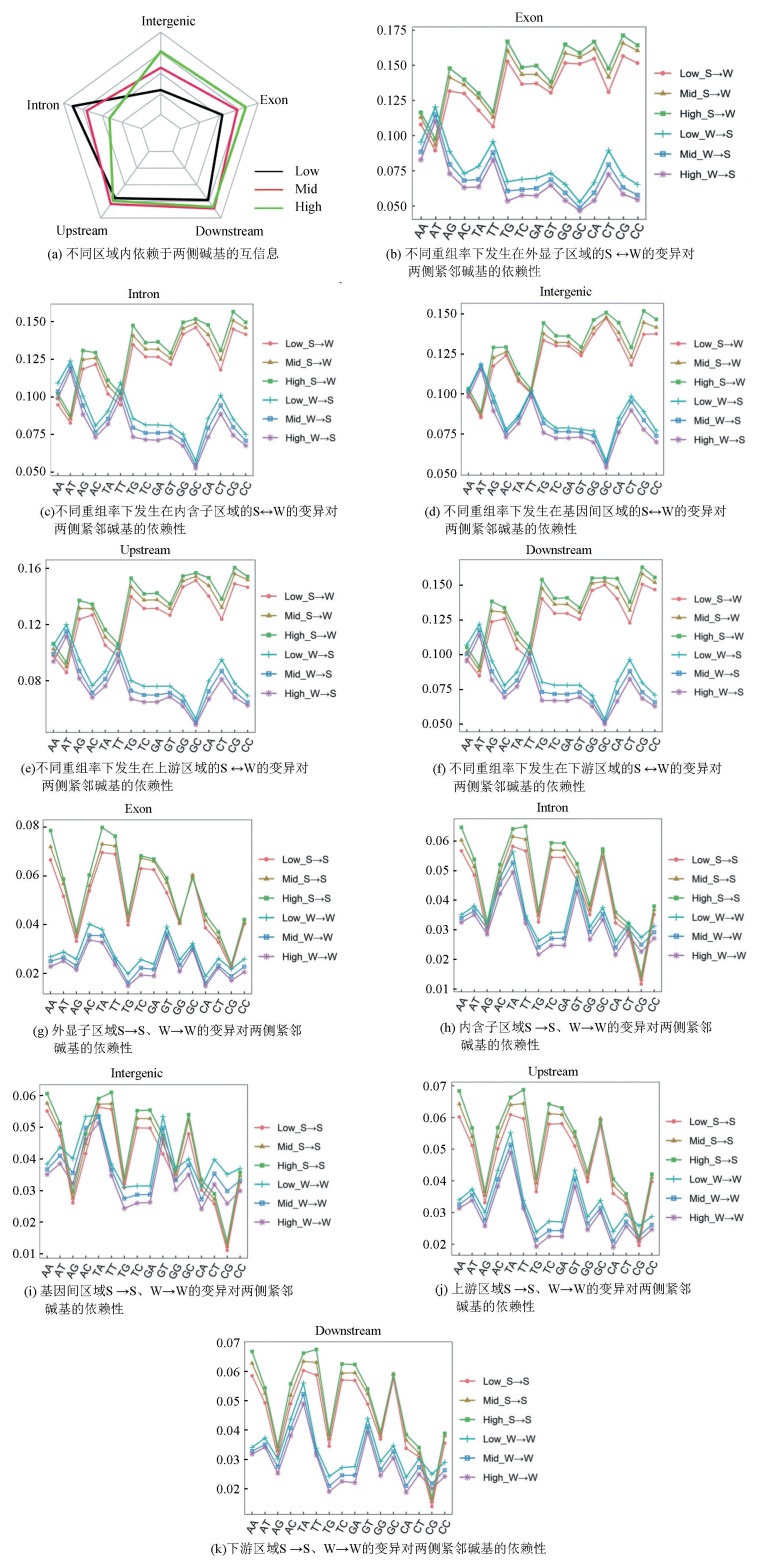
|
图 5 基因组不同功能区域内依赖于两侧碱基的条件互信息 Figure 5 Two-flanking-bases-dependent conditional mutual information in different functional regions of the genome 注:图b-k中的数据表示两侧碱基确定的条件下SNP位点上发生特定类型突变的条件概率. |
| 表 1 不同区域内突变对两侧碱基的依赖程度 Table 1 Dependence of mutations on the two flanking bases in different genomic regions |
对于SNP具体的突变偏好,我们将12种突变类型分成了W>S、S>W、S>S、W>W四种(见图 5b-5k),这里S代表G或C, W代表A或T。发现S>W对两侧碱基的依赖性随重组率升高而增强。W>S对紧邻碱基的依赖性受到高重组率的抑制(见图 5b-5f)。整体S>W类型的突变偏好性强于W>S类型,只有两侧为A/T碱基时,W>S的突变偏好性明显高于S>W。另外当两侧碱基为A/A时,除外显子区域S>W突变偏好性高于W>S外,其余区域二者偏好性相当。当两侧碱基为T/T时,只有内含子区域的W>S突变偏好性强于S>W(见图 5c)。我们还发现S>S对紧邻碱基依赖性随着重组率的增加而增强。整体的S>S类型的突变偏好性强于W>W(见图 5g-5k)。两侧碱基为C/G时,外显子区S>S突变偏好性强于W>W,其余区域恰好相反(见图 5g)。当两侧碱基为A/G、A/C、G/T、C/T时,只有基因间区的W>W突变偏好性强于S>S(见图 5i)。
在基因组不同的功能区域,突变对紧邻碱基的依赖性存在差异,而且重组率对该依赖性调控方式不尽相同。在外显子区域、以及基因上下游区域具有最强突变偏好性的SNP两侧碱基为C/G、C/T、A/G。在内含子、基因间区具有最强突变偏好性的SNP两侧碱基为T/T、A/A。在基因间区和外显子区域,SNP突变对紧邻碱基的依赖性与重组率呈正相关,而在内含子区域随着重组率的升高,其紧邻碱基依赖的突变偏好性受到抑制。另外,计算基因组不同区域时,样本大小在维持同一水平的情况下,SNP突变对紧邻碱基的依赖性与重组率的相关性结果与之前一致(见图 5、图 6、图 7、表 2),这意味着我们的计算不受样本大小不平衡的影响。
|
图 6 不同区域内依赖于上游碱基的条件互信息和部分条件互信息(相同样本大小) Figure 6 Upstream base-dependent conditional mutual information and partial conditional mutual information in different functional regions of the genome (Same sample size) 注:由于外显子区域样本最小(大约40万个SNP位点),因此我们以外显子区域的样本大小作为标准,各区域统一各取40W数据,使不同区域样本大小维持同一水平。在基因间区和外显子区域的条件互信息随重组率增大而增大,而在内含子区域的条件互信息与重组率呈负相关(见图6a)。部分条件互信息的结果显示外显子和基因上下游区域内SNP突变对上游紧邻碱基的依赖性由强到弱的顺序为C>A>T>G,在内含子和基因间区内突变对上游紧邻碱基的依赖性由强到弱的顺序为A>C>T>G(见图6b). |

|
图 7 不同功能区域内依赖于两侧碱基的条件互信息(相同样本大小) Figure 7 Two-flanking-bases-dependent conditional mutual information in different functional regions of thegenome (same sample size) 注:突变对两侧碱基的依赖性与上游碱基的依赖性一致。重组率越高,外显子与基因间区SNP的突变偏好性越强;而内含子区域SNP的突变偏好受到高重组率的抑制. |
| 表 2 不同区域内突变对两侧碱基的依赖程度(相同样本大小) Table 2 Dependence of mutations on the two flanking bases in different genomic regions (Same sample size) |
在人类基因组计划完成的早期,Zhao等[15, 17-18]利用基因组序列数据对“邻近碱基对突变的影响”进行了广泛的研究。但在评估紧邻碱基对突变的影响时,单纯考虑突变位点两侧的碱基含量是不够的,因为这很难捕获到两侧碱基与具体突变之间的内在关联性,而且两侧碱基的含量在很大程度上受到基因组整体碱基含量的影响。即使是通过用全基因组平均水平的碱基组分对突变两侧的碱基含量进行标准化[15],也不能做到“捕获两侧碱基与具体突变之间的内在关联性”的目的。“对突变类型一一单独分析”是一种富含信息量的分析手段,但这种方法在比较不同的物种、不同功能区域时又缺乏一种合理的统一指标来评估紧邻碱基对突变偏好性的“整体影响程度”。本文中定义的条件互信息能够定量评估紧邻碱基对突变偏好性的整体影响,也能够通过部分互信息等指标细化具体的紧邻碱基对突变偏好性的影响。而且,条件互信息的计算中已经考虑了“SNP两侧碱基组分”的影响,无需再进行标准化。期待在后续工作中将该指标应用于不同基因组之间的比较,探索“考虑紧邻碱基效应的突变谱”及其在系统发生领域中的潜在应用价值。
3 结论1) 突变对紧邻碱基的依赖性在高重组区得到加强。上游碱基对突变偏好性的影响顺序为A>C>T>G。上游碱基A或T对SNP突变偏好性的影响随着重组率增加减弱,而上游碱基C或G的影响随着重组率增加而增强。
2) 上下游紧邻碱基对突变偏好性的影响顺序为TT>AA>CT>AG>AT>CG>TA>CC>GG>CA>TG>GT>AC>TC>GA>GC,其中较为明显的是SNP两侧为AG、CG或CT时突变偏好性受重组率的正调控,而两侧为AT、AA、TT时受到负调控。
| [1] |
KEENEY S, LANGE J, MOHIBULLAH N. Self-Organization of meiotic recombination initiation: general principles and molecular pathways[J]. Annual Review of Genetics, 2013, 48(1): 187-214. DOI:10.1146/annurev-genet-120213-092304 (  0) 0) |
| [2] |
BROWN M S, BISHOP D K. DNA Strand exchange and RecA homologs in meiosis[J]. Cold Spring Harbor Perspectives in Biology, 2015, 7(1): a016659. DOI:10.1101/cshperspect.a016659 (  0) 0) |
| [3] |
HUNTER N. Meiotic Recombination: The essence of heredity[J]. Cold Spring Harbor Perspectives in Biology, 2015, 7(12): a016618. DOI:10.1101/cshperspect.a016618 (  0) 0) |
| [4] |
GRELON M. Meiotic recombination mechanisms[J]. Comptes Rendus Biologies, 2016, 339(8): 247-251. DOI:10.1016/j.crvi.2016.04.003 (  0) 0) |
| [5] |
NEALE M, KEENEY S. Clarifying the mechanics of DNA strand exchange in meiotic recombination[J]. Nature, 2006, 442(7099): 153-158. DOI:10.1038/nature04885 (  0) 0) |
| [6] |
WANG K J, CHEN P L, ZHU Q, et al. Recent advances in genes involved in DNA damage repair and meiosis undergoing premature ovarian insufficiency[J]. Journal of International Reproductive Health/Family Planning, 2021, 40(3): 221-225. DOI:10.12280/gjszjk.20200624 (  0) 0) |
| [7] |
CHEN Z J, TIAN Q J, QIAO J, et al. Clinical diagnosis and treatment of premature ovarian insufficiency by chinese expert consensus[J]. Chinese Journal of Obstetrics and Gynecology, 2017, 52(9): 577-581. DOI:10.3760/cma.j.issn.0529-567x.2017.09.001 (  0) 0) |
| [8] |
GALTIER N, PIGANEAU G, MOUCHIROUD D, et al. GC-content evolution in mammalian genomes: The biased gene conversion hypothesis[J]. Genetics, 2001, 159(2): 907-912. DOI:10.1002/gene.1080 (  0) 0) |
| [9] |
DURET L, ARNDT P F. The impact of recombination on nucleotide substitutions in the human genome[J]. PLoS Genetics, 2008, 4(5): e1000071. DOI:10.1371/journal.pgen.1000071 (  0) 0) |
| [10] |
WEBER C C, BOUSSAU B, ROMIGUIER J, et al. Evidence for GC-biased gene conversion as a driver of between-lineage differences in avian base composition[J]. Genome Biology, 2014, 15(12): 549-565. DOI:10.1186/s13059-014-0549-1 (  0) 0) |
| [11] |
HERNANDEZ R D, WILLIAMSON S H, BUSTAMANTE C D. Context dependence, ancestral misidentification, and spurious signatures of natural selection[J]. Molecular Biology & Evolution, 2007, 24(8): 1792-1800. DOI:10.1093/molbev/msm108 (  0) 0) |
| [12] |
HWANG D G, GREEN P. Bayesian Markov chain Monte Carlo sequence analysis reveals varying neutral substitution patterns in mammalian evolution[J]. Proceedings of the National Academy of Sciences of the United States of America, 2004, 101(39): 13994-14001. DOI:10.1073/pnas.0404142101 (  0) 0) |
| [13] |
HERNANDEZ R D, WILLIAMSON S H, ZHU L, et al. Context-dependent mutation rates may cause spurious signatures of a fixation bias favoring higher GC-content in humans[J]. Molecular Biology and Evolution, 2007, 24(10): 2196-2202. DOI:10.1093/molbev/msm149.Epub2007Jul26 (  0) 0) |
| [14] |
ARNDT P F, BURGE C B, HWA T. DNA sequence evolution with neighbor-dependent mutation[J]. Journal of Computational Biology, 2003, 10(4): 313-322. DOI:10.1089/10665270360688039 (  0) 0) |
| [15] |
ZHAO Z M, ERIC B. Neighboring-nucleotide effects on single nucleotide polymorphisms: a study of 2.6 million polymorphisms across the human genome[J]. Genome Research, 2002, 12(11): 1679-1685. DOI:10.1101/gr.287302 (  0) 0) |
| [16] |
SAHAKYAN A B, BALASUBRAMANIAN S. Single genome retrieval of context-dependent variability in mutation rates for human germline[J]. BMC Genomics, 2017, 18(1): 81-98. DOI:10.1186/s12864-016-3440-5 (  0) 0) |
| [17] |
ZHAO Hui, LI Qizhai, ZENG Changqing, et al. Neighboring-nucleotide effects on the mutation patterns of the rice genome[J]. Genomics Proteomics Bioinformatics, 2005, 3(3): 158-168. DOI:10.1016/S1672-0229(05)03021-4 (  0) 0) |
| [18] |
ZHANG Fengkai, ZHAO Zhongming. The influence of neighboring-nucleotide composition on single nucleotide polymorphisms (SNPs) in the mouse genome and its comparison with human SNPs[J]. Genomics, 2004, 84(5): 785-795. DOI:10.1016/j.ygeno.2004.06.015 (  0) 0) |
| [19] |
LIU Guoqing, LI Hong. The correlation between recombination rate and dinucleotide bias in Drosophila melanogaster[J]. Journal of Molecular Evolution, 2008, 67(4): 358-367. DOI:10.1007/s00239-008-9150-0 (  0) 0) |
| [20] |
LIU Guoqing, LI Hong. Effect of meiotic recombination on the dinucleotide bias in human genome[J]. Chinese Science Bulletin, 2009, 54(4): 448-456. DOI:10.1360/csb2009-54-4-448 (  0) 0) |
| [21] |
BHERER C, CAMPBELL C L, AUTON A. Refined genetic maps reveal sexual dimorphism in human meiotic recombination at multiple scales[J]. Nature Communications, 2017, 8: 14994-15003. DOI:10.1038/ncomms14994 (  0) 0) |
| [22] |
CINGOLANI P, PLATTS A, WANG L L, et al. A program for annotating and predicting the effects of single nucleotide polymorphisms, SnpEff: SNPs in the genome of Drosophila melanogaster strain w1118; iso-2; iso-3[J]. Fly (Austin), 2012, 6(2): 80-92. DOI:10.4161/fly.19695 (  0) 0) |
| [23] |
冯桂, 林其伟, 陈东华. 信息论与编码技术[M]. 北京: 清华大学出版社, 2007. FENG Gui, LIN Qiwei, CHEN Donghua. Information theory and coding technology[M]. Beijing: Qinghua University Press, 2007. (  0) 0) |
| [24] |
HORNAKOVA A, LIST M, VREEKEN J, et al. JAMI: Fast computation of conditional mutual information for ceRNA network analysis[J]. Bioinformatics, 2018, 34(17): 3050-3051. DOI:10.1093/bioinformatics/bty221 (  0) 0) |
| [25] |
YU S N, LEE M Y. Conditional mutual information-based feature selection for congestive heart failure recognition using heart rate variability[J]. Comput Methods Programs Biomed, 2012, 108(1): 299-309. DOI:10.1016/j.cmpb.2011.12.015 (  0) 0) |
| [26] |
GAO Wei, TIAN Zheng. Graphical models for multivariate time series based on conditional mutual information[J]. Control Theory & Applications, 2008, 25(2): 257-262. DOI:10.3901/JME.2008.06.095 (  0) 0) |
| [27] |
MAO Y C, CAO H, PING P, et al. Feature selection based on maximum conditional and joint mutual information[J]. Journal of Computer Applications, 2019, 39(3): 734-741. DOI:10.11772/j.issn.1001-9081.2018081694 (  0) 0) |
| [28] |
QUINLAN A R, HALL I M. BEDTools: A flexible suite of utilities for comparing genomic features[J]. Bioinformatics, 2010, 26(6): 841-842. DOI:10.1093/bioinformatics/btq033 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


