2. 解放军医学院,北京 100853;
3. 中国人民解放军总医院 第二医学中心血液科,国家老年疾病临床医学研究中心,北京 100853;
4. 山西医科大学 基础医学院,山西 晋中 030606
2. Chinese PLA Medical School, Beijing 100853, China;
3. National Clinical Research Center for Geriatric Diseases, Department of Hematology of the Second Medical Center, Chinese PLA General Hospital, Beijing 100853, China;
4. Basic Medical College, Shanxi Medical University, Jinzhong 030606, Shanxi, China
AML是一种高度异质性的血液系统恶性疾病,其特征是造血干/祖细胞的恶性克隆扩增和分化停滞。其发病率随着年龄的增长而增加,尤其是65岁以上患者死亡率高达90%[1]。由于复杂的肿瘤微环境以及不断演变的恶性增殖,AML患者预后差异明显,耐药性和疾病复发是预后不佳的主要原因,潜在机制可能与遗传、表观遗传和蛋白质组学的改变导致恶性克隆存在的抗死亡作用有关[2],但确切的机制仍不明确。铁在多种生物过程中是必不可少的,包括线粒体呼吸、免疫监视、细胞增殖和代谢活动等,然而,体内大量铁的积累会导致铁依赖性程序性细胞死亡,即铁死亡,其特征是细胞内活性氧的积累及脂质过氧化反应[3],这是另外一种类型的程序性细胞死亡。越来越多的证据表明铁死亡参与各种疾病的病理过程,但其在AML中的调节机制尚未阐明。因此,本研究旨在通过深入和全面的生物信息学分析确定AML中与铁死亡相关的潜在的生物标志物,并探讨其潜在作用,有助于判断AML患者的预后,为优化治疗策略提供指导。
1 材料和方法 1.1 数据来源与预处理UCSC Xena数据库中包含TCGA、GTEx、TARGET三个数据库合并后批次矫正处理的转录组表达谱数据[4],其中表达量的数据类型为原始count,数据库对其进行了log2(count+1)正态化处理(https://toil-xena-hub.s3.us-east-1.amazonaws.com/download/TcgaTargetGtex_gene_expected_count.gz),纳入TCGA数据库中173例AML患者mRNA数据作为实验组[5],纳入GTEx数据库337例正常人外周血mRNA数据作为对照组[6],排除12例AML中随访时间为0天的患者,排除10例随访状态不明的10例患者,得到151例AML患者。患者平均随访时间为1.66年,最长随访时间为8年。使用R语言(4.1.2版本)clusterProfiler包[7]和org.Hs.eg.db包[8]将ENSEMBL ID转换为GENE SYMBOL。对表达量进行反对数转换,使用floor函数对基因表达量进行取整,使用na.omit函数去除NA行。合并肿瘤样本和正常样本的表达量矩阵,利用既往研究人员实验证实的60个参与调控铁死亡的基因组成新的表达谱矩阵[9-12],将结果用于后续分析。
1.2 筛选AML铁死亡相关预后DEGs利用AML患者的生存时间和生存状态数据,对AML测序数据中的60个铁死亡相关基因进行单因素COX回归分析,以P < 0.05为阈值,筛选出AML铁死亡相关预后基因。同时,使用R语言中wilcox.test算法对AML患者组与健康对照组中的60个铁死亡相关基因进行差异基因表达分析,以|logFC|>0.5,FDR < 0.05为阈值,筛选出AML中铁死亡相关DEGs。最后,将AML中铁死亡相关的DEGs与预后基因取交集,得到AML铁死亡相关预后DEGs,作为风险基因。
1.3 构建风险基因的蛋白互作网络将风险基因上传至STRING数据库在线分析平台[13],进行蛋白互作(Protein-Protein Interaction,PPI)网络分析,以置信度>0.7作为阈值筛选出强关联性的蛋白互作关系。
1.4 构建基于基因标志物的预后模型提取AML风险基因的表达谱,结合LASSO回归算法,使用R包glmnet包来建立预后模型。为了最小化过拟合的风险,采用交叉验证拟合方法的同时,最小化变量个数,选择MSE最小的λ值构建基因标志物预后模型,其中,基因标志物指在AML患者与正常人之间差异表达的,能够帮助预测疾病预后风险的特定基因。基于基因标志物对AML患者风险进行分类,可以作为判断患者风险状态的预后生物标志物。根据每个基因的标准化表达水平及其相应的回归系数计算患者的风险评分。公式建立如下:riskScore=Sum(预后模型中基因相关系数×每个基因的表达量)。
将AML患者高风险组患者和低风险组患者,对模型中涉及的特征基因进行了进一步分析:(1)使用R语言stats包中prcomp函数执行PCA分析,以分析探索不同分组中样本的分布情况。(2)根据中位风险评分将AML患者分为高风险组和低风险组。然后用Kaplan-Meier法进行生存分析。(3)用SurvivalROC软件包进行ROC分析,计算1年、2年和3年生存率的AUC值。(4)使用AML患者的生存时间和生存状态数据,依次进行单因素和多因素Cox回归分析。
1.5 BeatAML数据库验证基因标志物的预后影响纳入beatAML(http://www.vizome.org/aml/)数据库中420例原发性AML患者的转录组数据,并进一步分析基因标志物对AML患者预后的影响,以评估模型的可靠性。
2 结果本研究的的分析按图 1进行,最终纳入了TCGA数据库中的151例AML患者和GTEx数据库中的337例正常样本。AML患者详细临床特征见表 1。
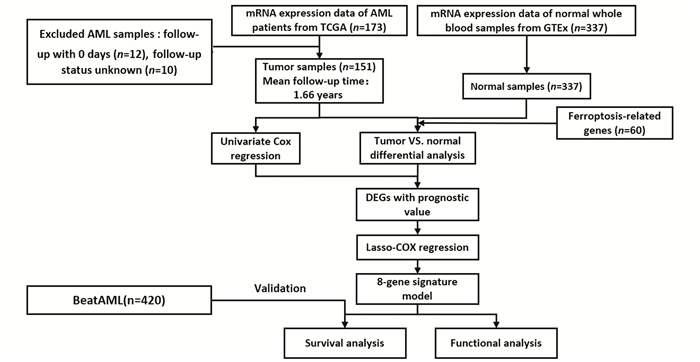
|
图 1 数据收集和分析流程图 Figure 1 Flow chart of data collection and analysis |
| 表 1 AML患者的临床特征 Table 1 Clinical features of AML patients |
对AML测序结果中的60个铁死亡基因进行单因素COX回归分析,以P < 0.05为阈值,筛选出与AML相关铁死亡相关预后基因17个(见图 2a)。在差异基因表达分析中,使用Wilcoxon方法,以|logFC|>0.5,FDR < 0.05为阈值,筛选出AML患者样本与正常人外周血样本之间的AML中铁死亡相关DEGs 51个,其中上调37个,下调14个(见表 2)。将AML中铁死亡相关的17个预后基因与51个DEGs取交集,得到13个AML铁死亡相关预后DEGs(风险基因)及基因热图(见图 2b)。
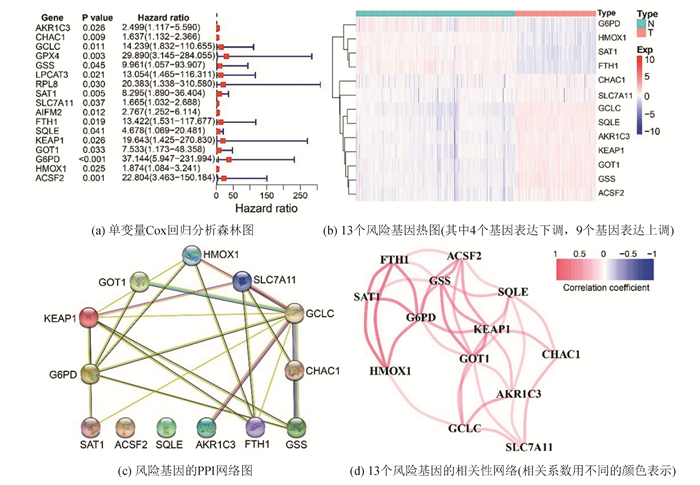
|
图 2 AML铁死亡相关基因的预后分析及关系网络构建 Figure 2 Prognostic analysis and interaction network construction of ferroptosis genes in AML |
| 表 2 前10个差异表达的基因列表(FDR值排序) Table 2 List of top ten differentially expressed genes (sorting by FDR) |
将预后相关DEGs上传至STRING在线分析平台,进行蛋白互作网络分析,共得到13个蛋白,21条相互作用关系,平均点度为3.23,这些基因之间的相互作用网络表明,GCLC、HMOX1、KEAP1、G6PD是中枢基因(见图 2c),这些基因的相关性网络(见图 2d)。
2.3 构建基于基因标志物的预后模型通过Lasso回归算法,构建了8个基因标志物的预后模型(见图 3a,3b),对其生存风险进行评估,以综合探讨这8个基因与AML预后的关系。根据样本中这8个基因的表达水平,构建了风险分值公式:riskScore=sum(预后模型中基因相关系数×每个基因的表达量),每个基因的相关系数(见表 3)。然后,用风险评分公式对AML患者的存活率进行分类和评估。在这里,风险得分>19.285的样本被定义为高风险患者,而风险得分 < 19.285的样本被认为是低风险患者(见图 4a)。为了进行进一步验证,使用PCA分析高、低风险组样本的分布情况,发现二者分布有明显区别,提示通过预后模型对AML患者风险进行分类,能够进行显著区分2组患者(见图 4b);展示AML患者的总体生存(Overall Survival,OS)状态、OS时间和风险评分分布情况,发现高风险患者生存期比低风险患者更容易死亡(见图 4c);使用Kaplan-Meier(KM)方法进行生存分析发现,低风险组患者生存率显著高于高风险组患者(见图 4d);通过ROC曲线评估OS风险评分的预测性能,ROC曲线下面积在1年时达到0.749,2年时达到0.776,3年时达到0.771,证明模型预测性能良好(见图 4e)。
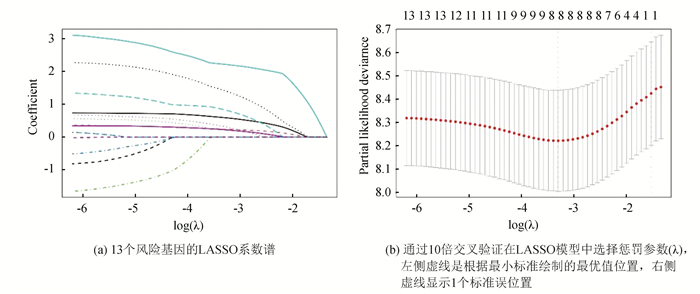
|
图 3 TCGA队列中8个基因标志物预后模型的构建 Figure 3 Construction of a prognostic model for eight gene signatures in TCGA cohort |
| 表 3 预后模型中基因的关联系数 Table 3 Correlation coefficients of genes in prognostic model |
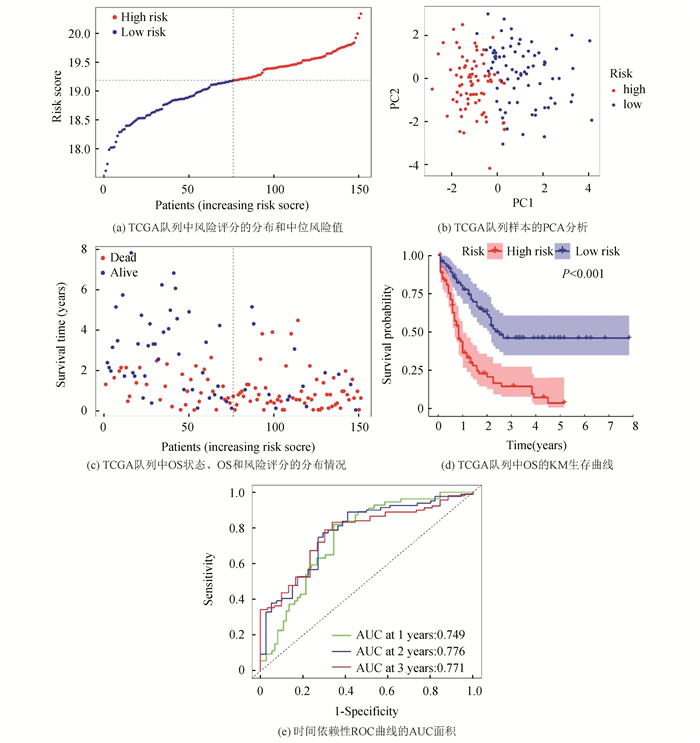
|
图 4 预后模型评估 Figure 4 Evaluation of prognostic model |
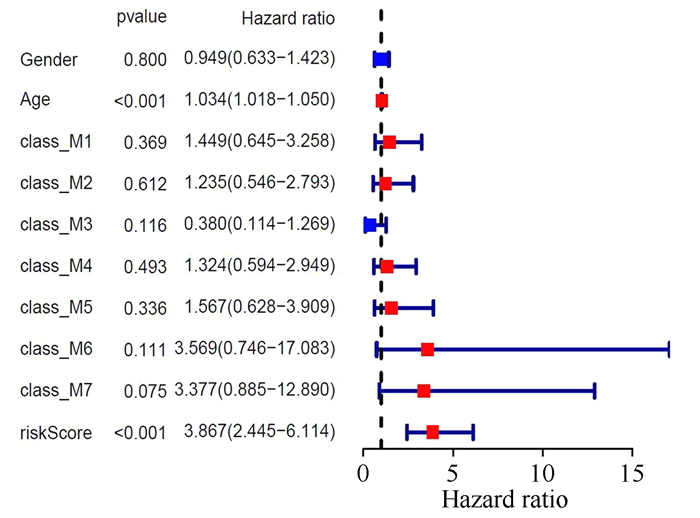
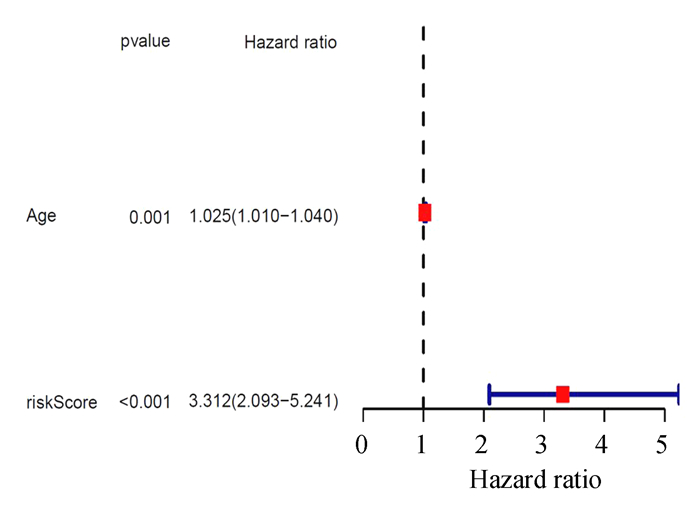
对临床数据和风险评分(RiskScore)进行单因素和多因素Cox回归分析,以确定风险评分是否是OS的独立预后指标。单变量Cox回归分析发现,年龄和风险评分均与OS显著相关(见图 5)。校正P>0.05的混杂因素后,在多因素Cox回归分析中,风险评分仍被证明是OS的独立预后因素(见图 6),证明了风险评分对于预后评估有一定的参考价值。
|
图 5 TCGA队列中OS的单因素Cox分析结果 Figure 5 Results of univariate Cox analyses regarding OS in TCGA cohort |
|
图 6 TCGA队列中OS的多因素Cox回归分析结果 Figure 6 Results of multivariate Cox regression analyses regarding OS in TCGA cohort |
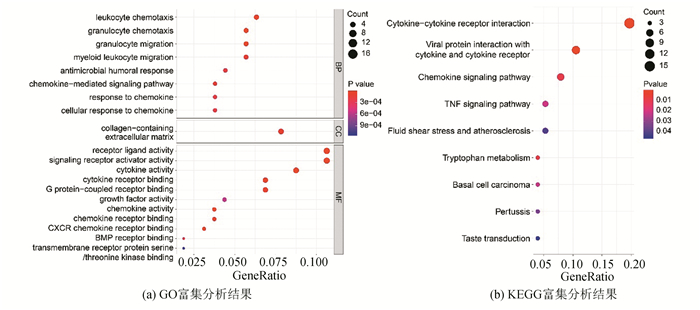
筛选AML患者高风险组患者和低风险组患者间的DEGs,以FDR < 0.05为阈值筛选出预后风险相关DEGs 384个,其中上调345个,下调基因39个,分别进行GO功能富集分析和KEGG通路富集分析。GO功能富集分析共得到20条结果,其中包括生物学过程(Biological Process,BP)8条,细胞组分(Cellular Components,CC)1条,分子功能(Molecular Function,MF)11条。在生物学过程方面,富集的基因大多与中性粒细胞和白细胞的趋化与迁移、趋化因子介导信号通路、体液免疫等相关,如CXCL1、CXCL6、CXCL9、CXCL10、CCL22、IL34等。在细胞组分方面,富集的基因主要集中于细胞外基质,如SDC2、SRPX2、HMCN1、HTRA1、P3H2、C1QA、IL7、GDF15、PF4、THSD4、KAZALD1、EFNA5。在分子功能方面,富集的基因大多参与细胞因子和趋化因子活性、G蛋白偶联受体结合、BMP受体结合等过程(见图 7a)。KEGG分析共富集通路9条,富集的通路主要与色氨酸代谢、趋化因子信号通路、细胞因子-细胞因子受体相互作用有关(见图 7b)。
|
图 7 AML铁死亡相关风险DEGs富集分析结果 Figure 7 Results of enrichment analysis of DEGs for ferroptosis-related risk in AML 注:(a)中横坐标表示每个GO中富集基因的比例,纵坐标表示GO的名称:BP(生物学过程)、CC(细胞组分),MF(分子功能). (b)中横坐标表示各信号通路富集基因的比例,纵坐标表示信号通路名称. |
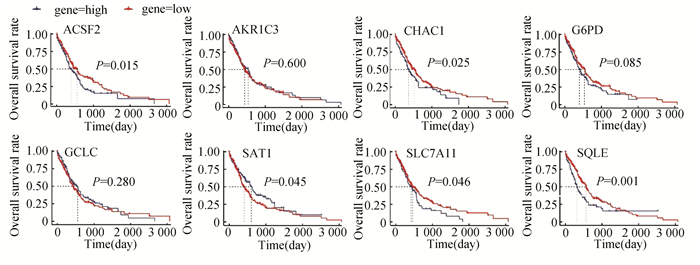
通过对BeatAML数据库中420例AML患者进行生存分析,结果显示,8个基因标志物中,ACSF2、CHAC1、SAT1、SLC7A11、SQLE基因高表达的患者OS显著低于其低表达的患者,验证了8个预后基因的预后模型在其他数据集中的良好性能(见图 8)。
|
图 8 AML患者中高、低表达铁死亡相关基因的KM生存曲线 Figure 8 KM survival curves of AML patients with high and low expression of ferroptosis-related genes 注:横坐标表示生存时间(年); 纵坐标表示患者生存率; 红色曲线代表基因低表达的患者,蓝色曲线代表基因高表达的患者. |
既往研究针对AML预后建立了相关模型,但均是针对临床或个别突变的研究,而对于转录组的研究较少,且效果不佳[14-18]。随着转录组研究的不断深入,Schmitz等人通过转录组分析建立了淋巴瘤预后模型,说明转录组分析可作为评估恶性血液病预后的一种手段[19]。因此,利用AML转录组数据确定预后相关的基因标志物对于优化AML的分层及后期治疗选择至关重要。
过去AML预后模型准确性差可能在于对死亡机制认识不足,铁死亡是新近发现的一种依赖铁信号的程序性死亡形式,其特征在于活性氧(ROS)和脂质过氧化的铁依赖性积累[3]。既往研究表明,铁死亡诱导剂erastin是一种RAS选择性致死化合物,可增强AML细胞(HL-60/NRASQ61L)对化疗药物的敏感性,从而证实诱导铁死亡可能是治疗AML的一种新型策略[20]。然而,目前还没有关于铁死亡相关基因与AML预后之间关系的报道。
本研究通过从TCGA数据库中提取AML转录组测序数据,采用单因素Cox、LASSO回归系统分析8个与AML预后密切相关的基因标志物,构建风险评估模型并进行验证。该模型可将患者分为高风险组与低风险组,模型显示在预测1年,3年和5年生存率方面表现良好,可能对AML患者预后预测及靶向治疗策略具有临床实用性。
本研究中用于构建预后风险评估模型的8个铁死亡相关基因(AKR1C3、CHAC1、GCLC、SAT1、SLC7A11、SQLE、G6PD、ACSF2)大多数都已被证实与癌症发展过程密切相关,其中大多数基因已被报道为通过调节脂质氧化和能量代谢来调节铁死亡进程。醇醛酮还原酶家族1成员C3(Aldo-Keto reductase 1C3,AKR1C3)是一种可溶性单体NADPH依赖性氧化还原酶,在各亚型的AML细胞及高风险T-ALL中过度表达[21]。AKR1C3可通过前列腺素F2α受体和下游丝裂原活化蛋白激酶级联的激活,将前体前列腺素PGD 2转化为11β-PGF2α,从而诱导白血病母细胞增殖[22]。Kshitij等[23]研究发现,AKR1C3抑制剂可控制髓样细胞增殖、分化和凋亡,在AML细胞系中AKR1C3抑制剂和依托泊苷或柔红霉素联合治疗可使后者化疗药物的细胞毒性增强达6至10倍以上,证明对于AML多种亚型可通过抑制AKR1C3表达从而抑制铁死亡进行干预治疗。阳离子转运调控样蛋白1(Glutathione Specific Gamma-Glutamylcyclotransferase 1,CHAC1)是一种位于ATF4-ATF3-CHOP级联反应的下游促凋亡ER应激蛋白,作为内质网应激诱导基因可参与γ-谷氨酰循环,其过表达导致谷胱甘肽的大量消耗从而促进AML细胞导致高水平的氧化应激和铁死亡发生[24]。已有研究表明,CHAC1的过度活化可促进eIF2α磷酸化,抑制AML细胞的耗氧率,从而导致生长停滞和细胞凋亡,表明上调CHAC1表达对依赖氧化磷酸化生存的化疗耐药AML原始细胞具有特殊意义[25]。AML细胞可通过促进谷胱甘肽代谢从而增强细胞的增殖和存活,既往研究表明,ROS积累和GSH依赖性防御系统在AML发展中起关键作用,谷氨酸-半胱氨酸连接酶催化亚基(Glutamate-Cysteine Ligase Catalytic,GCLC)是GSH合成过程中的限速酶,其表达量与AML耐药和肿瘤进展相关,抑制GCLC表达不仅可以诱导AML细胞脂质过氧化及铁死亡发生,还可诱导细胞凋亡,从而抑制AML生长和进展[26-27]。亚精胺/精胺N1-乙酰转移酶1(spermidine/spermine N1-acetyltransferase 1,SAT1)作为肿瘤抑制基因P53的靶点,是多胺分解代谢中的限速酶,Ou等[28]发现,SAT1激活可诱导脂质过氧化并使细胞在活性氧诱导的应激下发生铁死亡,表明了铁死亡可抑制肿瘤生长,但仍需大量研究评估铁死亡在SAT1介导的肿瘤抑制中的作用。溶质载体家族7成员11(solute carrier family 7 member 11,SLC7A11)是铁死亡的关键调节因子,P53可作为SLC7A11的转录抑制因子参与铁死亡过程,抑制半胱氨酸输入,从而促进AML细胞死亡[29]。在铁死亡激活剂Erastin、索拉非尼等药物的作用下可抑制SLC7A11表达,消耗胞内的谷胱甘肽,破坏氧化代谢平衡,细胞内ROS募集出现致死性损伤,最终导致细胞死亡[30]。胆固醇代谢参与铁死亡过程,阻碍胆固醇的合成和摄取可阻断肿瘤增殖。角鲨烯单加氧酶(squalene epoxidase,SQLE)作为胆固醇生物合成中的关键限速酶,可通过p53肿瘤抑制途径失活和β-连环蛋白致癌途径激活,临床前研究揭示SQLE具有促进癌细胞增殖特性,抑制SQLE表达可抑制肺鳞癌、前列腺癌、胰腺癌的生长和侵袭,且SQLE过表达的AML与AML患者接受异基因HSCT的时间较长,与临床分期不良和预后差显著相关[31-33]。AML细胞的增殖依赖戊糖磷酸途径的氧化分支,敲除6-磷酸葡萄糖脱氢酶(Glucose-6-phosphate dehydrogenase,G6PD)可降低AML细胞中NADPH水平,减少GSH的生成,并在一定程度上增加AML细胞对脂质的需求,从而调节铁死亡过程,可在未来作为药物靶点治疗AML[34]。酰基辅酶A合成酶家族成员2(Acyl-CoA synthetase family member 2,ACSF2)作为铁死亡驱动基因,与线粒体脂肪酸代谢调节相关,已有研究证实依赖谷氨酰胺代谢和ACSF2的脂质合成途径可以提供铁死亡所需的特定脂质前体[35],但其在AML中的预后价值有待进一步研究加以证实。
现有研究对肿瘤与铁死亡易感性的相关机制进行了广泛探索,本研究通过GO富集和KEGG通路分析显示,在高风险患者组,基因功能不仅主要与氨基酸代谢、脂质代谢途径及免疫调节相关,还与炎症反应、免疫系统过程的负反应、氧化应激反应存在联系,表明机体代谢异常和体内平衡紊乱可导致AML恶性转化和疾病进展。此外,铁死亡可介导中性粒细胞募集、迁移以及释放细胞核内DNA后裂解死亡,导致免疫反应激活及中性粒细胞减少,导致AML预后差。先前报道表明,早期诱导肿瘤细胞铁死亡可有效增强免疫反应,诱导Treg细胞铁死亡可能是改善AML治疗的一种治疗策略[36]。Wang等[37]发现免疫疗法激活的CD8+T细胞增强了肿瘤细胞中铁死亡特异性的脂质过氧化作用,并且增加的铁死亡有助于免疫疗法的抗肿瘤功效。因此,使用铁死亡诱导剂可能有助于提高AML患者的治疗效果。
本研究通过整合并分析铁死亡相关预后基因标志物在AML中的潜在作用,建立铁死亡相关预后模型并进行验证,表明8个铁死亡相关预后基因对AML患者预后预测及靶向治疗策略具有临床实用性,但本研究建立的预后模型是基于公开的回顾性数据,仍需进行前瞻性研究来验证其预测性能。其次,铁死亡与AML预后间的潜在机制仍缺乏相关细胞实验及临床队列研究佐证,具体机制有待进一步研究。
4 结论本研究发现部分铁死亡相关基因在AML中的表达具有显著差异,基于8个基因标志物构建模型来预测AML患者的预后风险,表现出良好的预测性能,预后风险相关DEGs主要在趋化因子、细胞因子、TNF等炎性和免疫杀伤信号通路中显著富集。但本研究仅限于转录组水平的模型构建,仍需利用多组学数据加以分子生物学实验来证实DEGs和信号通路在AML中的功能。
| [1] |
ALDOSS I, MARCUCCI G. More options for older patients with acute myeloid leukemia: Venetoclax in combination with low dose cytarabine[J]. Chinese Clinical Oncology, 2019, 8(S1): S25-S25. DOI:10.21037/cco.2019.09.03 (  0) 0) |
| [2] |
HU Linjun, GAO Yuling, SHI Zhan, et al. DNA methylation-based prognostic biomarkers of acute myeloid leukemia patients[J]. Annals of Translational Medicine, 2019, 7(23): 737. DOI:10.21037/atm.2019.11.122 (  0) 0) |
| [3] |
DIXON S J, LEMBERG K M, LAMPRECHT M R, et al. Ferroptosis: An iron-dependent form of nonapoptotic cell death[J]. Cell, 2012, 149(5): 1060-1072. DOI:10.1016/j.cell.2012.03.042 (  0) 0) |
| [4] |
GOLDMAN M J, CRAFT B, HASTIE M, et al. Visualizing and interpreting cancer genomics data via the Xena platform[J]. Nature Biotechnology, 2020, 38(6): 675-678. DOI:10.1038/s41587-020-0546-8 (  0) 0) |
| [5] |
TOMCZAK K, CZERWIŃSKA P, WIZNEROWICZ M. The Cancer Genome Atlas (TCGA): An immeasurable source of knowledge[J]. Contemporary Oncology, 2015, 19(1A): A68. DOI:10.5114/wo.2014.47136 (  0) 0) |
| [6] |
GTEx Consortium. The Genotype-Tissue Expression (GTEx) pilot analysis: Multitissue gene regulation in humans[J]. Science, 2015, 348(6235): 648-660. DOI:10.1126/science.1262110 (  0) 0) |
| [7] |
WU Tianzhi, HU Erqiang, XU Shuangbin, et al. ClusterProfiler 4.0: A universal enrichment tool for interpreting omics data[J]. The Innovation, 2021, 2(3): 100141. DOI:10.1016/j.xinn.2021.100141 (  0) 0) |
| [8] |
CARLSON M. Genome wide annotation for Human[DB/OL]. 2023-03-08. DOI: 10.18129/B9.bioc.org.Hs.eg.db.
(  0) 0) |
| [9] |
STOCKWELL B R, ANGELI J P F, BAYIR H, et al. Ferroptosis: A regulated cell death nexus linking metabolism, redox biology, and disease[J]. Cell, 2017, 171(2): 273-285. DOI:10.1016/j.cell.2017.09.021 (  0) 0) |
| [10] |
HASSANNIA B, VANDENABEELE P, BERGHE T V. Targeting ferroptosis to iron out cancer[J]. Cancer Cell, 2019, 35(6): 830-849. DOI:10.1016/j.ccell.2019.04.002 (  0) 0) |
| [11] |
BERSUKER K, HENDRICKS J M, LI Zhipeng, et al. The CoQ oxidoreductase FSP1 acts parallel to GPX4 to inhibit ferroptosis[J]. Nature, 2019, 575(7784): 688-692. DOI:10.1038/s41586-019-1705-2 (  0) 0) |
| [12] |
DOLL S, FREITAS F P, SHAH R, et al. FSP1 is a glutathione-independent ferroptosis suppressor[J]. Nature, 2019, 575(7784): 693-698. DOI:10.1038/s41586-019-1707-0 (  0) 0) |
| [13] |
SZKLARCZYK D, FRANCESCHINI A, WYDER S, et al. STRING v10: Protein-protein interaction networks, integrated over the tree of life[J]. Nucleic Acids Research, 2015, 43(D1): D447-D452. DOI:10.1093/nar/gku1003 (  0) 0) |
| [14] |
JAHN N, TERZER T, STRÄNG E, et al. Genomic heterogeneity in core-binding factor acute myeloid leukemia and its clinical implication[J]. Blood Advances, 2020, 4(24): 6342-6352. DOI:10.1182/bloodadvances.2020002673 (  0) 0) |
| [15] |
李剑雄, 刘恒, 盛宏霞, 等. 基于高通量测序技术的急性髓系白血病患者的基因突变谱和预后分析[J]. 中国实验血液学杂志, 2021, 29(2): 353-362. LI Jianxiong, LIU Heng, SHENG Hongxia, et al. Mutational spectrum and prognosis analysis of aml patients based on high-throughput sequencing[J]. Journal of Experimental Hematology, 2021, 29(2): 353-362. DOI:10.19746/j.cnki.issn.1009-2137.2021.02.008 (  0) 0) |
| [16] |
YUAN Xiaoqing, CHEN Peng, DU Yinxiao, et al. Influence of DNMT3A R882 mutations on AML prognosis determined by the allele ratio in Chinese patients[J]. Journal of Translational Medicine, 2019, 17(1): 1-10. DOI:10.1186/s12967-019-1959-3 (  0) 0) |
| [17] |
CHEN Shuo, WANG Chunmeng, ZHU Ruochen, et al. Predicting prognosis in acute myeloid leukemia patients by surface-enhanced Raman spectroscopy[J]. Nanomedicine, 2021, 16(21): 1873-1885. DOI:10.2217/nnm-2021-0199 (  0) 0) |
| [18] |
SINGH A, LOCKE S C, WOLF S P, et al. The relationship between emotional well-being and understanding of prognosis in patients with acute myeloid leukemia (AML)[J]. Supportive Care in Cancer, 2022, 30(1): 897-906. DOI:10.1007/s00520-021-06499-w (  0) 0) |
| [19] |
SCHMITZ R, WRIGHT G W, HUANG Dawei, et al. Genetics and pathogenesis of diffuse large B-cell lymphoma[J]. New England Journal of Medicine, 2018, 378(15): 1396-1407. DOI:10.1056/NEJMoa1801445 (  0) 0) |
| [20] |
YU Yan, XIE Yangchun, CAO Lizhi, et al. The ferroptosis inducer erastin enhances sensitivity of acute myeloid leukemia cells to chemotherapeutic agents[J]. Molecular & Cellular Oncology, 2015, 2(4): e1054549. DOI:10.1080/23723556.2015.1054549 (  0) 0) |
| [21] |
DESMOND J C, MOUNTFORD J C, DRAYSON M T, et al. The aldo-keto reductase AKR1C3 is a novel suppressor of cell differentiation that provides a plausible target for the non-cyclooxygenase-dependent antineoplastic actions of nonsteroidal anti-inflammatory drugs[J]. Cancer Research, 2003, 63(2): 505-512. (  0) 0) |
| [22] |
SALES K J, MILNE S A, WILLIAMS A R W, et al. Expression, localization, and signaling of prostaglandin F2α receptor in human endometrial adenocarcinoma: Regulation of proliferation by activation of the epidermal growth factor receptor and mitogen-activated protein kinase signaling pathways[J]. The Journal of Clinical Endocrinology & Metabolism, 2004, 89(2): 986-993. DOI:10.1210/jc.2003-031434 (  0) 0) |
| [23] |
VERMA K, ZANG Tianzhu, GUPTA N, et al. Selective AKR1C3 inhibitors potentiate chemotherapeutic activity in multiple acute myeloid leukemia (AML) cell lines[J]. ACS Medicinal Chemistry Letters, 2016, 7(8): 774-779. DOI:10.1021/acsmedchemlett.6b00163 (  0) 0) |
| [24] |
WANG Ning, ZENG Guangzhi, YIN Junlin, et al. Artesunate activates the ATF4-CHOP-CHAC1 pathway and affects ferroptosis in Burkitt's Lymphoma[J]. Biochemical and Biophysical Research Communications, 2019, 519(3): 533-539. DOI:10.1016/j.bbrc.2019.09.023 (  0) 0) |
| [25] |
STEVENS A M, XIANG M, HEPPLER L N, et al. Atovaquone is active against AML by upregulating the integrated stress pathway and suppressing oxidative phosphorylation[J]. Blood Advances, 2019, 3(24): 4215-4227. DOI:10.1182/bloodadvances.2019000499 (  0) 0) |
| [26] |
BANSAL A, SIMON M C. Glutathione metabolism in cancer progression and treatment resistance[J]. Journal of Cell Biology, 2018, 217(7): 2291-2298. DOI:10.1083/jcb.201804161 (  0) 0) |
| [27] |
LIN Chiouhong, VU J P, YANG Chenyen, et al. Glutamate-cysteine ligase catalytic subunit as a therapeutic target in acute myeloid leukemia and solid tumors[J]. American Journal of Cancer Research, 2021, 11(6): 2911. (  0) 0) |
| [28] |
OU Yang, WANG Shangjui, LI D, et al. Activation of SAT1 engages polyamine metabolism with p53-mediated ferroptotic responses[J]. Proceedings of the National Academy of Sciences, 2016, 113(44): E6806-E6812. DOI:10.1073/pnas.1607152113 (  0) 0) |
| [29] |
GALLUZZI L, PEDRO B S, KROEMER G. Ferroptosis in p53-dependent oncosuppression and organismal homeostasis[J]. Cell Death & Differentiation, 2015, 22(8): 1237-1238. DOI:10.1038/cdd.2015.54 (  0) 0) |
| [30] |
DAHLMANNS M, YAKUBOV E, CHEN D, et al. Chemotherapeutic xCT inhibitors sorafenib and erastin unraveled with the synaptic optogenetic function analysis tool[J]. Cell Death Discovery, 2017, 3(1): 1-11. DOI:10.1038/cddiscovery.2017.30 (  0) 0) |
| [31] |
XU Huanji, ZHOU Sheng, TANG Qiulin, et al. Cholesterol metabolism: New functions and therapeutic approaches in cancer[J]. Biochimica et Biophysica Acta (BBA)-Reviews on Cancer, 2020, 1874(1): 188394. DOI:10.1016/j.bbcan.2020.188394 (  0) 0) |
| [32] |
XU Fangshi, ZHANG Zhengliang, ZHAO Yidi, et al. Bioinformatic mining and validation of the effects of ferroptosis regulators on the prognosis and progression of pancreatic adenocarcinoma[J]. Gene, 2021, 795: 145804. DOI:10.1016/J.GENE.2021.145804 (  0) 0) |
| [33] |
MA Yanqing, LIU Yuchen, YIN Chengliang, et al. Value of SQLE expression in prognostic evaluation of patients with acute myeloid leukemia[J]. Journal of Experimental Hematology, 2021, 29(1): 9-16. DOI:10.19746/J.CNKI.ISSN.1009-2137.2021.01.002 (  0) 0) |
| [34] |
XU Shuangnian, WANG Tianshi, LI Xi, et al. SIRT2 activates G6PD to enhance NADPH production and promote leukaemia cell proliferation[J]. Scientific Reports, 2016, 6(1): 1-13. DOI:10.1038/srep32734 (  0) 0) |
| [35] |
WANG Huan, CHENG Qi, CHANG Kaikai, et al. Integrated analysis of ferroptosis-related biomarker signatures to improve the diagnosis and prognosis prediction of ovarian cancer[J]. Frontiers in Cell and Developmental Biology, 2021, 9: 807862. DOI:10.3389/FCELL.2021.807862 (  0) 0) |
| [36] |
EFIMOVA I, CATANZARO E, VAN DER MEEREN L, et al. Vaccination with early ferroptotic cancer cells induces efficient antitumor immunity[J]. Journal for Immunotherapy of Cancer, 2020, 8(2): e001369. DOI:10.1136/jitc-2020-001369 (  0) 0) |
| [37] |
WANG Weimin, GREEN M, CHOI J E, et al. CD8+T cells regulate tumour ferroptosis during cancer immunotherapy[J]. Nature, 2019, 569(7755): 270-274. DOI:10.1038/s41586-019-1170-y (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


