目前,在已知致病基因中约一半的变异为单核苷酸变异(Single-nucleotide Variants, SNVs)[1],这意味着开发高效校正SNVs的方法,具有广泛的科学研究价值和遗传病基因治疗潜力。CRISPR/Cas9系统是目前广泛应用的基因编辑工具之一,但其主要通过切割DNA双链,借助细胞中DNA双链断裂修复机制来编辑基因,难以精确校正单个核苷酸位点的突变。单碱基编辑系统(Base Editor, BE)是在CRISPR/Cas9系统的基础上开发得到的新型碱基编辑工具,主要有胞嘧啶碱基编辑器(C to T)和腺嘌呤碱基编辑器(A to G)两类。单碱基编辑系统能够实现单核苷酸水平的精准突变,其不但具有高效性和特异性,并且在细胞、小鼠模型和植物的基因组中产生插入缺失的比例一般低于1%[2-5]。因此,单碱基编辑系统在研究等位基因位点对基因功能影响,SNV相关遗传病的致病机制和治疗方案等方面具有独特优势。
影响单碱基编辑系统编辑效率的因素包括靶基因的选择、sgRNA序列设计、脱靶效应和单碱基编辑系统向细胞内的递送方式等,sgRNA序列设计是其中的关键因素之一[6]。有研究报道,sgRNA序列活性与Cas9蛋白序列偏好性[7-9],以及脱氨酶的序列偏好性[5, 10-12]有着密切联系。此外,若基因组上存在与设计的sgRNA序列相似度较高的片段,容易发生脱靶效应,在基因组靶位点之外产生编辑。因此,优化sgRNA设计有助于提高编辑效率,降低脱靶情况的发生。
sgRNA设计工具的实现方式主要有三大类:一是基于序列比对的方法,即sgRNA和基因组的比对结果;二是基于概率假设的方法,即利用sgRNA上的一些特征(如GC含量)进行设计;三是基于学习的方法,利用大型数据集训练机器学习和深度学习模型来设计sgRNA。目前针对单碱基编辑系统的sgRNA设计工具主要基于序列比对和sgRNA序列特征,且无法预测编辑效率。
本研究使用Song等人[13]发表的高通量单碱基编辑系统测序数据,利用卷积神经网络(Convolutional Neural Network,CNN)模型,结合Cas-OFFinder脱靶位点预测工具[14],开发了一个sgRNA序列设计工具BEguider。该工具能针对人基因序列进行sgRNA设计、靶向编辑效率预测以及寻找sgRNA在基因组上可能的脱靶位点。
1 数据与方法 1.1 数据本研究中,单碱基编辑器BE4-NGG和ABE7.10-NGG的CNN模型的训练数据来自于Song等人[13]发表的高通量外源递送系统测序数据。数据被划分为训练集,验证集和测试集,比例为8∶1∶1。
内源性位点数据来自于NCBI BioProject数据库的公开项目PRJNA476544。
人类hg38参考基因组序列来自于Ensembl数据库。
1.2 方法 1.2.1 BEguider分析预测流程首先,对输入的基因序列进行预处理,筛选候选sgRNA序列。分别在输入基因上的正链与负链上寻找是否存在NGG PAM序列。定位到NGG PAM序列后,向序列5’方向搜索,寻找编辑活性窗口内(即sgRNA第4到8位碱基)是否存在可编辑的底物核苷酸——A或C。若存在,则提取PAM序列前的20 nt核苷酸序列与4 nt PAM序列作为候选sgRNA序列。
其次,对于候选的sgRNA进行独热编码(One-hot Encoding)。为了使计算机能够识别并处理sgRNA序列,需要对每条序列进行独热编码,碱基A用向量[1, 0, 0, 0]表示,碱基C用向量[0, 1, 0, 0]表示,碱基G用向量[0, 0, 1, 0]表示,碱基T用向量[0, 0, 0, 1]表示。因此,每条长为L nt的sgRNA的序列可以被表示为一个L×4的矩阵Xinput∈RL×4。
最后,对候选sgRNA序列的编辑效率进行预测并输出。CNN模型优势在于能够很好地学习训练样本的局部特征,达到准确的预测效果。神经网络会对输入的每个sgRNA独热编码矩阵进行预测分析,经过两层二维卷积层与两层全连接层处理,输出对应的预测概率(见表 1)。在卷积层与全连接层之间,以及两层全连接层之间各有一层随机失活层,随机失活比例为0.3,防止CNN在较小的数据集上发生过拟合现象。
| 表 1 BEguider中的卷积神经网络结构 Table 1 CNN structure of BEguider |
CNN模型的评价指标有均方误差MSE,Pearson相关系数,Spearman相关系数。
全流程如图 1c所示。
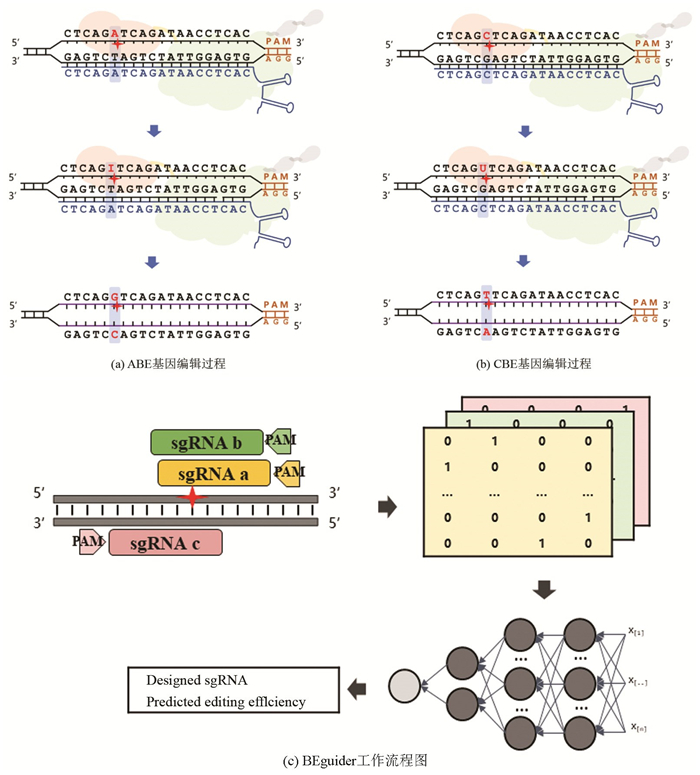
|
图 1 单碱基编辑器编辑示意图与BEguider工作流程图 Figure 1 Editing process of base editors and the workflow of BEguider 注:(a) ABE将腺嘌呤脱氨得到次黄嘌呤,细胞内DNA复制时次黄嘌呤会与胞嘧啶配对,经DNA复制,A: T配对被替换为G: C配对;(b) CBE将胞嘧啶脱氨得到尿嘧啶,细胞内DNA复制时尿嘧啶会与腺嘌呤配对,经DNA复制,C: G配对被替换为T: A配对(浅绿色为Cas9n核酸酶,淡橘色为DNA脱氨酶,浅灰色为尿嘧啶糖基化酶抑制剂,土黄色为DNA上的PAM序列,红色为点突变位点,蓝色为sgRNA序列,紫色为新合成的DNA双链);(c) sgRNA a,sgRNA b和sgRNA c表示针对同一个基因位点可以设计多条sgRNA。 |
BEguider的代码已上传至gitee仓库,可通过git clone命令下载。使用BEguider前,需要预先安装运行依赖的深度学习框架TensorFlow 2与第三方python程序包,建议通过开源软件包管理系统Conda进行安装(requirements_BEguider.txt已在软件中提供)。命令如下:
git clone https://gitee.com/gaojingjing123/beguider
conda create --name<env>--file requirements_BEguider.txt
BEguider支持三种输入形式(见图 2):

|
图 2 BEguider的命令行截图说明 Figure 2 Screenshot for commands of BEguider |
(1) 输入基因名或序列ID与对应的基因序列。
(2) 输入靶位点所在的染色体号与染色体坐标。
(3) 输入靶位点的rsID。
对于ABE7.10-NGG和BE4-NGG,BEguider支持两种sgRNA设计模式(见图 2):
(1) 指定单碱基编辑器模式:输入编辑器名称,指定ABE7.10-NGG或BE4-NGG作为编辑工具。
(2) ALL模式:BEguider给出最佳的单碱基编辑器与sgRNA组合。
BEguider整合Cas-OFFinder脱靶位点搜索工具,提供可选的预测脱靶位点功能。
1.2.3 BEguider在内源性位点的预测效果评估将内源性sgRNA序列输入BEguider,得到预测编辑效率,用Pearson相关系数和Spearman相关系数评估预测编辑效率与实验编辑效率之间的相关性。其中,ABE7.10-NGG的sgRNA序列有94条,BE4-NGG的sgRNA序列有102条。
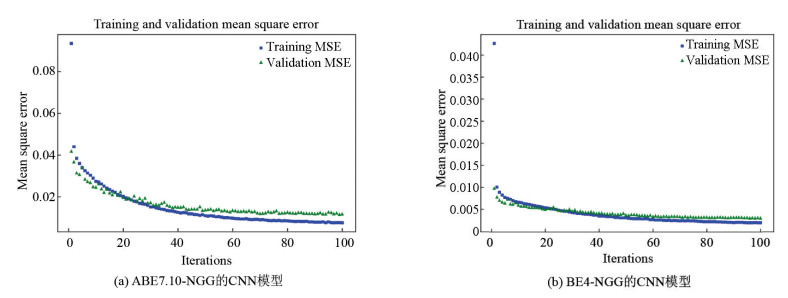
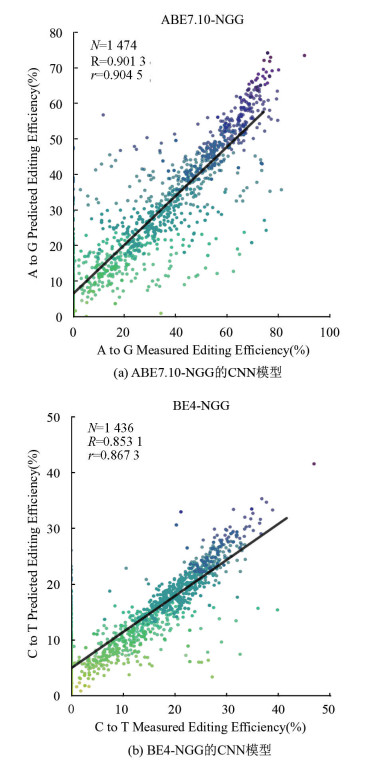
2 结果 2.1 BEguider神经网络模型训练结果BEguider的训练数据来自于外源靶序列,即慢病毒将靶序列整合进宿主细胞基因组,再由单碱基编辑器对靶位点编辑,最后PCR扩增出靶位点片段进行高通量测序获得编辑结果。BEguider中包括两个CNN模型,分别为ABE7.10-NGG和BE4-NGG提供预测功能。图 3为BEguider在训练集与验证集上MSE随训练轮次的变化。在模型训练过程中验证集的数据特征可能会泄露到模型中,因此模型评价需要在模型从未接触的测试集数据中进行。在测试集中ABE7.10-NGG的MES为0.007 8,Spearman相关系数为0.901 3,Pearson相关系数为0.904 5(见图 4a);BE4-NGG的MES为0.001 9,Spearman相关系数为0.853 1,Pearson相关系数为0.867 3(见图 4b)。这说明BEguider能够较好地预测未知sgRNA的编辑效率。
|
图 3 BEguider训练集与验证集上的MSE Figure 3 MSE of BEguider on training set and validation set 注:(a)、(b) 横坐标表示训练轮次,纵坐标表示每个轮次结束计算的MSE |
|
图 4 BEguider在测试集上预测结果与测序结果的相关性 Figure 4 Correlation between predicted efficiencies of BEguider and measured efficiencies on the test set 注:(a)、(b) 横坐标表示测序得到的编辑效率,纵坐标表示BEguider预测的编辑效率。N为纳入评估的sgRNA的数量,R为Spearman相关系数,r为Pearson相关系数. |
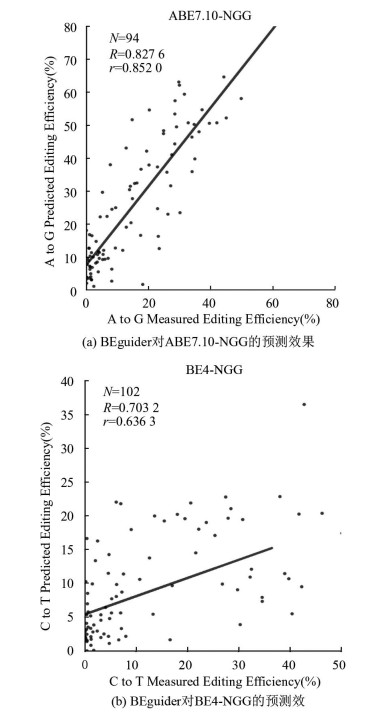
为了扩大BEguider的应用范围,需要评估BEguider对内源基因组位点编辑效率预测的效果。对于ABE7.10-NGG,BEguider的预测编辑效率与实验编辑效率的Spearman相关系数0.827 6,Pearson相关系数为0.852,MSE为0.021(见图 5a)。对于BE4-NGG,BEguider的预测编辑效率与实验编辑效率的Spearman相关系数0.703 2,Pearson相关系数为0.636 3,MSE为0.026 4(见图 5b)。说明BEguider能够准确提取sgRNA序列特征,建立sgRNA序列组成与编辑效率之间的联系,对于内源性位点的编辑效率也有较好的预测效果。
|
图 5 BEguider对内源性位点预测编辑效率的效果评估 Figure 5 Evaluation of the predictive performance of BEguider at endogenous sites 注:N为纳入评估的内源性位点数量,R为Spearman相关系数,r为Pearson相关系数。 |
以输入形式为rsID为例,选取10个SNV位点,将rsID输入BEguider,使用推荐单碱基编辑器与sgRNA组合的模式,默认不开启脱靶位点预测,在linux系统中运行命令如下:
python BEguider.py -s SNP_example.txt -b ALL -o./temp。
BEguider一共设计出12条sgRNA,输出信息包括单碱基编辑器、SNV的相关信息、sgRNA所在的正负链情况、设计的sgRNA序列以及对应的预测编辑效率。结果如表 2所示。
| 表 2 以10个SNV位点为例的sgRNA设计结果 Table 2 Output results of sgRNAs for ten SNVs using BEguider |
将BEguider与5种目前可用的碱基编辑设计工具进行比较(见表 3)。Benchling、BE-Designer和beditor主要功能是在指定的单碱基编辑器与点突变上给出编辑结果,而BEable-GPS、BE-FF能找到合适的碱基编辑器来还原给定点突变的变化。BEguider的优势在于提供了靶序列的多种输入方式,能够根据需要选择编辑野生型基因位点或突变型基因位点,同时批量为多个基因位点设计sgRNA,预测每条sgRNA的编辑效率。
| 表 3 多个sgRNA设计工具的比较 Table 3 Comparison of different sgRNA design tools |
传统的基因编辑工具,如锌指蛋白核酸酶(Zinc Finger Nuclease, ZFN),转录激活样效应因子核酸酶(Transcription Activator-Like Effector Nuclease, TALEN)等,需要通过改造蛋白结构域才能在新的靶位点进行编辑,过程复杂,而单碱基编辑系统仅需要合成新sgRNA序列即可靶向新的基因位点,因此单碱基编辑系统的高效性、精确性、稳定性和易操作性,使其成为目前最为流行的基因编辑工具之一。
单碱基编辑系统提高了在细胞内功能性SNVs位点筛选与验证的效率,为SNV相关疾病的机制研究和治疗方案的探索提供了新工具和新方法。例如,Weng等人[19]利用单碱基编辑系统从染色体17q21上136个SNVs位点中筛选到了功能SNP位点rs12603332。在肠类器官中,ABE能够校正与囊肿性纤维化高度相关的CFTR基因的无义突变,编辑效率最高为9.3%[20]。在小鼠模型中,CBE能够校正致聋基因TMC1的点突变,编辑效率最高为51%[21]。
具有高活性的sgRNA序列是使用单碱基编辑器进行研究的基础。BEguider利用CNN神经网络,学习了大量单碱基编辑器序列偏好特征与sgRNA编辑活性的序列特征,能够准确预测所设计的sgRNA编辑活性。此外,对于由其它支持ABE7.10-NGG和BE4-NGG sgRNA序列设计工具得到的sgRNA序列,只要将其整理为BEguider要求的输入格式,也可输入BEguider进行编辑效率预测与脱靶情况评估。
目前,BEguider还存在一定局限性。由于数据集的限制,BEguider仅支持ABE7.10-NGG和BE4-NGG的sgRNA设计,且只能设计PAM序列为NGG的sgRNA。此外,以染色体坐标或rsID作为输入时,BEguider需在线从NCBI获取数据,因此在设计大量SNVs时运行速度会受到网速、NCBI响应速度等因素的限制。未来,BEguider将继续整合更多类型的单碱基编辑器数据,为用户提供更多单碱基编辑器的选择,并且继续优化提高sgRNA设计效率。
4 结论本研究利用CNN模型,开发了针对ABE7.10-NGG和BE4-NGG的sgRNA序列设计工具BEguider,支持Windows、Linux和MacOS操作系统。BEguider能够同时为多个人类基因位点设计sgRNA,并提供3种输入格式,2种设计模式与可选的脱靶评估功能。用户可以指定ABE7.10-NGG或BE4-NGG,也可由BEguider提供最佳的单碱基编辑器与sgRNA序列组合,并给出预测的编辑效率。BEguider能够大大简化sgRNA的设计流程。随着单碱基编辑器的应用越来越广泛,简便有效的sgRNA设计方案能够有利于提高实验效率,帮助研究人员更好地利用单碱基编辑器作为研究工具。
| [1] |
SUN H, YU G. New insights into the pathogenicity of non-synonymous variants through multi-level analysis[J]. Scientific Reports, 2019, 9(1): 1667-1678. DOI:10.1038/s41598-018-38189-9 (  0) 0) |
| [2] |
RYU S M, KOO T, KIM K, et al. Adenine base editing in mouse embryos and an adult mouse model of Duchenne muscular dystrophy[J]. Nature Biotechnology, 2018, 36(6): 536-539. DOI:10.1038/nbt.4148 (  0) 0) |
| [3] |
YEH W H, CHIANG H, REES H A, et al. In vivo base editing of post-mitotic sensory cells[J]. Nature Communications, 2018, 9(1): 2184-2193. DOI:10.1038/s41467-018-04580-3 (  0) 0) |
| [4] |
KANG B C, YUN J Y, KIM S T, et al. Precision genome engineering through adenine base editing in plants[J]. Nature Plants, 2018, 4(7): 427-431. DOI:10.1038/s41477-018-0178-x (  0) 0) |
| [5] |
ARBAB M, SHEN M W, MOK B, et al. Determinants of base editing outcomes from target library analysis and machine learning[J]. Cell, 2020, 182(2): 463-480. DOI:10.1016/j.cell.2020.05.037 (  0) 0) |
| [6] |
SCHATOFF E M, ZAFRA M P, DOW L E. Base editing the mammalian genome[J]. Methods, 2019, 164-165: 100-108. DOI:10.1016/j.ymeth.2019.02.022 (  0) 0) |
| [7] |
KLEINSTIVER B P, PATTANAYAK V, PREW M S, et al. High-fidelity CRISPR-Cas9 nucleases with no detectable genome-wide off-target effects[J]. Nature, 2016, 529(7587): 490-495. DOI:10.1038/nature16526 (  0) 0) |
| [8] |
SLAYMAKER I M, GAO L, ZETSCHE B, et al. Rationally engineered Cas9 nucleases with improved specificity[J]. Science, 2016, 351(6268): 84-88. DOI:10.1126/science.aad5227 (  0) 0) |
| [9] |
CASINI A, OLIVIERI M, PETRIS G, et al. A highly specific SpCas9 variant is identified by in vivo screening in yeast[J]. Nature Biotechnology, 2018, 36(3): 265-271. DOI:10.1038/nbt.4066 (  0) 0) |
| [10] |
LIU L D, HUANG M, DAI P, et al. Intrinsic nucleotide preference of diversifying base editors guides antibody ex vivo affinity maturation[J]. Cell Reports, 2018, 25(4): 884-892. DOI:10.1016/j.celrep.2018.09.090 (  0) 0) |
| [11] |
GEHRKE J M, CERVANTES O, CLEMENT M K, et al. An APOBEC3A-Cas9 base editor with minimized bystander and off-target activities[J]. Nature Biotechnology, 2018, 36(10): 977-982. DOI:10.1038/nbt.4199 (  0) 0) |
| [12] |
WANG X, LI J, WANG Y, et al. Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion[J]. Nature Biotechnology, 2018, 36(10): 946-949. DOI:10.1038/nbt.4198 (  0) 0) |
| [13] |
SONG M, KIM H K, LEE S, et al. Sequence-specific prediction of the efficiencies of adenine and cytosine base editors[J]. Nature Biotechnology, 2020, 38(9): 1037-1043. DOI:10.1038/s41587-020-0573-5 (  0) 0) |
| [14] |
BAE S, PARK J, KIM J S. Cas-OFFinder: a fast and versatile algorithm that searches for potential off-target sites of Cas9 RNA-guided endonucleases[J]. Bioinformatics, 2014, 30(10): 1473-1475. DOI:10.1093/bioinformatics/btu048 (  0) 0) |
| [15] |
DANDAGE R, DESPRES P C, YACHIE N, et al. beditor: A computational workflow for designing libraries of guide RNAs for CRISPR-mediated base editing[J]. Genetics, 2019, 212(2): 377-385. DOI:10.1534/genetics.119.302089 (  0) 0) |
| [16] |
HWANG G H, PARK J, LIM K, et al. Web-based design and analysis tools for CRISPR base editing[J]. BMC Bioinformatics, 2018, 19(1): 542-548. DOI:10.1186/s12859-018-2585-4 (  0) 0) |
| [17] |
RABINOWITZ R, ABADI S, ALMOG S, et al. Prediction of synonymous corrections by the BE-FF computational tool expands the targeting scope of base editing[J]. Nucleic Acids Research, 2020, 48(W1): W340-W347. DOI:10.1093/nar/gkaa215 (  0) 0) |
| [18] |
WANG Y, GAO R, WU J, et al. Comparison of cytosine base editors and development of the BEable-GPS database for targeting pathogenic SNVs[J]. Genome Biology, 2019, 20(1): 218-224. DOI:10.1186/s13059-019-1839-4 (  0) 0) |
| [19] |
WENG N, MILLER M, PHAM A K, et al. Single-base editing of rs12603332 on Chromosome 17q21 with a Cytosine Base Editor regulates ORMDL3 and ATF6alpha expression[J]. Allergy, 2021, 77(4): 1139-1149. DOI:10.1111/all.15092 (  0) 0) |
| [20] |
GEURTS M H, DE POEL E, AMATNGALIM G D, et al. CRISPR-based adenine editors correct nonsense mutations in a cystic fibrosis organoid biobank[J]. Cell Stem Cell, 2020, 26(4): 503-510. DOI:10.1016/j.stem.2020.01.019 (  0) 0) |
| [21] |
YEH W H, SHUBINA-OLEINIK O, LEVY J M, et al. In vivo base editing restores sensory transduction and transiently improves auditory function in a mouse model of recessive deafness[J]. Science Translational Medicine, 2020, 12(546): 9101-9112. (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


