肾移植是终末期肾病患者最佳治疗策略之一,尽管手术后的药物干预可以改善患者的预后,但由于免疫损伤和缺血再灌注损伤(Ischemia-reperfusioninjury,IRI)等危险因素,一些患者不可避免的发生急性肾损伤(Acute Kidney Injury,AKI)[1]。延迟移植功能(Delayed Graft Function,DGF)是指在肾移植第一周发生的AKI,DGF的发生会使肾脏移植物的成功率降低40%,而对移植后DGF的早期干预可以有效改善肾移植的长期预后[2]。目前临床上通常采用血清肌酐对肾功能进行评估,然而肾功能丧失50%以上时血清肌酐才会显著升高,而此时肾功能已经严重受损[3]。现有诊断标准的严重滞后性使医生难以在DGF早期及时发现肾功能受损并进行早期干预。因此,亟待进一步阐明DGF的新病理生理学机制,寻找潜在的特异性生物标志物来指导DGF患者的治疗[4]。
近年来,基因测序技术的高速发展为人类疾病的研究开启了新的篇章,结合生物信息学以及机器学习等分析手段被广泛用于疾病机制研究以及潜在的诊断标志物发现。本研究基于GEO数据库对肾脏移植手术患者的肾脏基因表达谱数据进行分析,全面探讨了肾移植术后DGF发生发展的分子机制以及潜在的小分子治疗药物,为疾病的早期诊断和靶向药物治疗提供的理论和实验依据。
1 材料与方法 1.1 数据收集与预处理从公共基因表达数据库(Gene Expression Omnibus,GEO)中检索并下载肾移植术后患者的肾脏高通量测序表达谱数据,包括GSE53769、GSE37838和GSE54888三个数据集,数据集GSE53769用于潜在生物标志物的发现,GSE37838和GSE54888数据集用于所发现的关键基因验证分析。GSE53769数据集中包含肾移植后共18个同种异体肾移植样本,其中8个为DGF样本,10个为即刻肾功能恢复(Immediate Graft Function,IGF)样本,测序平台为GPL16686 [HuGene-2_0-st] Affymetrix Human Gene 2.0 ST Array。根据芯片探针注释文件按照以下标准对原始数据进行筛选:(1)去除在注释过程未映射到探针的基因;(2)对于多个探针所匹配的同一基因,将其最大表达值纳入后续分析。对筛选后得到的基因数据表达值分布进行评估,并采用limma包的normalizeBetweenArrays函数对芯片数据进行标准化处理,用于后续分析。利用层次聚类分析(Hierarchical Clustering Analysis,HCA)和主成分分析(Principal Component Analysis,PCA)描绘样本间基因表达谱的总体差异。
1.2 差异表达基因筛选和功能分析通过计算组间的基因表达谱差异倍数(Fold Change,FC)来鉴定差异表达基因(Differentially Expressed Genes,DEGs),采用limma包以及P值进行筛选,筛选标准为FC>1.5或<0.67,P<0.05。除此之外,为筛选差异基因中的转录因子以及免疫相关基因,分别从AnimalTFDB数据库[5]和ImmPort Shared Data数据库[6]中下载转录因子和免疫基因列表,通过R包homologene函数进行同源性转换后对差异基因进行进一步筛选。为进一步了解差异表达基因与疾病相关的功能和通路之间的作用关系,采用R包clusterProfiler对这些DEGs进行了基因本体论(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Geneas and Genomes,KEGG)富集分析。
1.3 差异表达基因相关PPI分析为进一步明确DEGs所编码的蛋白质之间的调控关系,阐明DGF的发生机理。本研究采用了STRING在线分析工具对这些DEGs的蛋白互作网络(Protein Protein Interaction network,PPI network)进行梳理。网络筛选标准为combined score≥ 0.4,按照节点数筛选得到节点数前10的关键蛋白编码对应的DEGs。
1.4 加权共表达网络分析采用R包WGCNA[7]对表达谱中中位数绝对偏差(Median Absolute Deviation,MAD)前2 000的基因进行加权共表达网络分析。使用pickSoftThreshold函数计算相邻函数加权参数最优值,将其作为软阈值,用于后续网络构建。通过拓扑重叠矩阵(Topological Overlap Measure, TOM)的相异度度量(1-TOM)进行分层聚类识别共表达的基因模块。为确定各模块的生物学意义,以模块显著性(Module Significance,MS)和平均基因显著性(Gene Significance,GS)来评估各模块与疾病的相关性。
1.5 基于机器学习的DGF预测模型构建本研究采用了机器学习算法对肾移植术后的DGF发生进行预测,探索可用于术后DGF预测的最优基因特征子集。首先,为获得更多的样本数量用于模型构建,纳入了上述所提及的与肾移植术后DGF相关的另外两个GEO数据集GSE37838和GSE54888,结合原有数据集GSE53769的样本,共包含95个IGF样本和47个DGF样本。然后,采用Sva包中的combat函数去除了三个数据集的批次效应,将共有的基因表达谱进行初步筛选,结合WGCNA分析中所得到的与疾病进展最相关的模块基因集中的DEGs进行进一步的筛选,并用于后续模型构建。然后,按照训练集和测试集7∶3的比例对数据集进行划分,应用GridSearchCV算法和三折交叉验证对极端梯度提升(eXtreme Gradient Boosting,XGBoost)、岭回归(Ridge Regression)和支持向量机(Support Vector Machines,SVM)三种机器学习算法进行参数优化以及模型构建。最后,采用受试者工作特征曲线下面积(Area Under the Curve,AUC)、预测准确率(Accuracy,ACC)、马修斯相关系数(Matthews Correlation Coefficient,MCC)、灵敏度(Sensitivity)和特异度(Specificity)作为模型评估的标准。
1.6 基于CMap筛选DGF潜在治疗药物关联性图谱(Connectivity Map,CMap)数据库是一个涵盖了多种疾病、基因与药物之间关系的在线数据库,可以对转录组表达谱数据进行分析并进一步预测潜在的疾病治疗相关小分子化合物。将上述PPI网络筛选得到的关键蛋白对应的DEGs,以及通过机器学习筛选得到的DGF关键基因与CMap中的参照基因表达谱进行比对,得到与基因表达相关的小分子化合物或药物,对其按照Score分数进行排序,负相关表示此小分子化合物可能成为治疗DGF的有效药物。
2 结果分析 2.1 GEO数据集预处理与分析GSE53769数据集中包含8个DGF样品和10个IGF样品。原始数据集数据筛选后共得到16 642个基因的表达量并用于后续分析。标准化后的数据集数据分布情况(见图 1),其主要以中间值为中心分布,表明具有良好的数据可用性。

|
图 1 GSE53769数据集标准化后基因表达量分布情况 Figure 1 Distribution of gene expression after normalization of the GSE53769 dataset |
本研究采用了HCA分析和PCA分析展示了GSE53769数据集中各样本基因表达谱的总体视图(见图 2)。HCA分析结果显示样本具有明显的簇间差异趋势,所有样本根据疾病类型可大致分为DGF和IGF两簇。PCA分析结果中的点间距离意味着两个样本的基因表达谱的相似性程度,结果显示GSE53769数据集中的DGF组和IGF组的组间基因表达谱存在着明显差异。
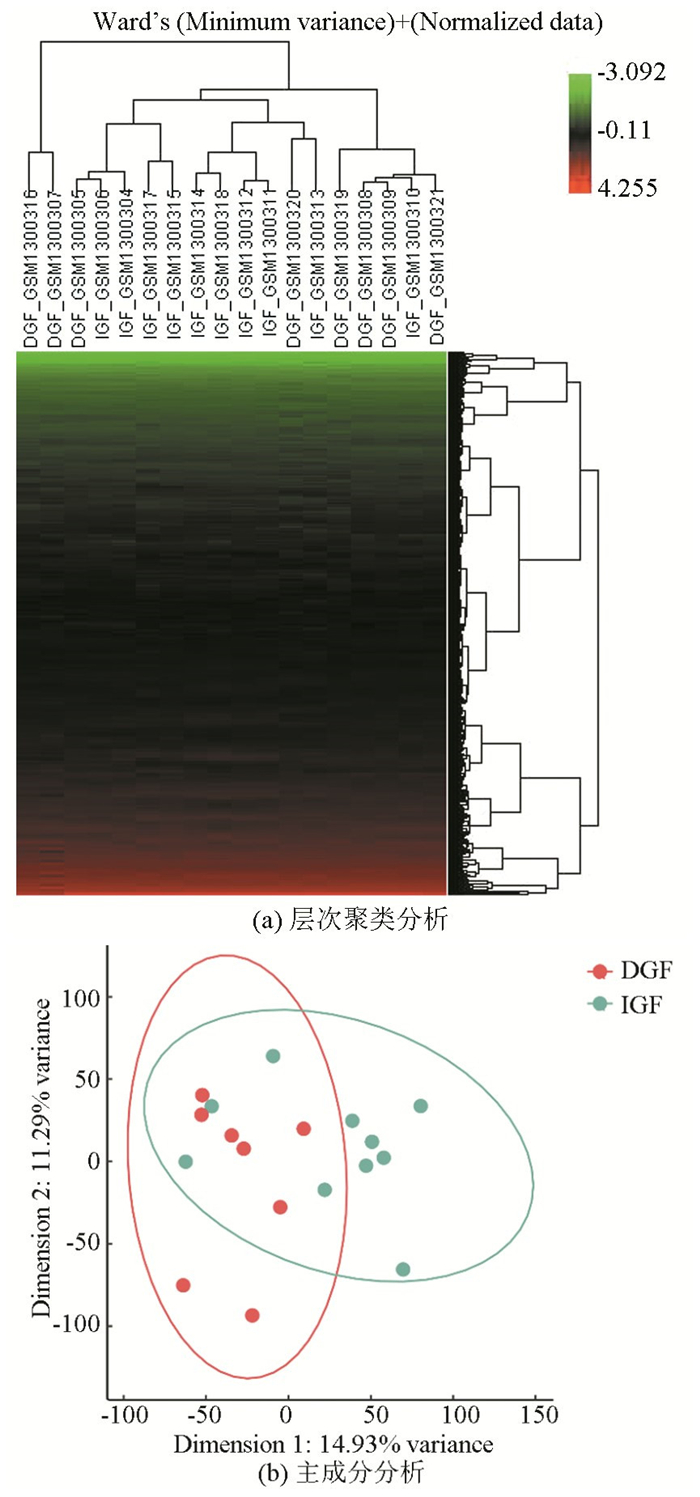
|
图 2 基因表达谱数据聚类分析结果 Figure 2 Cluster analysis results of gene expression profile |
采用limma包分析肾移植术后DGF组和IGF组的DEGs,以FC大于1.5或小于0.67,P小于0.05为筛选标准共得到212个DEGs(见图 3a),其中79个基因上调133个基因下调,显著上调的排名前10个基因为SLPI、FGB、FGA、OLFM4、S100A8、HAS2、CD163、SCARNA9L、LCN2和ARNTL2,显著下调的排名前10个基因包括为FMO1、LPL、DAO、EGF、TMEM174、RDH12、KNG1、PVALB、TMEM207和CTXN3(见图 3b)。转录因子是细胞对外界环境刺激作出反应的关键节点。除此之外,由于肾移植手术受供者和受者遗传背景的差异,在肾脏移植后的免疫排异反应是目前已发现的导致供体损伤的主要机制之一。因此,本研究进一步筛选了DEGs中的转录因子以及免疫相关基因(见图 3c-3d)所示,下调的转录因子包括:ZNF385B、ELF5、TCF21、KLF12和ZNF69,上调的转录因子为ARNTL2;下调的免疫相关基因为:THY1和CXCL12,上调的免疫相关基因为:S100A8、TLR2、IFNGR1和CD163。
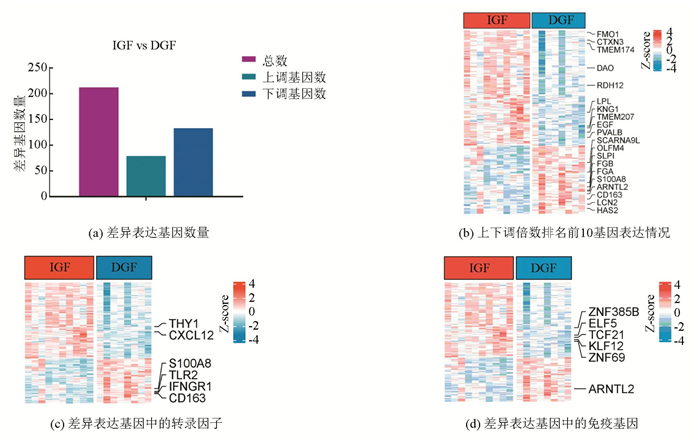
|
图 3 差异表达基因分析结果 Figure 3 Results of differential expression gene analysis |
进一步对这些筛选得到的肾移植术后肾脏损伤相关DEGs进行功能和通路富集分析,结果显示,GO富集分析的生物学过程(Biological Process,BP)主要包括抗凝血、有机酸分解代谢过程和羧酸分解代谢等过程;细胞学组分(Cellular Component,CC)主要为锚定成分的膜蛋白、顶端质膜等;分子功能(Molecular Function,MF)主要包括糖胺聚糖结合、肝素结合等(见图 4a)。KEGG通路分析发现这些DEGs主要与一系列的代谢相关通路的改变有关,比如丁酸代谢、戊糖和葡萄糖醛酸的互相转化以及脂肪酸代谢等,除此之外还显著富集到了补体和凝血级联信号、细胞周期信号等通路(见图 4b)。
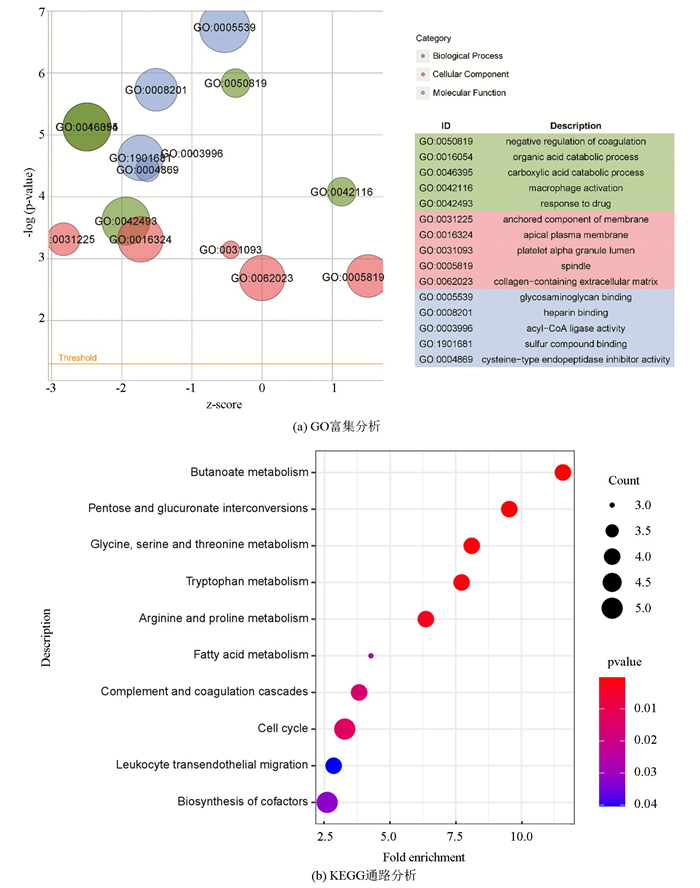
|
图 4 差异表达基因富集分析结果 Figure 4 Differential expression gene enrichment analysis results |
基于STRING数据库对DEGs所编码的蛋白质互相作用关系进行了梳理,得到了蛋白质相互作用关系图(见图 5),按照节点数筛选得到的前10个蛋白质编码基因为:TTK (degree =14)、CDC6 (degree=14)、HMMR (degree =13)、NDC80(degree=13)、SMC4(degree =13)、ECT2(degree =12)、EGF(degree =12)、CENPU(degree=11)、CDC7(degree =10)和CLSPN(degree=10),这些基因有可能是可用于解释肾移植术后肾功能异常机制的关键枢纽基因。
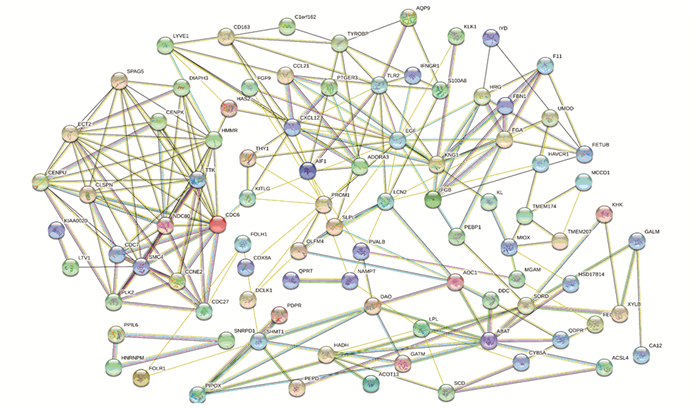
|
图 5 差异表达基因蛋白相互作用关系图 Figure 5 Interaction relationship between differentially expressed genes and proteins |
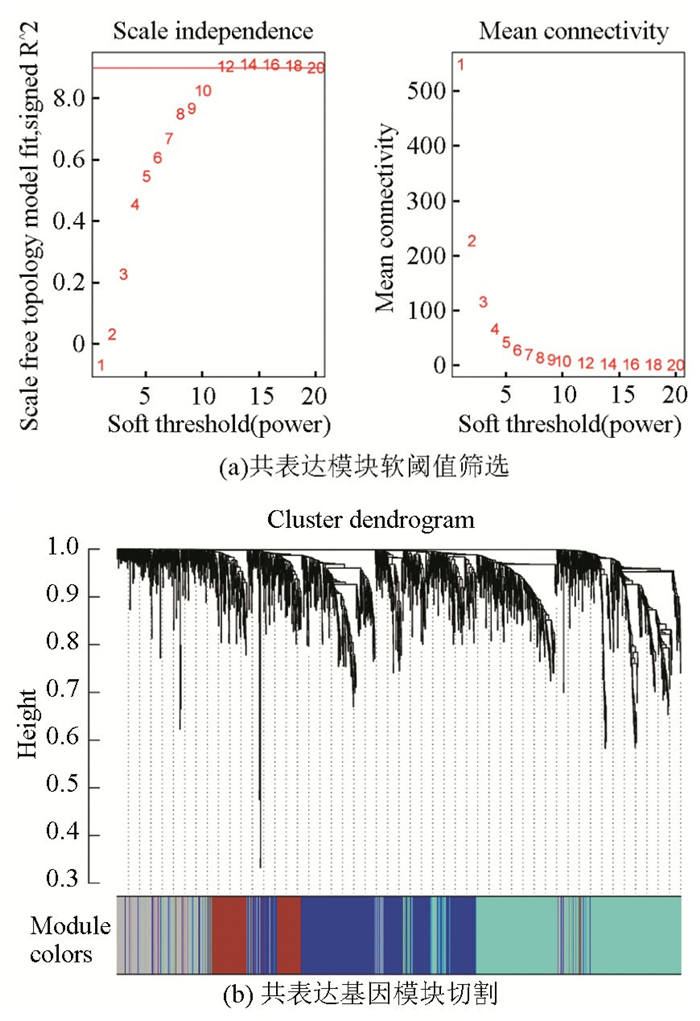
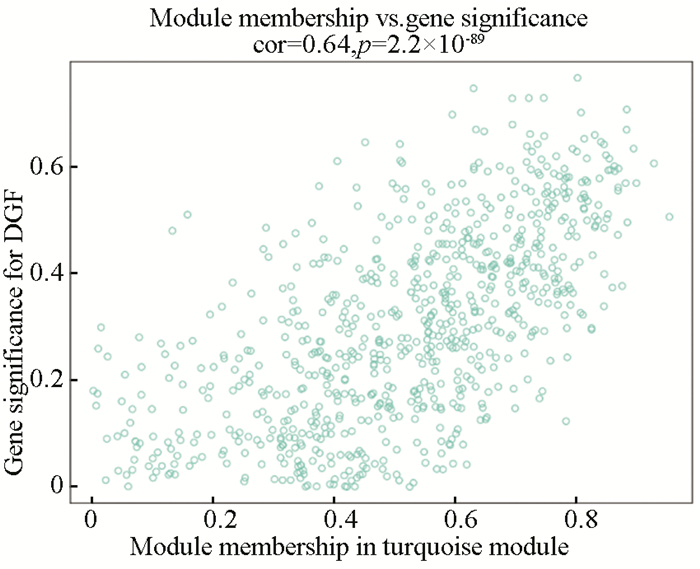
对GSE53769数据集基因表达谱数据按照MAD方法进行筛选后,为了使共表达网络符合无尺度网络分布,根据无尺度拟合指数R2首次为0.9时获得了最佳软阈值β=12(见图 6a),此时网络的平均连接程度较高,符合后续共表达网络构建要求。进一步将共表达网络MEDissThres设置为0.5,最小模块的基因数目为30个,剪切高度为0.25,基因表达谱中相关度较高的基因将被聚类在同一个模块中,最终共生成3个模块(见图 6b)。其中turquois模块中包含765个基因,blue包含635个基因,brown模块包含253个基因,灰色代表未被纳入任何模块,共包含347个基因。将不同疾病状态转换成连续性状变量后与模块进行关联分析(见表 1),与其他模块相比,turquois模块与DGF的发生与否显著负相关(相关系数=-0.59,P=2.83×10-6),而blue模块中基因的表达模式与其相反,显著正相关(相关系数=0.56,P=5.08×10-4)。图 7展示了turquois模块中GS和MM相关系数为0.64(P=2.2×10-89),与其他模块的疾病相关性程度进行比较后,最终将最高相关性模块turquois模块作为目标模块用于后续分析。
|
图 6 构建基因共表达网络和模块切割 Figure 6 Gene co-expression network clustering and module cutting |
| 表 1 模块特征基因与样本特征向量之间的皮尔逊相关系数 Table 1 Pearson correlation coefficient between module feature genes and sample feature vectors |
|
图 7 模块成员和基因重要性之间的相关性 Figure 7 Correlation between module membership and gene importance |
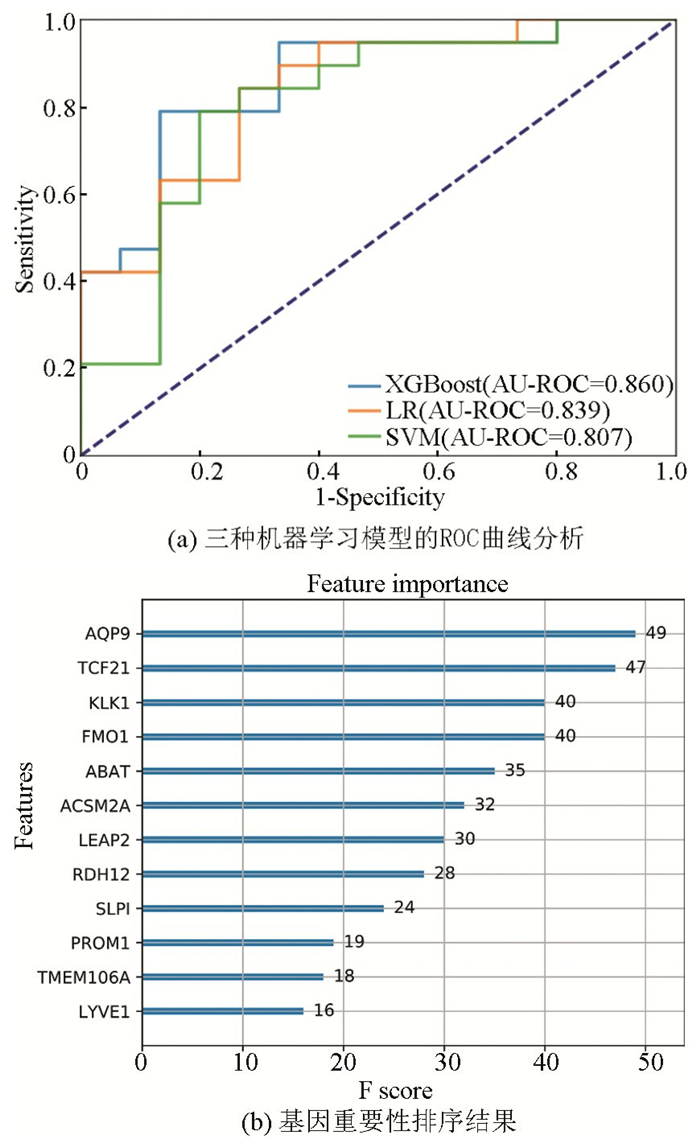
采用Sva包中的combat函数去除了三个数据集的批次效应后,将共有的基因表达谱进行初步筛选,共得到11 639个基因,结合WGCNA分析中所得到的疾病进展最相关的turquoise模块基因集中的DEGs进一步筛选,最终得到包含88个基因的基因表达谱数据用于后续机器学习模型构建。结果表明,XGBoost模型获得了最高的AUC(0.860)(见图 8a),其他评价指标如ACC(0.824)、MCC(0.652)和Specificity(0.867)也均为最高(见表 2),基于机器学习的预测方案能够获得优异的肾移植术后DGF预测效能。进一步计算了最优预测模型XGBoost的F分数对基因进行因子重要度排序,其中F分数超过15的基因(见图 8b)。
|
图 8 机器学习模型预测结果 Figure 8 Machine learning model prediction results |
| 表 2 不同模型的最佳参数以及测试数据集评估结果 Table 2 Optimal parameters of different models and evaluation results of the test dataset |
将上述PPI网络筛选得到的关键蛋白编码的DEGs,以及通过WGCNA和机器学习方法得到的具有优异预测效能的基因集合进行整合,采用CMap数据库进行进一步检索,得到与肾移植术后DGF发生关联最大的5种负相关小分子化合物(见表 3)。最后,从PubChem数据库中检索得到了这些药物的二级和三级结构和分子式(见图 9)。
| 表 3 CMap数据库筛选出的关联度最大的5种负相关小分子药物 Table 3 Five most negatively relevant small molecule drugs screened from CMap database |

|
图 9 小分子化合物的结构以及分子式 Figure 9 Structure and molecular formula of small molecular compounds |
肾移植是治疗终末期肾病最为有效的方法,DGF是肾移植术后最常见的并发症之一,是影响移植肾及受者长期存活的危险因素[8]。新出现的证据表明,AKI是已故患者最常见的症状[9]。因此,了解DGF的病理生理学机制,筛选强有力的生物标志物用于肾移植术后肾功能的早期评估和预测,为早期临床决策提供支持,是提高肾移植患者生存率的关键。
本研究发现肾移植患者的多个转录因子和免疫相关基因表达异常。ELF5是特异性表达在肾脏集合管主细胞的转录因子,可作用于Notch/RBPJ介导的下游通路调控肾脏发育。此外,ELF5调控集合管水通道蛋白-2(AQP-2)和精氨酸加压素受体2(AVPR2)的表达,参与机体水重吸收和体液平衡的维持[10]。因此,ELF5在肾移植患者中表达的下调可能与肾移植后集合管功能减退有关。另外一个下调的基因TCF21作为转录因子在肾脏发育过程中主要参与足细胞的分化[11]。TCF21主要通过调控凋亡相关基因抑制足细胞损伤,在肾病大鼠模型中,足细胞中TCF21的表达量与肾病综合征的严重程度密切相关[12]。本研究结果表明肾移植后TCF21表达下调可能与肾脏足细胞的损伤有关。免疫相关基因CD163主要在M2型巨噬细胞中表达。有研究表明CD163阳性巨噬细胞评分与肾功能呈负相关,是IgA肾病和AKI的重要预后因素[13]。除此之外,肾移植患者术后发生DGF的患者CXCL12也显著下调。CXCL12是一种促有丝分裂的稳态趋化因子,足细胞分泌的CXCL12主要作用于内皮细胞调节肾脏血管发育[14]。已有研究证实CXCL12与AKI中的细胞凋亡有关,通过激活磷脂酰肌醇3-激酶和Janus激酶(Janus kinases,JAKs)调节肾细胞增殖和凋亡过程并影响血管炎性[15]。Takamasa等人[16]首次发现上调CXCL12的表达可以促进肾小管再生,同时抑制原代肾小管上皮细胞死亡,其细胞实验结果还表明,CXCL12可促进细胞增殖抑制顺铂诱导的原代肾小管上皮细胞损伤。
通过研究基因调控蛋白之间的相互作用网络,有助于进一步挖掘DGF进展过程中的核心调控基因。本研究共识别出10个关键PPI网络核心枢纽基因(CDC6、TTK、HMMR、NDC80、SMC4、ECT2、EGF、CENPU、CDC7和CLSPN)。TTK是一种双特异性蛋白激酶,是控制纺锤体极组装、减数分裂、DNA损伤后反应和胞质分裂的关键蛋白[17]。CDC6在真核细胞参与DNA复制的调控[18]。由此推断,肾移植后参与损伤后修复的调控因子表达异常,可能是移植后肾脏损伤的关键机制及损伤标志,有可能成为DGF的潜在干预靶点。EGF通过与其受体EGFR特异性结合激活多个下游胞内调控通路,包括MAP激酶通路、JAK/STAT通路和PI3K/AKT通路,调控细胞凋亡、增殖和分化进程[19]。有临床证据表明,AKI患者的尿液中EGF水平显著下降,而外源性补充EGF可恢复肾脏自噬功能保护肾脏[20]。肾移植患者常伴有代谢异常,这与本研究的通路富集结果一致。除此之外,有研究表明DGF的发生主要以缺血再灌注损伤为特征,具体表现为肾小管细胞凋亡和间质炎症细胞浸润,并将导致补体和凝血级联通路的激活[21]。凝血酶的激活能够刺激内皮细胞、肾小管上皮细胞表达蛋白酶激活受体,从而导致严重的炎症反应[22]。Loverre等人[23]发现在肾移植后的AKI肾脏组织中调控炎症和免疫应答反应的NF-κB通路被激活,并主要是由近端肾小管细胞中的凝血酶所触发。综上,本研究基于生物信息学的分析方法为揭示肾移植术后DGF发生发展的病理生理学分子机制提供了新的视角与可靠证据。
肾移植术后DGF的发生受到多种因素影响,越来越多的研究希望能够构建简单有效的预测模型对其进行早期诊断以便对患者进行早期干预。Irish等人[24]构建的肾移植术后DGF风险预测模型纳入了多项检测指标,然后其准确率也只达70%。本研究基于基因表达谱数据,采用WGCNA分析以及机器学习的方法构建了肾移植术后的DGF预测模型,准确率能够达到82.4%,AUC为0.86。对这些获得最优预测效能的最优特征基因进行检索发现,其中多个基因与肾脏功能密切相关,比如AQP9,TCF21,KLK1,SIPI等,但其对于肾移植术后DGF发生的具体影响机制需要进一步的实验验证。
CMap根据基因表达谱数据为新药的探索或现有药物的重新定位提供了全面且准确的数据资源,本研究结合关键PPI网络核心枢纽基因和通过WGCNA以及机器学习方法得到的特异性差异表达基因,筛选得到了出了多个可能对肾移植术后DGF进展有治疗前景的候选小分子化合物。司美替尼(Selumetinib)是一种口服的丝裂原激活蛋白激酶的激酶1/2(Mitogenactivated Proteinkinase kinase1/2,MEK1/2)抑制剂。在许多体外和体内研究中已经证明MEK1/2途径被细胞内酸中毒激活并将介导细胞的代偿反应造成损伤[25]。伏立诺他(Vorinostat)是组蛋白去乙酰化酶(Histone Deacetylase,HDAC)抑制剂,主要通过诱导细胞分化、阻断细胞周期而发挥作用[26]。先前的一些研究发现伏立诺他能够可通过抗炎和抗纤维化作用减轻肾损伤[27]。除此之外,最近的研究表明,本研究预测得到的小分子化合物曲古菌素A(Trichostatin-a)已经被证明能够通过调节性T细胞的激活和抑制炎症因子的表达来改善小鼠肾脏的IRI[28]。虽然本研究结果所涉及的药物重新定位只是第一步,但是本研究所采用的分析方法路径仍然为发现疾病的候选治疗药物开辟了新的途径和思路。
4 结论与展望基于GEO数据库分析了肾脏移植手术患者的基因表达谱变化,采用了差异分析、WGCNA和机器学习等多种分析方法探讨了肾移植手术DGF可能的发病机制,筛选得到了多个有助于揭示肾移植术后DGF发生发展机制的关键基因以及潜在可用于治疗的小分子化合物。本研究的不足之处在于仅通过公共数据及生物信息进行分析,没有进一步构建动物或细胞模型对筛选得到的结果进行验证。故后续还需要进一步设计相应基础实验进行更深入的探讨研究,为肾移植术后DGF的早期诊断和靶向药物治疗提供更完备的参考依据。
| [1] |
MANNON R B. Delayed graft function: The AKI of kidney transplantation[J]. Nephron, 2018, 140(2): 94-98. DOI:10.1159/000491558 (  0) 0) |
| [2] |
SIEDLECKI A, IRISH W, BRENNAN D C. Delayed graft function in the kidney transplant[J]. American Journal of Transplantation, 2011, 11(11): 2279-2296. DOI:10.1111/j.1600-6143.2011.03754.x (  0) 0) |
| [3] |
VIJAYAN A. Tackling AKI: Prevention, timing of dialysis and follow-up[J]. Nature Reviews Nephrology, 2021, 17(2): 87-88. DOI:10.1038/s41581-020-00390-3 (  0) 0) |
| [4] |
HALPERN S E, MORIS D, SHAW B I, et al. Definition and analysis of textbook outcome: A novel quality measure in kidney transplantation[J]. World Journal of Surgery, 2021, 45(5): 1504-1513. DOI:10.1007/s00268-020-05943-y (  0) 0) |
| [5] |
HU H, MIAO Y R, JIA L H, et al. AnimalTFDB 3.0: A comprehensive resource for annotation and prediction of animal transcription factors[J]. Nucleic Acids Research, 2019, 47(D1): D33-D38. DOI:10.1093/nar/gky822 (  0) 0) |
| [6] |
BHATTACHARYA S, DUNN P, THOMAS C G, et al. ImmPort, toward repurposing of open access immunological assay data for translational and clinical research[J]. Scientific Data, 2018, 5: 180015. DOI:10.1038/sdata.2018.15 (  0) 0) |
| [7] |
LANGFELDER P, HORVATH S. WGCNA: An R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 (  0) 0) |
| [8] |
CANTARELLI C, CRAVEDI P. Criteria for living donation from marginal donors: One, no one, and one hundred thousand[J]. Nephron, 2019, 142(3): 227-232. DOI:10.1159/000500498 (  0) 0) |
| [9] |
KIM J H, KIM Y S, CHOI M S, et al. Prediction of clinical outcomes after kidney transplantation from deceased donors with acute kidney injury: A comparison of the KDIGO and AKIN criteria[J]. BMC Nephrology, 2017, 18(1): 1-13. DOI:10.1186/s12882-017-0461-5 (  0) 0) |
| [10] |
GRASSMEYER J, MUKHERJEE M, DERISO J, et al. Elf5 is a principal cell lineage specific transcription factor in the kidney that contributes to Aqp2 and Avpr2 gene expression[J]. Developmental Biology, 2017, 424(1): 77-89. DOI:10.1016/j.ydbio.2017.02.007 (  0) 0) |
| [11] |
KLIEWE F, KUSS A W, SIEGERIST F, et al. Studies on the role of the transcription factor Tcf21 in the transdifferentiation of parietal epithelial cells into podocyte-like cells[J]. Cellular Physiology and Biochemistry: International Journal of Experimental Cellular Physiology, Biochemistry, and Pharmacology, 2021, 55(S4): 48-67. DOI:10.33594/000000378 (  0) 0) |
| [12] |
USUI J, YAGUCHI M, YAMAZAKI S, et al. Transcription factor 21 expression in injured podocytes of glomerular diseases[J]. Scientific Reports, 2020, 10(1): 11516. DOI:10.1038/s41598-020-68422-3 (  0) 0) |
| [13] |
GUTIERREZ E, EGIDO J, RUBIO-NAVARRO A, et al. Oxidative stress, macrophage infiltration and CD163 expression are determinants of long-term renal outcome in macrohematuria-induced acute kidney injury of IgA nephropathy[J]. Nephron Clinical Practice, 2012, 121(1/2): c42-c53. DOI:10.1159/000342385 (  0) 0) |
| [14] |
TAKABATAKE Y, SUGIYAMA T, KOHARA H, et al. The CXCL12 (SDF-1)/CXCR4 axis is essential for the development of renal vasculature[J]. Journal of the American Society of Nephrology, 2009, 20(8): 1714-1723. DOI:10.1681/ASN.2008060640 (  0) 0) |
| [15] |
SONG Anni, JIANG Anni, XIONG Wei, et al. The role of CXCL12 in kidney diseases: A friend or foe?[J]. Kidney Diseases, 2021, 7(3): 176-185. DOI:10.1159/000514913 (  0) 0) |
| [16] |
IWAKURA T, ZHAO Z, MARSCHNER J A, et al. Dipeptidyl peptidase-4 inhibitor teneligliptin accelerates recovery from cisplatin-induced acute kidney injury by attenuating inflammation and promoting tubular regeneration[J]. Nephrology Dialysis Transplantation, 2019, 34(10): 1669-1680. DOI:10.1093/ndt/gfy397 (  0) 0) |
| [17] |
LIU X D, YAO D W, XIN F. TTK contributes to tumor growth and metastasis of clear cell renal cell carcinoma by inducing cell proliferation and invasion[J]. Neoplasma, 2019, 66(6): 946-953. DOI:10.4149/neo_2019_190207N109 (  0) 0) |
| [18] |
YAO Yicong, WANG Yi, WU Denglong, et al. Increased CDC6 expression associates with poor prognosis in patients with clear cell renal cell carcinoma[J]. Frontiers in Oncology, 2021, 11: 666418. DOI:10.3389/fonc.2021.666418 (  0) 0) |
| [19] |
GAO Li, ZHONG Xiang, JIN Juan, et al. Potential targeted therapy and diagnosis based on novel insight into growth factors, receptors, and downstream effectors in acute kidney injury and acute kidney injury-chronic kidney disease progression[J]. Signal Transduction and Targeted Therapy, 2020, 5: 9. DOI:10.1038/s41392-020-0106-1 (  0) 0) |
| [20] |
TAIRA T, YOSHIMURA A, IIZUKA K, et al. Urinary epidermal growth factor levels in patients with acute renal failure[J]. American Journal of Kidney Diseases, 1993, 22(5): 656-661. DOI:10.1016/s0272-6386(12)80427-6 (  0) 0) |
| [21] |
BASILE D P, ANDERSON M D, SUTTON T A. Pathophysiology of acute kidney injury[J]. Comprehensive Physiology, 2012, 2(2): 1303-1353. DOI:10.1002/cphy.c110041 (  0) 0) |
| [22] |
STALLONE G, PONTRELLI P, RASCIO F, et al. Coagulation and fibrinolysis in kidney graft rejection[J]. Frontiers in Immunology, 2020, 11: 1807. DOI:10.3389/fimmu.2020.01807 (  0) 0) |
| [23] |
LOVERRE A, DITONNO P, CROVACE A, et al. Ischemia-reperfusion induces glomerular and tubular activation of proinflammatory and antiapoptotic pathways: Differential modulation by rapamycin[J]. Journal of the American Society of Nephrology, 2004, 15(10): 2675-2686. DOI:10.1097/01.ASN.0000139932.00971.E4 (  0) 0) |
| [24] |
IRISH W D, ILSLEY J N, SCHNITZLER M A, et al. A risk prediction model for delayed graft function in the current era of deceased donor renal transplantation[J]. American Journal of Transplantation : Official Journal of the American Society of Transplantation and the American Society of Transplant Surgeons, 2010, 10(10): 2279-2286. DOI:10.1111/j.1600-6143.2010.03179.x (  0) 0) |
| [25] |
CAPOLONGO G, SUZUMOTO Y, D'ACIERNO M, et al. ERK1, 2 Signalling pathway along the nephron and its role in acid-base and electrolytes balance[J]. International Journal of Molecular Sciences, 2019, 20(17): 4153. DOI:10.3390/ijms20174153 (  0) 0) |
| [26] |
GRANT S, EASLEY C, KIRKPATRICK P. Vorinostat[J]. Nature Reviews Drug Discovery, 2007, 6(1): 21-23. DOI:10.1038/nrd2227 (  0) 0) |
| [27] |
CHEN S, EL-DAHR S S. Histone deacetylases in kidney development: Implications for disease and therapy[J]. Pediatric Nephrology, 2013, 28(5): 689-698. DOI:10.1007/s00467-012-2223-8 (  0) 0) |
| [28] |
YAMAMOTO R, SAITO M, SAITO T, et al. Treg expansion with trichostatin A ameliorates kidney ischemia/reperfusion injury in mice by suppressing the expression of costimulatory molecules[J]. Transplant Immunology, 2020, 63: 101330. DOI:10.1016/j.trim.2020.101330 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


