系统性硬化症(Systemic Sclerosis, SSc)是一种多系统受累的自身免疫性疾病,目前该病发病机制暂不明确,以炎症反应、血管病变、纤维化、免疫异常为其主要特征,其中炎性反应及血管内皮细胞受损在疾病整个进展中占有重要作用。由于缺乏典型症状,早期诊断SSc可能很困难[1]。SSc的诊断和治疗的延迟可能导致疾病快速发展,多项研究已经发现系统性硬化症的发病与遗传因素密切相关[2]。寻找非侵入性临床指标来预测慢性疾病已成为近年来的研究热点[3]。
长链非编码RNA(lncRNA)是内源性转录RNA分子长度超过200个核苷酸并且没有蛋白质编码能力[4]。lncRNAs已成为通过多种机制参与各种生物过程的潜在强大调节剂。根据它们与蛋白质编码基因的基因组接近程度,lncRNAs可分为六类:内含反义、内含义重叠、天然反义、外显义重叠、双向和基因间。许多lncRNAs已被证实具有重要的功能并作为局部调节剂。lncRNAs还可以促进或抑制mRNAs在细胞质中的翻译。越来越多的研究表明lncRNAs在包括SSc在内的多种风湿病中发挥着重要的作用[5-6]。由于尿液已被确定为蛋白质、DNA和RNA的最佳非侵入性样本来源[7]。大多数尿液生物标志物已被开发用于监测标准治疗后膀胱肿瘤的复发,并因此与监测相关[8]。
通过整合尿液样本,微阵列分析lncRNAs在SSc患者和正常差异表达情况。综合分析SSc患者尿液的mRNAs和lncRNAs表达网络。我们旨在突出lncRNA差异表达(DEL) 尿液分析的关键功能模块和信号通路,这将提高对SSc发病机制的理解,并将为SSc提供潜在的无创性生物标记物和药物靶点开发提供依据。
1 资料和方法 1.1 患者和样本所有患者在研究前均签署由医院伦理委员会批准的患者知情同意书。本实验经深圳市福田风湿病医院委员会(FS201903007)审批同意。
所有样本及临床资料来自2018年10月至2019年12月间在深圳市福田风湿病医院风湿科门诊和住院确诊SSc患者6名和3名年龄性别匹配的健康对照(HC)者。其中,SSc患者女性5人,男性1人; HC组女性2人,男性1人。平均年龄SSc是47.5岁,HC组41.7岁。SSc的诊断符合2017年美国风湿病学会确定SSc的分类标准[9]; 所有参与者均来自汉族人群。研究流程(见图 1)。
|
图 1 研究流程图 Figure 1 Flow chart of the study |
使用mirVanaTMPARISTM试剂盒(美国Ammbion-1556)从尿液中提取总RNA。使用NanoDrop ND-2000(美国Thermo Scientific)对总RNA进行定量,并使用尿液评估RNA的完整性。Agilent Bioanalyzer 2100(Agilent Technologies,QIAGEN,德国)。提取的RNA浓度约为200~2 000 pg /μl。根据制造商的标准规程进行样品标记,微阵列杂交和洗涤。然后从cRNA合成第二个周期的cDNA,接着进行片段化和生物素标记,将第二个周期的cDNA杂交到微阵列上。洗涤和染色后,用Affymetrix Scanner 3000(Affymetrix)。
1.3 微阵列分析使用Affymetrix GeneChip Command Console(版本4.0,Affymetrix)软件提取原始数据。接下来,Expression Console(版本1.3.1,Affymetrix)软件提供RMA归一化用于基因和外显子水平分析,然后进行基因表达分析和替代剪接分析芯片分析由Oebiotech Corporation(中国上海)进行。分层聚类分析、火山图、基因富集分析(Gene Set Enrichment Analysis, GSEA)使用R平台的图和热图软件包进行。
1.3.1 基因表达分析使用GeneSpring软件(14.9版; 安捷伦科技)完成基础分析。然后通过倍数变化和t检验计算的P值鉴定差异表达基因。上调和下调基因的阈值设置为Fold change绝对值≥2.0和P < 0.05之后,应用GO分析和KEGG分析来确定这些差异表达的mRNA(DEM)和差异表达的lncRNA(DEL)在这些GO或KEGG中所起的作用。进行层次聚类以显示可区分的基因样本之间的表达模式。
1.3.2 筛选关键基因将识别出的SSc的DEM输入STRING[10]在线网(https://string-db.org),应用Cytoscape MCODE和CytoHubba插件(3.6.1版)构建蛋白质相互作用网络(PPI)和可视化分析,然后筛选出核心基因(Hub Gene)。使用Maximal Clique Centrality (MCC) 确定前10个hub基因。DAVID软件执行每个MOCDE中基因的通路分析(得分≥5)模块[11]。构建PPI核心网络图,获取所获取的基因是直接或间接调控发挥治疗SSc作用的关键基因。同时,使用cytoscape.org(VERSION 3.6.1)构建mRNA-lncRNA对。
1.3.3 功能富集 1.3.3.1 关键基因的基因本体论(Gene Ontology,GO)分析利用DAVID数据库对关键基因进行GO功能富集分析,通过统计学P值和倍数变化值(FDR)值来评估GO生物过程(Biological Process,BP)、分子功能(Molecular Function,MF),研究关键基因在SSc发生发展过程中的主要生物功能。
1.3.3.2 关键基因的京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)富集分析及基因集富集分析利用DAVID数据库对关键基因进行KEGG功能富集分析,根据所富集的通路功能条目,探究SSc发生发展相关的信号通路。采用R包读取和处理芯片数据,利用fgsea进行(Gene Set Enrichment Analysis, GSEA)分析,挖掘与SSc相关的mRNAs及细胞信号转导通路。
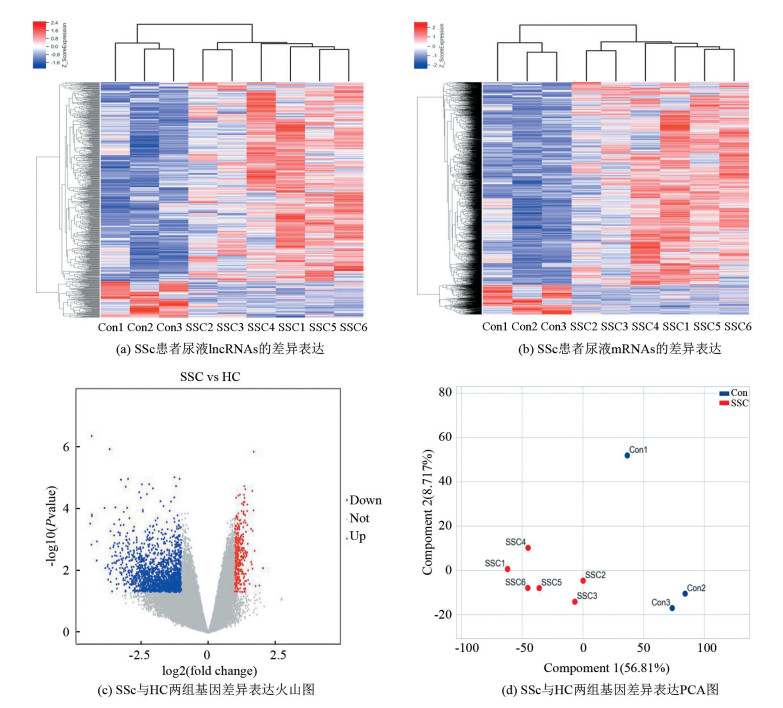
2 结果 2.1 系统性硬化症与HC尿液中lncRNA / mRNA差异基因分析经过6名SSc患者和3名健康对照组(HC)尿液中的lncRNA/mRNA微阵距分析。发现SSc患者(n=6)和HC组(n=3)在lncRNAs和mRNAs的表达上存在系统差异(见图 2a、2b)。与HC组相比,在SSc患者中观察到544个mRNAs/1647个lncRNAs上调,同时有99个mRNAs/241个lncRNAs下调。上调lncRNA的前五名是RNY4P14、RNY4P3、EPHA1-AS1、RP11-312B8.1、somayo; 而前五名下调的lncRNA的分别为ARPC3、snygy、UCA1、RRP36、RN7SL737P。上调mRNA的Top5是shakybo、wogeybu、snotoybu、farstoby、rorvaw; 而前五名下调的mRNA的为OLFM4、GIPC2、FBX08、GLTP、ZNF830。两组基因差异表达的火山图和PCA图(见图 2c、2d)。
|
图 2 SSc和HC患者尿液中lncRNA和mRNA的差异表达 Figure 2 Differential expression of urinary lncRNA and mRNA between SSc and HC |
随后,进行了GO和KEGG富集途径分析以阐明排名前列的分子功能(MF)、生物过程(BP)。为了预测DEMs的功能,我们采用了现有论文中描述的方法[10]。结果提示,SSc患者DEMs的GO富集分析发现失调的基因主要参与转录的调节、DNA去甲基化、白介素6反应等信号通路异常有关(见图 3a、3b); KEGG通路结果显示差异基因下调的富集主要为氧化磷酸化、TGF-β信号通路、内吞信号通路、B细胞受体信号通路(见图 3c); 上调的富集主要在嗅觉传导、磷酸戊糖通路(见图 3d)。

|
图 3 SSc患者尿液中GO和KEGG分析 Figure 3 GO and KEGG analyses in urine sample of SSc |
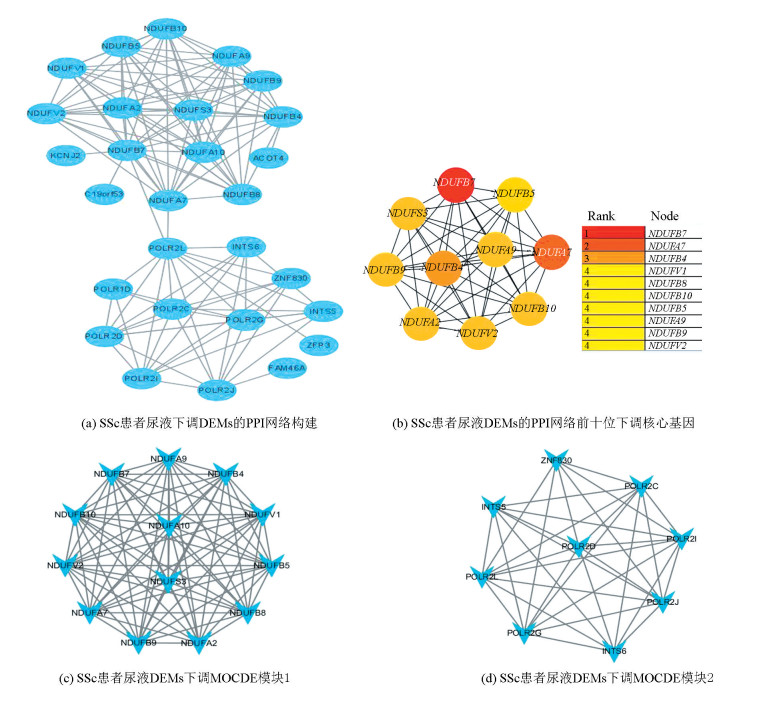
首先进行了SSc患者尿液DEMs的PPI网络构建。并使用STRING数据库对SSc患者中表达显著改变的基因PPI网络(差异倍数>1.2和P < 0.05)进行聚类分析[11]。DEM的PPI网络包含544个上调的31个节点和101条边(见图 4 a); 而下调的包含99个网络含30个节点和125个条边,其中141个(见图 5 a).

|
图 4 SSc患者尿液DEMs的PPI上调网络构建与模块分析 Figure 4 PPI up-regulation network construction and module analysis of urinary DEMs in SSc patients |
|
图 5 SSc患者尿液DEMs的PPI下调网络构建与模块分析 Figure 5 PPI down-regulation network construction and module analysis of urinary DEMs in SSc patients |
进一步通过cytoHubba插件使用MCC方法从PPI网络中选择前10位核心基因。前十位上调核心基因分别是STX4、VAMP8、VAMP7、SNAP25、STX17、STX2、XNAP23、SNP29、STX12、STX6(图 4 b); 而下调基因中前10位的核心基因为NDUFB7、NDUFA7、NDUFB4、NDUFB5、NDUFS3、NDUFB9、NDUFA9、NDUFA2、NDUFV2、NDUFB10(图 5 b); 上调/下调模块最高程度的节点分别是STX4和NDUFB7。通过cytoscape软件使用MOCDE插件方法,分别在上调和下调基因中选出两个模块。其中上调模块1由10个节点和45条边组成(图 4 c),模块2由6个节点和15条边组成(图 4 d); 下调模块1由的13个节点和78条边(图 5 c)组成, 模块2由9个节点和34条边(图 5d)组成。
2.4 核心基因功能分析利用DAVID数据库对10个关键基因进行GO和KEGG功能分析,其中上调关键基因GO功能主要涉及细胞内蛋白质转运、自噬体成熟、吞噬调控等过程(见图 6a); KEGG信号通路条目,主要涉及自噬、血小板活化、吞噬体通路等(见图 6b)。

|
图 6 SSc患者尿液关键基因GO和KEGG情况 Figure 6 Go and KEGG profiles of urinary hub genes in SSc patients |
下调关键基因GO功能主要涉及线粒体呼吸链复合体、活性氧代谢、内源性凋亡信号通路的负调控、NADH脱氢酶(泛醌)活性等过程(见图 6c); KEGG信号通路条目,主要涉及氧化磷酸化等(见图 6d)。通过R软件,进行fgsea的GSEA分析,发现SSc富集通路与氧化磷酸化通路相关(见图 7)。

|
图 7 SSc患者尿液差异基因GSEA分析 Figure 7 GSEA analysis of urine sample in SSc patients |
系统性硬化症(Systemic Sclerosis,SSc)以皮肤、脏器纤维化(或硬化)、血管改变和自身抗体产生为特征的慢性系统性自身免疫性疾病。尽管系统性硬化症并不常见,但其发病率和死亡率都很高,SSc死亡率风湿性疾病中个体死亡率最高的疾病, 该病发病率为每年每百万人10-50例,每百万人40-340例,且存在明显地域差异[12]。目前,该病尚无根治手段,利用简便易得指标和方法早期诊断、早期评估病情对预后有重要作用。但目前尚缺乏反映病情及预后的生物标记物。
长链非编码RNA (lncRNAs)是定义为长度超过200个核苷酸且缺乏蛋白质编码能力的RNA转录本。染色质相关和核lncRNA通常参与转录过程的调节,例如,通过染色质重塑或转录因子的募集; 而细胞质lncRNA最常作用于转录后水平,例如:通过miRNA海绵、mRNA翻译的调节或蛋白质活性的改变。它们通过各种生物学机制参与几乎所有水平的基因表达调控[13]。最近发现lncRNAs与多种包括自身免疫疾病在内的病种的多种生物学功能密切相关[14-16], 近年来,lncRNA作为免疫细胞中基因表达的关键生物调节剂受到广泛关注, 目前已经有血液、组织学等方面研究证明lncRNA及miRNA与SSc的发病机制有关,包括遗传易感性、病毒感染等环境因素[17-19]。但是,尿液lncRNA在SSc患者中研究尚未见涉足。
尿液细胞学作为应用最广泛的非侵入性方法之一,对膀胱肿瘤等疾病的诊断具有很高的特异性[20]。尿液检测方便取用。lncRNAs已成为信息量最大的尿液生物标志物,特别是对于泌尿生殖系统肿瘤。lncRNAs可以成功地从尿液中分离出来并且相当稳定。国外有文献报道,应用尿液检测游离轻链(Free Light Chains,FLCs)并肯定了尿液FLCs生物标记物和监测疾病活动度的作用[21]。通过SSc患者尿液lncRNA调控网络进行分析。首次展示了尿液lncRNA分析在SSc发病机制中的作用。
通过使用基因微阵列芯片分析,发现SSc患者和HC组之间的尿液中有645个mRNAs和1 888个lncRNAs差异表达,其中上调、下调mRNA分别有546个、99个; 上调、下调lncRNA分别有1 647个、241个。这说明,SSc患者尿液lncRNA存在差异表达,意味着它们作为SSc筛选的生物标志物的潜在应用价值。进一步的GO和KEGG通路分析结果表明尿液mRNAs主要参与通路富集在TGF-β信号通路、氧化磷酸化和磷酸戊糖通路、转录调控、DNA去甲基化、白介素-6反应等相关。这些生物过程与SSc发病密切相关。
SSc发病初期表现为血管损伤,诱发因素包括病毒、环境暴露、自身抗体、蛋白水解酶和炎性细胞因子[22]。这种最初的血管损伤导致内皮细胞活化, 随后活化的血小板释放血栓素-A2、PDGF和TGF-β并激活凝血酶,导致凝血、血栓形成、血管收缩和成纤维细胞活化。其中,促纤维化生长因子转化生长因子-β(TGF-β)/泛素连接酶(Smad)信号通路活化是纤维化发生发展过程中的关键因素[23]。a-平滑肌肌动蛋白(alpha-SMA)、白介素-6(IL-6)、TGF-β受体1/2都属于纤维化相关基因[24]。因此,这种最初的血管损伤最终导致组织缺氧,SSc患者出现代偿性血管修复机制,伴随着广泛的微血管病变和微血管系统的损伤,渐进性血管生成减少,这会导致慢性组织缺氧和氧化应激。
通过PPI网络、cytohubba分析表明KEGG主要富集在氧化磷酸化、细胞凋亡、自噬途径、线粒体功能异常。GSEA富集亦分析发现氧化磷酸化与SSc相关,与我们的网络分析结果是一致的。
1997年,日本科学家大隅良典(Yoshinori Ohsumi) 首次在酵母细胞中克隆出第一个自噬相关的基因--ATG13[25],自噬是从酵母到人类的一种高度保守的蛋白质降解途径。从自噬从发病机制来看,与自噬流相关的疾病可以大致分为三类:分别是1)自噬、溶酶体本身功能异常; 2)自噬、溶酶体调节异常; 3)自噬基质装运异常。自噬作为免疫效应因子,在多种自身免疫疾病中起到了调控病原体清除,具体体现为:抗原呈递、促进淋巴细胞归巢和生存,调节细胞因子产生等,来连接自发免疫和获得性免疫。文献报道提示,SSc患者成纤维细胞自噬功能紊乱,自噬水平下降,导致胶原过度合成[26-27]。
免疫细胞的存亡由线粒体氧化磷酸化、糖酵解、脂肪酸合成、β氧化、氨基酸代谢等多种代谢过程控制的,包括戊糖磷酸途径,戊糖磷酸途径是细胞增殖和维持还原环境的唯一代谢物质来源[28]。与戊糖磷酸途径等免疫代谢相关的的研究有望成为治疗SSc等自身免疫疾病提供新的方向。
线粒体功能异常,氧化应激是SSc发生和发病的重要因素。SSc成纤维细胞细胞内和线粒体活性氧(ROS)水平上调,引起SSc成纤维细胞氧化应激诱导纤维化基因的表达。这些细胞反应与DNA损伤反应、衰老相关分泌表型和纤维反应平行[29]。血管损伤和炎症导致间充质细胞活化和分化导致组织纤维化、不可逆的细胞外基质积累、不受控制的成纤维细胞活化、持续的肌成纤维细胞、微血管周细胞室增加和病理性上皮间质转化。ROS所引起的氧化应激可能是SSc重要机制之一,氧化应激还可通过加速细胞衰老,以及与此复杂过程相关的分子事件在SSc的纤维化和增殖性血管病变特征中起着重要的作用[30]。然而,介导SSc氧化应激发展的确切机制及其致病作用尚未完全阐明。
在下调差异基因中,发现多个NDUF基因为SSc核心基因,而且富集通路为还原型烟酰胺腺嘌呤二核苷酸(NADH)通路、氧化磷酸化相关通路。NADH是人体代谢、应激、细胞分化、线粒体功能等各种生物过程中必不可少的,而补充NADH可以恢复体内的NAD+水平,除了激活沉默信息调节因子(SIRT1)外,NADH还可能通过调节其他途径降低老年动物的氧化应激,改善血管功能、保护细胞、降低细胞死亡率[31]。总之,SSc目前病因未明,国内外学者也在积极寻求新的生物标记物。
4 结论1) 生物信息分析方法可为包括SSc在内的自身免疫疾病进行基因组个体化诊疗提供有力证据。
2) 通过使用微阵列揭示了SSc样本尿液中lncRNA和mRNA的表达水平,这可能直接反映关键基因在SSc中整合识别的作用,为SSc早期诊断和预后提供新的非侵入性潜在生物标志物检测手段。也说明了SSc的发病是多基因、多靶点参与的复杂过程。
3) 通过关键基因的分析也是研究SSc的突破点,可为药物干预疾病提供较为可靠的路径和作用靶点。
| [1] |
BELLANDO R S, MATUCCI C M. Very early systemic sclerosis[J]. Best Practice & Research Clinical Rheumatology, 2019, 33(4): 101428. DOI:10.1016/j.berh.2019.101428 (  0) 0) |
| [2] |
ORLAMDI M, BARSOTTI S, LEPRI G, et al. One year in review 2018: systemic sclerosis[J]. Clinical and Experimental Rheumatology, 2018, 38(S125): 3-23. (  0) 0) |
| [3] |
JING J, GAO Y. Urine biomarkers in the early stages of diseases: current status and perspective[J]. Discovery Medicine, 2018, 25(136): 57-65. (  0) 0) |
| [4] |
YANG Z, JIANG S, SHANG J, et al. LncRNA: Shedding light on mechanisms and opportunities in fibrosis and aging[J]. Ageing Research Reviews, 2019, 52: 17-31. DOI: 10.1016/j.arr.2019.04.001. Epub 2019 Apr 4.
(  0) 0) |
| [5] |
CHEN W, LIU D, LI Q Z, et al. The function of ncRNAs in rheumatic diseases[J]. Epigenomics, 2019, 11(7): 821-833. DOI: 10.2217/epi-2018-0135. Epub 2019 Jun 7.
(  0) 0) |
| [6] |
ASLANI S, SSBHANI S, GHARIBDOOST F, et al. Epigenetics and pathogenesis of systemic sclerosis; the ins and outs[J]. Human Immunology, 2018, 79(3): 178-187. DOI: 10.1016/j.humimm.2018.01.003. Epub 2018 Jan 9.
(  0) 0) |
| [7] |
LOPEZ B A, CHENG L, GEVAERT T, et al. Current and emerging bladder cancer biomarkers with an emphasis on urine biomarkers[J]. Expert Review of Molecular Diagnostics, 2020, 20(2): 231-243. DOI: 10.1080/14737159.2020.1699791. Epub 2019 Dec 11.
(  0) 0) |
| [8] |
SANTONI G, MMRELLI M B, AMANTINI C, et al. Urinary markers in bladder cancer: An update[J]. Frontiers in Oncology, 2018, 362. DOI:10.3389/fonc.2018.00362 (  0) 0) |
| [9] |
KPWAL B O, FRANSEN J, AVOCAC J, et al. Eustar coauthors. update of eulAR recommendations for the treatment of systemic sclerosis[J]. Annals of Rheumatology Disease, 2017, 76(8): 1327-1339. DOI: 10.1136/annrheumdis-2016-209909. Epub 2016 Nov 9.
(  0) 0) |
| [10] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: Quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Research, 2017, 45(D1): D362-D368. DOI:10.1093/nar/gkw937 (  0) 0) |
| [11] |
BADER G D, HOGUE C W. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, 4: 2. DOI: 10.1186/1471-2105-4-2. Epub 2003 Jan 13.
(  0) 0) |
| [12] |
CALDERON L M, POPE J E. Scleroderma epidemiology update[J]. Current Opinion in Rheumatology, 2021, 33(2): 122-127. DOI:10.1097/BOR.0000000000000785 (  0) 0) |
| [13] |
HOMBACH S, KRETZ M. Non-coding RNAs: Classification, biology and functioning[J]. Advances in Experimental Medicine and Biology, 2016, 937: 3-17. DOI:10.1007/978-3-319-42059-2_1 (  0) 0) |
| [14] |
STATELLO L, GUO C J, CHEN L L, et al. Gene regulation by long non-coding RNAs and its biological functions[J]. Nature Reviews Molecular Cell Biology, 2021, 22(2): 96-118. DOI:10.1038/s41580-020-00315-9 (  0) 0) |
| [15] |
TSAI C Y, SHEN C Y, LIU C W, et al. Aberrant non-coding rna expression in patients with systemic lupus erythematosus: consequences for immune dysfunctions and tissue damage[J]. Biomolecules, 2020, 10(12): 1641. DOI:10.3390/biom10121641 (  0) 0) |
| [16] |
MIHAYLOVA G, VASILEV V, KOSTURKOVA M B, et al. Long non-coding rnas as new biomarkers in lupus nephritis: A connection between present and future[J]. Cureus, 2020, 12(7): e9003. DOI:10.7759/cureus.9003 (  0) 0) |
| [17] |
MARIOTTI B, SERVAAS N H, ROSSATO M, et al. The long non-coding RNA NRIR drives IFN-response in monocytes: implication for systemic sclerosis[J]. Frontier of Immunology, 2019, 10: 100. DOI:10.3389/fimmu.2019.00100 (  0) 0) |
| [18] |
VAN DER KROEF M, CASTELLUCCI M, MOKRY M, et al. Histone modifications underlie monocyte dysregulation in patients with systemic sclerosis, underlining the treatment potential of epigenetic targeting[J]. Annals of Rheumatology Disease, 2019, 78(4): 529-538. DOI: 10.1136/annrheumdis-2018-214295. Epub 2019 Feb 6.
(  0) 0) |
| [19] |
SZABO I, MUNTEAN L, CRISAN T, et al. Novel concepts in systemic sclerosis pathogenesis: Role for miRNAs[J]. Biomedicines, 2021, 9(10): 1471. DOI:10.3390/biomedicines9101471.PMID:34680587;PMCID:PMC8533248 (  0) 0) |
| [20] |
BRISOTTO G, GUERRIERI R, COLIZZI F, et al. Long noncoding RNAs as innovative urinary diagnostic biomarkers[J]. Methods Molecular Biology, 2021, 2292: 73-94. DOI:10.1007/978-1-0716-1354-2_7 (  0) 0) |
| [21] |
GIGANTE A, PELLICANO C, LEODORI G, et al. Serum and urine free light chains measurements in patients with systemic sclerosis: novel biomarkers for disease activity[J]. Clinical & Experimental Immunology, 2021, 205(2): 135-141. DOI: 10.1111/cei.13611. Epub 2021 Jun 2.
(  0) 0) |
| [22] |
VAN CAAM A, VONK M, VAN DEN HOOGEN F, et al. Unraveling SSc pathophysiology; The myofibroblast[J]. Frontier of Immunology, 2018, 9: 2452. DOI:10.3389/fimmu.2018.02452 (  0) 0) |
| [23] |
FINNSON K W, ALMADANI Y, PHILIP A. Non-canonical (non-SMAD2/3) TGF-β signaling in fibrosis: Mechanisms and targets[J]. Seminars in Cell & Developmental Biology, 2020, 101: 115-122. DOI:10.1016/j.semcdb.2019.11.013 (  0) 0) |
| [24] |
ARVIA R, MARGHERI F, STINCARELI, et al. Parvovirus B19 activates in vitro normal human dermal fibroblasts: a possible implication in skin fibrosis and systemic sclerosis[J]. Rheumatology (Oxford), 2020, 59(11): 3526-3532. DOI:10.1093/rheumatology/keaa230 (  0) 0) |
| [25] |
FUNAKOSHI T, MATSUURA A, OHSUMI Y. Analyses of APG13 gene involved in autophagy in yeast, Saccharomyces cerevisiae[J]. Gene, 1997, 192(2): 207-213. DOI:10.1016/s0378-1119(97)00031-0 (  0) 0) |
| [26] |
JIN M, ZHANG Y. Autophagy and autoimmune diseases[J]. Advances in Experimental Medicine and Biology, 2020, 1207: 405-408. DOI:10.1007/978-981-15-4272-5_28 (  0) 0) |
| [27] |
MORI T, TAMURA N, WAGURI S, et al. Autophagy is involved in the sclerotic phase of systemic sclerosis[J]. Fukushima Journal of Medicine Science, 2020, 66(1): 17-24. DOI: 10.5387/fms.2019-28. Epub 2020 Apr 10.
(  0) 0) |
| [28] |
STATHOPOULOU C, NIKOLERI D, BERTSIAS G. Immunometabolism: an overview and therapeutic prospects in autoimmune diseases[J]. Immunotherapy, 2019, 11(9): 813-829. DOI:10.2217/imt-2019-0002 (  0) 0) |
| [29] |
MANCINI O K, ACEVEDO M, FAZEZ N, et al. Oxidative stress-induced senescence mediates inflammatory and fibrotic phenotypes in fibroblasts from systemic sclerosis patients[J]. Rheumatology (Oxford), 2021, 61: 1265-1275. DOI:10.1093/rheumatology/keab477 (  0) 0) |
| [30] |
DORIDOT L, JELJELI M, CHENE C, et al. Implication of oxidative stress in the pathogenesis of systemic sclerosis via inflammation, autoimmunity and fibrosis[J]. Redox Biology, 2019, 25: 101122. DOI: 10.1016/j.redox.2019.101122. Epub 2019 Jan 25. PMID: 30737171; PMCID: PMC6859527.
(  0) 0) |
| [31] |
PECLAT T R, SHI B, VARGA J, et al. The NADase enzyme CD38: an emerging pharmacological target for systemic sclerosis, systemic lupus erythematosus and rheumatoid arthritis[J]. Current Opinion of Rheumatology, 2020, 32(6): 488-496. DOI:10.1097/BOR.0000000000000737 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


