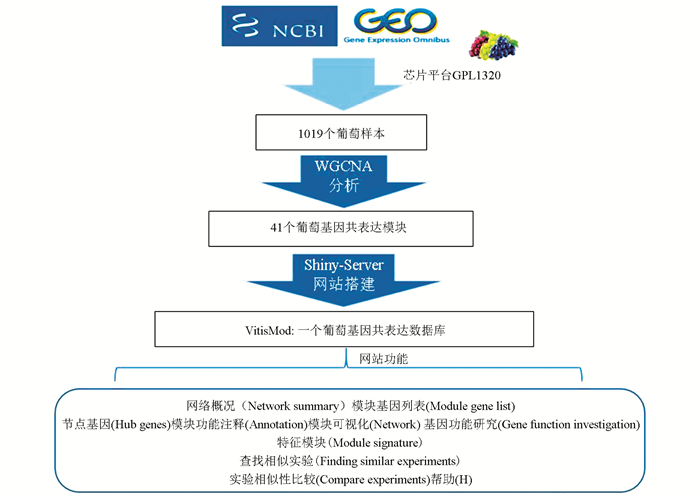
葡萄(Vitis vinifera L.)是一种营养丰富的水果,也常用于酿酒。基因表达芯片技术十分成熟,价格低廉,可以在基因组水平对基因表达进行测量[1]。大量葡萄基因表达芯片数据保存于公共数据库,如美国国立生物技术信息中心(NCBI)的基因表达数据库(GEO)[2]。通过生物信息学工具对这些高通量数据重新分析以挖掘基因功能,并获得基因表达与实验条件之间的关系,如温度和干旱等非生物胁迫,白粉病菌等生物胁迫和组织特异性,可以为葡萄抗病和育种提供有用的信息。传统的差异基因表达分析只适合小样本数据分析,而不能有效分析来自不同实验或条件下的大量异质性样本[3]。在高通量数据分析中,降维是一种常用的数据简化方法。显然,应根据研究目的选择合适的分析技术。基因共表达网络(GCN)分析方法被广泛应用于转录组的全局和时空表达分析。权重基因共表达网络分析(WGCNA)是一种常用的GCN方法,其已被应用到拟南芥、水稻、玉米和棉花等[4-7]。目前关于葡萄基因共表达的研究不多。5-氮杂胞苷可促进葡萄浆果发育,研究者对5个发育时期5-氮杂胞苷处理葡萄浆果进行基因共表达分析,鉴定到8个和发育时期相关的模块及一些枢纽基因[8]。葡萄白腐病是一种重要病害,研究者使用WGCNA对葡萄白腐病菌侵染12 h和36 h后叶片的基因表达进行分析,鉴定得到4个模块,其中1个模块可分为50个基因簇,其中一个基因簇可能和葡萄抗性相关[9]。对7个不同品系来源的葡萄皮基因共表达分析,共鉴定64个模块,和葡萄糖累积、节律等相关,提示这些功能可能在葡萄浆果成熟过程中发挥作用[10]。所有这些研究都包括几十个特定条件下的样本,只鉴定和特定条件相关的模块,这可能限制共表达分析的能力。因此,为鉴定更多的模块,需要更多来自不同研究的样本。组织来源或实验处理可被认为是一种扰动,这是我们整合不同研究数据集的原因。它有助于识别跨实验的共有模块,这不同于样本仅限于特定条件的模块分析[11]。我们收集了来自35个实验的1 019个葡萄转录组样本数据,应用WGCNA将数据维度从16 437个探针集减少到41个模块,并对模块基因进行功能注释。通过关联推定(Guilt-by-association)来注释功能未知的基因。模块与性状的关系进行关联分析。最后,建立一个简单实用的网页工具,为用户提供分析得到的模块信息、连接度信息,也提供样本比较等功能。分析框架如图 1所示。
|
图 1 VitisMod数据库构建流程图 Figure 1 Workflow for VitisMod database construction |
从NCBI GEO数据库下载葡萄基因表达芯片数据,平台为GPL1320,该平台目前具有最多的葡萄转录组数据。该平台使用Affymetrix Vitis vinifera (Grape) Genome芯片(http://www.afymetrix.com)。根据制造商提供的注释文件(Release 36),阵列包含16 437个探针集,代表 9 192个Entrez基因。使用MAS5方法在Expression Console(v1.4.1.46)中分析了原始基因芯片数据,得到探针水平的基因表达数据。去除对照探针后,在R(v3.3.1)中使用Normalization.Quantiles函数进行分位数归一化,并保留所有探针以保证最大的信息。为了方便,文中将探针集称为其对应的基因。
1.2 加权基因共表达网络分析(WGCNA)使用Bioconductor WGCNA软件包(v1.63)进行共表达网络分析,参数如下:networkType = 'signed',softPower = 16,minModuleSize = 30,deepSplit = 4,cutHeight = 0.99,MEDissThres = 0.2。根据基因表达矩阵构建符号共表达网络(Signed Network)。首先计算每对基因间的Pearson相关系数,获得相关系数矩阵; 相关矩阵进行幂指数加权将其转换为邻接矩阵(Adjacency Matrix)[12]; 其次,幂指数设置为16,以满足无标度网络拓扑标准。两个基因之间的关系不仅取决于它们的相关性,还取决于它们共享的邻域。WGCNA使用拓扑重叠网络(Topological Overlap,TO)来度量基因间关系。以1-TO为基因间距离进行分层聚类,鉴定高度共表达的基因[12]; 最后,使用动态树切割算法鉴定基因共表达模块[13]。每个模块通过奇异值分解获得模块特征基因表达(Module Eigengene,ME),因此每个模块的表达由其第一个主成分表示。ME解释了模块表达水平的最大变化量,被认为是模块中最具代表性的基因表达。设置模块中包含的基因个数最小为30。考虑到样本量大,为了鉴定尽可能多的模块,将划分模块的敏感度deepSplit设为最高值4。为减少冗余模块,对相似性高于0.8的模块进行合并,因此MEDissThres设为0.2。测试模块稳定性的方法为:原始连接度与随机采样1 000次的一半样本的连接度之间的平均相关性[14]。对于每个样本,每个模块都有1个ME值。最终原始基因表达数据降维到样本数×模块数。
1.3 模块的功能注释使用g: Profiler对共表达模块进行功能富集分析[15]。它将基因映射到已知功能信息源,如基因本体论(GO)数据库,并检测与模块基因集相关联的特定GO术语和背景分布是否存在差异,计算出统计显著性。本研究中,显著性阈值设定为校正后的P值小于0.05。
1.4 基于R Shiny的数据库构建VitisMod数据库构建主要使用R shiny包和shiny server搭建[16]。和该流程相关的代码和文件可在以下链接免费获取:http://bioinformatics.fafu.edu.cn/shiny-grape.zip。
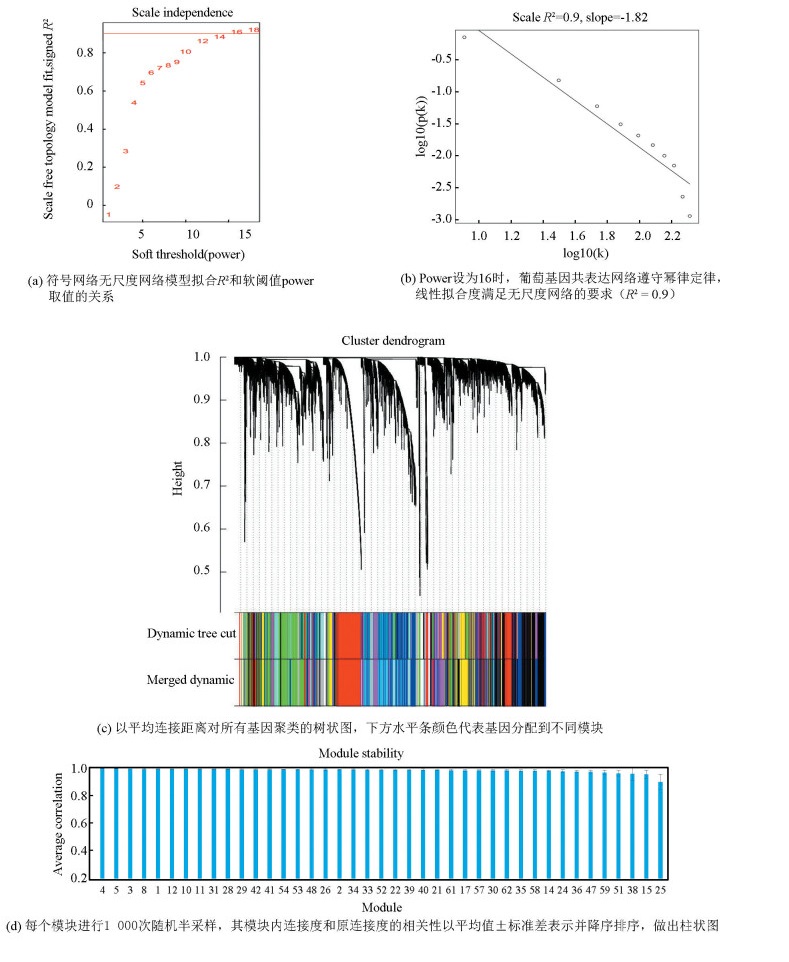
2 结果与分析 2.1 葡萄基因共表达网络构建使用WGCNA分析1 019个葡萄样本数据,power设置为16,以构建无标度(Scale-free)的基因共表达网络(见图 2a,2b)。通过动态树切割法对网络基因进行聚类和切割(见图 2c)。为了方便,每个模块都分配一个数字编号。相似簇合并后形成41个基因共表达模块。通过计算原始连接度与从每个模块的1 000个半采样连接度值计算出连接度相关性,来测试模块的稳定性(见图 2d)。结果表明除了M25,所有模块的平均连接度相关性都大于0.95(模块命名为M加上模块编号,例如M4。在WGCNA期间会合并一些相似的模块,因此编号不是连续的。)。M4的模块稳定性最高,而M25的模块稳定性最低。利用Shiny server[16]建立了一个网页工具来浏览模块基因和模块功能注释信息(http://bioinformatics.fafu.edu.cn/grape)。在“Genelist”选项卡中,用户可以浏览模块的连接度信息(见图 3)。
|
图 2 从葡萄基因共表达网络中鉴定到41个模块 Figure 2 Forty-one modules were identified from grape gene co-expression network |

|
图 3 VitisMod网络工具截图 Figure 3 Snapshots of the web tool VitisMod 注:在“Genelist”选项卡中,用户可以浏览选定模块的信息,包括探针集ID、基因模块间和模块内连接度、Entrez数据库ID、基因符号及Ensembl数据库ID。 |
在网络中,基因的连接度(Connectivity)定义为其与所有其他基因的连接之和。枢纽基因(Hub Gene)通常在模块中具有高连接度,因此可能更重要。在WGCNA中,连接度分2种类型。全局连接度(Global Connectivity)是整个网络内的连接度,而模块内连接度(Intramodule Connectivity)是模块内的连接度。全局连接度中排名前100的基因显著富集于叶绿体(Chloroplast,P= 5×10-28)。第一名高连接度的基因是VIT_00031272001(Chloroplast stem-loop binding protein of 41 kDa b),第二名高连接度的基因是VIT_00010012001(Thioredoxin M-type, chloroplastic-like)。
模块内连接度表示特定模块内基因间的关系。模块间连接度(Intermodule Connectivity)等于全局连接度减去模块内连接度,表明基因在模块间协调功能中的贡献。基于模块的基因连接度分析显示,M2模块间连接度比例最低,而M53模块间连接度比例最高。M2参与脂质应答。M53无明确注释,但其枢纽基因的共表达基因富集二甲基烯丙基二磷酸酯代谢。用户可在“Hub gene”选项卡中浏览每个模块的前3个枢纽基因。例如,M4主要和质体相关,其枢纽基因是磷酸甘油酸激酶。
2.3 将模块与实验条件/特征相关联鉴定的模块通过其第一主成分ME来概括模块基因的表达。计算每个样品中的每个模块表达,获得的ME向量可用来表征实验条件/特征。可以在“Module signature”选项卡中查看模块高表达的30个样本。例如,节律性光照处理的葡萄浆果具有最高的M58表达,M58和DNA拓扑异构酶有关,这和已有报道一致[17]。缺水12天的葡萄茎尖具有最高的M42表达,功能富集分析提示M42和细胞壁合成有关。这些模块可作为特定条件的特征。M35富集热响应基因,根据“Module signature”选项卡,该模块在转色期绿果阶段表达最高,而在更成熟阶段如红果阶段表达相对下调,提示热响应基因在葡萄浆果成熟过程发挥重要作用。
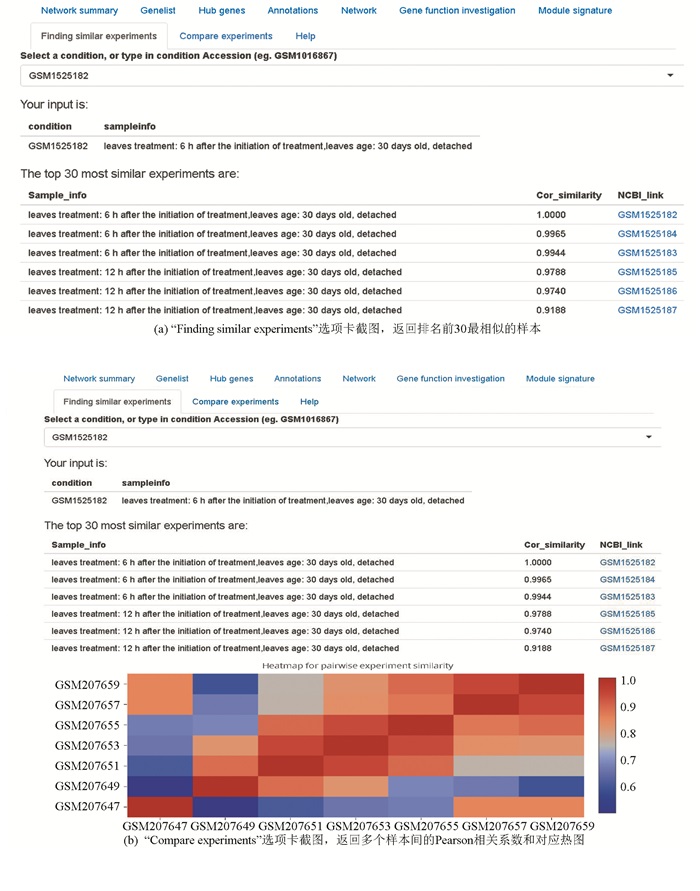
在“Find similar experiments”选项卡中提供了一个功能,通过ME向量的Pearson相关性来计算不同实验/条件的相似性,即用户可选择一个感兴趣的实验,VitisMod返回30个最相似的实验(见图 4a)。关于实验的详细信息,可点击实验的超链接来访问查看。例如,如果选择GSM1525182(紫外辐射C处理10 min后6 h采样的叶片),该工具将返回GSM1525184和GSM1525183,这是除其自身外的最相似实验。此外,用户可通过“Compare experiment”选项卡,选择感兴趣的数据来查看任意多个样本间的相似性(见图 4b)。例如,如果选择样本GSM207647、GSM207649、GSM207651、GSM207653、GSM207655、GSM207657和GSM207659,这些样本对应转色前浆果热处理0 h、1 h、4 h、7 h、9 h、14 h和24 h,结果发现相邻时间点间,4 h和7 h的处理、14 h和24 h的处理更为相似,均为0.94,这表明7 h和24 h提供冗余的信息。因此,4 h和14 h可能是更关键的采样时间点。如果选择三个生物学重复样本(GSM1016867,GSM1016868和GSM1016869),发现这些重复非常相似,相关性均高于0.99,表明实验重复性非常好。
|
图 4 VitisMod工具中基于模块表达的2项功能截图 Figure 4 Snapshots of two functions of VitisMod tool based on module eigengene (ME) expression |
基因共表达分析可用于基因功能预测[18],它基于“关联推定”的原理,即共表达的基因在功能上相关,将功能分配给同一模块中注释较差的基因。一个例子是M59,这是一个只有39个基因的小模块,注释为冰冻应答。M59枢纽基因包括VIT_08s0007g07520、VIT_07s0205g00010和一个21 kDa蛋白。VIT_08s0007g07520是PPR(Pentatricopeptide repeat)蛋白家族成员,在冷胁迫等非生物胁迫下发挥重要作用[1]。基因组中,VIT_07s0205g00010和VIT_08s0007g07520基因表达相关性最高(Pearson相关系数r=0.9, P < 0.01),但注释为功能未知蛋白质。其在拟南芥的同源基因为AT5G65660,是一个富含羟脯氨酸糖蛋白(Hydroxyproline-rich glycoproteins),具有感应温度变化的功能[19]。21 kDa蛋白没有功能注释,但其序列含有一个PMEI-like_3保守结构域,PEMI最近被证实和冰冻耐受有关[20]。
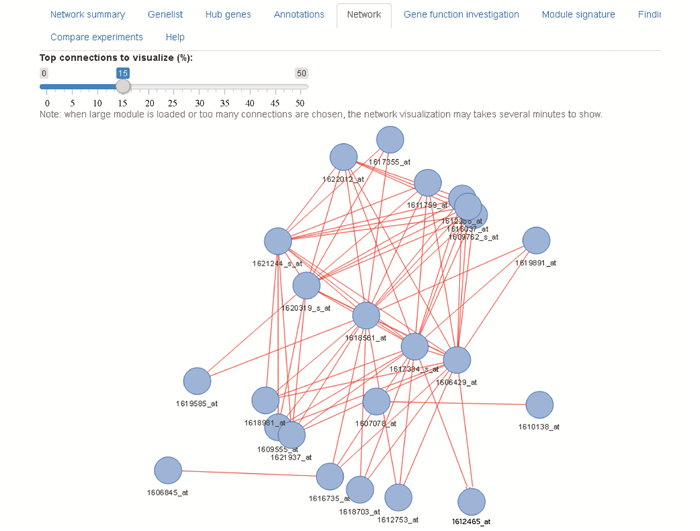
2.5 网络模块可视化模块通常以网络视图显示,基因间关系简明。在“Network”选项卡中,用户可选择感兴趣的模块并可视化高度连接的基因(见图 5)。由于可视化过程消耗大量计算机资源,VitisMod仅绘制了最相关的基因。用户可通过左侧的“Download Module”和“Download Genelist”两个按钮分别下载模块信息和模块的基因列表。所有的模块基因及其连接度信息均可以下载。
|
图 5 选项卡“Network”截图,返回不同比例的高连接度基因的共表达网络 Figure 5 Snapshots of tab "Network". Gene co-expression network visualized by choosing different percentages of high-connectivity genes |
基因表达芯片是一种成熟的技术,检测成本较低。公共数据库中有大量基于基因表达芯片的转录组数据,这些宝贵的数据可用于知识挖掘。由于多重比较负担和样本量的变化,传统的成对差异基因表达分析不可行[21]。尽管RNA-Seq比基因表达芯片更具优势,但二代测序的文库类型多样,测序深度不同和仪器类型差异,导致难以合并来自不同研究的数据[3]。有研究表明基因表达芯片数据更适合于基因共表达网络分析,数据越多,鉴定模块越多[22-23]。另外,特定条件下转录组通常是静态的,如果分析局限于某一条件,鉴定出的共表达模块也仅限于该条件[4]。因此,不能期望从一种条件下的样本中识别出许多模块。如果将条件/扰动视为输入,将模块视为输出,则重复或相似的输入可能会导致相同或相似的输出。因此,数据集整合有助于解决此问题,当更多的扰动或条件下的数据添加到分析中时,将鉴定出更多显著性强的模块[24]。因此,大样本数有助于提高WGCNA的统计效能。有研究整合了374个葡萄基因芯片数据,构建基因共表达网络,鉴定得到557个模块,但没有提供一个可使用的数据库[25]。VTCdb是一个葡萄基因共表达数据库,它整合了约800个样本的基因表达芯片数据,为基因模块鉴定、基因功能注释及候选基因排序等定性分析提供工具[26]。本文整合了更多个数据集并使用WGCNA对不同实验条件下的葡萄转录组进行分析。其次,进行了深入分析以挖掘更为精确的定量信息,这些信息包括基因连接度,模块枢纽基因,条件的特征模块和基因功能预测。最后,提供了一个简单的网络工具,以帮助葡萄研究人员探索这些信息或产生可检验的假设。
通过模块功能富集分析和模块稳定性测试验证了所鉴定模块的有效性。确定了41个具有生物学意义的模块。使用者可通过模块功能或高度相关基因的功能来推断基因功能。模块中的基因由于其连接度差异而对模块贡献不同,可从其模块内连接度高低推断基因的重要性。枢纽基因通常是重要的高连接度基因,这些信息有助于选择候选基因进行下一步的实验验证。使用者可在“Network”选项卡中可视化模块网络。
WGCNA是一种降维方法,有助于减少数据冗余[12]。每个样本都可由41个模块表示。通过ME值对样本进行排名可为特定实验提供潜在的特征模块。特征模块中的枢纽基因是该样本的潜在生物标志物,值得验证。此外,研究者需要了解样本之间的相似性,相似的样本可能表明相似的表达模式或机制。样本的ME向量使样本间比较或发现相似的样本成为可能。
本研究构建了一个简单易用的数据库,可用于葡萄研究中候选基因的筛选或者功能未知基因的功能预测。例如实验获得了差异基因列表,但哪个基因更重要并值得验证是未知的,此时可以在本数据库获取基因的连接度信息,模块内连接度高的基因往往更重要。在Gene function investigation选项卡中输入要查询的基因,会获得和该基因表达相关系数高的基因列表及其功能富集信息,同时可以查看该基因位于哪个模块。Module signature选项卡可查看该模块和哪个实验条件最相关,为实验验证提供重要信息。
4 结论本分析提供了在不同条件下葡萄转录组的系统视图,鉴定到41个稳定的又具有不同功能的基因共表达模块,发现高全局连接度基因显著富集于叶绿体、模块内高连接度基因可以用于基因功能预测,挖掘到和实验相关的特征模块,如M58和节律性光照有关,M42和缺水处理有关,M35和热响应有关。开发了一个简单的葡萄基因共表达网络数据库来帮助研究者探索与模块基因相关的信息,如模块基因连接度、模块基因网络可视化、模块功能富集情况、模块在不同实验基因表达情况和基于模块的样本相似性等,为实验验证产生新的假设。
| [1] |
CHEN G, ZOU Y, HU J, et al. Genome-wide analysis of the rice PPR gene family and their expression profiles under different stress treatments[J]. BMC Genomics, 2018, 19(1): 720. DOI:10.1186/s12864-018-5088-9 (  0) 0) |
| [2] |
BARRETT T, WILHITE S E, LEDOUX P, et al. NCBI GEO: archive for functional genomics data sets-update[J]. Nucleic Acids Research, 2013, 41: D991-995. DOI:10.1093/nar/gks1193 (  0) 0) |
| [3] |
刘伟, 李立, 叶桦, 等. 权重基因共表达网络分析在生物医学中的应用[J]. 生物工程学报, 2017, 33(11): 1791-1801. LIU Wei, LI Li, YE Hua, et al. Weighted gene co-expression network analysis in biomedicine research[J]. Chinese Journal of Biotechnology, 2017, 33(11): 1791-1801. DOI:10.13345/j.cjb.170006 (  0) 0) |
| [4] |
LIU W, LIN L, ZHANG Z, et al. Gene co-expression network analysis identifies trait-related modules in Arabidopsis thaliana[J]. Planta, 2019, 249(5): 1487-1501. DOI:10.1007/s00425-019-03102-9 (  0) 0) |
| [5] |
杨宇昕, 桑志勤, 许诚, 等. 利用WGCNA进行玉米花期基因共表达模块鉴定[J]. 作物学报, 2019, 45(2): 161-174. YANG Yuxin, SANG Zhiqin, XU Cheng, et al. Identification of maize flowering gene co-expression modules by WGCNA[J]. Acta Agronomica Sinica, 2019, 45(2): 161-174. DOI:10.3724/SP.J.1006.2019.83053 (  0) 0) |
| [6] |
李旭凯, 李任建, 张宝俊. 利用WGCNA鉴定非生物胁迫相关基因共表达网络[J]. 作物学报, 2019, 45(009): 1349-1364. LI Xukai, LI Renjian, ZHANG Baojun. Identification of rice stress-related gene co-expression modules by WGCNA[J]. Acta Agronomica Sinica, 2019, 45(9): 1349-1364. DOI:10.3724/SP.J.1006.2019.82061 (  0) 0) |
| [7] |
巨飞燕, 张思平, 刘绍东, 等. 利用WGCNA进行棉花果枝节间伸长相关基因共表达模块鉴定[J]. 棉花学报, 2019, 31(5): 403-413. JU Feiyan, ZHANG Siping, LIU Shaodong, et al. Identification of co-expression modules of genes related to internode elongation of cotton fruiting branches by WGCNA[J]. Cotton Science, 2019, 31(5): 403-413. DOI:10.11963/1002-7807.jfypcy.20190904 (  0) 0) |
| [8] |
GUO D L, LI Q, JI X R, et al. Transcriptome profiling of 'Kyoho' grape at different stages of berry development following 5-azaC treatment[J]. BMC genomics, 2019, 20(1): 825. DOI:10.1186/s12864-019-6204-1 (  0) 0) |
| [9] |
ZHANG Y, YAO J L, FENG H, et al. Identification of the defense-related gene VdWRKY53 from the wild grapevine Vitis davidii using RNA sequencing and ectopic expression analysis in Arabidopsis[J]. Hereditas, 2019, 156: 14. DOI:10.1186/s41065-019-0089-5 (  0) 0) |
| [10] |
GHAN R, PETEREIT J, TILLETT R L, et al. The common transcriptional subnetworks of the grape berry skin in the late stages of ripening[J]. BMC plant biology, 2017, 17(1): 94. DOI:10.1186/s12870-017-1043-1 (  0) 0) |
| [11] |
HE F, MASLOV S. Pan- and core-network analysis of co-expression genes in a model plant[J]. Scientific Reports, 2016, 6: 38956. DOI:10.1038/srep38956 (  0) 0) |
| [12] |
LANGFELDER P, HORVATH S. WGCNA: An R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 (  0) 0) |
| [13] |
LANGFELDER P, ZHANG B, HORVATH S. Defining clusters from a hierarchical cluster tree: the Dynamic Tree Cut package for R[J]. Bioinformatics, 2008, 24(5): 719-720. DOI:10.1093/bioinformatics/btm563 (  0) 0) |
| [14] |
YE H, LIU W. Transcriptional networks implicated in human nonalcoholic fatty liver disease[J]. Molecular Genetics and Genomics: MGG, 2015, 290(5): 1793-1804. DOI:10.1007/s00438-015-1037-3 (  0) 0) |
| [15] |
REIMAND J, ARAK T, ADLER P, et al. G: Profiler-a web server for functional interpretation of gene lists (2016 update)[J]. Nucleic Acids Research, 2016, 44(W1): W83-W89. DOI:10.1093/nar/gkw199 (  0) 0) |
| [16] |
CHANG W, CHENG J, ALLAIRE J J, et al. Shiny: Web application framework for R[Z]. R Package Version 0.11, 2015, 1(4): 106.
(  0) 0) |
| [17] |
KUPRIYANOVA E V, ALBERT E V, BLIZNINA A I, et al. Arabidopsis DNA topoisomerase I alpha is required for adaptive response to light and flower development[J]. Biology Open, 2017, 6(6): 832-843. DOI:10.1242/bio.024422 (  0) 0) |
| [18] |
VAN DAM S, VOSA U, VAN DER GRAAF A, et al. Gene co-expression analysis for functional classification and gene-disease predictions[J]. Briefings in Bioinformatics, 2018, 19(4): 575-592. DOI:10.1093/bib/bbw139 (  0) 0) |
| [19] |
PINSKI A, BETEKHTIN A, SALA K, et al. Hydroxyproline-rich glycoproteins as markers of temperature stress in the leaves of brachypodium distachyon[J]. International Journal of Molecular Sciences, 2019, 20(10). DOI:10.3390/ijms20102571 (  0) 0) |
| [20] |
CHEN J, CHEN X, ZHANG Q, et al. A cold-induced pectin methyl-esterase inhibitor gene contributes negatively to freezing tolerance but positively to salt tolerance in Arabidopsis[J]. Journal of Plant Physiology, 2018, 222: 67-78. DOI:10.1016/j.jplph.2018.01.003 (  0) 0) |
| [21] |
DALMASSO C, BROET P, MOREAU T. Multiple comparison procedures: principles, limits. Applications to microarray phenotype-genotype analysis[J]. Revue D'epidemiologie et de Sante Publique, 2004, 52(6): 523-537. DOI:10.1016/s0398-7620(04)99092-x (  0) 0) |
| [22] |
GIORGI F M, DEL FABBRO C, LICAUSI F. Comparative study of RNA-seq- and microarray-derived coexpression networks in Arabidopsis thaliana[J]. Bioinformatics, 2013, 29(6): 717-724. DOI:10.1093/bioinformatics/btt053 (  0) 0) |
| [23] |
SONG L, LANGFELDER P, HORVATH S. Comparison of co-expression measures: mutual information, correlation, and model based indices[J]. BMC Bioinformatics, 2012, 13(1): 328. DOI:10.1186/1471-2105-13-328 (  0) 0) |
| [24] |
SCHAEFER R J, MICHNO J M, MYERS C L. Unraveling gene function in agricultural species using gene co-expression networks[J]. Biochimica et Biophysica Acta. Gene Regulatory Mechanisms, 2017, 1860(1): 53-63. DOI:10.1016/j.bbagrm.2016.07.016 (  0) 0) |
| [25] |
LIANG Y H, CAI B, CHEN F, et al. Construction and validation of a gene co-expression network in grapevine (Vitis vinifera. L.)[J]. Horticulture Research, 2014, 1: 14040. DOI:10.1038/hortres.2014.40 (  0) 0) |
| [26] |
WONG D C, SWEETMAN C, DREW D P, et al. VTCdb: a gene co-expression database for the crop species Vitis vinifera (grapevine)[J]. BMC Genomics, 2013, 14: 882. DOI:10.1186/1471-2164-14-882 (  0) 0) |
 2023, Vol. 21
2023, Vol. 21


